基于转录组数据的马氏珠母贝EST-SSR位点的信息分析及其多态性检测*
王忠良 丁 燏 许尤厚 简纪常 鲁义善王 蓓 陈 刚 吴灶和,
(1. 广东海洋大学水产学院 湛江 524088; 2. 广东海洋大学南海水产经济动物增养殖重点实验室 湛江 524088; 3. 广西北部湾海洋生物多样性养护重点实验室(钦州学院) 钦州 535000; 4. 广东省水产经济动物病原生物学及流行病学重点实验室湛江 524088; 5. 仲恺农业工程学院 广州 510225)
马氏珠母贝(Pinctada martensii)又称合浦珠母贝(Pinctada fucata), 是人工培育海水珍珠的主要母贝。自 1965年我国成功开展马氏珠母贝人工育苗以来,海水珍珠养殖业发展迅速, 尤其是进入20世纪90年代后, 海水珍珠养殖已成为广东、广西和海南沿海地区的支柱产业(史兼华等, 2006; Wang et al, 2009)。然而, 由于长期的人工养殖和近亲繁殖, 马氏珠母贝种质已呈现明显的退化现象, 如种苗活力下降、死亡率增加、育珠贝变小、珍珠质量差等(Miyazaki et al, 1999;Tomaru et al, 2001; He et al, 2008)。为改善这一状况,马氏珠母贝的遗传改良和优良品种培育是当前海水珍珠养殖业的首要任务, 而筛选稳定可靠的分子标记评价其遗传多样性对马氏珠母贝良种选育和种质资源保护都具重要的指导意义。
简单重复序列(Simple Sequence Repeats, SSR),又称微卫星DNA, 是指由1-6个核苷酸组成的简单串联重复DNA序列(Powell et al, 1996; Liu et al, 2013)。与诸多分子标记相比, 具有数量丰富、多态性高、多等位性、共显性和可重复性高等优点, 已广泛应用于动植物遗传图谱构建、种质鉴定、基因定位、遗传多样性、分类与进化及分子标记辅助育种等研究领域(赵莹莹等, 2006; 徐美佳等, 2009; 李云峰等, 2010;孙国华等, 2010; 刘博等, 2012; 王东等, 2014)。根据来源, SSR标记又可分为基因组 SSR(Genomic SSR,gSSR)和表达序列标签 SSR(Expressed sequence tag SSR, EST-SSR)两种。与gSSR标记相比, EST-SSR标记无需构建基因组文库, 节省了大量人力物力, 开发成本较低, 且来源于基因的转录区, 其多态性与基因功能可能直接相关, 同时在相近物种间具良好的通用性(Varshney et al, 2005; 李琪, 2006; 张琼等,2010)。
近年来, 随着EST和cDNA大规模测序技术的快速发展, 多种海产经济贝类 SSR标记的开发利用研究已相继展开(李红蕾等, 2003; Sato et al, 2005; 李琪,2006; 李云峰等, 2010), 但现有的SSR标记还非常有限, 仍不能满足进一步研究的需要。为此, 本文基于前期Illumina高通量测序技术获得的马氏珠母贝转录组数据(未发表), 利用计算机软件辅助进行大规模EST-SSR标记的发掘, 并对其组成、分布及特征等信息进行了分析, 同时对 SSR标记的多态性进行了评价, 以期为马氏珠母贝遗传多样性、遗传图谱构建及分子辅助育种研究提供有效工具, 促进马氏珠母贝种质资源保护、优良品种培育和珍珠养殖业的健康发展。
1 材料与方法
1.1 马氏珠母贝的暂养及血细胞采集
马氏珠母贝(平均壳长 70mm)购自广东省湛江市徐闻迈陈珍珠贝养殖场, 暂养于室内玻璃钢水槽中(200L水体), 每槽饲养20只; 水温25°C, 盐度28, 饲养期间连续充气; 暂养过程中以螺旋藻粉为饵料投喂,每天换水(100%)一次; 室内暂养一周后采集血淋巴。
利用 1mL无菌注射器于马氏珠母贝闭壳肌处采集血淋巴, 每只 0.5mL; 采集的血淋巴分装至 1.5mL离心管中, 每管1mL; 4°C, 800g 离心10min 收集血细胞, 并立即进行DNA提取。
1.2 马氏珠母贝转录组数据来源
马氏珠母贝转录组数据来源于课题组前期利用Illumin/Hiseq-2000高通量测序平台对马氏珠母贝血细胞进行的全转录组测序。转录组测序共获得70407878条原始读数(Raw reads), 经去除含有接头、重复及测序质量较低的原始读数后, 获得 56345139条纯净读数(Clean reads)。使用转录组de novo组装软件Trinity(Grabherr et al, 2011)对Clean reads进行组装,并进行去冗余处理和进一步拼接, 共得到74007条非冗余独立基因(Unigene)。(以上转录组数据尚未发表,转录组Raw reads已提交至NCBI的SRA数据库, 登录号为SRP041567。)
1.3 马氏珠母贝转录组EST-SSR位点筛选
为检测马氏珠母贝转录组中 EST-SSR位点, 使用软件MIcroSAtellite (MISA) (Lu et al, 2013)对组装得到的Unigenes序列进行SSR 搜索和定位。所检测SSR 位点共6类, 分别为单核苷酸重复、二核苷酸重复、三核苷酸重复、四核苷酸重复、五核苷酸重复和六核苷酸重复, 其筛选标准为: 单核苷酸重复≥10次; 二核苷酸重复≥6次; 三核苷酸重复、四核苷酸重复、五核苷酸重复和六核苷酸重复≥5次。
1.4 马氏珠母贝转录组EST-SSR引物设计
使用Primer3软件进行SSR引物设计, 每条SSR产生3对引物; 引物设计的主要参数设置如下: 引物长 18—23bp, 最适长度 23 bp; PCR扩增产物长度100—300bp; GC含量40%—65%, 最适含量50%; 退火温度55—65°C, 最佳退火温度55°C。
1.5 马氏珠母贝转录组EST-SSR多态性检测
1.5.1 模板DNA的制备 采用Universal Genomic DNA Mini-Isolation Kit试剂盒(上海生工生物工程有限公司), 并按照说明书方法进行血细胞 DNA的提取。经紫外分光光度计定量后, –20°C保存备用。
1.5.2 PCR扩增及产物检测 为了筛选和验证所设计的SSR引物, 随机挑选80对引物(引物由上海生工生物工程有限公司合成), 以上述血细胞DNA为模板进行PCR扩增。PCR反应体系为20 μL, 包括2×ES Taq MIX 10 μL, 上下游引物(10 μmol/L)各 1 μL, 模板DNA 1 μL, ddH2O补足至 20 μL。PCR扩增程序为:94°C预变性5 min, 30个循环(94°C变性30s, 最佳Tm退火 30s, 72°C延伸 30s), 最后 72°C 延伸 5 min。PCR扩增产物用8%非变性聚丙烯酰胺凝胶电泳结合银染法显色进行检测。
2 结果与分析
2.1 马氏珠母贝转录组EST-SSR的分布及频率
利用MISA软件对马氏珠母贝转录组中的74007条Unigenes序列进行搜索, 共检测到9872个SSR位点, 分布于8135条Unigenes序列中。SSR 发生频率(含SSR的Unigene数/Unigene总数)为10.99%, SSR出现频率(检出SSR位点数/Unigene总数)为13.34%;平均每5102bp含有1个SSR位点(检测序列总碱基数50368155 bp)。其中, 1350条Unigene含有1个以上SSR位点, 复合型SSR数目为518个。
马氏珠母贝 EST-SSR重复类型丰富, 除六核苷酸重复外, 1—5核苷酸重复均有发现。其中, 单核苷酸重复有4种类型, 二核苷酸重复有11种类型, 三核苷酸重复有 52 种类型, 四核苷酸重复有 58种类型,五核苷酸重复有7种类型(表1)。从各类型SSR位点数量看, 出现最多的为 1—3核苷酸重复, 占总 SSR位点数的98.32%。其中, 单核苷酸重复比例最高, 占81.46%; 其次为二核苷酸重复和三核苷酸重复, 分别占10.48%和6.38%; 4—5核苷酸重复SSR数量较少,共占 1.68%(表 1)。

表1 马氏珠母贝EST-SSRs不同重复基元分布情况Tab.1 Distribution of different repeat motifs of EST-SSRs in P.martensii transcriptome
马氏珠母贝转录组中 EST-SSR的长度在 12—20bp的有3677条, 其中单核苷酸重复SSR共计2226条; 长度超过20bp的SSR有336条, 并以单核苷酸重复SSR最多, 达129条(表2)。SSR位点重复次数方面, 以重复 10次(3883)最多, 占总 SSR位点的39.33%; 其次为重复11次和12次, 分别占17.12%和8.88%; 重复5—9次、13—17次及18—24次的SSR位点个数分别为1671、1279和472(图1)。
2.2 马氏珠母贝转录组EST-SSR的特征
在马氏珠母贝转录组9872个SSR位点中, 共检测到 132种重复基元(表 1)。其中, 以单核苷酸重复基元 A/T和 C/G最多, 分别占总 SSR的 71.27%和10.19%, 发生频率分别为 9.507%和 1.359%; 其次为二核苷酸重复基元AT/AT和AG/CT, 分别占4.66%和4.24%; 三核苷酸重复单元中, 以 ATC/ATG、AAG/CTT和AAC/GTT出现次数最多, 分别占总SSR的2.07%、1.29%和1.16%; 四核苷酸和五核苷酸重复基元种类较多, 但数量较少, 频率较低, 共占SSR总数的 1.67%(表 3)。
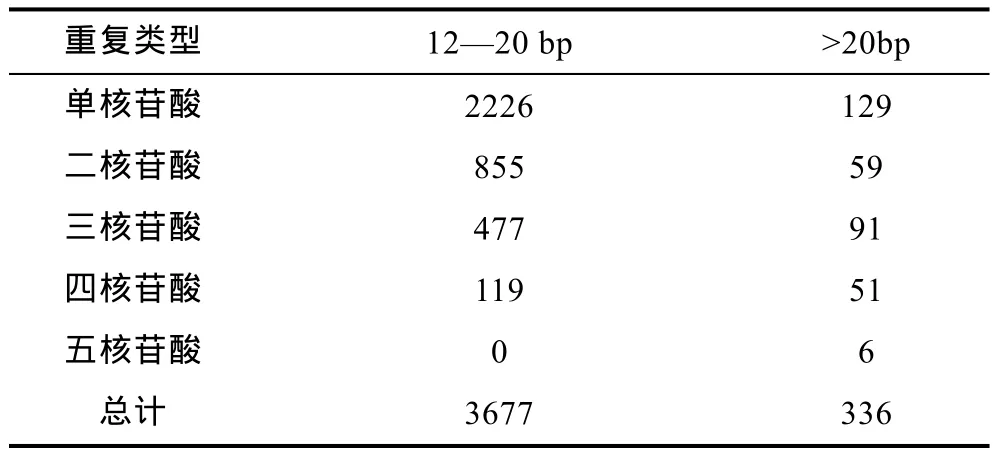
表2 马氏珠母贝EST- SSRs长度分布情况Tab.2 Lengthdistribution of EST-SSRs in P. martensii transcriptome

图1 马氏珠母贝EST-SSRs重复次数分布图Fig.1 Distribution of EST-SSR repeats frequency in P.martensii transcriptome
2.3 马氏珠母贝转录组EST-SSR引物设计及多态性检测
为更好地利用马氏珠母贝 SSR位点, 本文应用Primer3软件对上下游序列均不小于 150 bp的EST-SSR设计引物, 每条序列产生 3对引物。经SSRFinder校验及除去不符合条件的引物后, 共为5922条EST-SSR序列成功设计出17766对引物, 占马氏珠母贝SSR总数的59.99%。
从上述设计引物中随机挑选 80对引物, 并以随机挑选的20只健康马氏珠母贝血细胞基因组DNA为模板进行PCR扩增以验证设计的EST-SSR引物。结果表明, 共有 62对引物成功扩增出稳定条带, 占引物总数的 77.5%, 但其中有 23对引物扩增产物片段与预期产物片段大小不符; 在62个SSR位点中, 以二碱基、三碱基重复类型的 SSR位点扩增成功率较高; 此外, 17对 EST-SSR引物(占扩增引物总数的21.25%)表现出个体多态性(表 4), 多态性比率为27.42%; 17对多态性引物所在SSR位点中, 有7个为二碱基重复, 6个为三碱基重复, 2个为单碱基重复,四碱基重复和五碱基重复各1个。
3 讨论
目前, 有关贝类微卫星标记的研究主要是通过构建cDNA文库来获得含有微卫星的序列, 并且设计引物对微卫星标记进行筛选(Sato et al, 2005; 李春艳等, 2009)。如石耀华等(2008)从马氏珠母贝9个组织的SMART cDNA文库中共获得6979条EST序列, 并筛选到268个SSR位点, SSR出现频率为3.48%; 同样, 李云峰等(2010)从虾夷扇贝(Patinopecten yessoensis) cDNA文库中发现了74个SSR重复序列(2007条EST序列), SSR出现频率为3.69%。与上述传统 EST-SSR标记开发技术相比, 基于高通量转录组测序的 SSR标记开发技术不仅可以借助更全面的数据信息, 提高遗传多样性研究的准确性, 而且开发的SSR标记同样具有EST-SSR标记的诸多优点。王学颖(2012)从马氏珠母贝珍珠囊转录组的99127条无冗余EST序列中共发现了6595个SSR位点, SSR出现频率为6.65%; 同样, 本文利用 MISA软件从马氏珠母贝转录组74007条Unigenes中共检索出9872个SSR位点, 分布于8135条Unigenes中, SSR出现频率高达 13.34%。因此, 结合其它动植物 SSR开发相关文献和本研究结果表明, 基于转录组数据筛选 SSR标记是发掘非模式生物 EST-SSR的一种快速、高效的方法, 应用前景广阔。

表3 马氏珠母贝EST-SSRs重复基元的类型及频率Tab.3 EST-SSR repeats motifs and the frequency in P. martensii transcriptome
根据现有报道, 虾夷扇贝和马氏珠母贝的EST-SSR均以二核苷酸重复基元为主, 其所占比例分别为 40.54%和 48.5% (石耀华等, 2008; 李云峰等,2010); 缢蛏(Sinonovacula constricta)SSR以三核苷酸重复SSR比列最高, 为37.13%(刘博等, 2012); 此外,基于马氏珠母贝珍珠囊转录组的 SSR分析中, 四碱基重复基元占总EST-SSR的40%(石耀华等, 2008)。而本研究中以单核苷酸重复基元为主, 比列达到81.46%。造成以上差异的原因, 除EST序列获取方法不同外(王学颖, 2012), 在一定程度上与搜索 SSR时设置的参数有很大的关系(Wei et al, 2011)。Aggarwal等(2007)发现, 如果改变 SSR搜索标准(提高二核苷酸重复基元的重复次数, 或降低三核苷酸重复基元的重复次数), 原本二核苷酸重复 SSR占优势的研究中, 便会出现三核苷酸重复SSR占优势的情况; 同样,若本研究中提高单核苷酸 SSR的重复次数, 则 SSR将以二核苷酸重复基元为主。因此, 亟需建立一个统一的 SSR检测参数标准以便有效比较不同研究中同一或相近物种SSR分布的类型与特点。

表4 17对多态性EST-SSRs引物序列及扩增参数Tab.4 Sequences and amplification parameters of the 17 EST-SSRs primers
研究表明, SSR标记可用性的重要依据——多态性的高低, 主要取决于的其长度大小(李珊等, 2010)。当SSR长度≥20bp时其多态性较高, 长度在12—20bp之间的SSR则呈现中等多态性, 而长度<12bp时多态性较低(杨华等, 2011)。本研究中, 长度在 12—20bp之间的SSR有3677条(占SSR总数的37.25%), 这些SSR具中等多态性; 而长度≥20bp的SSR有336条(占SSR总数的 3.4%), 此类SSR呈较高的多态性。此外, 本研究中20bp以上的低级重复基元(一、二、三核苷酸重复基元)较多, 共计 279条(占 20bp以上SSR的83.04%)。由此可预见马氏珠母贝转录组来源的 SSR具有较高的多态性潜能, 在分子标记研究方面将具有较高的利用价值(Dreisigacker et al, 2004)。
为检测转录组中EST-SSR的多态性, 随机挑选了80对引物进行PCR扩增, 其中62对引物成功扩增出稳定条带, 但仍有18条引物未能扩增出产物。这可能是由于所设计的引物序列位于两个外显子上, 或者基因组对应序列含有内含子而不具备SSR序列特征等原因造成(Varshney et al, 2005)。此外,本研究出现了 23对引物扩增产物片段与预期产物片段大小不符的现象, 而该现象同样出现在栉孔扇贝和缢蛏的 EST-SSR扩增产物中(李红蕾等, 2003;刘博等, 2012)。究其原因, 有学者认为其与引物的非特异性结合(错配)或基因组 DNA序列含有内含子等相关(李红蕾等, 2003; Saha et al, 2004)。17对多态性引物所在SSR位点中, 多为二、三核苷酸重复, 这从某种程度上也验证了低级重复基元多态性高于高级重复基元多态性的推断(Dreisigacker et al,2004)。
4 结论
本研究利用马氏珠母贝的转录组数据, 筛选获得了9872个EST-SSR位点, 位点出现频率达13.34%;同时, 共为5922条EST-SSR序列成功设计出17766对引物; 随机选择的 80对扩增引物中, 共有 17对EST-SSR引物表现出个体多态性, 多态性比率为27.42%。以上研究结果对于丰富马氏珠母贝分子标记类型、加速功能基因资源的利用、探究种群遗传结构、分析遗传多样性、保护种质资源和培育优良品种均具有重要意义。
王 东, 曹玲亚, 高建平, 2014. 党参转录组中SSR位点信息分析. 中草药, 45(16): 2390—2394
王学颖, 2012. 马氏珠母贝微卫星引物的批量开发及选育系和珍珠囊的标记分析. 湛江: 广东海洋大学硕士学位论文
石耀华, 洪 葵, 郭希明等, 2008. 马氏珠母贝EST微卫星的筛选. 水产学报, 32(2): 174—181
史兼华, 何毛贤, 黄良民等, 2006. 马氏珠母贝选育系遗传变异的AFLP分析. 高技术通讯, 16(5): 534—538
刘 博, 邵艳卿, 滕爽爽等, 2012. 缢蛏(Sinonovacula constricta)EST-SSR分布特征及引物开发利用. 海洋与湖沼, 43(1): 132—137
孙国华, 杨建敏, 宋志乐等, 2010. 刺参(Apostichopus japonicus)EST 序列中微卫星分布分析及其标记的筛选.海洋与湖沼, 41(1): 133—139
李 珊, 周天华, 赵桂仿等, 2010. 马蹄香表达序列标签资源的SSR信息分析. 中草药, 41(3): 464—468
李 琪, 2006. 海洋贝类微卫星 DNA标记的开发及其在遗传学研究中的应用. 中国水产科学, 13(3): 502—509
李云峰, 刘卫东, 高祥刚等, 2010. 虾夷扇贝外套膜和肾脏组织cDNA文库构建以及EST 的初步分析. 中国水产科学,17(3): 578—585
李红蕾, 宋林生, 王玲玲等, 2003. 栉孔扇贝EST中微卫星标记的筛选. 高技术通讯, 13(12): 72—75
李春艳, 丁 君, 常亚青等, 2009. 虾夷扇贝微卫星标记的分离及其养殖群体的遗传结构分析. 中国水产科学, 16(1):39—46
杨 华, 陈 琪, 韦朝领等, 2011. 茶树转录组中SSR位点的信息分析. 安徽农业大学学报, 38(6): 882—886
张 琼, 刘小林, 李喜莲等, 2010. EST-SSR分子标记在水生动物遗传研究中的应用. 水产科学, 29(5): 302—306
赵莹莹, 朱晓琛, 孙效文等, 2006. 虾夷扇贝的多态性微卫星座位. 动物学报, 52(1): 229—233
徐美佳, 张 妍, 鲁翠云等, 2009. 利用 EST-SSR座位对鲤鱼 4种生长性状的单标记回归分析. 水产学杂志, 22(1): 15—18
Aggarwal R K, Hendre P S, Varshney R K et al, 2007.Identification, characterization and utilization of EST-derived genic microsatellite markers for genome analyses of coffee and related species. Theor Appl Genet,114(2): 359—372
Dreisigacker S, Zhang P, Warburton M L et al, 2004. SSR and pedigree analyses of genetic diversity among CIMMYT wheat lines targeted to different megaenvironments. Crop Sci, 44(2): 381—388
Grabherr M G, Haas B J, Yassour M et al, 2011. Full-length transcriptome assembly from RNA-Seq data without a reference genome. Nat Biotechnol, 29(7): 644—652
He M X, Guan Y Y, Yuan T et al, 2008. Realized heritability and response to selection for shell height in the pearl oyster Pinctada fucata (Gould). Aquac Res, 39(8): 801—805
Liu T M, Zhu S Y, Fu L L et al, 2013. Development and characterization of 1, 827 expressed Sequence tag-derived simple sequence repeat markers for ramie (Boehmeria nivea L. Gaud). PLoS One, 8(4): e60346
Lu X, Wang H X, Liu B Z et al, 2013. Three EST-SSR markers associated with QTL for the growth of the clam Meretrix meretrix revealed by selective genotyping. Mar Biotechnol,15(1): 16—25
Miyazaki T, Goto K, Kobayashi T et al, 1999. Mass mortalities associated with a virus disease in Japanese pearl oysters Pinctada fucata martensii. Dis Aquat Org, 37(1): 1—12
Powell W, Machray G C, Provan J, 1996. Polymorphism revealed by simple sequence repeats. Trends Plant Sci, 1(7):215—222
Saha M C, Rouf Mian M A, Eujayl I et al, 2004. Tall fescue EST-SSR markers with transferability across several grass species. Theor Appl Genet, 109(4): 783—791
Sato M, Kawamata K, Zaslavskaya N et al, 2005. Development of microsatellite markers for Japanese scallop (Mizuhopecten yessoensis) and their application to a population genetic study. Mar Biotechnol, 7(6): 713—728
Tomaru Y, Kawabata Z, Nakano S, 2001. Mass mortality of the Japanese pearl oyster Pinctada fucata martensii in relation to water temperature, chlorophyll a and phytoplankton composition. Dis Aquat Org, 44(1): 61—68
Varshney R K, Graner A, Sorrells M E et al, 2005. Genic microsatellite markers in plants: features and applications.Trends Biotechnol, 23(1): 48—55
Wang Z L, Wu Z H, Jian J C et al, 2009. Cloning and expression of heat shock protein 70 gene in the haemocytes of pearl oyster (Pinctada fucata, Gould 1850) responding to bacterial challenge. Fish Shellfish Immunol, 26(4): 639—645
Wei W L, Qi X Q, Wang L H et al, 2011. Characterization of the sesame (Sesamum indicum L.) global transcriptome using Illumina paired-end sequencing and development of EST-SSR markers. BMC Genomics, 12: 451

