HDAC2磷酸化区域突变体的构建及其对小鼠成纤维细胞增殖的影响*
肖 勇,黄 宏,郭 韡,邢 伟,李向云,袁培淞,徐 祥
(第三军医大学附属大坪医院野战外科研究所/创伤、烧伤与复合伤国家重点实验室第一研究室,重庆400042)
组蛋白去乙酰化酶(histone deacetylases,HDACs)是一类锌离子依赖性金属蛋白酶,是参与表观遗传乙酰化修饰调控的关键蛋白酶,特别是对组蛋白乙酰基团的去除在染色体重塑调控中起重要作用[1]。HDAC2是由488个氨基酸构成的肽链,正常表达于多种器官与组织,特别表达于如愈合创面的肉芽组织和增殖性肠腺细胞等增殖度相对活跃的组织和细胞中,在维持正常组织器官细胞增殖和存活及抑制分化方面起重要作用[2]。已证实,HDAC2去乙酰化结构域位于其N 端的第100~323个氨基酸内。同时,研究还发现,在其羧基端还含有一个独立的不依赖于其去乙酰化酶活性的Sumo-E3连接酶功能结构域[3]。然而关于HDAC2 的Sumo-E3 连接酶活性的功能、结构和生物学意义等仍不清楚。另外,分析发现HDAC2的磷酸化修饰区域正好位于其Sumo-E3 连接酶的结构域内[4],且磷酸化修饰和Sumo修饰之间存在密切的联系[5-6]。为此,推测HDAC2的自身磷酸化在其Sumo-E3连接酶功能的发挥中起重要的调节作用,但有待证实。为证实这一推测,本研究在前期获得的HDAC2Sumo-E3连接酶功能结构域的基础上[7],进一步运用片段缺失突变技术获得HDAC2Sumo-E3连接酶磷酸化区域缺失的突变体,鉴定HDAC2自身磷酸化对其Sumo-E3连接酶的调节作用。
1 材料与方法
1.1 主要材料 pcDNA3.1/HDAC2(DEL100-322AA)质粒和L929 细胞由本实验室保存。5×Phusion High-Fidelity DNA Polymerase购自Thermo公司,10 MM dNTPs购自申能博彩生物科技有限公司。DpnI等酶购自宝生物工程(大连)有限公 司,Annealing Buffer购自Beyotime公司,胎牛血清DMEM 高糖培养基购自Gibco公司,Lipofectamine 2000转染试剂盒购自Invtro-gen公司,LUC 报告基因检测试剂盒购自Promega公司。ODC和C-MYC抗体购自Santa Cruz公司。
1.2 引物设计 设计引物(Ft Rs或Rt Fs),每对引物完成突变体DNA 一条单链的扩增。FT:5′-CCA GAG GAT GCT GTT CAT GAA GAC GAG GAT GAA GGT GAA GGA GGT-3′;RS:5′-GTC TTC ATG AAC AGC ATC CTA-3′;FS:5′-GAG GAT GAA GGT GAA GGA GGT-3′;RT:5′-ACC TCC TTC ACC TTC ATC CTC GTC TTC ATG AAC AGC ATC CTA-3′。
1.3 突变体的构建 取两管0.12μL pcDNA3.1/HDAC2(DEL100-322AA)为模板,分别于其中一只管中加入对应的FT 1μL和RS 1μL,另一只管中加入对应的FS 1μL和RT 1 μL,然后各管分别加入10 MM dNTPs 0.4μL,5×Phusion High-Fidelity Buffer 4μL,DMSO 0.6μL,Phusion DNA Polymerase 0.2μL,最后加入双蒸水12.68μL 补足20μL 体系。放入PCR 仪中设置98 ℃预变性30s;98 ℃变性30s,50 ℃退火30s,72 ℃延伸6min,共35个循环;72 ℃终循环10 min。扩增后的每管中目的片段是一条单链。将对应的两管各取10 μL,加入5×Annealing Buffer for DNA oligos 10μL,最后加入20μL ddH2O 补足反应体系至50μL。体系加好后在沸水中煮5min,然后待其自然冷却过夜退火。最后用甲基化切割酶DpnI去除带有甲基化修饰的模板,在退火产物中加入DpnI酶1μL,10×T buffer 6μL,用3μL ddH2O 补足体系至60μL。体系加好后放入37 ℃水浴锅中酶切5h。将构建好的载体进行乙醇沉淀纯化后,转化入TOP10感受态菌中铺氨苄板筛选。挑取单克隆扩增菌液40mL,然后用质粒抽提试剂盒(购自北京百泰克生物技术有限公司)进行质粒抽提,并进行浓度的测定,BamH I和Xho I双酶切初步鉴定后,测序确定。
1.4 荧光素酶报告基因检测磷酸化区域突变体对蛋白质翻译的影响 分别将荧光素酶报告基因同空载体、野生型和构建好的片段突变体进行共转染入L929 细胞,转染方法见Invtrogen 公司的LipofectamineTM2000 转染试剂盒说明书,48h后裂解细胞,加入荧光素酶底物(购自Promega公司),测定化学发光值,作为评价荧光素酶翻译水平的指标。
1.5 Western blot检测L929细胞中HDAC2Sumo-E3连接酶介导的靶蛋白的表达 采用以上相同的转染方法将空载体、野生型和构建好的片段突变体转染入L929细胞以后,培养48 h,使用细胞裂解液提取各组细胞总蛋白,用考马斯亮蓝染色法测定蛋白含量,取50μg蛋白在10%SDS-PAGE胶上电泳,后转移至PVDF膜上,用TBST 配置5%脱脂奶分别加入一抗c-Myc,ODC抗体(1∶500)4 ℃孵育过夜,二抗(1∶10 000)常温孵育4h,胶片曝光显影,以β-actin 为内参照进行免疫印迹检测。
1.6 MTT 检测突变体对细胞的影响 将L929 细胞培养后按照每孔0.4×104个细胞铺板,将空载体、野生型和构建好的片段突变体转染入细胞,待培养细胞密度至70%~80%以后,加入50μL MTT(5mg/mL)继续培养4h,然后终止培养,小心吸弃上清液,每孔加入150μL DMSO,置于摇床上低速摇10 min,使结晶物充分溶解。采用比色分析法,应用自动酶联免疫检测仪测定波长490nm 处的各孔吸光值。
1.7 统计学处理 采用SPSS17.0 软件处理数据,各分组所得计量资料采用±s表示,两组间均数比较用t检验。检验水准α=0.05,P<0.05为差异有统计学意义。
2 结 果
2.1 磷酸化区域缺失突变体的构建与鉴定 用BamH I和Xho I 对pcDNA 3.1/HDAC2(DEL100-322AA)和pcDNA 3.1/HDAC2(DEL-PSD)(DEL 394-424)进行双酶切后,用1%的琼脂糖凝胶电泳分析所得到的酶切片段分别是5.5kb pcDNA 3.1载体片段、800bp的模板片段及700bp的片段缺失突变体片段,同预期的片段大小一致。
2.2 荧光素酶报告基因检测磷酸化区域突变体E3连接酶功能活性 根据野生型、突变体组与空载体组发光值比值计算出活化倍数,反应突变体对HDAC2Sumo-E3连接酶功能活性的影响。LUC报告基因检测片段突变体对Sumo-E3 连接酶功能活性较野生型提高了4.24 倍。由此表明磷酸化可能抑制HDAC2Sumo-E3连接酶的活性,从而影响蛋白质的翻译,见图1。

图1 HDAC2磷酸化片段缺失突变体对LUC的影响
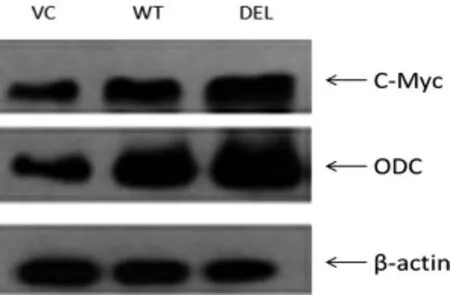
图2 Western blot检测ODC和c-Myc蛋白的表达
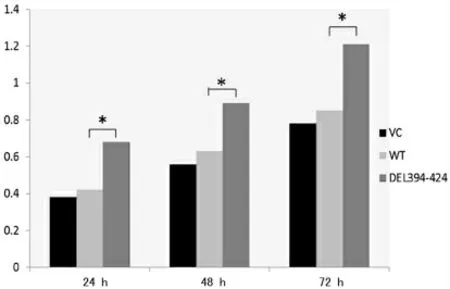
图3 MTT 检测磷酸化突变体对细胞增殖影响
2.3 Western blot检测磷酸化突变体对L929细胞中HDAC2 Sumo-E3连接酶介导的靶蛋白的表达 Western blot检测结果显示,与野生型组相比,突变体组明显促进了ODC 和c-Myc蛋白的表达,见图2。
2.4 MTT 检测磷酸化突变体对成纤维细胞增殖的影响 MTT 检测结果显示,与野生型相比,突变体组明显促进了细胞的增殖,并且两组吸光度值差异有统计学意义(P<0.05),见图3。
3 讨 论
在真核细胞中,有95%的蛋白合成都是通过帽依赖性蛋白mRNA 的翻译。其翻译起始的关键步骤是真核翻译起始因子eIF4F 复 合物(eIF4E、eIF4A 和eIF4G)的形成[8-9]。而eIF4E的Sumo化可以促进eIF4F复合物的形成。Sumo化是一种可以逆转的共价结合在其底物的蛋白修饰分子。人类基因组编码了4种Sumo分子,分别是Sumo1、Sumo2、Sumo3和Sumo4[10]。蛋白质的Sumo化受到激活(E1),结合(E2)和连接(E3)酶的影响[11]。Ubc9是唯一的Sumo-E2结合酶,然而Sumo-E3连接酶已经鉴定有数种[12]。只有HDAC4和HDAC7被报道有E3连接酶活性。本课题组前期研究发现,HDAC2的羧基末端还具有不依赖其去乙酰化酶功能的Sumo-E3连接酶功能结构域[3,13]。通过此功能介导,其可在翻译水平上调如ODC、c-Myc、Survivin和Bcl-2 等与细胞生长、增殖和抗凋亡相关基因的表达,而去除羧基末端的片段缺失突变体则丧失此能力。同时本课题组证实其主要机制是:通过上调翻译起始因子eIF4E 的Sumo 化修饰,而促进翻译起始因子eIF4E、eIF4G 和eIF4A 间的相互作用,促进蛋白质翻译起始复合物eIF4F的形成,最终在翻译水平上调与细胞增殖、生长和抗凋亡相关基因的表达,促进细胞快速生长。但此新功能的调控机制未知,且磷酸化修饰和Sumo 修饰之间存在密切联系[5-6]。
为了研究HDAC2的磷酸化区域对其Sumo-E3连接酶活性的影响,本研究以pcDNA3.1/HDAC2(DEL100-322AA)为模板构建HDAC2 磷酸化区域缺失片段突变体pcDNA3.1/HDAC2(DEL-PSD)(DEL 394-424AA)。通过双酶切和测序鉴定的结果提示,成功构建了pcDNA3.1/HDAC2(DEL-PSD)片段突变体。从帽依赖性mRNA 翻译蛋白报告基因LUC 检测的结果提示,HDAC2的磷酸化区域被去除以后的目的片段使LUC的表达增加了4.24倍。反映了其磷酸化区域被去除以后明显促进了蛋白质的翻译。为了研究其促进哪一类蛋白质的翻译,进行Western blot检测了HDAC2Sumo-E3连接酶介导的eIF4E相关的基因的表达,发现ODC 和c-Myc表达增加,和本课题组前期研究一致[5]。其机制可能是去除磷酸化位点的HDAC2片段能增加其Sumo-E3连接酶活性,从而增强eIF4E的Sumo化修饰,在翻译水平选择性激活eIF4E 介导的细胞增殖、生长和抗凋亡相关基因的表达。最后本研究将构建好的突变体转入L929细胞中进行MTT 检测发现,磷酸化区域被去除以后的目的片段可以明显促进L929 细胞的增殖。因L929细胞在创伤愈合过程中发挥了重要作用,蛋白质翻译的增加也可促进创伤修复,加快愈合过程,所以本研究结果也提示其对创面愈合有促进作用。本实验过程中研究的是磷酸化区域的片段突变体,所有已知的磷酸化位点都被去除掉。具体是哪个磷酸化位点起主要作用?还是整体位点在其中起作用,尚待进一步的深入探讨。从而为进一步研究HDAC2Sumo-E3连接酶活性的调控机制奠定基础,同时也为促进创面愈合提供新的药物靶点。
[1] Witt O,Deubzer HE,Milde T,et al.HDAC family:What are the cancer relevant targets?[J].Cancer Lett,2009,277(1):8-21.
[2] Nakagawa M,Oda Y,Eguchi T,et al.Expression profile of class I histone deacetylases in human cancer tissues[J].Oncol Rep,2007,18(4):769-774.
[3] Xu X,J Vatsyayan,C Rao,et al.HDAC2promotes eIF4E sumoylation and activates mRNA translation gene specifically[J].J Biolgi Chem,2010,285(24):18139-18143.
[4] Karwow ka-Desaulniers P,Ketko A,Kamath N,et al.Histone deacetylase 1phosphorylation at S421and S423 is constitutive in vivo,but dispensable in vitro[J].Biochem Biophys Res Commun,2007,361(2):349-355.
[5] Segre CV,Chiocca S.Regulating the regulators:the posttranslational code of classⅠHDAC1and HDAC2[J].J Biomed Biotechnol,2011:837-848.
[6] Deribe YL,Pawson T,Dikic I.Post-translational modifications in signal integration [J].Nat Struct Mol Biol,2010,17(6):666-672.
[7] 郭韡,熊渊,黄宏,等.组蛋白去乙酰化酶HDAC2突变体构建及其SUMO 修饰E3连接酶功能研究[J].现代生物医学进展,2013,33(6):1005-1008.
[8] Sonenberg N,Hinnebusch AG.Regulation of translation initiation in eukaryotes:mechanisms and biological targets[J].Cell,2009,136(4):731-745.
[9] Gingras AC,Raught B,Sonenberg N.eIF4initiation factors:effectors of mRNA recruitment to ribosomes and regulators of translation [J].Annu Rev Biochem,1999(68):913-963.
[10] Johnson E.Protein modification by SUMO[J].Annu Rev Biochem,2004(73):355-382.
[11] Di Bacco A,Gill G.SUMO-specific proteases and the cell cycle.An essential role for SENP5in cell proliferation[J].Cell Cycle,2006,5(20):2310-2313.
[12] Geiss-Friedlander R,Melchior F.Concepts in sumoylation:a decade on[J].Nat Rev Mol Cell,2007,8(12):947-956.
[13] Xu X,Vatsyayan J,Rao C,et al.Sumoylation of eIF4Eactivates mRNA translation[J].EMBO Rep,2010,11(4):299-304.

