长三角及邻近地区138种草本植物DNA C-值测定及其生物学意义
郭水良,于 晶,李丹丹,周 平,方 其,印丽萍
1 上海师范大学, 生命与环境科学学院, 200234 2上海市出入境检验检疫局, 200135
长三角及邻近地区138种草本植物DNA C-值测定及其生物学意义
郭水良1,于 晶1,李丹丹1,周 平1,方 其1,印丽萍2,*
1 上海师范大学, 生命与环境科学学院, 200234 2上海市出入境检验检疫局, 200135
为了评估DNA C-值和基因组大小(genome size)在植物入侵性评估中的价值,应用流式细胞仪测定了长三角及邻近地区138种草本植物的核DNA含量,其中111种为首次报道。在此基础上比较了不同植物类群这两个值的差异,特别是入侵性与非入侵性植物这两个值的差异。结果表明:(1)138种草本植物平均DNA C-值为1.55 pg,最大者是最小者的37.17倍。127个类群平均基因组大小为1.08 pg,最大者是最小者的34.11倍;(2)统计了菊科(Asteraceae)、禾本科(Poaceae)、石竹科(Caryophyllaceae)、十字花科(Brassicaceae)、玄参科(Scrophulariaceae)、蓼科(Polygonaceae)、唇形科(Labiatae)和伞形科(Umbelliferae)的DNA C-值和基因组大小,发现禾本科植物的这两个值显著地大于其他7个科(P< 0.01)。单子叶的DNA C-值和基因组极显著地大于双子叶植物(P< 0.01);(3)杂草比非杂草具有更低的DNA C-值(P< 0.01)和基因组大小(P< 0.001);与DNA C-值相比,基因组大小在这两个类群之间的差异更为明显(P<0.001),这种现象也体现在菊科植物中。随着基因组(X1)和DNA C-值(X2)由大变小,植物的杂草性(入侵性,Y)由弱变强,两者关系分别符合:Y=2.2334-1.2847 ln(X1)(r=0.4612,P< 0.01)和Y=2.4421-0.7234 ln(X2)(r=0.2522,P< 0.01),DNA C-值和基因组大小可以作为植物入侵性评估的一个指标;(4)多倍体杂草的基因组极明显地小于二倍体杂草(P< 0.01),前者为后者的0.63倍。在非杂草中,多倍体基因组比二倍体的略小,前者仅为后者的0.84倍,差异不显著(P> 0.5)。菊科植物中多倍体杂草的基因组也显著地小于二倍体杂草(P< 0.1)。基因组变小和多倍体化相结合,进一步增强了植物的入侵性。在多倍体植物入侵性评估中,基因组大小比DNA C-值更有价值。
流式细胞术;DNA C-值;基因组大小;杂草;入侵性;倍性
DNA C-值指的是一个物种配子核中没有复制时的DNA含量,基因组大小是指单个染色体组中的DNA含量,采用DNA 2C-值除以其倍性的方法计算[1]。该值对每种生物而言基本上是恒定的,具有种的特征[2]。
生物由简单到复杂,低级向高级的进化,遗传信息肯定会增加,遗传信息的增加伴随着DNA C-值的增加。但是,人们发现亲缘关系相近的种类,或一些复杂性相似的植物,却有极大的DNA C-值差异[3]。
传统的观点认为,DNA C-值具有物种的特异性。但是随着研究工作的深入,人们发现DNA C-值在某些物种也有一定程度的变异。DNA C-值在种上和种下水平的变异,具有重要的生态学、分类学、进化学上的意义[4]。
随着DNA C-值数据的积累,人们开始应用统计方法解析它们在不同类群的分布特点及其可能的生物学意义。Bennett[5]对已有的数据统计发现,156种重要的世界性杂草比其它植物具有更低的核DNA C-值,恶性杂草的核DNA C-值比一般性杂草的要低;在澳大利亚的千里光属(Senecia)和印度的金合欢属(Acacia)植物中,外来入侵种比同属其它种类均具有较低的核DNA 含量[6-7]。郭水良等[8]对中国境内有分布的539中被子植物的DNAC-值和基因组大小在单子叶植物与双子叶植物、不同生活型、不同科、不同倍性,特别是在杂草与非杂草之间的差异进行了统计;陈国奇等[9]以世界范围内2006年以前积累的3676种被子植物DNA C-值和基因组大小为材料,作了类似的统计工作,均得出了这样一个重要结论:杂草或入侵性植物的DNA C-值极显著地小于非杂草,随着植物DNA C-值和基因组由大变小,它们的杂草性增强,杂草中多倍化类群的基因组又极显著地小于二倍体类群。为探讨DNA C-值为植物入侵性之间的关系,Rejmánek和Richardson[10]对松属(Pinus)的DNA C-值与入侵性之间的关系进行了研究,发现与非入侵性种相比,具有入侵性的物种具有低的核DNA C-值,从而使其种子相对较小,世代时间也较之本地种缩短,这些均增强了其入侵能力。
另外,DNA C-值与海拔、经纬度、温度等也有一定联系[11-12]。研究发现DNA含量与被子植物物种的多样性、物种灭绝的风险性以及对生境多样性的适应能力等都有一定关系[13]。
DNA C-值在种上和种下变异丰富多样,还没有找到明显的规律性。关于DNA C-值生态环境适应上的作用,至今的研究结论也不尽相同。付改兰和冯玉龙[14]等研究认为,作为整体,外来入侵植物的平均核DNA C-值显著低于本地种和外来非入侵种,但是具体到某一特定类群,核DNA C-值与植物入侵性无必然联系。Rejmánek & Richardson[10]对松属的入侵性木本植物种研究显示出核DNA C-值与生活史、种子大小、幼苗生长时间长短有关。尽管陈国奇等[9]对多达3676种被子植物的DNA C-值和基因组大小进行了统计分析,发现在整个草本植物类群,以及在二倍体、多倍体、不同生活型,以及在单子叶、双子叶和菊科、禾本科、毛茛科和鸭跖草科中,杂草的DNA C-值和基因组均显著地小于非杂草,然而在豆科、苋科、茄科和葱科中杂草与非杂草的两项指标上没有显著差异。而且还有众多科属由于样本太少无法进行统计检验。由于DNA C-值和基因组大小在被子植物的不同类群中差异极大,评估目的植物的入侵性时,必须与来自同一类群的植物进行比较。因此,要充分利用核 DNA C-值和基因组大小进行植物入侵性评估,需要通过测定获得更多植物的DNA C-值和基因组大小数据。
陈国奇等[9]统曾建立了植物入侵性与基因组大小之间的函数关系,发现不同的类群往往有不同的了回归公式。因此,在评估目的植物的入侵性前,应该在相同科或属的背景下建立入侵性与核DNA含量的定量关系。但是,相对于几十万种高等植物,目前已知DNA C-值的种类还非常有限[9]。郭水良等[8]统计发现,我国境内有分布的近3万种种子植物中仅有539种有DNA C-值的资料,而且绝大部分是国外学者测定。因此,需要在更多的植物类群中进行核DNA含量的测定,丰富草本植物的核DNA含量数据库,为不同类群入侵性的评估打下基础。
另外,虽然陈国奇等[9]和郭水良等[8]统计过DNA C-值和基因组大小在不同类群中的分布特点,但是这些数据均是基于不同学者,采用不同测定方法所得的数据,因此用这些数据进行分析,从实验设计上讲也不是十分的严谨。这是因为由于流式细胞仪生产厂家不同,同一厂家的型号不同,不同的细胞核悬液可能会影响植物DNA C-值的测定结果[15-16]。前人在测定植物DNA C-值时一般采用鸡血红细胞,Johnston等[17]研究发现使用鸡血红细胞作为标样时,从不同的鸡采取血液其2C-值在2.49 pg至3.01 pg之间变化,这又会影响实验结果。
杂草是某一自然地理区域,其种群主要分布在人为干扰的环境、本身又不是目的植物的类群[18]。因此,杂草是具有入侵性的植物类群。有研究表明,植物的入侵性与其杂草性(在原产地的分布特点和扩散程度)关系最为密切[19];Sutherland也曾以杂草为对象开展北美植物的入侵性研究[20]。
本文的目的,以长三角及邻近地区的部分杂草和非杂草为对象,1)应用流式细胞术在相同的条件下测定它们的DNA C-值;2)对获得的数据进行统计分析,了解其在不同类群中的变异特点,进一步评估DNA C-值、基因组大小和植物的杂草性之间的关系,进而探讨细胞核的这两个值在植物入侵性评估中的价值。本文工作也将丰富我国DNA C-值数据库含量,为更深入地了解DNA C-值的生物学、生态学和分类学意义提供基础数据和资料。
1 材料与方法
1.1 材料
材料采自长三角地区的上海、浙江和南通郊区野外空地、田埂和废弃工地等环境,共计39科102属134种4变种144个居群。每个分类群采集3份样品进行测定。为比较同一种植物不同形态差异的种群在DNA C-值上的差异,其中蒲公英(Taraxacummongolicum)、北美车前(Plantagoviriginica)、三色堇(Violatricolor)、春飞蓬(Erigeronphiladelphicus)、铜锤草(Oxaliscorymbosa)和半夏(Pinelliaternata)分别采集了两个不同性状的居群(表1)。
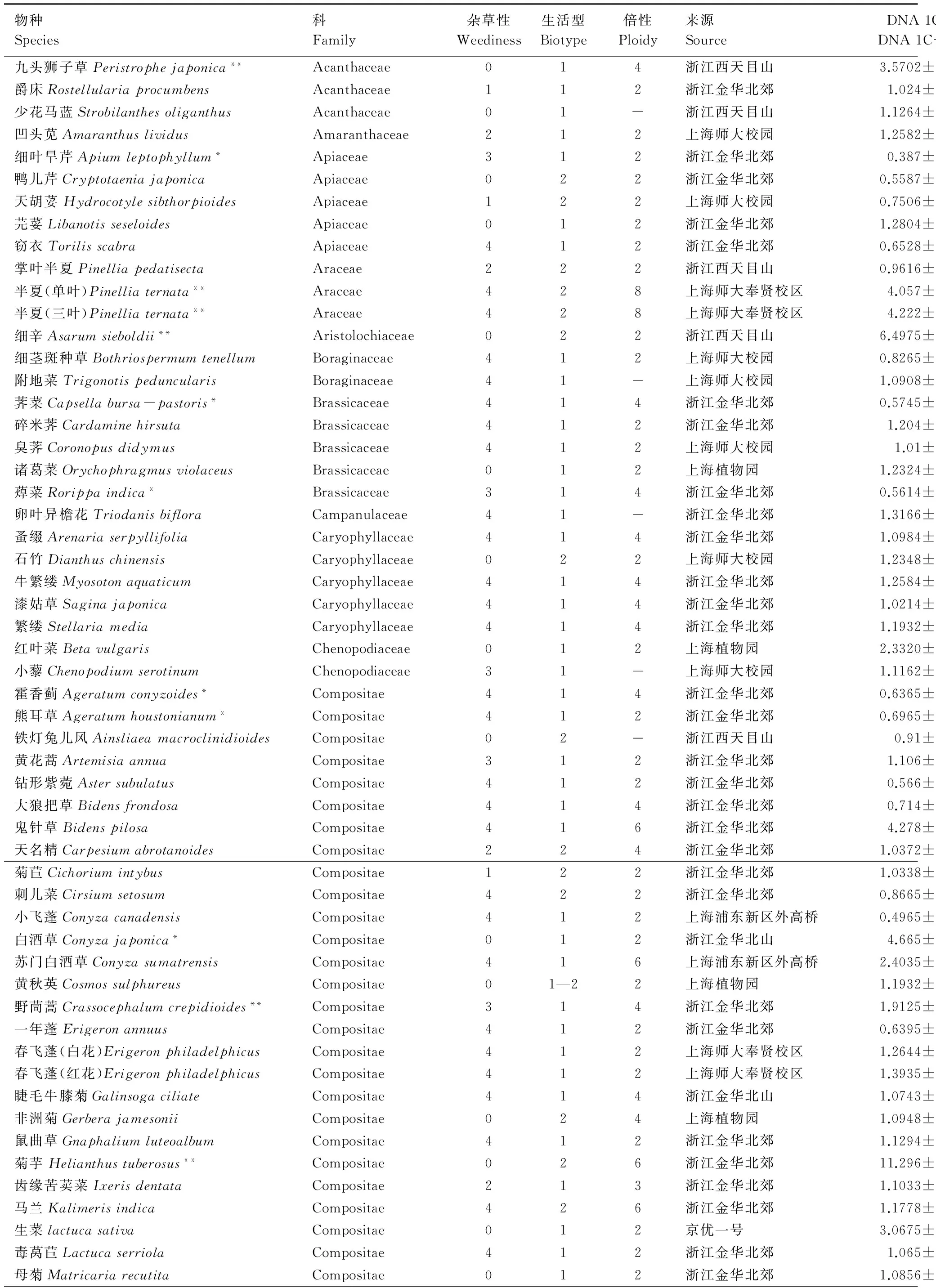
表1 138种草本植物DNA C-值和基因组大小
材料都直接从野外采集其新鲜幼嫩叶片,在两个小时内带回实验室测量C-值,如果无法短时间带回实验室测定的材料,将植株移植于实验室,待其恢复生长后取新鲜叶片实验。凭证标本存放于上海出入境检验检疫局植物与粮谷实验室。
小米(Setariaitalica)种子来源于山东,高粱(Sorghumbicolor)、假高粱(S.halepense)、黑高粱(S.almum)、苏丹草(S.sudanense)、拟高粱(S.propinquum)种子来自上海出入境检验检疫局,经萌发后置于20 ℃光照培养箱中培养获得实验材料。
1.2 方法
采用美国BD公司的FACSCalibur流式细胞仪测定,方法见宋平根和李素文文献[21]。
Otto缓冲液,包括Otto I和Otto II(Otto I:100 mmol/L 柠檬酸,0.5%(体积分数)Tween20,用去离子水定容至200 mL,pH值约2.3,4度保存;Otto II:0.4 mol/L Na2HPO4·12H2O 28.65 g, 用去离子水定容至200 mL,室温保存);PI-Rnase(基因公司 550825);双面刀片(飞鹰牌);50 μm尼龙网;离心机等。
选择大豆(2.4pg/2C)[22]、六倍体小麦(31pg/2C)[23]和油菜(1.4pg/2C)[24]为标样。
通过仪器自带ModFit LT 软件分析直方图,得到样品的G0/G1峰值即荧光值,和G0/G1峰的变异系数(CV%=标准差/平均值×100)[25],CV值小于3%说明结果准确,CV值小于5%基本正确,CV值大于8%的不能采用[26]。 峰值及CV值均能从ModFit LT 软件上直接读出,数据出现在直方图的右边。最后通过比较样品与标样的G0/G1峰值(即荧光值)来计算测试样核DNA C-值[27]。
在测定获得DNA 1C-值的基础上,查阅相关文献和植物染色体数目数据库(http://www.tropicos. org/Project/IPCN/),获得相应的倍性信息,从而计算基因组大小。基因组大小指单组染色体中的DNA含量,采用DNA 2C-值除以其倍性的方法计算。
为了解DNA C-值、基因组大小与植物杂草性之间的关系,根据Baker[18]关于杂草的定义,将138种植物首先分成杂草和非杂草两大类。Holm等[28]将世界范围内的杂草进一步细可分为普通杂草、主要杂草、恶性杂草和危害性不明的杂草。李扬汉[29]“中国杂草志”中对中国境内的杂草分布和危害也作了详细记载。结合以上资料和相关植物在长三角及邻近地区不同生态系统的分布特点,按入侵性(杂草性)由强到弱将138种植物分成0、1、2、3、4五个等级:
0级 主要见于天然植被环境、极少见于人为干扰环境中的非杂草植物;
1级 主要见于天然植被环境,偶见于人为干扰环境中,入侵性弱的杂草;
2级 多见于人为干扰环境,但是很少形成大面积入侵种群,少见于农田中的杂草;
3级 为主要杂草,常见于为人干扰环境,在农田环境也常见的杂草;
4级 多为恶性杂草,主要见于农田或人为干扰环境,能够形成成片优势种群。
不同类群间的显著性差异均通过SPSS11.0统计软件中的Independent-samples T-Test比较获得,数据用平均值±标准误表达。
2 结果
2.1 测定植物的DNA C-值和基因组大小
138个分类群的DNA-C值和基因组大小见表1,平均DNA C-值为1.55 pg,燕麦的最大(11.2115 pg),婆婆纳的最小(0.322 pg),两者差37.17倍。在能够计算基因组大小的127个类群中,平均基因组大小为1.08 pg,细辛的最大(6.4975 pg),最小者为蛇莓(0.1905 pg),两者差34.11倍。上述类群中,有111种的核DNA数量为首次报道。
2.2 测定的8个科的DNA C-值和基因组大小比较
菊科(Compositae)共计测定了39种,平均DNA C-值为1.6363pg,最大者为菊芋(11.296 pg),最小为小飞蓬(0.4965 pg);平均基因组大小为1.18418 pg,最大者为白酒草(4.665 pg),霍香蓟的最小(0.3183 pg)。
禾本科(Gramineae)测定了16种,平均DNA C-值为3.3329 pg,燕麦的最大(11.21115 pg),苏丹草的最小(0.7775 pg);平均基因组大小为1.5994 pg,最大者为燕麦(3.73720 pg),最小者为棒头草(0.3763 pg)。
在测定的5种石竹科(Caryophyllaceae)植物中,平均的DNA C-值为1.1612 pg,最大者为牛繁缕(1.2584 pg),最小为漆菇草(1.0214 pg);基因组的平均大小为0.7041 pg,最大者为石竹(1.2348 pg),最小为漆菇草(0.5107 pg)。
测定的五种十字花科(Cruciferae)植物的DNA C-值和基因组大小与石竹科的相近,它们平均的DNA C-值为0.9165 pg,最大者为诸葛菜(1.2324 pg),最小者为蔊菜(0.5614 pg);基因组大小平均值为0.8029 pg,最大也为诸葛菜(1.2324 pg),最小为荠菜(0.2807 pg)。
共测定了玄参科(Scrophulariaceae)的7种植物,平均的DNA C-值和基因组大小分别为0.9164 pg和0.6314 pg,前者最大者为通泉草(1.5955 pg),最小者为婆婆纳(0.322 pg),基因组最大者为弹刀子菜(1.1693 pg),最小者为蚊母草(0.2708 pg)。
测定的6种蓼科(Polygonaceae)植物平均的DNA C-值为1.0003 pg,最大者为金线蓼(1.4172 pg),最小的为蚕茧蓼(0.6436 pg);平均的基因组大小为0.7064 pg,最大者为愉悦蓼(1.4116 pg),最小的为蚕茧蓼(0.2415 pg)。
测定的5种唇形科(Lamiaceae)植物平均的DNA C-值和基因组大小分别为0.7990 pg和0.7164 pg,野芝麻的DNA C-值和基因组均为最大,为1.1117 pg,宝盖草的DNA C-值最小,为0.5956 pg,基因组最小的为风轮菜,仅为0.413 pg。
伞形科(Umbelliferae)5种植物的平均DNA C-值和基因组均为0.7259 pg,以栽培植物芫荽为最大(1.2804 pg),以细叶旱芹为最小(0.387 pg)。
就以上8个科测定的88种植物中,禾本科的DNA C-值最大,为2.6803 pg,伞形科的最小,为0.7259 pg,禾本科与其他7个科的DNA C-值均存在极显著的差异,但是其他科之间的DNA C-值没有统计学上的差异;基因组大小在8个科之间没有显著差异(表2)。
统计还表明,DNA C-值和基因组在单子叶和双子叶植物中存在极显著的差异(P< 0.01)(表3)。
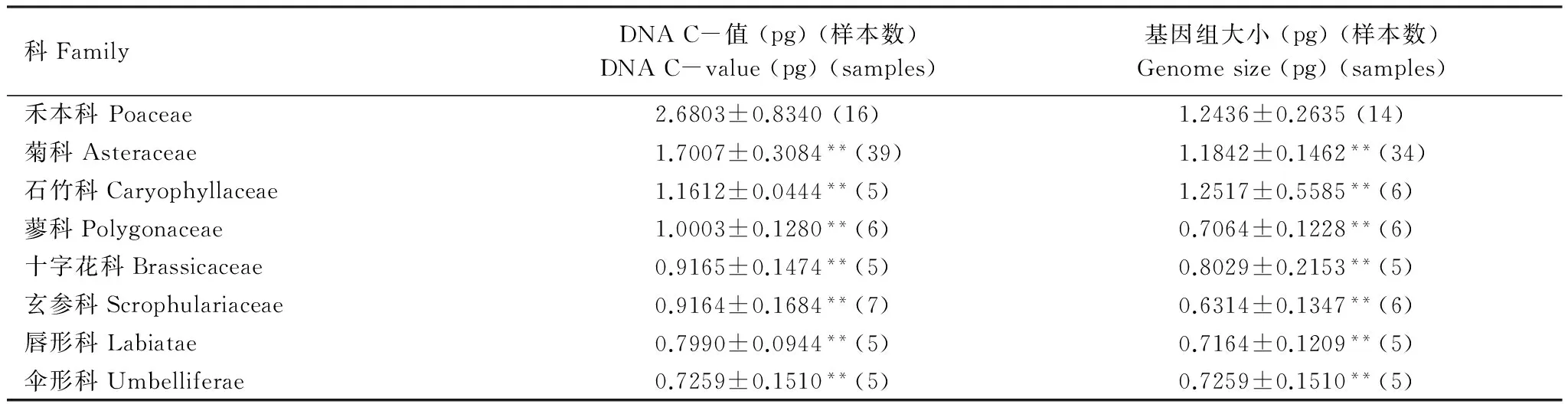
表2 八个科的DNA C-值和基因组大小

表3 单子叶和双子叶植物DNA C-值和基因组大小
2.3 DNA C-值和基因组大小在杂草和非杂草之间的比较
138种植物中,93种为杂草,45种非杂草。比较发现,与非杂草相比,杂草具有更低的DNA C-值(P<0.01)和基因组大小(P< 0.001),与DNA C-值相比,基因组大小在这两个类群之间的差异更为明显(表4)。
而且,随着基因组(X1)和DNA C-值(X2)由大变小,植物的杂草性(Y)均由弱变强,两者关系分别符合:Y=2.2334-1.2847 ln(X1) (r=0.4612,P< 0.01)和Y=2.4421-0.7234 ln(X2) (r=0.2522,P< 0.01)(图1)。
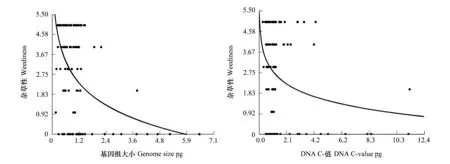
图1 核DNA含量和植物杂草性之间的关系Fig.1 Relationship of weediness with nuclear DNA amount
在测定的37种菊科植物中,杂草的DNA C-值和基因组大小也明显地小于非杂草(表5)。

表5 菊科杂草与非杂草DNA C-值和基因组大小
在菊科测定的36种杂草中,随着基因组(X1)和DNA C-值(X2)由大变小,植物的杂草性(Y)由弱变强,两者关系分别符合:Y=3.219-1.55 ln(X1) (r=0.4386,P< 0.01)和Y=3.532-1.05ln (X2) (r=0.3225,P< 0.1)。
比较还发现,在二倍体植物类群中,杂草比非杂草具有更小的基因组大小(P< 0.01);在多倍体类群中,杂草的基因组也显著地小于非杂草(P< 0.01)(表6)。
2.4 基因组大小在二倍体与多倍体植物之间的比较
本文检测的92种杂草中,二倍体有49种,多倍体有35种。统计发现,多倍体杂草的基因组极明显地小于二倍体杂草,前者为后者的0.63倍,差异极显著(P< 0.01)。而在非杂草中,多倍体基因组比二倍体的略小,前者仅为后者的0.84倍,差异不显著(P> 0.5)。菊科植物中多倍体杂草的基因组也显著地小于二倍体杂草(P< 0.1)(表7)。

表6 二倍体和多倍体基因组大小在杂草和非杂草之间的差异比较

表7 菊科杂草中二倍体和多倍体DNA C-值、基因组大小的比较
2.5 基因组大小和DNA C-值在不同生活型植物之间的比较
一、二年生草本植物的DNA C-值和基因组大小均小于多年生植物,但是这种差异没有达到统计上的显著相关(表8)。在96份杂草样品中,多年生杂草的基因组比一、二年生的要小,这种差异统计上也不显著(表9)。
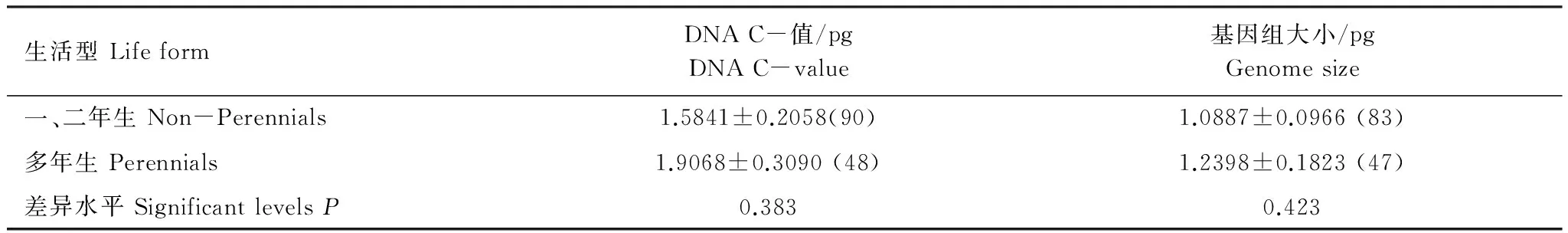
表8 DNA C-值和基因组大小在不同生活型草本植物之间的比较
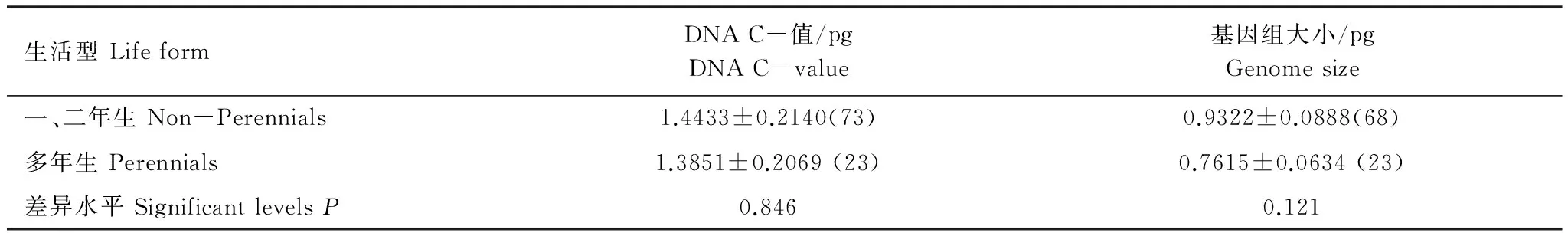
表9 DNA C-值和基因组大小在不同生活型杂草之间的比较
3 讨论
植物DNA C-值和基因组大小既有种的特征,又具有生态适应上的意义。本文基于138种草本植物的DNA C-值测定,结合倍性、生活型、杂草性等信息,发现DNA C-值和基因组大小在杂草与非杂草、不同分类群之间、不同倍性的类群之间以及在不同生活型中存在差异,有的达到显著或极显著差异。
陈国奇等[9]对世界范围的3676种被子植物、郭水良等[8]对中国境内有分布的539种被子植物的统计表明,DNA C-值和基因组大小在不同植物类群中差异极大,其平均值在单子叶植物中显著高于双子叶植物,多年生植物显著高于非多年生植物。在大多数类群中DNA C-值(基因组大小)都与被子植物的入侵性显著相关,如:草本、单子叶、双子叶、多年生、非多年生、二倍体、多倍体、以及禾本科和菊科中。因此,DNA C-值和基因组大小在植物入侵性评估中有一定的应用价值。本文研究得出了相同的结论。但是,前期的两篇论文数据来自于不同的文献,这些数据是通过不同测定方法获得的,用这类数据进行分析也不十分严谨。本文在同样条件下进行测定,数据的可比性更强,因此得出的结论更科学,尽管本文的结论与前期工作的相同。
在本文测定的已知倍性信息的138种植物中,84种杂草中41.67%的为多倍体,而36种非杂草中16.67%的种类为多倍体。前人研究也表明,多倍化是形成杂草的重要途径[53]。另一方面,在84种杂草中,35种多倍体种类的基因组又极明显地小于二倍体种类。可以看出,基因组变小和多倍体化相结合,进一步增强了植物的杂草性。因此,在植物入侵性评估中,目的植物的染色体倍性信息非常重要,对于多倍体而言,基因组大小更适合用于被子植物的入侵性评估。
与非杂草性植物相比,杂草种类具有更低的DNA C-值和基因组大小,随着基因组和DNA C-值由大变小,植物的杂草性由弱变强,两者关系极为显著。陈国奇等[9]统计也发现,当植物的基因组大于DNA 1C-值≤1.4 pg时,其成为杂草的概率是26.06%,成为恶性杂草的概率为7.50%;当该值超过35 pg时,杂草的概率为1.24%,而统计中没有草本恶性杂草的DNA 1C值高于35 pg。然而,不同的类群往往有不同的回归公式,因此在评估目的植物的入侵性前应该选择一个最有效的公式。因此,需要统计更多的研究,丰富草本植物的核DNA含量数据库,为不同类群入侵性的评估打下基础。
DNA C-值和基因组小的植物具有更强的杂草性有一定的生物学基础。DNA C-值低的植物,其核基因组中就含有较少的非编码序列和重复序列,其有丝分裂周期短,细胞分裂速率快、世代缩短、幼苗生长速率高,这最终决定了其能在有限的时间内顺利完成生活周期,从而得以成功适应新的环境。一个物种的最短世代时间决定着其在特定有限时间环境中的生存和繁殖能力,而DNA C-值可以限制一个物种的生活史类型[54-55]。因此DNA C-值与植物的杂草性相联系[5,9,56]。
DNA C值的检测方法主要有化学分析法、孚尔根染色法[57]、复性动力学法[58]和流式细胞术等4种方法。前三种方法目前已很少使用。流式细胞术是近年来受到重视的测定植物DNA C-值的方法。在应用流式细胞术测定植物的DNA C-值过程中,需要选择适用的缓冲液。细胞核分离缓冲液的组成不仅与DNA含量分辨率有关,并且会影响细胞核从细胞质中释放、分离后细胞核的完整性,以及DNA的染色效果。选择的缓冲液通常由多种成分组成,要保持一定浓度的Mg2+,需要螯合剂柠檬酸钠等;为了使溶液的pH值与DNA荧光染料的一致,还需要添加TRIS和MOPS等有机物,从而使缓冲液pH值保持相对稳定。本文选择Otto缓冲液,实验结果比较稳定。前人在测定植物DNA C值时一般采用鸡血红细胞,但是文献报道的作为内标的鸡血红细胞2C-值并不恒定。另外,由于动物材料中存在的性染色体,所以不同性别的材料会存在 DNA C-值差异[59]。植物材料取材方便,制备过程简便。本文选择标样时,考虑到测定的目标植物的DNA C-值与标样的值比较接近为原则,以使测定结果更加可靠。
陈国奇等[9]统计了3676种被子植物的DNA C-值和基因组大小。发现草本被子植物平均的DNA C-值和基因组大小分别为7.04 pg和5.21 pg。本文测定的138种植物的DNA C-值和基因组大小明显地要小。可能的原因是,陈国奇等[9]统计的3676种植物中,非杂草占了84.98%,单子叶植物占了41.84%中,而本文中涉及的138种植物中,非杂草占的比例仅34.78%,单子叶植物为15.22%。由于非杂草的DNA C-值和基因组明显地大于杂草,单子叶植物的这两个值也极显著地大于双叶子植物;其次由于不同的科属的DNA C-值和基因组大小差异极大,例如陈国奇等[9]统计的3676种植物中,DNA C-值大的达127.4 pg, 小的仅为0.1 pg, 大小相差1274倍,而基因组最大的有74.45 pg,最小的为0.07 pg,大小相差1064倍。取材上的差异,也会造成测定数据的差异。
真核生物的核DNA由编码和非编码两部分组成,而非编码DNA则占了绝大部分,构成了细胞染色体的主体框架[60]。细胞核中的编码DNA可以通过编码蛋白质直接影响生物的性状,同时核DNA本身的重量及体积也可以影响生物的表型[61]。已有资料表明,DNA C-值与与被子植物物种的多样性、物种灭绝的风险性以及对生境多样性的适应能力等都有一定关系[13]。因此,今后应该进一步开展植物DNA C-值和基因组大小的测定,丰富被子植物DNA C-值数据库,为深入地揭示DNA C-值的生物学意义积累数据资料。
[1] Leitch I J, Bennett M D. Genome downsizing in polyploid plants. Biological Journal of the Linnean Society, 2004, 82(4): 651-663.
[2] Mishiba K I, Ando T, Mii M, Watanabe H,Kokubun H,Hashimoto G, Marchesi E. Nuclear DNA content as an index character discriminating taxa in the genusPetuniasensuJussieu (Solanaceae). Annals of Botany, 2000, 85(5): 665-673.
[3] 苏筱娟. 非编码DNA到底有没有用? 国外科技动态, 2000, (11): 32-33.
[4] 郭水良, 周平, 印丽萍, 娄玉霞. 植物DNA C-值在种间和种内的变异及其生物学意义. 上海师范大学学报: 自然科学版, 2011, 40(1): 102-110.
[5] Bennett M D. Plant genome values: How much do we know? Proceedings of the National Academy of Sciences of the United States of America, 1998, 95(5): 2011-2016.
[6] Lawrence M E.SenecioL. (Asteraceae) in Australia: nuclear DNA amounts. Australian Journal of Botany, 1985, 33(2): 221-232.
[7] Hughes C E, Styles B T. The benefits and potential risks of woody legume introductions. International Tree Crops Journal, 1987, 4(4): 209-248.
[8] 郭水良, 陈国奇, 毛俐慧. DNA C-值与被子植物入侵性关系的数据统计分析——以中国境内有分布的539种被子植物为例. 生态学报, 2008, 28(8): 3698-3705.
[9] Chen G Q, Guo S L, Yin L P. Applying DNA C-values to evaluate invasiveness of angiosperms: validity and limitation. Biological Invasions, 2010, 12(5): 1335-1348.
[10] Rejmánek M, Richardson D M. What attributes make some plant species more invasive? Ecology, 1996, 77(6): 1655-1661.
[11] 李国旗, 安树青, 陈兴龙, 程晓莉. 生物的C值矛盾与其生态适应性的关系初探. 大自然探索, 1999, 18(2): 61-66.
[12] Bancheva S, Greilhuber J. Genome size in BulgarianCentaureas. l. (Asteraceae). Plant Systematics and Evolution, 2006, 257(1/2): 95-117.
[13] Kraaijeveld K. Genome size and species diversification. Evolutionary Biology, 2010, 37(4): 227-233.
[14] 付改兰, 冯玉龙. 外来入侵植物和本地植物核 DNA C-值的比较及其与入侵性的关系. 生态学杂志, 2007, 26(10): 1590-1594.
[15] 方其, 印丽萍, 郭水良. 应用流式细胞术测定植物DNA C-值的实验方法研究. 植物检疫, 2011, 25(2): 40-44.
[16] Doležel J. Flow cytometric analysis of nuclear DNA content in higher plants. Phytochemical Analysis, 1991, 2(4): 143-154.
[17] Johnston J S, Bennett M D, Rayburn A L, Galbraith D W, James H. Reference standards for determination of DNA content of plant nuclei. American Journal of Botany, 1999, 86(5): 609-613.
[18] Baker H G. The evolution of weeds. Annual Review of Ecology and Systematics, 1974, 5: 1-24.
[19] Williamson M H, Fitter A. The characters of successful invaders. Biological Conservation, 1996, 78(1/2): 163-170.
[20] Sutherland S. What makes a weed a weed: life history traits of native and exotic plants in the USA. Oecologia, 2004, 141(1): 24-39.
[21] 宋平根, 李素文. 流式细胞术的原理和应用. 北京: 北京师范大学出版社, 1992.
[22] Bennett M D, Leitch I J. Nuclear DNA amounts in angiosperms. Annals of Botany, 1995, 76(2): 113-176.
[23] Hanson L, McMahon K A, Johnson M A T, Bennett M D. First nuclear DNA C-values for 25 angiosperm families. Annals of Botany, 2001, 87(2): 251-258.
[24] Bennett M D, Leitch I J. Nuclear DNA amounts in angiosperms-583 new estimates. Annals of Botany, 1997, 80(2): 169-196.
[25] 金亮, 张宪银, 薛庆中. 分离缓冲液对水稻细胞核悬液 DNA 分辨率效应的比较. 浙江农业学报, 2007, 19(2): 93-96.
[26] 杜立颖, 冯仁青. 流式细胞仪. 北京: 北京大学出版社, 2008.
[27] Seker M, Tuzcu O, Ollitrault P. Comparison of nuclear DNA content of citrus rootstock populations by flow cytometry analysis. Plant Breeding, 2003, 122(2): 169-172.
[28] Holm L G, Pancho J V, Herberger J P. A Geographical Atlas of World Weeds. New York: John Wiley and Sons, 1979.
[29] 李扬汉. 中国杂草志. 北京: 中国农业出版社, 1998.
[30] 葛传吉, 李岩坤, 周月, 徐炳声. 山东地区药用植物染色体数目的观察(III). 云南植物研究, 9(3): 333-338.
[31] 李名旺, 顾德兴, 刘友良, 徐炳声. 半夏属的染色体数目、倍性与珠芽发生的关系. 植物分类学报, 1997, 35(3): 208-214.
[32] 张守栋, 王庆亚, 顾德兴. 繁缕和无瓣繁缕六个居群的数值分析. 云南植物研究, 2002, 24(2): 185-190.
[33] 张延昌, 张永明. 鬼针草的研究现状与应用前景. 山东教育学院学报, 2004, 19(4): 79-83.
[34] 鄢本厚, 尹祖棠. 蓟属两种植物的染色体研究. 广西植物, 1995, 15(2): 172-175.
[35] 何文奇. 翅果菊属PterocypselaShih (菊科—菊苣族)分类学研究[D]. 郑州: 郑州大学, 2012.
[36] 李国泰, 刘海滨. 蒲公英染色体核型分析. 中国林副特产, 2004, (6): 11-12.
[37] 葛传吉, 李岩坤, 徐炳声. 苍耳XanthiumstrumariumL. 染色体数目和核型的观察. 武汉植物学研究, 1985, 3(4): 433-437.
[38] 严寒, 周胜梅, 田志宏. 马蹄金染色体的核型分析. 长江大学学报: 自然科学版, 2005, 2(2): 25-27.
[39] Chen R Y, Song W Q, Li X, Li M X, Liang G, Chen C B. Chromosome alas of major economic plants genome in China, Vol. 3, chromosome alas of garden flowering plants in China. Beijing: Science Press, 2003.
[40] 杨德奎, 周俊英. 山东10种植物的核型分析. 广西植物, 1999, 19(4): 349-354.
[41] 方永鑫, 杨斌生, 欧善华. 黄精属几个种的染色体研究. 上海师范学院学报: 自然科学版, 1984, (1): 66-76.
[42] 时丽冉, 高汝勇, 李会芬, 李慧凝. 紫茉莉染色体数目及核型分析. 草业科学, 2010, 27(1): 52-55.
[43] Xu B S, Weng R F, Zhang M Z. Chromosome numbers of Shanghai plants I. Investigatio Et Studium Naturae, 1992, 12: 48-65.
[44] 王丰, 郭继明, 郭灵云. 北美车前染色体核型研究. 植物学通报, 1993, 10(2): 57-58.
[45] 杨瑞武, 周永红, 郑有良. 鹅观草(Roegneriakamoji)的染色体C带分析. 广西科学, 2002, 9(2): 138-141.
[46] 李娜, 印丽萍, 郭水良. 高粱属4种植物的核型及其与入侵性关系探讨. 植物检疫, 2009, 23(6): 6-9.
[47] 许崇梅. 山东蓼族植物染色体和基于 ITS 序列探讨杠板归的系统位置的研究[D]. 济南: 山东师范大学, 2004.
[48] Liao L, Xu L L, Chen Y, Fang L. Studies of karyotypes ofRanunculuscantoniensispolyploid complex and its allied species. Acta Phytotaxonomica Sinica, 1995, 33(3): 230-239.
[49] 袁艺, 王莲, 王小娟. 不同居群鱼腥草的核型比较. 园艺学报, 2008, 35(9): 1377-1383.
[50] 杨永年, 张海洋, 吴国宜. 中国龙葵复合种细胞学分析和地理分布的研究. 植物研究, 1994, 14(2): 208-213.
[51] 陈又生. 中国堇菜属的分类修订[D]. 北京: 中国科学院研究生院 (植物研究所), 2006.
[52] 孔继君, 刘刚. 姜科植物研究进展. 西北植物学报, 2005, 25(8): 1692-1699.
[53] 李君, 强胜. 多倍化是杂草起源与演化的驱动力. 南京农业大学学报, 2012, 35(5): 64-76.
[54] Smith J B, Bennett M D. DNA variation in the genusRanunculus. Heredity, 1975, 35(2): 231-239.
[55] Bennett M D. Variation in genomic form in plants and its ecological implications. New Phytologist, 1987, 106(S1): 177-200.
[56] Rejmánek M. Invasive plants: approaches and predictions. Austral Ecology, 2000, 25(5): 497-506.
[57] Cavalier-Smith T. Eukaryote gene numbers, non-coding DNA and genome size // Cavalier-Smith T. The Evolution of Genome Size. Chichester: John Wiley & Sons, 1985: 69-103.
[58] 陈建华. C-值悖论及其机理. 洛阳大学学报, 1994, 9(2): 14-20.
[59] Bennett M D, Smith J B. Nuclear DNA amounts in angiosperms. Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences, 1976, 274(933): 227-274.
[60] Britten R J, Graham D E, Neufeld B R. Analysis of repeating DNA sequences by reassociation // Grossman L, Moldave K. Methods in Enzymology. New York: Academic Press, 1974, 29: 363-418.
[61] Vindel D L L, Christensen I J, Jensen G, Nissen N I. Limits of detection of nuclear DNA abnormalities by flow cytometric DNA analysis. Results obtained by a set of methods for sample-storage, staining and internal standardization. Cytometry, 1983, 3(5): 332-339.
DNA C-values of 138 herbaceous species and their biological significance
GUO Shuiliang1,YU Jing1, LI Dandan1,ZHOU Ping1,FANG Qi1,YIN Liping2,*
1.CollegeofLifeandEnvironmentSciences,ShanghaiNormalUniversity,Shanghai200234,China2.ShanghaiEntry-ExitInspectionandQuarantineBureau,Shanghai200135,China
The DNA C-value is an important biological concept and it has been used in many areas of biological research. The C-value can be used as an index for evaluating angiosperm invasiveness. However, it is still uncertain how the DNA C-value influences plant invasiveness because there is a paucity of systematic experiments that test all the important aspects of this question. There is also a lack of data that identify the relationships between DNA C-value and invasiveness using invaders and non-invaders from the same genus or family. Therefore, expanding the database of plant DNA C-values is important for elucidating the mechanism by which the DNA C-value influences plant invasiveness. Using a cytometry method, we determined the DNA C-values for 138 herbaceous species collected from the Yangtze River Delta and its neighboring area. Among these species, 111 were newly reported. According to related literature and the plants′ distributions in artificial habitats, we also evaluated the weediness of these 138 species, and classified them using five grades: 0, 1, 2, 3, and 4. Based on these data, we calculated the genome sizes of 127 species with known ploidy levels and compared the differences in the two nuclear values (DNA C-value and genome size) between families, monocots and dicots, polypoid and diploid species, and invasive and non-invasive groups. The results showed that: (1) The average DNA C-value of the 138 herbaceous species was 1.55 pg, with the maximum 37.17 times greater than the minimum. The average genome size of the 127 species with known ploidy was 1.08 pg, with the maximum 34.11 times greater than the minimum; (2) We compared the average DNA C-value/genome size (pg) among Poaceae (2.6803/1.2436), Asteraceae (1.7007/1.2436), Caryophyllaceae (1.1612/1.1842), Brassicaceae (0.9165/0.8029), Scrophulariaceae (0.9164/0.6314), Polygonaceae (1.0003/0.7064), Labiatae (0.7990/0.7164), and Umbelliferae (0.7259/0.7259), and found that these two values in Poaceae were significantly higher than in those in the other seven families (P< 0.01). Additionally, the two values in monocots were significantly higher than those in dicots; (3) The average DNA C-value of weeds was significantly lower than that of non-weeds (P< 0.01), while the difference in genome sizes between the two groups was more significant than that of the DNA C-values (P< 0.001). A similar situation was found for Asteraceae. With the decrease of genome size (x1) and DNA C-value (x2) in the focal plants, their weediness (or invasiveness,y) increased, followingy=2.2334-1.2847ln(x1) (r=0.4612,P< 0.01) andy=2.4421-0.7234ln(x2) (r=0.2522,P< 0.01). We suggest that the DNA C-value and genome size could be used as indicators to evaluate plant invasiveness and; (4) The genome sizes of polyploid weeds were significantly lower than those of diploid weeds (P< 0.01), with the former 0.63 times greater than the latter. The genome size of non-weedy polyploid plants was not significantly lower than that of diploid plants (P> 0.5). In Asteraceae weeds, the genome sizes for polyploids were significantly lower than those for diploids (P< 0.1). Our analyses showed that genome downsizing with polyploidy enhances plant invasiveness. In the evaluation of polyploid plant invasiveness, knowing the genome size is more valuable than knowing the DNA C-value. The expansion of the plant DNA C-values database is also useful for comprehensively understanding the variation of plant DNA C-values among different taxa, and it has possible uses in taxonomic, systematic, ecological, and evolutionary studies.
flow cytometry; DNA C-value; genome size; weeds; invasive; ploidy
国家自然科学基金项目(31070479); 上海市科委基础研究重点项目(10JC1412100); 国家出入境检验检疫局科研项目(200810787); 上海市教委科技创新重点项目(06ZZ20, NO.10ZZ81)
2014-06-11;
2014-12-05
10.5846/stxb201406111208
*通讯作者Corresponding author.E-mail: yinliping@hotmail.com
郭水良,于晶,李丹丹,周平,方其,印丽萍.长三角及邻近地区138种草本植物DNA C-值测定及其生物学意义.生态学报,2015,35(19):6516-6529.
Guo S L,Yu J, Li D D,Zhou P,Fang Q,Yin L P.DNA C-values of 138 herbaceous species and their biological significance.Acta Ecologica Sinica,2015,35(19):6516-6529.

