HaCaT 细胞中FUT4表达与其启动子区甲基化的相关性分析
李洪艳,佟少明,燕秋
1. 辽宁师范大学生命科学学院,大连 116081;2. 大连医科大学基础医学院,生物化学与分子生物学教研室,大连 116044
HaCaT 细胞中FUT4表达与其启动子区甲基化的相关性分析
李洪艳1,佟少明1,燕秋2
1. 辽宁师范大学生命科学学院,大连 116081;2. 大连医科大学基础医学院,生物化学与分子生物学教研室,大连 116044
岩藻糖基转移酶Ⅳ(Fucosyltransferase Ⅳ,FUT4)在正常细胞中表达量很低,但其低表达的调控机制以及是否受其启动子甲基化调控并不十分清楚。文章采用Western blot、免疫荧光和Real-time PCR的方法检测正常人永生化表皮细胞系HaCaT细胞FUT4的表达,观察DNA甲基转移酶抑制剂5-aza-dC处理对FUT4表达的影响。应用甲基化特异性PCR方法分析HaCaT细胞中FUT4启动子甲基化状态。结果表明,HaCaT细胞中FUT4的表达水平明显低于人表皮鳞癌细胞A431和SCC12。5 µmol/L的5-aza-dC处理72 h的HaCaT细胞,其FUT4 mRNA水平明显升高,并且与未经5-aza-dC处理的对照组相比,U引物扩增检测到的产物量增加,M 引物扩增检测到的产物量明显减少。这些结果表明,HaCaT细胞中FUT4的低表达可能与其启动子区CpG岛甲基化有关。
岩藻糖基转移酶Ⅳ;低表达;甲基化;HaCaT细胞
细胞表面的糖蛋白或糖脂是细胞与细胞之间、细胞与细胞外基质之间传递信号的纽带,而糖蛋白和糖脂上的寡糖链是其行使功能的重要组成部分。其中,Lewis 寡糖如Lewis a (Lea)、sLewis a (sLea)、Lewis b (Leb)、Lewis X (LeX)、sLewis X (sLeX) 和Lewis Y (LeY)是寡糖中的一类,其共同特点是都含有岩藻糖分子 Fucose,因此这些寡糖又可称为岩藻糖寡糖[1]。
岩藻糖基转移酶(Fucosyltransferase,FUT)负责催化岩藻糖寡糖的合成,迄今为止,共发现 11个FUT,分别命名为FUT1-FUT11。其中FUT4是合成LeY 寡糖的关键酶,它以[Fucα1→2Galβ1→4Glc-NAcβ1→R]为受体,以α-1,3糖苷键将岩藻糖基(Fucose)连接到 N-乙酰葡萄糖上形成[Fucα1→2Galβ1→4(Fucα1→3)GlcNAcβ1→R],即 LeY寡糖分子。该寡糖存在于多种蛋白分子上,如表皮生长因子受体EGFR,从而完成蛋白分子之间、细胞之间的相互识别和作用。研究表明,FUT4和LeY在细胞生理及病理条件下的细胞信号转导过程中都发挥着重要作用[2,3]。FUT4在上皮来源的癌中表达水平升高[4~8]。用FUT4的过表达质粒转染A431细胞,发现细胞中FUT4表达明显增加,对细胞的生长具有促进作用;而用FUT4 RNA干扰质粒转染A431细胞,FUT4的表达明显下调,细胞的增殖及移植瘤的生长均被抑制[1,9,10]。肿瘤组织中的LeY寡糖表达水平比正常组织中的表达水平明显升高。不同的肿瘤以及肿瘤的不同分期,LeY的表达量也不同,因此LeY的表达水平成为肿瘤相关性的诊断、治疗、预后的一种标志物[11,12]。用LeY的特异性抗体可以封闭表皮生长因子受体介导的信号转导途径,从而抑制癌细胞的增殖和肿瘤的生长[13,14]。
然而,FUT4在肿瘤细胞和正常细胞中差异表达的调控机理并不十分清楚,本实验室前期研究已经发现FUT4在人表皮鳞癌细胞A431和SCC12细胞中的表达水平与启动子的不同甲基化水平有关[l5]。本研究旨在探讨正常细胞中 FUT4的表达水平及其是否也受启动子甲基化调控,为 FUT4在不同细胞中表达水平的差异性调控提供更多的实验依据,同时为临床上癌症的诊断和治疗提供新思路。
1 材料和方法
1.1 细胞
人永生化表皮细胞系HaCaT细胞和人表皮鳞癌细胞系A431细胞均购自美国ATCC细胞库,人表皮鳞癌细胞系SCC12细胞由哈佛大学医学院皮肤病学系Dr. James Rheinwald 惠赠。所有细胞均培养于添加10%的胎牛血清、100 U/mL青霉素、50 μg/mL链霉素的DMEM/F12培养基中(Gibibco公司),培养条件5%CO2、37℃。
1.2 方法
1.2.1 Real-time PCR
应用Trizol 试剂(Invitrogen公司)提取HaCaT、A431和SCC12细胞的总RNA,具体操作如下:6孔板培养的细胞加入1 mL Trizol,反复吹打混匀3~5 min,将细胞裂解液收集到 1.5 mL离心管里,冰上放置10 min。每管加入氯仿0.2 mL,充分振荡15 s,室温下静置3~5 min。4℃、10 000 r/min离心15 min,将上清移入另外的离心管中。加入0.5 mL的异丙醇(或与上清等体积的异丙醇),-20℃放置30 min。4℃、10 000 r/min离心10 min。弃异丙醇,用DEPC处理过的水配制70%乙醇洗涤沉淀。4℃、10 000 r/min离心5 min,弃乙醇,沉淀在空气中自然干燥,加入50 µL DEPC水溶解沉淀,琼脂糖凝胶电泳检测RNA的完整性。紫外分光光度计检测RNA纯度和含量,并将浓度调成1 µg/µL。
用DNA酶消化后按照M-MLV 反转录酶说明书进行常规反转录反应,获得 cDNA,然后采用染料法进行 Real-time PCR,扩增FUT4基因。FUT4扩增引物为:5′-CTCAGGCCGTGCTTTTCCA-3′(Forward)和5′-GTAGTCCAACACGCGCAGAT-3′(Reverse)。GAPDH扩增引物为:5′-ATGGGGAAGGTGAAGGTCG-3′(Forward)和5′-GGGGTCATTGATGGCAACAATA-3′(Reverse) (TaKaRa公司)。反应条件:95℃预变性30 s;95℃变性5 s,60℃复性10 s,45个循环,选取15~20个循环处的数据进行分析。按照以上程序,进行 3次重复实验。基因表达的定量分析采用比较CT值法,GAPDH作为内参基因,用以校正每个样品中总RNA量的偏差。将比较CT值法得到的数据绘制成柱形统计图,根据柱形图纵坐标的数值衡量 FUT4的相对表达量的高低及各样本之间表达倍数关系。
1.2.2 5-aza-dC对细胞的处理
5-aza-dC (Sigma公司) 用冰醋酸配成0.5 mol/L的保存母液,-80℃保存。由于该药极不稳定,需现用现配,每个药物处理周期前根据使用量取出适量母液稀释成0.05 mol/L,并分装成20 µL/管,-20℃保存。细胞铺板 24 h后,用终浓度为 5 µmol/L 的5-aza-dC处理细胞,每24 h更换一次培养液,并向培养液中重新加入新鲜的5-aza-dC,药物作用72 h后收取细胞,可用于提取总DNA,RNA或总蛋白。
1.2.3 甲基化特异性PCR(Methylated-specific PCR, MSP)
采用苯酚法提取总DNA。具体操作如下:6孔板培养细胞至90%以上的融合度,胰酶消化并离心。细胞沉淀中加入0.5 mL DNA 提取缓冲液,同时加入RNase,在37℃水浴中保温30 min。然后加入2 µL蛋白酶K,并在50℃下保温2 h。加入等体积的饱和酚,充分震荡混匀,10 000 r/min离心10 min。将上层水相转入另一个 Ep管中,加等体积氯仿/异戊醇(24:1),充分混匀后,10 000 r/min离心10 min。将上层水相转入新的Ep管中,加入3 mol/L的NaAC 50 µL,2倍体积的无水乙醇,离心后沉淀用 70%乙醇离心洗涤一次,保留沉淀,自然干燥,加适量的无菌水或TE溶解。琼脂糖电泳鉴定并用紫外分光光度计定量所提取的DNA,然后按照EZ DNA Methylation-Gold 试剂盒(ZYMO REASCHER公司)说明书操作,使非甲基化的CpG转变成UpG,而甲基化的CpG不变。以试剂盒处理过的DNA为模板进行甲基化特异性PCR。M对引物:5′-CGGGTTGTTTTTATAATTCGATC-3′和5′-AATAACGTCGACTTCCTACCGT-3′(TaKaRa公司)。反应条件:94℃预变性5 min;然后94℃ 30 s,48℃ 1 min,72℃ 30 s,35个循环;最后72℃延伸10 min。U 对引物:5′-TGGGTTGTTTTTATAATTTGATTGT-3′和5′-AAAATAACATCAACTTCCTACCATT-3′(TaKaRa公司)。反应条件:94℃预变性5 min;然后94℃ 30 s,45℃ 1min,72℃ 30 s,35个循环;最后72℃延伸10 min。PCR产物经1.5 %琼脂糖凝胶电泳检测。
1.2.4 Western blot
6孔板培养的细胞,PBS清洗后,加入适量的蛋白变性裂解液,用细胞刮刀将细胞收集到1.5 mL离心管中,4℃冰箱放置2 h,期间在振荡器上间歇震荡3次。离心后,以BSA为标准品,考马斯亮蓝法定蛋白含量。根据蛋白定量结果,调整每个样品的总蛋白上样量,进行SDS-PAGE电泳,电泳后的凝胶按照常规方法转移到 NC膜上。转膜后,将膜放于 5 %脱脂奶粉中进行封闭,封闭后根据分子量大小将膜剪成上下两部分,并分别与FUT4、β-actin单克隆或多克隆抗体(Proteintech公司)免疫杂交,经过夜结合,次日PBS清洗多余一抗后,进行二抗结合,PBS清洗掉多余二抗后,采用ECL法显示蛋白的表达[15]。
1.2.5 免疫荧光
在正常状态下爬片培养的细胞,经 PBS漂洗、冰丙酮固定、破膜、正常羊血清封闭,加入 FUT4特异性一抗孵育液(Santa Cruz公司),37℃孵育1 h 或4℃过夜。细胞经PBS漂洗并加入罗丹明标记的荧光二抗,37℃孵育1 h,PBS漂洗,封片后,细胞在荧光显微镜下扫描并拍照,检测 FUT4蛋白在细胞中表达情况[15]。
1.2.6 统计学分析
所有的实验均重复3次,数据采用T检验进行分析,当 P<0.05时具有显著性差异,当 P<0.01时具有极显著性差异。
2 结果与分析
2.1 FUT4在人永生化表皮细胞系 HaCaT细胞中的表达
采用Western blot的方法(图1A)对HaCaT细胞中 FUT4的表达水平进行了检测。结果表明,与作为对照的人表皮鳞癌细胞系A431和SCC12细胞相比,FUT4在HaCaT细胞中的表达量较低。
免疫荧光方法(图1B)显示,FUT4主要定位在细胞浆内,HaCaT细胞中荧光强度明显较A431细胞和SCC12细胞中弱,同样表明FUT4在HaCaT细胞中的表达水平较低。
进一步采用Real-time PCR方法(图2)对HaCaT细胞中 FUT4的表达进行了定量分析。结果显示A431和SCC12细胞中FUT4 mRNA的表达水平分别约是HaCaT细胞中的13倍和5倍。
2.2 5-aza-dC对HaCaT细胞中FUT4表达的影响
为了观察DNA甲基转移酶抑制剂5-aza-dC是否会对HaCaT 细胞中FUT4的表达水平有影响,将细胞用5 µmol/L的5-aza-dC 处理72 h,检测FUT4的表达变化。Real-time PCR结果表明,与未经药物处理组相比,5-aza-dC处理 HaCaT细胞后,FUT4 mRNA的表达明显增加,并与 A431细胞中的表达水平相当(图 2)。同样的药物处理也引起了 SCC12细胞中FUT4 mRNA表达明显增加,而A431细胞中FUT4 mRNA表达基本不变。这一结果提示,HaCaT细胞中FUT4的表达水平和细胞中DNA甲基化水平可能有关。
2.3 5-aza-dC对HaCaT细胞中FUT4启动子区的甲基化状态的影响
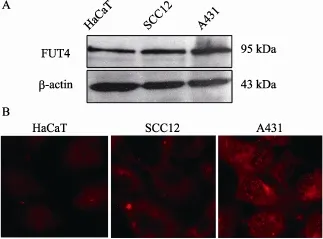
图1 FUT4在HaCaT细胞中的表达

图2 5-aza-dC对HaCaT细胞中FUT4 mRNA表达的影响
为了进一步确定HaCaT 细胞中FUT4的表达是否直接受FUT4启动子甲基化调控,利用软件Meth-Primer(http//www.urogene.org//methprimer/indexl.html) 对FUT4启动子区的CpG岛进行了预测,结果表明在转录起始位点上游-29~-725 bp处存在两个CpG岛[15],本文选择-429~-671 bp处的CpG岛,采用甲基化特异性PCR的方法对FUT4启动子的甲基化状态进行了分析。结果表明,5-aza-dC对HaCaT细胞处理前,使用M引物扩增检测到大量产物,U引物扩增未检测到产物。5 µmol/L的5-aza-dC处理HaCaT细胞72 h后,U引物扩增检测到大量产物,M引物扩增未检测到产物(图3)。SCC12细胞加药处理后也出现甲基化产物减少,非甲基化产物增加的趋势,但没有HaCaT细胞中的变化显著。这一结果提示,FUT4在HaCaT细胞中的低表达与其启动子区的甲基化状态相关。
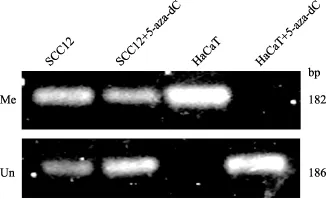
图3 HaCaT细胞中FUT4启动子甲基化状态分析
3 讨 论
细胞表面糖蛋白和糖脂上的岩藻糖寡糖LeY在精卵识别、胚胎着床及发育等生理过程和肿瘤等病理过程中起着非常重要的作用。FUT4是合成 LeY的关键酶,定位于高尔基体上。研究表明,FUT4 和LeY在相同组织来源的肿瘤细胞和正常细胞中以及不同组织来源的肿瘤细胞中其表达水平具有明显差异,但调控这种表达差异的原因还不是很清楚。
Taniguchi等[16]研究表明,在急性髓性白血病和结肠癌细胞系中,FUT4转录本的长短不一样,并确定了长短转录本的增强子。杨雪松等[17]报道,在乳腺癌细胞中 HSF1 和 Sp1两个转录因子调控了FUT4的表达,从而影响细胞的增殖。Withers等[18]则证明Elk-1是组织细胞淋巴瘤U937细胞中FUT4表达的关键转录因子,并且FUT4基因符合CpG岛的定义。近年来,基因启动子区CpG岛甲基化状态改变被认为是很多基因表达调控的重要手段[19~24],也有研究证明岩藻糖基转移酶基因FUT3、FUT7的表达受其启动子甲基化水平的调控[25,26]。在本实验室前期的研究中发现并报道了增殖能力弱的人表皮鳞癌细胞系SCC12细胞比增殖能力强的A431细胞中 FUT4启动子甲基化程度高,加入甲基化酶抑制剂5-aza-dC处理后,SCC12细胞中FUT4表达水平明显提高,启动子区甲基化水平显著降低[15]。
但 FUT4在正常细胞中的表达水平如何,是不是其表达也受启动子甲基化调控呢?本研究采用正常的人永生化表皮细胞系HaCaT细胞以及人表皮鳞癌细胞系A431和SCC12细胞对其进行了研究。结果发现,FUT4在HaCaT、SCC12和A431细胞中的表达水平依次升高。这一结果提示,在正常的细胞中FUT4处于基础表达水平,当细胞恶性转变以后,FUT4的表达明显增加。研究表明,FUT4的表达水平与细胞的增殖、迁移能力相关[1,4,27],少量的FUT4表达可以满足正常细胞正常生理活动的需要,而当细胞恶性生长后,就需要大量的 FUT4的表达,产生大量的LeY岩藻糖寡糖,从而加强细胞之间的识别,激活相关信号转导途径,促进肿瘤细胞的恶性发展。
为了证明HaCaT细胞中FUT4的低表达是否与甲基化有关,本研究用 5 µmol/L的 5-aza-dC处理HaCaT细胞72 h后,检测了基因表达水平及启动子区甲基化状态的变化,结果表明,HaCaT细胞中FUT4 mRNA的表达水平显著升高,说明HaCaT细胞中 FUT4的表达和细胞中的甲基化水平相关,但不能确定5-aza-dC作用后FUT4表达的变化是受其自身启动子甲基化状态的直接调控还是由于其他类似转录因子的基因表达变化从而间接引起的。通过软件对FUT4启动子区的CpG岛进行了预测,并选取其中的一个CpG岛进一步研究其甲基化情况。结果发现,FUT4表达升高的同时,该CpG岛的甲基化程度显著降低,这一结果暗示,FUT4的表达可能是和其自身启动子的甲基化程度相关。
一般认为,基因的启动子区甲基化抑制基因的表达,一方面是由于直接影响了转录因子与DNA的结合,另一方面是由于启动子甲基化以后会与甲基化结合蛋白(Methyl-CpG binding proteins)结合,进一步招募去已酰化酶(HDAC),共抑制蛋白(mSin3A),使染色质处于凝缩状态,致使RNA聚合酶、转录因子等不能结合到DNA上,从而转录被抑制。Zhu等[28]研究报道,p21Cip1基因的转录因子 Sp1/Sp3识别区周围序列的甲基化可以直接减少该转录因子的结合,从而影响基因转录。Zhang等[29]则发现甲基化酶抑制剂5-aza-dC处理癌细胞后,LHR基因的表达水平升高,并不是因为转录因子的活性或与启动子区的结合数量发生变化,而是因为促进了去乙酰化酶复合物HDAC1/HDAC2/mSin3A从LHR启动子区解离,提高了乙酰化水平,从而促进基因表达。在本研究中,与A431和SCC12两种恶性细胞相比,HaCaT细胞可能由于不需要太多FUT4的表达,因此FUT4启动子区 CpG岛需要处于更高程度的甲基化状态,启动子的高度甲基化致使转录因子不能与启动子区结合,进而大量减少 FUT4基因的转录,使 FUT4的表达维持在基础水平,满足细胞的正常生长需要。但 FUT4启动子区从甲基化到去甲基化,基因从低表达到高表达,其启动子区上所结合的蛋白究竟发生了怎样的转变还有待于进一步研究。
值得注意的是,SCC12细胞经5-aza-dC作用72 h后,FUT4 mRNA的表达量虽然有所提高,但并没有达到A431细胞中的表达水平。分析原因有以下几种可能:第一,FUT4的表达很有可能还受其他因素的影响,如转录因子的活性和结合数量以及组蛋白的乙酰化状态等,多种方式参与的调控也见于其他基因的研究中[29]。相关文献也表明,在不同的细胞中,细胞的不同状态下,对 FUT4的表达起主要调控作用的机制可能不同[16~18]。本文所选取的细胞是正常细胞和恶性程度不同的癌细胞,这种细胞之间的差异性很可能会形成相同的基因却存在不同的表达调控机制。第二,5-aza-dC是一种甲基化酶抑制剂,根据预实验并参考文献的报道,本文采用终浓度为5 μmol/L、作用时间72 h。由于细胞之间存在差异性,可能这个使用浓度和作用时间对SCC12细胞来讲并不是最佳条件,因而药物作用后虽然出现 FUT4启动子去甲基化,基因表达水平升高的趋势,但并没有达到完全去甲基化,基因的表达水平也没有达到最高。
本研究一方面为 FUT4在不同细胞中的表达调控机制提供了参考依据;另一方面,由于所选择的研究细胞是正常细胞和恶性程度不同的鳞癌细胞,在这些细胞中 FUT4的表达具有随着恶性程度的升高而表达量增加,甲基化程度降低的趋势。FUT4基因在临床病人的样本中如果也具有同样的甲基化调控趋势,那么检测 FUT4启动子的甲基化状态将有望成为临床上鳞癌的诊断及恶性程度分级的辅助手段,同时也为抗癌药物的研发提供新思路。
[1]Zhang ZB, Sun P, Liu JW, Fu L, Yan J, Liu YJ, Yu LH, Wang XQ, Yan Q. Suppression of FUT1/FUT4 expression by siRNA inhibits tumor growth. Biochim Biophys Acta, 2008, 1783(2): 287-296.
[2]Dettke M, Pálfi G, Pursch E, Fischer MB, Loibner H. Increased expression of the blood group-related Lewis Y antigen on synovial fluid granulocytes of patients with arthritic joint diseases. Rheumatology (Oxford), 2001, 40(9): 1033-1037.
[3]Ponnampalam AP, Rogers PAW. Expression and regulation of fucosyltransferase 4 in human endometrium. Reproduction, 2008, 136(1): 117-123.
[4]Ciolczyk-Wierzbicka D, Bodzioch M, Gil D, Zmudzińska D, Dembińska-Kiec A, Laidler P. Expression of fucosyltransferases contributes to melanoma invasive phenotype. Med Chem, 2007, 3(5): 418-424.
[5]Escrevente C, Machado E, Brito C, Reis CA, Stoeck A, Runz S, Marmé A, Altevogt P, Costa J. Different expression levels of alpha3/4 fucosyltransferases and Lewis determinants in ovarian carcinoma tissues and cell lines. Int J Oncol, 2006, 29(3): 557-566.
[6]Kudo T, Ikehara Y, Togayachi A, Morozumi K, Watanabe M, Nakamura M, Nishihara S, Narimatsu H. Up-regulation of a set of glycosyltransferase genes in human colorectal cancer. Lab Invest, 1998, 78(7): 797-811.
[7]Martín-Satué M, Marrugat R, Cancelas JA, Blanco J. Enhanced expression of alpha(1,3)-fucosyltransferase genes correlates with E-selectin-mediated adhesion and metastatic potential of human lung adenocarcinoma cells. Cancer Res, 1998, 58(7): 1544-1550.
[8]Petretti T, Schulze B, Schlag PM, Kemmner W. Altered mRNA expression of glycosyltransferases in human gastric carcinomas. Biochim Biophys Acta, 1999, 1428(2-3): 209-218.
[9]Yang XS, Zhang ZB, Jia S, Liu YJ, Wang XQ, Yan Q. Overexpression of fucosyltransferase IV in A431 cell line increases cell proliferation. Int J Biochem Cell Biol, 2007, 39(9): 1722-1730.
[10]Yang XS, Liu S, Liu YJ, Liu JW, Liu TJ, Wang XQ, Yan Q. Overexpression of fucosyltransferase IV promotes A431 cell proliferation through activating MAPK and PI3K/Akt signaling pathways. J Cell Physiol, 2010, 225(2): 612-619.
[11]Terracciano D, Di Carlo A, Papa P, Cicalese M, Maietta P, Cecere C, Mariano A, Macchia V. New approaches in the diagnostic procedure of malignant pleural effusions. Oncol Rep, 2004, 12(1): 79-83.
[12]Di Carlo A, Terracciano D, Mariano A, Oliva A, D'Armiento M, Macchia V. Role of cytokeratins, nuclear matrix proteins, Lewis antigen and epidermal growth factor receptor in human bladder tumors. Int J Oncol, 2003, 23(3): 757-762.
[13]Farhan H, Schuster C, Klinger M, Weisz E, Waxenecker G, Schuster M, Sexl V, Mudde GC, Freissmuth M, Kircheis R. Inhibition of xenograft tumor growth and down-regulation of ErbB receptors by an antibody directed against Lewis Y antigen. J Pharmacol Exp Ther, 2006, 319(3): 1459-1466.
[14]Boghaert ER, Sridharan L, Armellino DC, Khandke KM, DiJoseph JF, Kunz A, Dougher MM, Jiang F, Kalyandrug LB, Hamann PR, Frost P, Damle NK. Antibody-targeted chemotherapy with the calicheamicin conjugate hu3S193-N-acetyl γcalicheamicin dimethyl hydrazide targets Lewisyand eliminates Lewisy-positive human carcinoma cells and xenografts. Clin Cancer Res, 2004, 10(13): 4538-4549.
[15]Li HY, Tong SM, Liu JW, Han L, Yang XS, Hou HS, Yan Q, Wang XQ. Differential fucosyltransferase IV expression in Squamous carcinoma cells is regulated by promoter methylation. Cell Mol Biol Lett, 2012, 17(2): 206-216.
[16]Taniguchi A, Suga R, Matsumoto K. Expression and transcriptional regulation of the human alpha1, 3-fucosyltransferase 4 (FUT4) gene in myeloid and colon adenocarcinoma cell lines. Biochem Biophys Res Commun, 2000, 273(1): 370-376.
[17]Yang XS, Wang J, Liu S, Yan Q. HSF1 and Sp1 regulate FUT4 gene expression and cell proliferation in breast cancer cells. J Cell Biochem, 2014, 115(1): 168-178.
[18]Withers DA, Hakomori SI. Human alpha (1,3)-fucosyltransferase IV (FUTIV) gene expression is regulated by elk-1 in the U937 cell line. J Biol Chem, 2000, 275(51): 40588-40593.
[19]Gupta A, Godwin AK, Vanderveer L, Lu A, Liu J. Hypomethylation of the synuclein gamma gene CpG island promotes its aberrant expression in breast carcinoma and ovarian carcinoma. Cancer Res, 2003, 63(3): 664-673.
[20]Wu HJ, Chen YP, Liang J, Shi B, Wu G, Zhang Y, Wang D, Li RF, Yi X, Zhang H, Sun LY, Shang YF. Hypomethylation-linked activation of PAX2 mediates tamoxifen-stimulated endometrial carcinogenesis. Nature, 2005, 438(7070): 981-987.
[21]Kang CW, Song JJ, Lee J, Kim MY. Epigenetics: An emerging player in gastric cancer. World J Gastroenterol, 2014, 20(21): 6433-6447.
[22]Zhang GQ, Pradhan S. Mammalian epigenetic mechanisms. IUBMB Life, 2014, 66(4): 240-256.
[23]Huang B, Jiang C, Zhang R. Epigenetics: the language of the cell? Epigenomics, 2014, 6(1): 73-88.
[24]Jeltsch A, Jurkowska RZ. New concepts in DNA methylation. Trends Biochem Sci, 2014, 39(7): 310-318.
[25]Serpa J, Mesquita P, Mendes N, Oliveira C, Almeida R, Santos-Silva F, Reis CA, LePendu J, David L. Expression of Lea in gastric cancer cell lines depends on FUT3 expression regulated by promoter methylation. Cancer Lett, 2006, 242(2): 191-197.
[26]Syrbe U, Jennrich S, Schottelius A, Richter A, Radbruch A, Hamann A. Differential regulation of P-selectin ligand expression in naive versus memory CD4+ T cells: evidence for epigenetic regulation of involved glycosyltransferase genes. Blood, 2004, 104(10): 3243-3248.
[27]Yang XS, Liu S, Yan Q. Role of fucosyltransferase IV in epithelial-mesenchymal transition in breast cancer cells. Cell Death Dis, 2013, 4(7): e735.
[28]Zhu WG, Srinivasan K, Dai ZY, Duan WR, Druhan LJ, Ding HM, Yee L, Villalona-Calero MA, Plass C, Otterson GA. Methylation of adjacent CpG sites affects Sp1/ Sp3 binding and activity in the p21Cip1 promoter. Mol Cell Biol, 2003, 23(12): 4056-4065.
[29]Zhang Y, Fatima N, Dufau ML. Coordinated changes in DNA methylation and histone modifications regulate silencing/derepression of luteinizing hormone receptor gene transcription. Mol Cell Biol, 2005, 25(18): 7929-7939.
(责任编委: 方向东)
Correlation between FUT4 expression and its promoter methylation in HaCaT cells
Hongyan Li1, Shaoming Tong1, Qiu Yan2
1. College of Life Science, Liaoning Normal University, Dalian 116081, China; 2. Department of Biochemistry and Molecular Biology, Dalian Medical University, Dalian 116044, China
The expression level of fucosyltransferase Ⅳ (FUT4) is low in normal cells. The mechanism underlying regulation of FUT4 expression in normal cells remains elusive. In this study, Western blot, immunofluorescence and real-time PCR were used to analyze FUT4 expression in the immortalized human keratinocytes cells HaCaT. Methylated-specific PCR was used to investigate methylation status of FUT4 promoter. The results showed that the FUT4 expression level was significantly lower in HaCaT cells than squamous carcinoma cells A431 and SCC12. FUT4 mRNA expression was increased in HaCaT cells treated by 5-aza-dC (5 µmol/L), an inhibitor of DNA methyltransferase. Furthermore, using the primers to amplify the methylated fragment yielded PCR products and noproducts were yielded by the primers to amplify the unmethylated fragment in HaCaT cells. Unmethylated PCR products were obtained in HaCaT cells treated by 5-aza-dC, while methylated PCR products were not detected. These results suggest that the lower expression of FUT4 in HaCaT cells may be correlated with the methylation of CpG island in FUT4 promoter.
FUT4; low expression; DNA methylation; HaCaT cells
2014-07-09;
2014-08-19
李洪艳,博士,副教授,研究方向:肿瘤糖生物学,表观遗传学。Tel: 0411-85827090;E-mail: L-hongyan@163.com
燕秋,博士,教授,研究方向:糖生物学。E-mail: yanqiu63@126.com
10.16288/j.yczz.2015.01.007
时间: 2014-9-19 13:35:30
URL: http://www.cnki.net/kcms/detail/11.1913.R.20140919.1335.001.html

