基于小麦淀粉合成酶SSⅡa氨基酸序列的生物进化分析
韩俊杰,王昊龙,蔡曼,李咏,李卫华
(石河子大学农学院/新疆兵团绿洲生态农业重点实验室,石河子 832003)
小麦是人类最早种植的粮食作物,原产于亚洲西部,目前在世界范围内均有种植。在成熟的小麦籽粒中,淀粉是其主要的组成成分,其它还含有一些蛋白质、脂类、纤维和灰分等[1]。由于小麦是主要的粮食来源和工业原料,其用途非常广泛,因此,对小麦在生理生化上的研究一直是个热点。小麦淀粉的形成过程较为复杂,由多个基因共同调控完成。研究表明,小麦淀粉是在造粉体中形成的,在不同的阶段主要受到以下几种酶的调控:腺苷二磷酸葡萄糖焦磷酸化酶(AGPase)、淀粉合成酶(SS)、淀粉分支酶(SBE)和淀粉去分支酶(DBE)[2]。AGPase催化1磷酸葡萄糖和ATP形成ADPG,从而提供淀粉合成的底物[3];SS主要负责淀粉支链的合成,延长支链淀粉的侧链[4];SBE则主要催化α-1,6糖苷键的形成;DBE的作用是水解α-1,6糖苷键,去除支链淀粉中过多的分支[5]。在其合成过程中,以上4种调控酶都会影响到小麦淀粉粒的结构。普通小麦基因来源已比较明确,但大多数都集中在禾本科作物中,对其与其它非禾本科作物的亲缘关系还鲜见报道。目前,对生物有机体的进化框架已经有了较为明确的认识,但是生物的表观性状在进化的历程中是非常复杂的,对其进化历史研究很难构建清晰的图像。而蛋白质作为生命物质的承担者,其在漫长的进化过程中已经形成了独特的系统,因此,从蛋白的角度去研究生物的进化机制是很有意义的,这也是分子生物学发展的趋势。目前,很多的数据库都已经收录了大量的基因序列和氨基酸序列,这使得从生物大分子的角度去揭示生物进化机制成为可能。
因此,本研究选取小麦淀粉合成酶SSⅡa,通过克隆得到SSⅡa的cDNA序列,并获得其编码的氨基酸序列,对小麦淀粉合成酶氨基酸序列进行分析,另外通过与其他不同生物的淀粉合成酶序列比对分析,看其在系统进化过程中的表现,判断其与其它生物的亲缘进化关系远近,探究其在不同生物中的进化历程,以期对淀粉合成酶的进化机制提供一些素材。
1 材料与方法
1.1 材料氨基酸序列的获取
通过克隆测序得到其cDNA序列,经过翻译得到小麦淀粉合成酶SSⅡa氨基酸序列。其它47种生物的淀粉合成酶序列均来自 SwissProt(http://www.uniprot.org/)非冗余蛋白数据库。截止到2015-01-07的16:56pm,从20308个序列中搜索得到与小麦淀粉合成酶SSⅡa氨基酸序列有同源性的氨基酸序列。其中包括6种藻类、1种苔藓、1种蕨类、1种菌类和38种陆生被子植物。
1.2 方法
翻译得到的小麦淀粉合成酶氨基酸序列进行NCBI/BLASTp同源性比对,并将所得到的所有生物的同源性氨基酸序列编辑整理,将其成熟肽部分用Clustal W做比对分析,并用PAUP建树软件Bootsrap1000次做系统进化树分析。
2 结果与分析
2.1 小麦淀粉合成酶SSⅡa氨基酸序列
将克隆测序得到的小麦淀粉合成酶SSⅡa的cDNA的 ORF(Open Reading Frame,ORF)翻译得到其氨基酸序列如下:

该序列共有799个氨基酸残基,本研究利用SignalP(http://www.cbs.dtu.dk/services/SignalP/)分析发现,前端30个氨基酸为其信号肽序列,后面为其成熟肽序列(图1)。
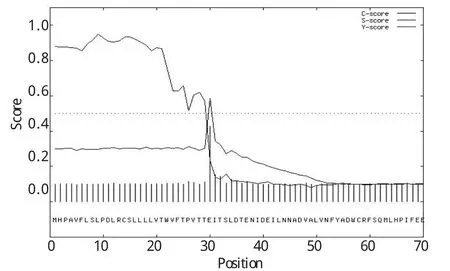
图1 氨基酸序列信号肽预测Fig.1 The signal peptide prediction of the amino acid sequence
2.2 淀粉合成酶SSⅡa氨基酸序列比较分析
将从SwissProt非冗余蛋白数据库获得的其它生物的氨基酸序列整理编辑,去除其信号肽序列,只保留成熟太序列,并将所有整理过的氨基酸序列进行Clustal W比对分析(结果在此未列出),结果表明,小麦淀粉合成酶SSⅡa氨基酸序列与其它生物的同源性为37%-99%,而在这47种生物种,其序列之间的同源性最小为24.17%,最大为94.97%,可见氨基酸序列的变异范围较大。对小麦和其它47种生物比对和这47种生物之间的比对发现,其中有108个氨基酸残基是固定不变的(结果未列出),在小麦中这些完全保守的氨基酸残基数占小麦淀粉合成酶SSⅡa氨基酸残基的13.52%,由此可见,淀粉合成酶SSⅡa氨基酸序列在进化的过程中具有很高的保守性。研究还发现,小麦与同属于禾本科(Poaceae)小麦族(Trib.Triticeae)的山羊草和大麦同源性分别为96%和90%,而对其它38种陆生被子植物氨基酸序列作同源性比对分析发现,它们的同源性为24.17%-74.97%,可见淀粉合成酶SSⅡa在这38种陆生被子植物中,其序列保守性较低。
2.3 基于淀粉合成酶SSⅡA氨基酸序列的不同生物的系统进化树的构建
由于淀粉合成酶SSⅡa氨基酸序列有较高的保守性和一定的变异性,因此,可以根据淀粉合成酶SSⅡa氨基酸序列做系统进化分析。利用Clustal W将这47种生物的氨基酸序列做比对分析后,用建树软件构建系统进化树,结果见图2。
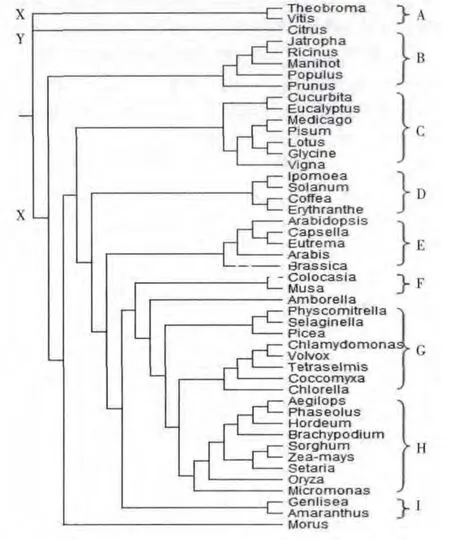
图2 基于淀粉合成酶SSⅡa氨基酸序列的系统进化树Fig.2 The phylogenitic tree based on the amino acid sequence of SSⅡa
图2为假定所有生物有一个共同的祖先所构建的有根树。由图2可见,这47个生物在进化史上分为3簇X、Y和Z,其中 X簇由1种苔藓、1种蕨类、1种菌类、5种藻类和其它陆生被子植物的氨基酸序列组成;Y簇则是芸香科 (Rutaceae)植物柑橘(Citrus reticulata Blanco),因此其单独成一簇;Z簇则由2种陆生被子植物组成。这些单独的分支也说明了淀粉合成酶基因进化较为复杂。然后,我们又把这3簇细分为A-I共9个簇,其中的5种藻类又归为G簇,3种大戟科(Euphorbiaceae)植物归为B簇,5种豆科(Leguminosae)植物归于C簇,十字花科(Cruciferae)植物归于 E簇,禾本科(Poaceae)作物则全在H簇。
为了探究同类的生物的进化关系,我们将6种藻类生物和38种陆生被子植物分别作系统进化树分析(图3、图4),2张图中均为无根树。
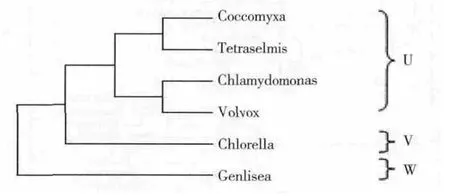
图3 基于淀粉合成酶SSⅡA氨基酸序列的藻类生物的系统进化树Fig.3 The phylogenitie based on the amino acid sequence of SSⅡA
由图3可知,按照这6种藻类淀粉合成酶氨基酸序列的同源性,可将其分为3簇。在图3的U簇中,同属于绿藻纲(Chlorophyceae)团藻目(Volvocales)、衣藻科(Chlorococcales)的绿藻(Coccomyxa)、衣藻(Chlamydomonas)和扁藻 (Tetraselmis),其分别属于衣藻属(Chlamydomonas)和扁藻属(Phacus),因而将其归为一簇。小球藻(Chorella fusca)是绿球藻目(Prochlorococcus) 小球藻科 (Chlorellales),而螺旋狸藻(Genlisea)属于双子叶植物纲、唇形目、狸藻科,因此它们可单独成一簇。这6种藻类均为真核生物,从系统发育来看,衣藻具有最简单的单细胞结构,在其顶端有2条等长的鞭毛,可以游动;小球藻是单细胞生物,其它则形成个体,属于陆生淡水藻类。因此,从淀粉合成酶氨基酸序列构建的进化树来看,这是可以用来反映生物系统进化的。但是,由于只有6种藻类生物,不足以说明其进化关系,而且另外38种陆生被子植物在进化上有一定的相近性,因此,根据这些陆生被子植物的淀粉合成酶氨基酸序列来构建进化树,以增加其进化过程中在不同生物亲缘关系远近的可靠性。
图4的无根树图中,我们将这38中陆生被子植物分为2大簇,并在此基础上细分为Ⅰ-Ⅵ簇,发现:与图2的47种氨基酸序列构建的进化树有所不同,其亲缘关系有所变化。其中簇C、E、G和H基本没变,A、B和D簇则形成新簇与之并列,其它各簇均有所变动。
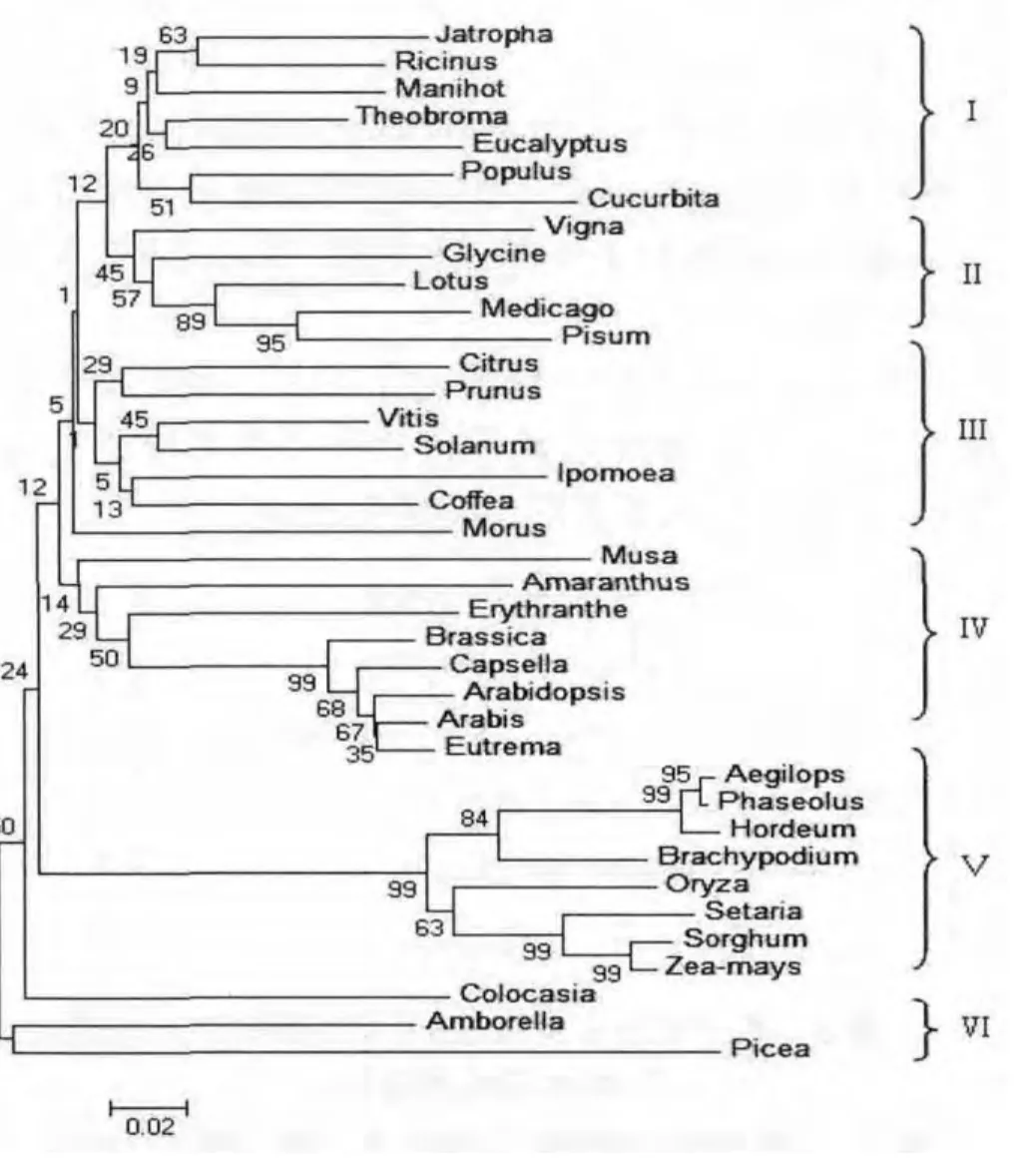
图4 38种植物淀粉合成酶氨基酸序列的同源性Fig.4 Homology analysis of Starch synthase amino sequence of 38 organisms
在这38中陆生被子植物中,根据生物系统进化分级,从较低级的分类阶元上,我们无法看出其归属关系,但从科这一级来看,这6簇的归属关系较为清晰,氨基酸序列的同源性很高,而在我们的分类中,处于同一科的生物基本都在同一个分支。如大戟科的麻风树(Jatrophacurcas)、蓖麻(Ricinus communisL.)和木薯(Manihot utilissima),它们都在簇Ⅰ中;豆科的豇豆(Vigna unguiculataWalp.)、大豆(Glycine maxMerr)、百脉根(Lotuscorniculatus)、苜蓿(MedicagosativaLinn)和豌豆(Pisum sativum)则共同形成簇Ⅱ;十字花科芥菜(Brassica juncea(L.)Czern.)、芸苔(BrassicacampestrisL.var.campestris)、荠菜(Capsella bursa-pastoris) 和拟南芥(Arabidopsis thaliana(L.)Heynh.)归属于簇Ⅳ,其同源性高达99%;另外,基本所有的禾本科作物,如山羊草(Aegilops)、大麦(Barley)、水稻(Oryza sativa)和玉米(Zea maysLinn.Sp.)共同组成了簇Ⅴ,其同源性为84%-99%。但是,在分类中也有一些特殊的情况,如豆科的菜豆(Phaseolus vulgarisLinn)则属于簇Ⅴ,未归于豆科形成的簇中;而蔷薇科的海棠(Malus spectabilis(Ait.)Borkh)和芭蕉科的芭蕉(Musa basjoo)则分在了簇Ⅳ中,在其它的不同簇中也各有其它科的植物,这在构建的有根树和无根树中均会出现,造成这种情况出现的原因需进一步分析。
3 讨论
(1)本研究中,47种不同的生物做系统进化树分析结果,大体上能够反映出不同生物的系统进化关系,但是其本身仍然存在缺陷。本研究的结果中,不同科的生物被分在同一个簇中,还有同属的生物并未在同一个簇中。造成此情况的原因主要有:首先是所用的软件本身存在缺陷,本实验中应用的是较为常用的软件 Clustal W、MEGA 6和 PAUP及DNAMAN。这些软件本身在计算方法上存在一定缺陷,这使得系统发育分析并不能得到十分精确的结果,但随着统计方法的进步和生物软件的更新,利用氨基酸序列来分析生物系统进化关系的准确性将会得到提高;另外,由于本研究中得到的氨基酸序列数目不多,其代表性不够,因此,其只能够在大体上反映生物的进化关系。假如能够获得更多的氨基酸序列,且在每个进化阶段均有代表出现,在做生物进化分析时,则能够更好的反映其亲缘进化关系。一般研究认为,近缘种的遗传物质非常相似[6-9],以上分类也说明了这一点。根据Cronquist[7-8]的系统发育理论对不同生物物种的分类结果可知,这些簇中包含了在系统进化上相近的种类。
(2)本研究的进化图中,苔藓、卷柏(蕨类)和藻类归为一簇,明显表现出更近的亲缘关系,与其它陆生植物关系较远。其原因是这2种生物进化上较为低级,而与进化上更为低级的藻类更为接近。如果能够增加一些趋于中间的类型,利用大量的氨基酸序列做出生物进化树图,则更能反映出这种亲缘进化关系。在前人的报道[10-18]中,根据大量生物的DNA序列和氨基酸序列做比对分析并构建了系统发育图,发现其主要类群分支与经典系统分类相符。随着大量氨基酸序列的提交和不同数据库的完善,今后利用氨基酸序列来分析生物进化将会更加完善。
(3)本研究中,处于同一科的近缘种并未完全被分在同一个分支中,究其原因可能是受其生长环境因素影响。环境的差异,导致其核苷酸序列出现置换、插入和缺失等,从而导致氨基酸序列的差异,还可能存在多基因家族。但是总体上,利用淀粉合成酶氨基酸序列构建的进化树基本上能反映出这47种不同生物的进化关系。同时也说明依据淀粉合成酶氨基酸序列做生物系统发育分析有一定的合理性,可为生物系统的进化提供一些信息。
4 结论
本研究分析的47种不同生物的淀粉合成酶氨基酸序列中,由构建进化树可见,在相同科属的植物中,其亲缘关系较近,基本全部被分在同一簇。在对氨基酸序列比对分析时发现,在多处氨基酸位点都表现出较高的保守性,在禾本科作物中,其保守性要高于其它生物。因此,利用淀粉合成酶SSⅡa氨基酸序列可以用来做系统进化分析,并作为判定亲缘关系远近的依据。
[1]廖金花,李纪明,吴瑜.小麦淀粉合成酶的研究进展[J].贵州农业科学,2014,42(2):18-22.LIAO Jinhua,LI Jiming,WU Yu.Research progress in starch synthase of wheat[J].Guizhou Agriculture Science,2014,42(2):18-22.
[2]Nakamura Y.Towards a better understanding of the metablic system for amylopectin biosynthesis in plants:rice endosperm as a model tissue[J].Plant Cell Physiology,2002,43(7):718-725.
[3]Rahman S,Li Z,Batey I,et al.Genetic alteration of starch functionality in wheat[J].Journal of Cereal Science,2000,31(1):91-110.
[4]Nakamura Y.Towards a better understanding of the metabolic system for amylopectin biosynthesis in plants:rice endosperm as a model tissue[J].Plant Cell Physiology,2002,43(7):718-725.
[5]Ball S G,van de Wal M H B J,Visser R G F.Progress in understanding the biosynthesis of amylose[J].Trends in Plant Science,1998,3(12):462-467.
[6]Wapinski I,Pfeffer A,Friedman N,et al.Natural history and evolutionary principles of gene duplication[J].Nature,2007,449,54-61.
[7]Cronquist A.The evolution and classification of flowering plants[M].New York:New York Botonical Garden Press,1988.
[8]Cronquist A.An integrated system of classification of flowering plants[M].Columbia:Columbia University Press,1981.
[9]Craig J,Lioyd JR,Tomlinson K,et al.Mutations in the gene encoding starch synthase II profoundly alter amylopectin structure in pea endosperm[J].Plant Cell,1998,10(3):413-426.
[10]赵阳.蒺藜苜蓿全基因组NBS类型抗病基因及其进化分析[D].合肥:安徽农业大学,2010.
[11]朱新霞,艾尼江,闫洁,等.同步提取棉花DNA和RNA的方法[J].石河子大学学报:自然科学版,2011,29(3):347-351.ZHU Xinxia,AI Nijiang,YAN Jie,et al.Amethod of simultaneous isolation of DNA and RNA from cotton[J].Journal of Shihezi University:Natural Science,2011,29(3):347-351.
[12]杨海波,贺志昊,刘昱成,等.羊口疮病毒新疆野毒株SHZ1、B2L和F1L基因的克隆及遗传进化分析[J].石河子大学学报:然科学版,2013,31(3):323-327.YANG Haibo,HE Zhihao,LI Yucheng,et al.Cloning and phylogenetic analysis of B2L and F1L genes of orf virus xinjiang wild strains SHZ1[J].Journal of Shihezi University:Natural Science,2013,31(3):323-327.
[13]张再超,乔军,蔡扩军,等.猪2型圆环病毒石河子流行株全基因组的克隆和序列分析[J].石河子大学学报:然科学版,2011,29(6):696-699.ZHANG Zaichao,QIAO Jun,CAI Kuojun,et al.Cloning and complete genome analysis of Shihezi strain of Porcine Circovirus Type 2[J].Journal of Shihezi University:Natural Science,2011,29(6):696-699.
[14]李峥,高玉红,毕胜,等.云南省丙型肝炎病毒基因型及3b型遗传进化树分析[J].国际检验医学杂志,2006,27(11):979-983.LI Zheng,GAO Yuhong,BI Sheng,et al.Hepatitis C virus genotype in Yunnan and phylogenetic tree analysis[J].International Journal of Laboratory Medicine,2006,27(11):979-983.
[15]Kaneko T,Nakamura Y,Wolk C P,et al.Complete genomic sequence of the filamentous nitrogen-fixing cyanobacterium Anabaena sp.strain PCC 7120[J].DNA Research,2001,8(5):205-213.
[16]李淼淼,南富波,李卫华,等.小麦SBEⅡa、SSⅡa基因片段的克隆及VIGS重组载体的构建[J].新疆农业科学,2013,50(11):1961-1966.LIMiaomiao,NAN Fubo,LIWeihua,et al.Cloning of wheat SBEⅡa and SSⅡa genes partial sequence and construction of their VIGS recombinant vectors[J].Xinjiang Agricultural Sciences,2013,50(11):1961-1966.
[17]侯磊,李家宝,罗小英,等.棉花ADP-ribosylation factor基因(GhARF1)的克隆与表达分析[J].作物学报,2007,33(8):1226-1231.HOU Lei,LI Jiabao,LUO Xiaoying,et al.Cloning,expression and characterization of an ADP-ribosylation factor gene from cotton(Gossypium hirsutumL.)[J].Acta Agronomica Sinica,2007,33(8):1226-1231.
[18]王金明,丁在松,张桂芳,等.家稗丙酮酸磷酸双激酶(PPDK)基因的克隆及序列分析[J].作物学报,2007,33(6):927-930.WANG Jinming,DING Zaisong,ZHANG Guifang,et al.Cloning and sequence analysis of pdk gene inEchinochloa crusgallivar.frumentaceaRoxb.WF Wight[J].Acta Agronomica Sinica,2007,33(6):927-930.

