亚洲棉全基因组中NAC类转录因子基因的鉴定与分析
赵灿,刘任重,柳展基,李菲,王立国,马骏骏,朱新霞
(1石河子大学生命科学学院农业生物技术重点实验室,石河子 832003;2山东棉花研究中心,济南 250100)
NAC转录因子是广泛存在于植物中的一类转录因子。1996年 Souer等[1]在矮牵牛(Pharbifis nilL.)中克隆了第一个NAC转录因子基因NAM(no apical meristem),其nam突变株不能形成顶端分生组织,大部分突变体在幼苗期便死亡,少数存活突变体的花器官发育异常。1997年Aida等[2]首先报道了NAC结构域,发现 NAM、ATAF1/2和 CUC2(cup-shaped cotyleton)基因编码蛋白的N末端有一段大约150个氨基酸的保守结构域,取此3个基因首字母命名为NAC。NAC蛋白N末端氨基酸序列相对保守,主要功能为结合DNA序列、细胞核定位及与其它NAC蛋白形成二聚体等,而C末端氨基酸序列高度变异,行使转录激活或转录抑制功能[3]。
NAC转录因子在植物生长发育、代谢调节、器官形成、激素响应等方面发挥重要作用。Mao等[4]在水稻(Oryza sativaL.)中过量表达OsNAC2基因,发现转基因水稻株高降低、分蘖能力增强且分蘖角度增大。Xie等[5]发现拟南芥(Arabidopsis thalianaL.)的NAC1基因受生长素诱导表达,通过激活2个下游生长素响应基因DBP和AIR3来促进侧根发育。Uauy等[6]研究发现普通小麦(Triticum aestivumL.)NAC转录因子NAM-B1基因能够加速植株衰老,促进叶片中的营养物质向发育中的籽粒转运,进而提高籽粒中的蛋白质、锌和铁的含量。Zhong等[7]发现拟南芥中NAC转录因子SND1为调控纤维次生壁合成的主要转录开关,在茎维管束纤维和木质部纤维特异表达,抑制或过表达SND1会导致纤维次生壁的缺失或加厚。在生物和非生物逆境响应方面,不同物种的NAC转录因子同样发挥重要作用[8-9]。
在拟南芥、水稻和雷蒙德氏棉(Gossypium raimondiiL.)等测序完成的基因组序列中,利用生物信息学方法已成功预测了大量NAC转录因子基因[11],其中拟南芥有117个、水稻基因组中有151个成员、烟草 (Nicotiana tabacumL.)152个、大豆(Glycine maxL.)152个、雷蒙德氏棉145个,为进一步研究相应物种的NAC转录因子的功能奠定了基础[11]。在雷蒙德氏棉NAC转录因子基因家族的分析中,Shang等[10]将145个NAC-TF基因划分为18个不同的亚家族,其中127个基因分布于13条染色体上,80(55%)个基因含2次重复,6个基因含3次重复。基因表达分析发现大部分NAC-TF基因呈时空和组织特异性表达模式。亚洲棉(Gossypium arboretumL.)被认为是最接近陆地棉A染色体组的供体,其基因组测序已经完成,相关结果发表在2014年5月18日出版的《Nature Genetics》上[12]。本研究利用生物信息学方法对亚洲棉全基因组的NAC基因成员、染色体定位、基因结构和系统进化等进行了分析,以期为进一步进行亚洲棉及陆地棉NAC转录因子基因的克隆和功能分析奠定基础。
1 材料与方法
1.1 亚洲棉NAC基因的鉴定
亚洲棉的基因组序列下载于中国农业科学院棉花研究所网站(http://cgp.genomics.org.cn/page/species/in dex)。将亚洲棉蛋白质序列构建至本地BLAST数据库,以Pfam数据库(http://pfam.xfam.org)中NAM结构域(PF02365)为探针进行BLASTP搜索(E≤ 10-5)。匹配的NAC候选序列经Clustal软件比对后,去除冗余序列,同时去除NAM结构编码氨基酸数目低于120的候选序列。根据亚洲棉NAC基因在染色体上的位置和分布,利用MapChart2.2绘制物理图谱。利用Pfam数据库分析NAC蛋白的保守结构域,存在NAM结构域的蛋白序列属于NAC蛋白家族。基因结构利用在线软件 Gene Structure Display Server(GSDS,http://gsds.cbi.pku.edu.cn/index.php)分析获得[13]。
1.2 亚洲棉NAC基因系统进化树的构建
利用Clustal对亚洲棉和部分功能已知的拟南芥、矮牵牛、小麦、水稻等的NAC蛋白序列进行氨基酸序列多重比对后,利用MEGA 6.0软件采用邻接法(neighbor joining,NJ)构建系统进化树,对构建的进化树进行自检,校验参数bootstrap值重复设为1000。拟南芥NAC蛋白序列来自于拟南芥数据库(http://www.arabidopsis.org)。其它物种的NAC蛋白序列下载自NCBI数据库。
2 结果与分析
2.1 亚洲棉NAC基因的鉴定
利用保守的NAM结构域检索亚洲棉基因的蛋白质序列,共发现138个匹配序列,经Pfam数据库分析,均含有保守的NAM结构域(PF02365)。根据其所在染色体位置及先后顺序进行了系统编号,命名为GaNAC001-GaNAC138。GaNAC基因在亚洲棉13条染色体上皆有分布,但数目不均,其中第2号染色体最少,仅1个,即GaNAC010,而第11号染色体最多,共有16个(图1)。有9条染色体上的GaNAC基因在10个以上。多数NAC基因在染色体上的位置分布较为均匀,但在 3、4、5、7、10、11和 13号染色体上均存在多个NAC基因紧密邻接的基因簇,其中 2重复基因 GaNAC014和 GaNAC015、GaNAC034和 GaNAC035、GaNAC092和 GaNAC093、GaNAC101和GaNAC102、GaNAC133和 GaNAC134相隔基本均在10 kb以内。在其它未进行染色体定位的18个scaffolds上均未检索到NAC基因。GaNAC基因的长度差异较大,平均长度为1957 bp,其中GaNAC099最长,为 7936 bp;GaNAC068最短,为 544 bp。GaNAC基因编码蛋白的长度为78-859个氨基酸,平均长度为326个氨基酸。

图1 GaNAC基因家族在亚洲棉染色体上的位置和分布(基因图距单位为Mb)Fig.1 The location and distribution of GaNAC gene family in Gossypium arboretum chromosomes(The gene map distance unit is Mb)
2.2 亚洲棉GaNAC基因结构的分析
分别下载138GaNAC基因的蛋白序列和DNA序列,利用在线软件GSDS对其基因结构进行了分析。如图2A所示,不同的GaNAC基因结构差异较大,分别含有0-8个内含子,其中GaNAC029没有内含子;9个GaNAC基因仅含1个内含子,占6.5%;88个GaNAC基因含有 2个内含子,占 62.4%;12个GaNAC基因含有3个内含子,占8.6%;其余28个GaNAC基因含有4-8个内含子,占20.2%。另外,内含子的长度差异也较大,最短的内含子长度仅为4 bp,为GaNAC084基因的第1个内含子;最长的内含子长度为4802 bp,为GaNAC097基因的第1个内含子。此外,还有15个GaNAC基因含有长度超过1500 bp的内含子。
138个GaNAC蛋白均含有NAM结构域,其中137个GaNAC的NAM结构域在N末端前150个氨基酸之内,仅有GaNAC059的NAM结构域位于N末端前150个氨基酸之后 (图 2B)。另外,GaNAC037、GaNAC085和GaNAC134、GaNAC095还各含有1个非特异识别 (Non-specific hit)的结构域,分别为DUF2756超家族、marB、Tox_ODYAM1。GaNAC097除含有NAM结构域外,还含有1个特异识别(Specific hit)的PA_subtilisin_lik(枯草杆菌蛋白酶)结构域(E值:9.95E-11)。

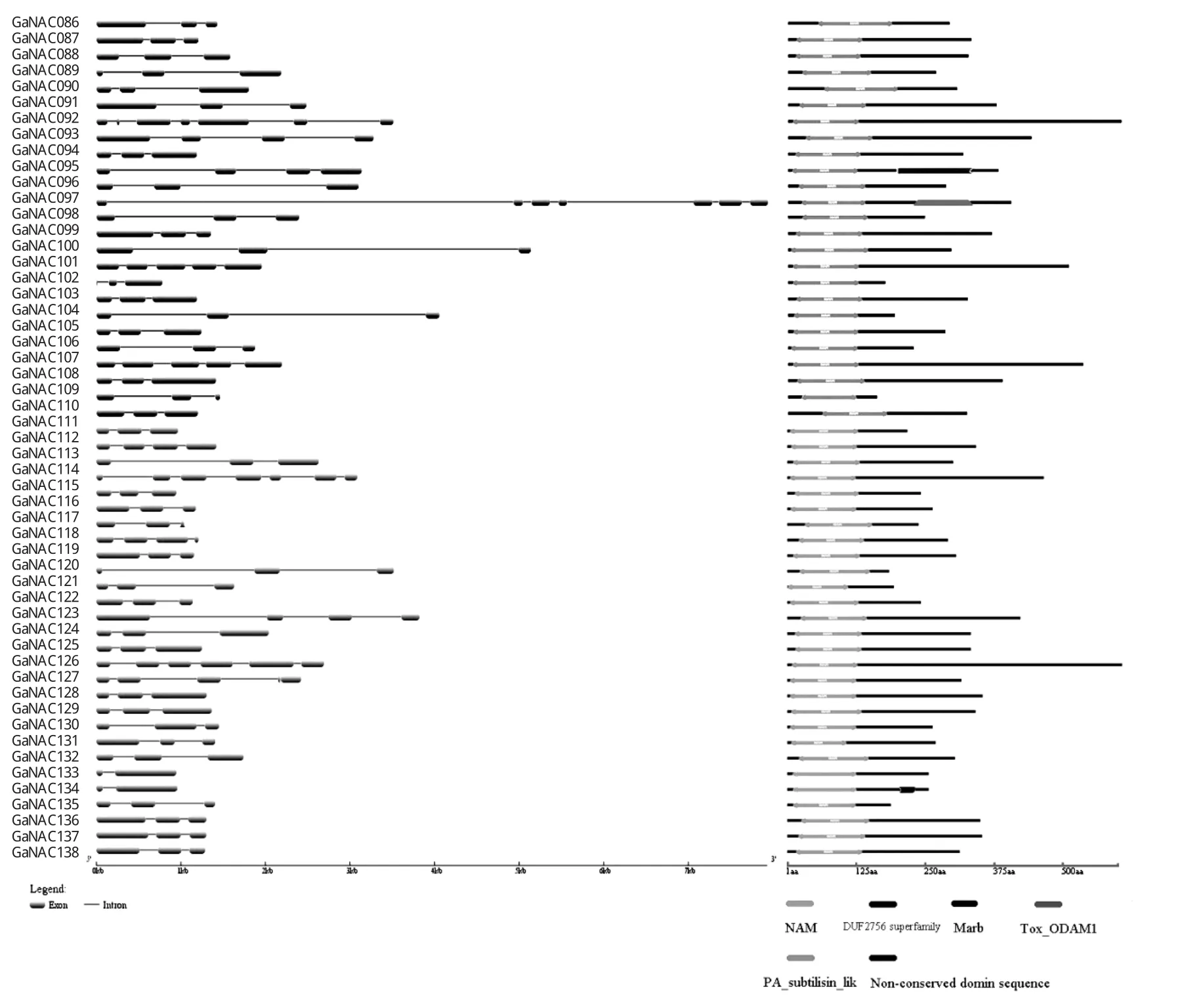
图2 138个亚洲棉NAC转录因子的基因结构及NAM结构域Fig.2 Gene structure and NAM domain of 138 GaNAC gene family
2.3 亚洲棉GaNAC蛋白的系统进化分析
在模式植物拟南芥和水稻中,部分NAC转录因子的功能已经明确,我们从中挑选了14个参与植物生长发育调控、激素信号转导、生物及非生物胁迫响应等功能已知的NAC蛋白,利用ClustalW软件对其与亚洲棉GaNAC蛋白序列进行多重比对,构建系统进化树。根据氨基酸序列的相似程度及其进化关系,将亚洲棉GaNAC蛋白分为11个亚家族(图3)。不同亚家族中的GaNAC蛋白数目差异很大,为4-25个。在第Ⅰ亚家族包含19个GaNAC蛋白,其中GaNAC022、GaNAC040、GaNAC043、GaNAC108、GaNAC 026、GaNAC068和 GaNAC053与拟南芥 SND1、NST1和NST2关系最近,SND1、NST1和NST2在调控次生壁合成中起重要作用[14],暗示这7个GaNAC转录因子的功能可能与次生细胞壁合成有关。第Ⅱ亚家族包含17个GaNAC蛋白,GaNAC023、GaNAC066、GaNAC124、GaNAC055和 GaNAC104等与拟南芥AtNAC1、CUC1/2、NAM共聚在一起 ,AtNAC1、CUC1/2、NAM与顶端分生组织形成有关,表明该亚家族成员可能与组织形成有关。第9染色体上的基因GaNAC084与第1染色体上的基因GaNAC005及GaNAC008旁系同源,两两之间蛋白质一致性高达74.8%以上。第Ⅲ亚家族仅包含4个GaNAC蛋白。第Ⅳ亚家族包含20个GaNAC蛋白,与NTL8聚在一起。在拟南芥种子萌发过程中,高盐可以通过赤霉素信号途径诱导NTL8的表达[15]。位于第11染色体上的基因GaNAC101和GaNAC102,外显子和内含子均不同,但含有完全相同的NAM结构域,编码的蛋白质一致性高达99.5%,可能在信号转导中有相同的功能。第Ⅴ亚家族由23个亚洲棉GaNAC蛋白组成,为亚洲棉最大的亚家族,其中位于第5染色体上的3个基因GaNAC033、GaNAC034和GaNAC035紧密相连,两两相隔约在8 kb以内,氨基酸序列一致性达83%以上,结构和功能相近 (图1-图3),明显是基因倍增的结果,为旁系同源基因。第Ⅵ亚家族包含11个GaNAC蛋白,其中GaNAC136和GaNAC137、GaNAC074和GaNAC105可能为旁系同源基因对,蛋白一致性分别为70.1%和80.5%,该家族蛋白与ATAF1/2、OsNAC6和RD26等生物和非生物胁迫响应转录因子聚在一起,表明这些GaNAC基因可能在逆境应答和防御中起作用。第Ⅶ亚家族包含19个GaNAC蛋白,与NAM-B1、AtNAC2聚在一起,NAM-B1能加速小麦植株衰老[6],AtNAC2与拟南芥的衰老有关[16],故推测该亚家族GaNAC蛋白可能参与植株衰老过程。第Ⅷ至Ⅺ亚家族各包含4、5、4、7个GaNAC蛋白。第8染色体上的2个基因对GaNAC077和 GaNAC078、GaNAC071和 GaNAC072为旁系同源基因对,蛋白质一致性分别为82.0%、92.9%,分别位于Ⅴ、Ⅶ亚家族,而位于第12染色体上的GaNAC115又与GaNAC071和 GaNAC072旁系同源,蛋白质一致性在81%以上。第9染色体上的基因GaNAC085与第13染色体上的基因GaNAC134及 GaNAC133旁系同源,两两之间蛋白质一致性高达92.1%以上,位于第Ⅹ亚家族。
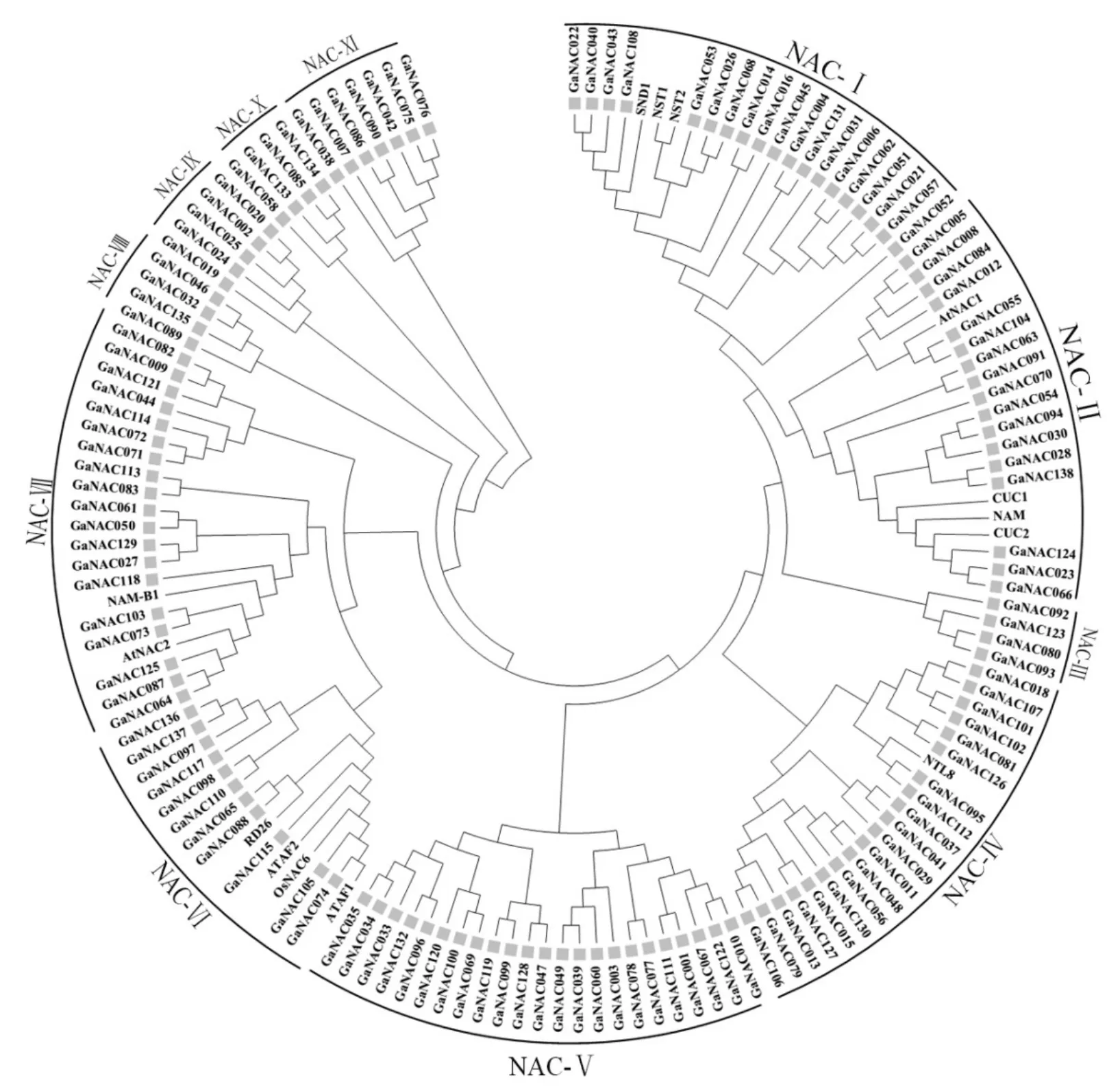
图3 GaNAC蛋白的系统进化分析Fig.3 Polygenetic tree analysis of GaNAC protein family
2.4 亚洲棉与陆地棉、雷蒙德氏棉NAC蛋白的同源性分析
利用138个亚洲棉NAC蛋白序列分别与145个雷蒙德氏棉[10]、78个已克隆的陆地棉[17-20]以及105个拟南芥的NAC蛋白氨基酸序列进行同源性分析。亚洲棉与雷蒙德氏棉NAC蛋白匹配程度最高,平均序列一致性达93.9%,其次为陆地棉,平均序列一致性达81.2%,拟南芥最低,平均一致性为54.9%。有58个直系同源基因的蛋白质序列在亚洲棉、雷蒙德氏棉和陆地棉之间一致性达到95%以上,E值多数为0,如 GaNAC031、GRNAC081和 GhNAC43蛋白序列一致性达100%,说明这些基因在棉属的进化过程中高度保守,对维持棉属的生命功能至关重要;有41个NAC基因在亚洲棉和雷蒙德氏棉之间高度保守,蛋白一致性在95%以上,但和陆地棉之间差异较大,有2个NAC基因在亚洲棉和陆地棉之间高度保守,但和雷蒙德氏棉之间差异相对较大,另有37个NAC基因在亚洲棉、雷蒙德氏棉和陆地棉之间差异均较大,说明这些基因在进化过程中变异程度较高,有些可能是生物体为了适应环境的变化产生了新的功能。
3 讨论
自2000年底拟南芥基因组序列发布以来,植物基因组研究发展很快,截止目前,已有70多个植物基因组测序完成,其中包括二倍体棉花亚洲棉(AA)和雷蒙德氏棉(DD)。基因组测序产生的大量数据为研究基因结构、可变剪切、物种进化和基因家族预测等提供了机会。本研究利用生物信息学方法在亚洲棉基因组中鉴定出了138个GaNAC基因,分布于亚洲棉13条染色体上,并结合部分功能已知的NAC蛋白构建了系统进化分析,为进一步研究GaNAC基因的功能奠定了基础。
(1)根据雷蒙德氏棉、可可(T.cacaoL.)、拟南芥和水稻共473个NAC蛋白序列的N末端NAM结构域的比对结果,Shang等[10]将NAC基因划分为18个亚家族。进一步根据145个雷蒙德氏棉NAC蛋白的多重比对结果,将雷蒙德氏棉NAC基因划分为13个亚家族[10]。在本研究中,我们结合拟南芥、水稻、矮牵牛和小麦中14个功能已知的NAC蛋白序列,将138个亚洲棉GaNAC基因划分为11个亚家族,与上述结果基本一致。一些结构和功能相近,且在染色体上位置紧密相邻的NAC基因,明显是基因倍增的结果,有的为2重复,有的为3重复,这些旁系同源基因体现出亚洲棉基因组中NAC基因的冗余现象。通过与陆地棉、雷蒙德氏棉NAC蛋白的同源性分析,发现一些直系同源的NAC基因在这些种间高度保守,另一些则变异性相对较大,体现出在棉属进化过程中的不同特性。NAC基因在同一棉种或不同棉种中的数目、结构或功能的变化,是由于棉属基因组在进化过程中经历的多轮染色体复制、重排或二倍体化造成的[21]。A、D基因组棉种于600万-730万年前开始分化[22],大约在130万-170万年前A基因组植株被D基因组植株授粉,形成异源四倍体的AD基因组棉种[22]。相对于其它物种,棉属的不同种间基因组同线性和共线性较强,基因组结构在进化上比较保守[23],本研究中发现亚洲棉与雷蒙德氏棉、陆地棉NAC蛋白序列同源性及一致性极高,进一步证实了这种观点。而相对于雷蒙德氏棉,亚洲棉与陆地棉NAC蛋白序列差异较大,可能由于分析中仅采用了陆地棉中的部分NAC蛋白序列,一些更加匹配的陆地棉NAC蛋白序列被排除在外。待陆地棉和海岛棉AD基因组测序结果发表后再进行分析,将会得到更加准确的信息。
(2)截止目前,陆地棉中已经克隆了近78个GhNAC基因[17-20],表达分析发现它们可能在叶片衰老、纤维发育、生物和非生物逆境等方面起作用。我们利用BlastP软件对GaNAC和GhNAC蛋白进行了分析,每一个GaNAC蛋白只选取最佳匹配的GhNAC蛋白,结果发现32个GaNAC蛋白与21个GhNAC匹配。叶片衰老通常被认为是基因和环境共同控制的结果[24]。拟南芥NAC类转录因子中超过1/5的基因(20/106)与叶片衰老有关[24]。Shah等[18]在陆地棉中发现了15个叶片衰老相关的GhNAC转录因子,本研究中22个GaNAC基因与其序列相似性较高,如GaNAC078与GhNAC18间的序列一致性高达99.5%,占亚洲棉总GaNAC的15.60%,与拟南芥中结果相似[24],这些基因为研究棉花的熟性机制及抗早衰机理研究提供了可能。
(3)棉花纤维发育经历了起始期、伸长期(初生壁形成)、次生壁形成和成熟期。Shah等[18]分析了GhNAC基因在陆地棉纤维发育过程中的表达变化,发现GhNAC31、GhNAC49、GhNAC73和GhNAC77在10DPA(days post anthesis)表达量最高,GhNAC53、GhNAC71、GhNAC72在15 DPA时表达量达到高峰,表达这些基因与棉花纤维发育早期伸长(7-20 DPA)有关,而GhNAC25和GhNAC27在25DPA时表达量最强,此时恰逢纤维次生壁合成开始。本研究共发现10个GaNAC基因与陆地棉纤维发育相关GhNAC基因匹配,因此推测它们可能在棉花纤维发育中起作用。
[1]Souer E,van Houwelingen A,Kloos D,et al.The No Apical Meristem gene of Petunia is required for pattern formation in embryos and flowers and is expressed at meristem and primordia boundaries[J].Cell,1996,85(2):159-170.
[2]Aida M,Ishida T,Fukaki H,et al.Genes involved in organ separation in Arabidopsis:an analysis of the cup-shaped cotyledon mutant[J].Plant Cell,1997,9(6):841-857.
[3]Duval M,Hsieh T F,Kim S Y,et al.Molecular characterization of AtNAM:A member of the Arabidopsis NAC domain superfamily[J].Plant Mol Biol,2002,50(2):237-248.
[4]Mao C,Ding W,Wu Y,et al.Overexpression of a NAC-domain protein promotes shoot branching in rice[J].New Phytologist,2007,176(2):288-298.
[5]Xie Q,Frugis G,Colgan D,et al.Arabidopsis NAC1 transduces auxin signal downstream of TIR1 to promote lateral root development[J].Genes&Development,2000,14(23):3024-3036.
[6]Uauy C,Distelfeld A,Fahima T,et al.A NAC gene regulating senescence improves grain protein,zinc,and iron content in wheat[J].Science,2006,314(5803):1298-1301.
[7]Zhong R,Demura T,Ye Z.SND1,a NAC domain transcription factor,is a key regulator of secondary wall synthesis in fibers of Arabidopsis[J].Plant Cell,2006,18:3158-3170.
[8]魏艳玲,贾跃腾,杨超凡,等.新疆无苞芥OpNAC083基因的克隆及表达分析[J].石河子大学学报:自然科学版,2015,33(2):90-97.WEI Yanling,JIA Yueteng,YANG Chaofan,et al.Molecular Cloning and Expression Analyes of OpNAC083 fromOlimaarabidopisisi pumilaL.[J].Journal of Shihezi University:Natural Science,2015,33(2):90-97.
[9]Meng C,Cai C,Zhang T,et al.Characterization of six novel NAC genes and their responses to abiotic stresses inGossypium hirsutumL.[J].Plant Science,2009,176(3):352-359.
[10]Shang H,Li W,Zou C,et al.Analyses of the NAC transcription factor gene family inGossypium raimondiiUlbr.:chromosomal location,structure,phylogeny,and expression patterns[J].Journal of Integrative Plant Biology,2013,55(7):663-676.
[11]Hu H H,Dai M Q,Yao J L,et al.Overexpressing a NAM,ATAF,and CUC(NAC)transcription factor enhances drought resistance and salt tolerance in rice[C].Proceedings of the National Academy of Sciences,USA,2006,103(35):12987-12992.
[12]Li F,Fan G,Wang K,et al.Genome sequence of the cultivated cottonGossypium arboreum[J].Nature Genetics,2014,46(6):567-572.
[13]Guo A Y,Zhu Q H,Chen X,et al.GSDS:a gene structure display server[J].Hereditas,2007,29(8):1023-1026.
[14]Zhong R,Richardson E A,Ye Z H.Two NAC domain transcription factors,SND1 and NST1,function redundantly in regulation of secondary wall synthesis in fibers of Arabidopsis[J].Planta,2007,225(6):1603-1611.
[15]Kim S G,Lee A K,Yoon H K,et al.A membrane-bound NAC transcription factor NTL8 regulates gibberellic acidmediated salt signaling in Arabidopsis seed germination[J].The Plant Journal,2008,55(1):77-88.
[16]Balazadeh S,Siddiqui H,Allu A D,et al.A gene regulatory network controlled by the NAC transcription factor ANAC 092/AtNAC2/ORE1 during salt-promoted senescence[J].The Plant Journal,2010,62(2):250-264.
[17]Shah S T,Pang C,Fan S,et al.Isolation and expression profiling of GhNAC transcription factor genes in cotton(Gossypium hirsutumL.)during leaf senescence and in response to stresses[J].Gene,2013,531(2):220-234.
[18]Shah S T,Pang C,Hussain A,et al.Molecular cloning and functional analysis of NAC family genes associated with leaf senescence and stresses inGossypium hirsutumL.[J].Plant Cell,Tissue and Organ Culture,2014,117(2):167-186.
[19]Huang G Q,Li W,Zhou W,et al.Seven cotton genes encoding putative NAC domain proteins are preferentially expressed in roots and in responses to abiotic stress during root development[J].Plant Growth Regulation,2013,71(2):101-112.
[20]赵凤利,范术丽,宋美珍,等.陆地棉转录因子GhNAC78基因的特征及功能分析[J].棉花学报,2014,26(4):283-289.Zhao F L,Fan S L,Song M Z,et al.Characterization and function analysis of GhNAC78,a transcription factor gene in upland cotton(Gossypium hirstumL.)[J].Cotton Science,2014,26(4):283-289.
[21]Wang K,Wang Z,Li F,et al.The draft genome of a diploid cottonGossypium raimondii[J].Nature Genetics,2012,44(10):1098-1103.
[22]Senchina D S,Alvarez I,Cronn R C,et al.Rate variation among nuclear genes and the age of polyploidy in Gossypium[J].Molecular Biology and Evolution,2003,20(4):633-643.
[23]Rong J,Abbey C,Bowers J E,et al.A 3347-locus genetic recombination map of sequence-tagged sites reveals features of genome organization,transmission and evolution of cotton(Gossypium)[J].Genetics,2004,166(1):389-417.
[24]Guo Y,Cai Z,Gan S.Transcriptome of Arabidopsis leaf senescence[J].Plant,Cell&Environment,2004,27(5):521-549.DOI:10.1111/j.1365-3040.2003.01158.x.

