小麦基部茎秆禾谷丝核菌DNA含量的定量检测
周淼平, 姚金保, 张 鹏, 杨学明, 余桂红, 马鸿翔
(江苏省农业科学院农业生物技术研究所/江苏省农业生物学重点实验室,江苏 南京 210014)
小麦纹枯病(Wheat sharp eyespot)主要由禾谷 丝核菌(Rhizoctonia cerealis vander hoeven)引起,是危害性较大的世界性小麦土传真菌病害[1-3],现已成为中国长江中下游麦区和黄淮麦区的主要病害,每年造成的产量损失严重[4]。抗病品种的培育和应用是控制该病害最经济和有效的途径,小麦抗性的准确鉴定是培育小麦抗病品种以及纹枯病抗性遗传分析的关键。目前已报道的小麦纹枯病抗性鉴定方法多样,鉴定标准不一,主要分为自然病圃鉴定和人工接种鉴定[5],人工接种鉴定根据接种方法的不同,又有玉米砂混法、菌丝外贴法、嵌入法、土表接种法和沟带接种法[6-8]。这些鉴定方法采用的接种菌株不完全相同,病害调查方法也不一致,并且鉴定结果易受环境影响,因而病害鉴定结果存在差异,影响了对小麦材料抗性的准确评价。另外,小麦根部和茎基部其他病害,如小麦根腐病、小麦全蚀病以及小麦茎腐病等,由于与纹枯病早期病征相似,也对抗性的准确评价造成干扰[1,9]。因此,建立准确和可靠的小麦纹枯病抗性鉴定方法非常必要。
为鉴别病原菌,一些基于普通PCR技术的检测方法,如 RAPD[10]、竞争性 PCR[11]以及rDNA的 ITS 序列差异[12]等,先后应用于禾谷丝核菌的检测,但这些方法尚不能对禾谷丝核菌进行定量评估。实时荧光定量PCR(Real-time PCR)技术很好地解决这一问题,通过在PCR体系中加入荧光基团,利用荧光信号累积实时监测整个PCR进程,最后通过标准曲线对未知模板进行总量分析或通过Ct值对模板进行相对定量,与普通PCR技术相比,其特异性更强,灵敏度更高。目前,该技术已经成功运用于土壤中纹枯病病菌DNA的定量检测[13-14],少量感病小麦茎秆材料也尝试采用该技术进行禾谷丝核菌DNA含量的定量测定[9],但小麦茎秆中禾谷丝核菌的DNA含量与小麦自身的纹枯病抗性的关系尚未有文献报道。
本研究的目的是建立小麦茎秆材料中禾谷丝核菌DNA含量的定量测定方法,探索小麦纹枯病抗性与其茎秆中禾谷丝核菌DNA含量的关系,为今后开展小麦纹枯病抗性鉴定和抗性QTL的定位研究奠定基础。
1 材料与方法
1.1 供试材料
小麦纹枯病抗病品种CI12633和感病品种扬麦158以及其他44份供试品种或品系(表1和表2),由江苏省农业科学院农业生物技术研究所小麦室保存和提供。小麦禾谷丝核菌R0301由江苏省农业科学院植物保护研究所陈怀谷研究员提供。
1.2 DNA提取和浓度测定
小麦禾谷丝核菌DNA和小麦茎秆(含叶鞘)DNA均采用Karroten DNA提取试剂盒(北京西美杰科技有限公司生产)提取,DNA浓度和质量采用Eppendorf Biophotometer测定。
1.3 小麦背景的禾谷丝核菌DNA浓度标准曲线的建立
1.3.1 Real-time PCR反应体系 Real-time PCR引物序列参考文献[13],正反向引物序列分别为5'-CCTAAATGAGTCTGGAGTAAGTC-3'和5'-GCTAGTGCGGTCAATGTATAG-3'。Real-time PCR采用SYBR®GreenI试剂(TaKaRa公司生产)在LightCycler2.0荧光定量PCR仪(Roche公司生产)上进行,反应总体积为10 μl,含2 ×SYBR ® Premix Ex TaqTM5 μl,5 μmol/L正反向引物各 1 μl,模板 DNA 3 μl。
1.3.2 标准曲线的建立 将禾谷丝核菌DNA按浓度梯度分别掺入未接种的宁麦13 DNA溶液(浓度15 ng/μl)中,使禾谷丝核菌DNA最终浓度分别为40 000.0 μg/L、4 000.0 μg/L、400.0 μg/L、40.0 μg/L、4.0 μg/L、0.4 μg/L。采用禾谷丝核菌 DNA浓度梯度样品为模板进行Real-time PCR,以禾谷丝核菌DNA量的对数为横坐标,Real-time PCR获得的Ct值为纵坐标,采用Excel软件建立标准曲线。
1.4 小麦禾谷丝核菌接种及其DNA含量测定
小麦禾谷丝核菌接种分别采用田间接种和室内幼苗接种2种方法。田间接种采用病麦粒嵌入法,于2014年3月小麦拔节期将带有禾谷丝核菌的麦粒嵌入到小麦植株贴近地面的基部第1~2叶鞘之间,喷雾保湿3 d。室内幼苗接种采用菌丝直接接种,小麦种子经萌发催芽,芽长5 mm左右,浸于经3 mm直径钢珠打碎的禾谷丝核菌菌丝悬浮液中5 min,每10粒接种后的种子呈直线均匀置于灭菌的湿润纸巾(24 cm×16 cm)上,芽的生长方向指向纸巾边缘,芽上端离纸巾边缘3 cm,将纸巾从一端向另一端卷起,置于泡沫盒中,20℃,保湿培养。
首先采用抗纹枯病品种CI12633和感纹枯病品种扬麦158探索合适的定量测定取样时间,于田间接种后2 d、4 d、6 d、8 d、10 d、12 d 和14 d 各取5 株小麦植株基部茎秆(1 cm,含叶鞘),混合,液氮磨碎,提取DNA,进行定量检测,根据标准曲线计算病原菌DNA含量,重复3次。室内接种后2 d、4 d、6 d、8 d、10 d和12 d取10株小麦基部茎秆(含叶鞘),检测方法同上。
其他供试小麦品种样品按同样方法接种,于接种后12 d取样,DNA提取和定量检测同上,重复3次。
1.5 小麦品种的纹枯病抗性鉴定
田间接种后于小麦乳熟期调查纹枯病发病情况,每个品种调查15~20个发病茎秆,计算平均病级。病级根据王裕中等[15]方法略作修改:1级:叶鞘有典型的纹枯病病斑,但不侵茎;2级:病菌侵入茎秆,病斑宽度环茎不超茎秆周长1/2;3级:茎秆上病斑环茎宽度为茎秆周长1/2~3/4;4级:茎秆上病斑绕茎秆3/4以上,或茎秆已软腐;5级:枯孕穗或枯白穗。
接种后的室内幼苗于取样当天调查纹枯病发病情况,根据病害严重度将病级分为5级:1级为第1叶鞘病斑长度小于1.5 cm;2级为第1叶鞘病斑长度1.5~3.0 cm;3级为第1叶鞘病斑长度大于3.0 cm,幼苗未萎焉;4级为幼苗出现萎蔫病症;5级为幼苗死亡,根据调查结果,计算平均病级。
1.6 统计分析
采用SAS 9.0软件分析不同小麦品种(系)基部茎秆中禾谷丝核菌DNA含量与其纹枯病抗性的相关性。
2 结果与分析
2.1 小麦禾谷丝核菌接种方法
小麦纹枯病发病的高峰期主要在苗期和拔节孕穗期[16],目前小麦纹枯病抗性评价方法多针对拔节孕穗期即成株期的抗性,而对小麦苗期的抗性评价方法鲜有报道。本研究在室内采用禾谷丝核菌直接接种的方法对小麦幼苗纹枯病抗性进行评价,该鉴定方法的温度和湿度可控,重复性好,操作简便。对成株期的抗性评价采用禾谷丝核菌病麦粒嵌入法在田间进行。
本研究有10份材料同时进行室内苗期和田间成株期抗性鉴定,对2种抗性鉴定结果进行相关性分析发现,相关系数只有0.41,表明幼苗的纹枯病抗性机制与小麦成株期可能不完全相同。
2.2 标准曲线的建立
采用Guo等[13]的方法根据禾谷丝核菌β-tublin基因序列设计的引物,对小麦DNA样品和掺有禾谷丝核菌DNA的小麦DNA背景样品扩增,只有掺有禾谷丝核菌DNA的小麦DNA样品扩增出预期的138 bp条带,表明小麦基因组中没有高同源性序列,可以用于小麦组织样品中禾谷丝核菌DNA的定量检测。优化 LightCycler2.0荧光定量 PCR仪Real-time PCR扩增条件,采用的扩增条件为:95℃30 s;95 ℃ 5 s,60 ℃ 10 s,72 ℃ 20 s,40个循环。
对禾谷丝核菌DNA浓度梯度掺入的小麦DNA样品进行Real-time PCR检测,发现除了0.4 μg/L样品未有扩增产品外,其余样品均能正常扩增,且重复性较好,表明该检测方法的最低检测量为4 pg。以样品中禾谷丝核菌DNA质量的对数值为横坐标,Real-time PCR测得的Ct值为纵坐标,构建标准曲线(图1),回归方程为Y=-3.562x+35.310,相关性较好,R2=0.999。
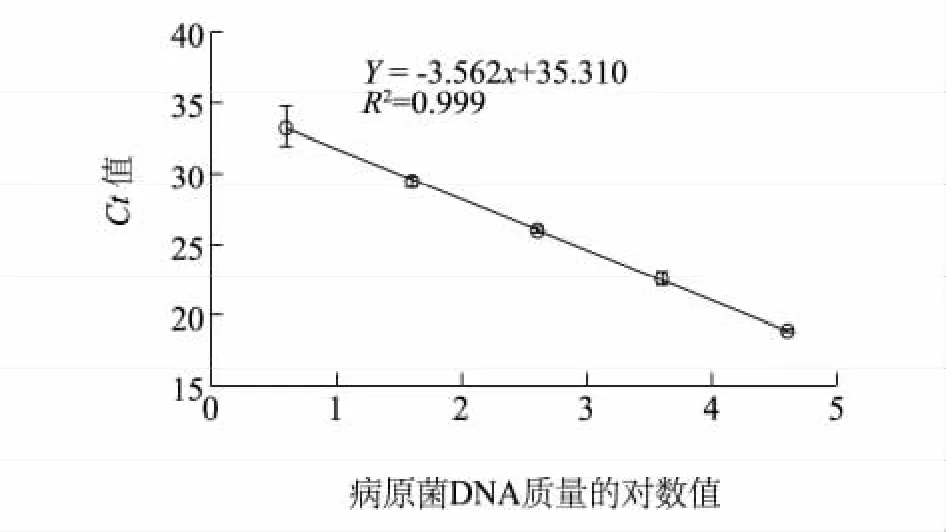
图1 标准曲线图Fig.1 Standard curve
2.3 小麦茎秆基部禾谷丝核菌DNA含量的定量检测
抗病品种和感病品种接种后,基部茎秆DNA样品作为模板进行Real-time PCR检测获得相应的Ct值,再根据图1建立的回归方程、DNA样品体积以及茎秆样品的质量可计算出基部茎秆中禾谷丝核菌DNA含量。由于室内幼苗接种菌丝的接种量大,接种2 d,抗纹枯病品种CI12633和感纹枯病品种扬麦158基部茎秆的禾谷丝核菌DNA含量分别为267.0 ng/g和373.7 ng/g,接种6 d,抗病品种和感病品种的差异非常明显,12 d后,抗病品种和感病品种基部茎秆中禾谷丝核菌DNA含量分别为1 723.9 ng/g和4 166.1 ng/g,抗病品种和感病品种基部茎秆中禾谷丝核菌DNA含量呈指数级增长(图2A)。
田间接种的抗病品种和感病品种,接种2 d也可检测出禾谷丝核菌DNA;接种8 d,基部茎秆中禾谷丝核菌DNA含量差别明显,分别为179.6 ng/g和595.2 ng/g;此时小麦纹枯病病征尚未出现,表明该方法在小麦纹枯病的早期检测中可以发挥重要作用;接种14 d,抗病品种和感病品种基部茎秆中禾谷丝核菌DNA含量分别为849.4 ng/g和3 855.1 ng/g,感病品种禾谷丝核菌DNA含量是抗病品种的4倍多;与室内接种相似,抗病品种和感病品种基部茎秆中禾谷丝核菌DNA含量也呈指数级增长(图2B)。
综合2种接种方法结果,后续品种定量测定均采用接种后12 d的样品。

图2 CⅠ12633和扬麦158接种禾谷丝核菌后,基部茎秆中禾谷丝核菌DNA含量变化Fig.2 The changes of Rhizoctonia cerealis DNA content in the stalk of CⅠ12633 and Yangmai158 after inoculation
2.4 小麦幼苗期纹枯病抗性与基部茎秆禾谷丝核菌DNA含量的相关性
对42份小麦品种(系)在室内采用禾谷丝核菌菌丝直接接种方法进行了纹枯病抗性鉴定,结果表明,纹枯病平均病级为2.4,变幅为1.5~3.3;荧光定量PCR分析结果显示,42份材料的基部茎秆禾谷丝核菌 DNA含量平均为4 101.0 ng/g,变幅为566.3~9 887.2 ng/g(表1)。相关性分析发现,幼苗基部茎秆禾谷丝核菌DNA含量与其纹枯病平均病级具有较高的相关性,相关系数为0.76(P<0.000 1),荧光定量PCR测定的基部茎秆禾谷丝核菌DNA含量可以作为判定小麦幼苗纹枯病抗性的重要指标(图3)。

表1 室内幼苗接种小麦纹枯病平均病级与茎秆禾谷丝核菌DNA含量Table 1 The average disease grade of wheat sharp eyespot and R.cerealis DNA content in the stalk by seedling inoculation
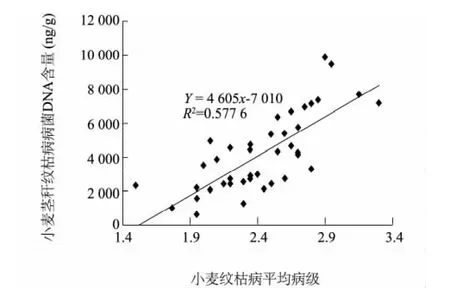
图3 小麦幼苗期茎秆禾谷丝核菌DNA含量与纹枯病平均病级的相关性Fig.3 Correlation between R.cerealis DNA content in wheat stalk and disease grade of sharp eyespot
2.5 小麦成株期纹枯病抗性与基部茎秆禾谷丝核菌DNA含量的相关性
在田间对12份小麦品种(系)采用病麦粒嵌入接种法进行抗性鉴定,同时对基部茎秆的禾谷丝核菌DNA含量进行定量测定(表2),相关性分析结果显示,田间小麦基部茎秆的禾谷丝核菌DNA含量与其纹枯病病级没有明显的相关性(r=-0.30,P=0.34),表明对拔节期小麦基部茎秆禾谷丝核菌DNA含量的定量分析结果对乳熟期小麦纹枯病抗性的判断帮助不大。主要原因是定量分析取样时间与纹枯病抗性调查时间相隔太长,期间小麦经历了孕穗期、抽穗期和开花灌浆期,并且田间可能发生禾谷丝核菌多次侵染,成株期的抗性评价与苗期抗性评价标准也不同,不仅涉及叶鞘的侵染,而且涉及茎秆的侵染。本研究接种后12 d测定的主要是叶鞘中禾谷丝核菌DNA含量,与乳熟期小麦纹枯病抗性相关不明显也很正常。
3 讨论
Real-time PCR既能进行定性分析,又能进行高灵敏度的定量分析,且操作简便,近十多年来在植物病原菌的鉴别和定量分析中应用广泛。Guo等[13]采用该方法,不仅将禾谷丝核菌与其他20余种土壤病原菌区分开,而且可以检测最低达100 fg的禾谷丝核菌DNA。本研究采用该方法,建立了小麦背景的禾谷丝核菌DNA测定标准曲线,最低可以检测到4 pg的禾谷丝核菌DNA,在4 μg/L至40 000 μg/L范围内有较好的线性关系(R2=0.999),对今后开展小麦茎秆的禾谷丝核菌DNA含量测定提供了很好的工具,特别对于小麦纹枯病病征尚未能明显区分的小麦材料间的纹枯病抗性鉴别,定量PCR结果可以作为指示小麦纹枯病是否发生的重要指标。

表2 小麦成株期纹枯病平均病级及茎秆禾谷丝核菌DNA含量Table 2 The average disease grade of wheat sharp eyespot and R.cerealis DNA content in wheat stalk by field inoculation
小麦田间纹枯病发病的高峰期主要在苗期和拔节孕穗期,目前的抗性鉴定方法多针对拔节孕穗期即成株期,对苗期的抗性和鉴定方法重视不够。本研究采用室内菌丝直接接种的方法进行幼苗的纹枯病抗性鉴定,室内鉴定温度和湿度容易控制,抗性鉴定结果重复性好,可靠性较高。研究发现,小麦幼苗基部茎秆禾谷丝核菌DNA含量与其纹枯病平均病级数有较高的相关性(r=0.76),因此小麦幼苗基部茎秆禾谷丝核菌DNA含量的多少可以用于预示小麦幼苗的纹枯病抗性。仪器测定可以减小人为的目测鉴定误差,从而提高小麦幼苗抗性鉴定的准确性。
小麦纹枯病与小麦白粉病类似,苗期抗性和成株期抗性不完全相同,苗期抗性只涉及叶鞘的侵染,成株期抗性不仅涉及叶鞘的侵染,对茎秆的侵染更为重要。研究也发现,拔节期接种12 d,小麦基部茎秆禾谷丝核菌DNA含量与成株期抗性相关不明显,本研究只调查了乳熟期小麦纹枯病的发生情况,其纹枯病平均病级与接种后12 d取样测定的小麦幼苗基部茎秆禾谷丝核菌DNA含量没有很好的对应关系。今后需对拔节期、孕穗期、抽穗期和开花灌浆期分别进行基部茎秆禾谷丝核菌DNA含量定量测定以及纹枯病病害调查,才能正确判定小麦基部茎秆禾谷丝核菌DNA含量与其成株期抗性的关系。
[1] HAMADA MS,YIN Y,CHEN H,et al.The escalating threat of Rhizoctonia cerealis,the causal agent of sharp eyespot in wheat[J].Pest Manag Sci,2011,67:1411-1419.
[2] 蒋彦婕,朱芳芳,蔡士宾,等.小麦纹枯病抗性QTL遗传分析[J]. 江苏农业学报,2014,30(6):1222-1226.
[3] 陈双喜,张乐乐,朱丽娟,等.小麦纹枯病生防菌的筛选和鉴定[J]. 江苏农业科学,2013,41(5):97-98.
[4] CHEN L,ZHANG Z Y,LIANG H X,et al.Overexpression of TiERF1 enhances resistance to sharp eyespot in transgenic wheat[J].Journal of Experimental Botany,2008,59:4195-4204.
[5] 李斯深,刘爱新,李 强.小麦纹枯病抗性研究进展[J].山东农业大学学报,1999,30(1):85-90.
[6] 史建荣,王裕中,陈怀谷,等.小麦纹枯病品种抗性鉴定技术及抗病资源的筛选与分析[J].植物保护学报,2000,27(2):107-112.
[7] 陈厚德,王彰明,倪桂花.用嵌入法接种小麦纹枯病菌的研究[J].江苏农业科学,2002(5):31-32.
[8] 颜 伟,蔡士宾,吴纪中,等.小麦纹枯病不同接种方法的比较[J].安徽科技学院学报,2007,21(5):8-12.
[9] HAMADA M S,YIN Y N,MA Z H.Detection of Rhizoctonia cerealis in wheat tissues by a real-time PCR assay [J].Journal of Plant Pathology,2012,94(1):215-217.
[10] NICOLSON P,PARRY D W.Development and use of a PCR assay to detect Rhizoctonia cerealis,the cause of sharp eyespot in wheat[J].Plant Pathol,1996,45:872-883.
[11] NICOLSON P,TURNER A S,EDWARDS S G,et al.Development of stem-base pathogens on different cultivars of winter wheat determined by quantitative PCR[J].Eur J Plant Pathol,2002,108:163-177.
[12]陈怀谷,方 正,陈厚德,等.小麦纹枯病菌的分子检测[J].植物保护学报,2005,32(3):261-265.
[13] GUO Y,LI W,SUN H,et al.Detection and quantification of Rhizoctonia cerealis in soil using real-time PCR[J].J Gen Plant Pathol,2012,78:247-254.
[14]徐娜娜,李鹏昌,于金凤.应用real-time PCR定量检测土壤中小麦纹枯病菌方法的建立[J].山东农业科学,2014,46(3):106-109.
[15]王裕中,吴志凤,史建荣,等.小麦纹枯病流行规律研究[J].江苏农业科学:麦类纹枯病专辑,1993:48-53.
[16]张会云,陈荣振,冯国华,等.中国小麦纹枯病的研究现状与展望[J].麦类作物学报,2007,27(6):1150-1153.

