武汉市丙酸杆菌对红霉素耐药与23S rRNA点突变和携带erm基因相关
曾宪玉 姜敏 付辰 董碧麟 吴卓漩 王玮蓁
武汉市丙酸杆菌对红霉素耐药与23S rRNA点突变和携带erm基因相关
曾宪玉 姜敏 付辰 董碧麟 吴卓漩 王玮蓁
目的探讨武汉市耐红霉素丙酸杆菌23S rRNA有无点突变以及携带ermX基因的Tn5432转座子是否转入了丙酸杆菌。方法从痤疮患者皮损中分离丙酸杆菌,E-test法检测分离株对红霉素和克林霉素的MIC值。PCR扩增耐药株23S rRNA、ermX、ermX(cj)、IS1249a、IS1249b并测序,并在基因库中比较。结果19株痤疮丙酸杆菌(P.acnes)和10株卵白丙酸杆菌(P.avidum)对红霉素均表现为高度耐药(MIC均>256 μg/ml)。19株P.acnes中,16株对克林霉素高度耐药(MIC>256 μg/ml),3株敏感;10株P.avidum对克林霉素高度耐药(MIC>256 μg/ml)。19株P.acnes耐药株中,7株在相当于E.coli23S rRNA 2058位点发现由A→G点突变,均对红霉素和克林霉素高度耐药;4株在相当于E.coli23S rRNA 2059位点发现由A→G点突变,其中1株对克林霉素耐药,3株敏感;另外8株P.acnes扩增ermX阳性,其序列与基因库中P.acnesermX基因100%同源。10株P.avidum中,2株ermX扩增阳性,其序列与P.acnesermX基因100%同源;另外8株扩增ermX(cj)得到预期片段PCR产物,与基因库中Corynebacterium jeikeiumermX(cj)序列99%同源,而与P.acnesermX基因仅有94%的同源性。10株扩增ermX基因阳性的菌株扩增IS1249a和IS1249b均阳性,而其余菌株均阴性。结论武汉市耐红霉素丙酸杆菌分别由相当于E.coli23S rRNA 2058、2059由A→G点突变、携带ermX的Tn5432传入以及ermX(cj)传入丙酸杆菌引起。
丙酸杆菌属;红霉素;克林霉素;23S rRNA;基因,erm
作者单位:430022武汉市第一医院皮肤科(曾宪玉、姜敏、付辰、董碧麟、吴卓璇、王玮蓁);湖南中医药大学(曾宪玉)
痤疮丙酸杆菌(P.acnes)是引起痤疮炎症反应的重要致病菌,随着红霉素和克林霉素制剂外用治疗痤疮,丙酸杆菌对红霉素和克林霉素耐药报道在欧洲、美洲、大洋洲和亚洲等地区相继出现[1-2]。国外研究表明:丙酸杆菌23S rRNA的点突变和携带红霉素耐药基因是其耐红霉素的机制,并根据基因点突变的不同位点将其分为4个不同的耐药表型[3]。我们在前期[4]实验中已从痤疮患者皮损中分离到耐红霉素丙酸杆菌,本研究拟了解耐药株23S rRNA有无点突变以及携带ermX基因的Tn5432转座子是否转入了丙酸杆菌。
资料与方法
一、资料
1.菌株:29株丙酸杆菌分离株均从2009—2010年武汉市第一医院皮肤科门诊痤疮患者皮损中分离,经革兰染色、23S rRNA测序鉴定为丙酸杆菌。29例中,15例在取材前服用过多西环素、米诺环素或罗红霉素至少2周以上;4例外用过克林霉素制剂;4例同时外用克林霉素和口服上述抗生素;6例无明确的抗生素治疗史。经鉴定的丙酸杆菌分离株[4]用LB溶液保存于-80℃冰箱。进行E-test药敏试验前至少转种厌氧培养两次。P.acnesATCC11827购自美国ATCC标准菌库。
2.主要试剂和仪器:强化布氏琼脂、布氏肉汤、红霉素和克林霉素E-test条、厌氧产气袋、厌氧试剂条、比浊仪均购于法国梅里埃公司,TaqDNA聚合酶和dNTP由上海invitrogen公司提供。PCR扩增仪购于德国Eppendorf公司。
二、方法
1.E-test药敏方法:参照美国临床和实验室标准协会第7版关于厌氧菌药敏试验指南和文献[2],采用E-test方法检测分离丙酸杆菌菌株红霉素和克林霉素药敏试验。主要步骤如下:按要求配置5%羊血强化布氏琼脂(每1 000 ml含43 g强化布氏琼脂、维生素K1溶液1 ml、氯化血红蛋白溶液1 ml),制备5%强化布氏琼脂血平板。-4℃保存备用。取2~3个转种2次以上的单菌落,用布氏肉汤调整至0.5麦氏比浊,分3个方向均匀涂布于5%强化布氏琼脂血平板,室温放置15min,放置E-test条。厌氧培养48 h,观察结果。P.acnesATCC11827作为阴性对照。
2.DNA提取:按文献进行操作[5],获得单菌落的分离菌株后,取单个菌落加入200 μl DB溶液,100℃10min,-20℃2 h以上,11 700×g离心3min,取上清,加200 μl 1/2 TE溶液,获得DNA模板。-20℃保存。
3.引物、PCR和测序:扩增丙酸杆菌23S rRNA、ermX、ermX(cj)、IS1249a、IS1249b 引 物 和PCR反应条件见表1。PCR扩增条件:每个25 μl PCR反应体系组成如下:5 μl DNA模板、正向引物和反向引物(100 pmol/μl)分别 0.25 μl,1.25 dNTP(各 2.5 mmol/L),2.5 μl 10 × 缓冲液、0.1 μl DNA Taq 聚合酶(5 U/μl),加水至 25 μl。 IS1249b 扩增体系中, 除dNTP为2.0 μl及0.2 μl DNA Taq聚合酶外,余体系同上。以H2O作为阴性对照。取6 μl PCR产物在1.2%凝胶上电泳判读结果。所有扩增产物均用上游引物单向测序。引物合成和测序均由武汉华大基因公司完成。

表1 引物序列和PCR扩增条件
结 果
一、分离株鉴定结果
29株菌23S rRAN引物扩增均得到预计片段大小的PCR产物(430 bp),序列在基因库中Blast结果显示:19株分离株的23S rRNA扩增序列与P.acnes23S rRNA序列(NR_076216.1)显示有98%~100%的一致性;结合革兰染色,鉴定为P.acnes。另外10株分离株的23S rRNA扩增序列与卵白丙酸杆菌(P.avidum)23S rRNA序列(基因库No:NR_103003.1)显示有99%~100%的同源性;结合革兰染色,鉴定为P.avidum。
二、药敏结果
19株P.acnes和10株P.avidum均对红霉素表现为高度耐药,MIC均 >256 μg/ml。19株P.acnes中,16株对克林霉素高度耐药(MIC>256 μg/ml),3株敏感;10株P.avidum对克林霉素高度耐药(MIC> 256 μg/ml)。
三、23S rRNA和erm基因测序结果
19株P.acnes中,7株在相当于E.coli23S rRNA2058点发现由A→G点突变(图1),均对红霉素和克林霉素高度耐药(MIC>256 μg/ml);4株在相当于E.coli23S rRNA 2059位点发现由A→G突变(图1),其中1株分离株对克林霉素高度耐药(MIC > 256 μg/ml),3 株敏感(MIC 1.5 ~ 2 μg/ml);另外8株23S rRNA未检测到点突变的分离株ermX扩增得到预期片段PCR产物(395 bp,图2),其序列均与基因库中P.acnesermX基因100%同源(基因库 No:AF411029.1)。10株P.avidum耐药株中,在相当于E.coli23S rRNA 2058、2059位点均未发现点突变;2株ermX扩增得到预期片段PCR产物(图2),其序列与基因库中P.acnesermX基因100%同源(基因库 No:AF411029.1);另外8株扩增ermX(cj)得到预期片段PCR产物(图2,394 bp),与基因库中Corynebacterium jeikeiumermX(cj)序99%同源(基因库No:AF338706.1),相差一个碱基,而与P.acnesermX基因仅有94%的同源性。所有耐药株在相当于E.coli23S rRNA 2057位点均未检测到碱基突变。见表2。
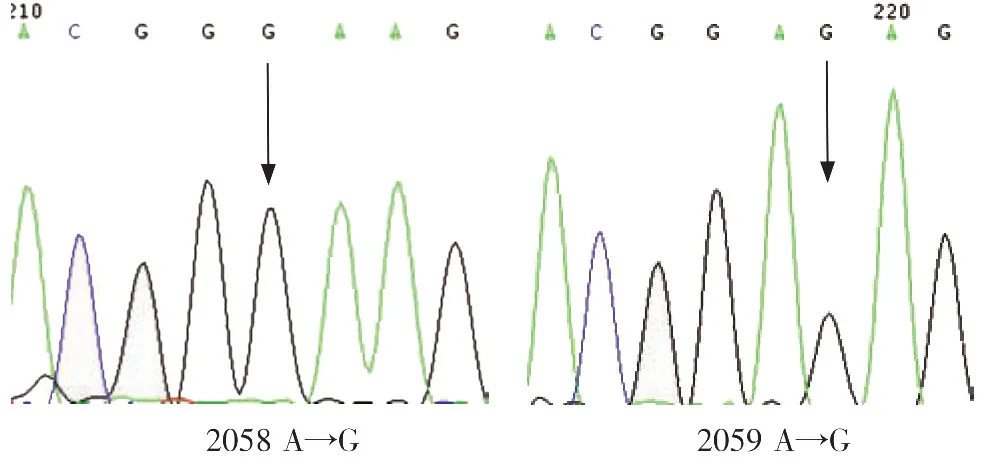
图1 扩增P.acnes分离株23S rRNA在相当于E.coli23S rRNA 2058及2059位点的由A→G突变
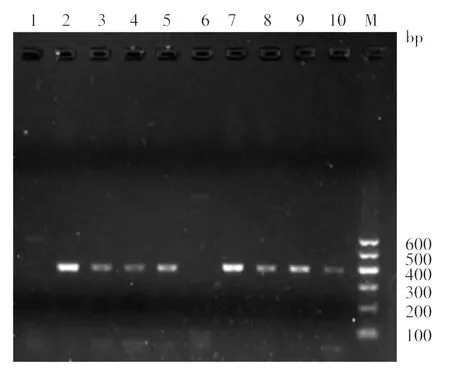
图2 丙酸杆菌ermX PCR和ermX(cj)PCR扩增结果 1:阴性;2~5:ermX(cj);6:阴性;7~10:ermX;M:标准参照物
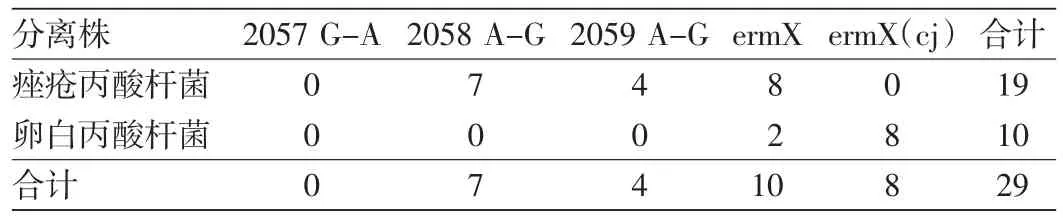
表2 红霉素耐药丙酸杆菌的耐药结果
四、IS1249a和IS1249b扩增结果
10株携带P.acnesermX的菌株均扩增出IS1249a及IS1249b的预期片段产物(分别为454 bp和1 105 bp,图3),序列Blast显示与基因库中Tn5432转座子中IS1249a和IS1249b分别具有100%的同源性(基因库No:AF411029.1)。23S rRNA 2058和2059点突变菌株和携带99%Corynebacterium jeikeiumermX(cj)基因的菌株IS1249a及IS1249b PCR扩增均为阴性。
讨 论
耐红霉丙酸杆菌菌株最初报道见于美国,后在欧洲和亚洲等地区相继出现[1-2]。Ross根据其对大环类酯类-林可霉素类-链阳性霉素B类抗生素(MLS)MIC的差别,分为4个耐药表型:Ⅰ型为对所有MLS类抗生素固有耐药;Ⅱ型为所有大环类酯抗生素固有低度耐药和诱导性高度耐药;Ⅲ型仅对红霉素耐药,对16环大环类酯抗生素敏感;Ⅳ型对所有的14环、15环和16环大环类酯类高度耐药,对克林霉素敏感[1]。随后研究证明Ⅰ、Ⅲ和Ⅳ型耐药分别由相当于E.coli23S rRNA的2058(A→G)、2057(G→A)、2059(A→G)的基因点突引起[3],而Ⅱ型由携带了位于转座子中的红霉素耐药基因ermX引起[6]。本研究中,共7株在相当于E.coli23S rRNA 2058位点出现由A→G的突变,均表现为对红霉素和克林霉素高度耐药,与文献报道耐药表型一致;4株在相当于E.coli23S rRNA 2059位点出现由A→G的突变,均对红霉素高度耐药,但3株对克林霉素敏感、另外1株耐药,其药敏表型与文献结果有差异,原因不明。10株携带ermX的P.acnes和P.avidum均对红霉素和克林霉素高度耐药(MIC>256 μg/ml)。ermX在其最早的宿主中呈诱导性表达[7],但本研究中所有携带ermX的分离株均对红霉素高度耐药,提示其为固有表达,与欧洲和日本报道结果相似[6,8]。这8株分离株均从有红霉素类或克林霉素类抗生素使用史的患者中分离,也可能是ermX固有表达的原因。
Tn5432转座子最先报告见于Tauch等[7]报道,其由两个拷贝的IS1249和红霉素耐药基因ermCX组成。2002年Ross报道在耐红霉素的丙酸杆菌菌株中发现了全长4 259 bp的Tn5432转座子,其由两个位于两侧的编码转座酶的IS1249序列、一个插入序列(ISCXI)和ermX基因构成;携带Tn5432的丙酸杆菌(包括P.acnes、P.avidum、P.granulosum)对所有MLS类均高度耐药[6]。2010年埃及也报道了由Tn5432转座子中的ermX基因的插入导致了耐MLS类抗生素P.acnes[9],本研究中8株P.acnes和2株P.avidum携带的ermX序列与P.acnesermX具有100%的同源性;且10株分离株均检测到与Tn5432序列一致的IS1249a和IS1249b,提示Tn5432转座子已在中国出现,且携带ermX进入P.acnes中引起红霉素耐药。本研究是首次在国内报道P.acnes携带ermX基因。
8株P.avidum携带ermX(cj),其序列与基因库中Corynebacterium jeikeium的ermX(cj)具有99%的同源性,而与P.acnesermX基因仅有94%的同源性,与从埃及痤疮患者皮损中分离的P.acnes携带的ermX(cj)序列一致[9];且所有携带ermX(cj)的P.acnes和P.avidum分离株中均未检测到IS1249a和IS1249b,提示ermX(cj)不是由Tn5432携带进入丙酸杆菌,其是否通过其他转座子进入丙酸杆菌有待进一步研究。本研究中,仅在P.avidum中检测到ermX(cj),后者是否仅携带于P.avidum中或在丙酸杆菌中均存在,因本研究分离的耐药菌株较少,尚待扩大样本量进一步研究。本研究在P.acnes和P.avidum中均检测到位于Tn5432中的ermX基因,提示Tn5432通过不同途径进入丙酸杆菌的至少两个种内,或在丙酸杆菌内转移。Tn5432是一个非融合性转座子,其最初的宿主中位于携带多个耐药基因的pPT10质粒中[10],既往研究在携带Tn5432和ermX的丙酸杆菌中未发现pPT10质粒存在的证据,Tn5432是通过其他质粒或整合到丙酸杆菌染色体中尚待研究。
大环类酯类和林可霉素类是治疗痤疮的常用抗生素,自从克林霉素和红霉素外用制剂用于治疗痤疮后,耐红霉素的P.acnes株在世界各地相继出现[1-2,11],P.acnes的耐药不断增多,是抗生素治疗痤疮疗效差或无效的主要原因。本研究表明,武汉市丙酸杆菌的耐药由23S rRNA点突变和erm基因两种不同的机制引起,前者提示丙酸杆菌面临高强度的抗生素压力,后者提示其他细菌携带的红霉素耐药基因已通过多种途径传入丙酸杆菌,值得临床关注。
[1]Ross JI,Snelling AM,Eady EA,et al.Phenotypic and genotypic characterization of antibiotic-resistantPropionibacteriumacnesisolated from acne patients attending dermatology clinics in Europe,the U.S.A.Japan and Australia[J].Br J Dermatol,2001,144(2):339-346.
[2]Ishida N,Nakaminami H,Noguchi N,et al.Antimicrobial susceptibilities ofPropionibacterium acnesisolated from patients with acne vulgaris[J].Microbiol Immunol,2008,52(12):621.
[3]Ross JI,Eady EA,Cove JH,et al.Clinical resistance to erythromycin and clindamycin in cutaneous propionibacteria isolated from acne patientsisassociated with mutationsin 23S rRNA[J].Antimicrob Agents Chemother,1997,41(5):1162-1165.
[4]曾宪玉,姜敏,付辰,等.武汉地区痤疮患者皮损中分离丙酸杆菌的耐药研究[J].中国皮肤性病学杂志,2012,28(2):131-134.
[5]Zeng X,Kong F,Wang H,et al.Simultaneous detection of nine antibiotic resistance-related genes inStreptococcus agalactiaeusing multiplex PCR and reverse line blot hybridization assay[J].Antimicrob Agents Chemother,2006,50(1):204-209.
[6]Ross JI,Eady EA,Carnegie E,et al.Detection of transposon Tn5432-mediated macrolide-lincosamide-streptogramin B(MLSB)resistance in cutaneous propionibacteria from six European cities[J].J Antimicrob Chemother,2002,49(1):165-168.
[7]Tauch A,Kassing F,Kalinowski J,et al.The corynebacterium xerosis composite transposon Tn5432 consists of two identical insertion sequences,designated IS1249,flanking the erythromycin resistance gene ermCX[J].Plasmid,1995,34(2):119-131.
[8]Nakase K,Nakaminami H,Noguchi N,et al.First report of high levels of clindamycin-resistantPropionibacterium acnescarrying erm(X)in Japanese patients with acne vulgaris[J].J Dermatol,2012,39(9):794-796.
[9]El-Mahdy TS,Abdalla S,El-Domany R,et al.Detection of a new erm (X)-mediated antibiotic resistance inEgyptian cutaneouspropionibacteria[J].Anaerobe,2010,16(4):376-379.
[10]Tauch A,Kassing F,Kalinowski J,et al.The erythromycin resistance gene of theCorynebacterium xerosisR-plasmid pTP10 also carrying chloramphenicol,kanamycin,and tetracycline resistancesiscapable oftransposition inCorynebacterium glutamicum[J].Plasmid,1995,33(3):168-179.
[11]Toyne H,Webber C,Collignon P,et al.Propionibacterium acnes(acnes)resistance and antibiotic use in patients attending Australian general practice[J].Australas J Dermatol,2012,53(2):106-111.
2013-09-13)
(本文编辑:尚淑贤 吴晓初)
Erythromycin resistance in propionibacteria isolated from patients with acne in Wuhan city is associated with mutations in 23S rRNA gene and presence of erm genes
Zeng Xianyu*,Jiang Min,Fu Chen,Dong Bilin,Wu Zhuoxuan,Wang Weizhen.*Department of Dermatology,Wuhan First Hospital,Wuhan 430022,China
Wang Weizhen,Email:weizhenwang2007@aliyun.com
ObjectiveTo determine whether erythromycin-resistant propionibacteria isolated from patients with acne in Wuhan city harbor 23S rRNA gene mutations as well as the transposon Tn5432 carrying ermX genes.MethodsTwenty-ninePropionibacteriumstrains isolated from outpatients with acne in Wuhan city were included in this study.The E-test method was used to determine the susceptibility of these strains to erythromycin and clindamycin.PCR was performed to amplify the 23S rRNA,ermX,ermX(cj),IS1249a and IS1249b genes from resistant strains followed by DNA sequencing and nucleotide alignment.ResultsAmong the 29Propionibacteriumstrains,19 were identified asP.acnesand 10 asP.avidum.All of thesePropionibacteriumstrains were resistant to erythromycin(MIC > 256 μg/ml)and clindamycin(MIC > 256 μg/ml),except for 3P.acnesstrains sensitive to clindamycin.SevenP.acnesstrains resistant to both antibiotics exhibited an A→G transition at a position cognate withEscherichia coli23S rRNA 2058.An A→G transition at a position cognate withE.coli23S rRNA 2059 was identified in one clindamycin-resitant and three clindamycin-sensitiveP.acnesisolates.The ermX gene was found in the remaining 8P.acnesisolates and 2P.avidumisolates,with the sequence 100%identical to the reference sequence of the ermX gene ofP.acnesin Genbank.Meanwhile,the ermX(cj)gene was successfully amplified from the other 8P.avidumisolates,which showed 99%sequence homology with the ermX(cj)gene ofCorynebacterium jeikeium,but 94%homology with the ermX gene ofP.acnesin Genbank.Both IS1249a and IS1249b genes were amplified in the 10 ermX gene-positivePropionibacteriumstrains,but not in the other ermX gene-negative strains.ConclusionsThe erythromycin resistance inPropionibacteriumisolates from Wuhan city may be associated with the A→G transition at theE.coliequivalent bases 2058 and 2059 of the 23S rRNA gene,as well as the presence of the erm X(transferred through the transposon Tn5432)and ermX(cj)genes.
Propionibacterium;Erythromycin;Clindamycin;23S rRNA;Genes,erm
10.3760/cma.j.issn.0412-4030.2014.06.003
王玮蓁,Email:weizhenwang2007@aliyun.com

