临床分离肺炎克雷伯菌Ⅰ类整合子的结构与功能
张跃进,常清利,汪倩,卢俊婉,2,王欢,李佩珍,应俊,包其郁,胡云良
1.温州医科大学检验医学与生命科学学院,温州 325000;2.丽水学院医学院,丽水 323000;3.温州医科大学护理学院,温州 325000
肺炎克雷伯菌(Klebsiella pneumoniae)是仅次于大肠埃希菌(Escherichia coli)的重要临床条件致病菌,主要引起医院内感染,能导致尿路感染、软组织感染及败血症等多种疾病,并被认为是医源性感染的一个指针[1]。世界范围内都有发现由多重耐药肺炎克雷伯菌所引起的医院内感染爆发,还有研究认为克罗恩氏病(Crohn's disease)及强直性脊柱炎也与克雷伯菌感染有关,肺炎克雷伯菌在新生儿病房是尤其危险的病源菌[2]。随着多重耐药菌株的增加,临床用药变得越来越复杂,肺炎克雷伯菌感染性疾病的致死率愈来愈高[3],极大地危害公共健康及安全。
整合子是一种可移动的DNA元件,含有位点特异重组系统和基因盒,能捕获外源基因(尤其是耐药性相关基因)并进行水平转移,导致耐药性播散。整合子在临床分离的革兰阴性耐药菌株中的检出率可高于50%[4],是革兰阴性菌耐药基因捕获和播散的主要机制。因此,对细菌耐药性相关整合子的监测,在分子水平研究细菌耐药性的产生和传播显得十分必要。本研究从临床多重耐药菌株着手,采用基因克隆等技术,探索整合子介导肺炎克雷伯菌耐药性的形成和播散的分子机制,为临床合理使用抗菌药物和预防耐药性播散提供理论依据。
1 材料和方法
1.1 材料
1.1.1 菌株来源
收集2002年~2007年间从温州医科大学附属第一医院临床送检的体液(如血液、腹水等)、分泌物(如痰液、脓液等),分离得到179株随机留取的肺炎克雷伯菌。所有菌株都经过生物梅里埃公司VITEK-32全自动微生物分析鉴定系统鉴定。其中分离自2006年的肺炎克雷伯菌454(携带dhfr17-orfF-aadA2基因盒)用于基因盒携带耐药性基因活性的研究。
1.2 方法
1.2.1 药物敏感性试验
采用纸片扩散法(K-B法,含药纸片购自中国药品生物制品检定所)检测肺炎克雷伯菌对氨苄西林(AMP)、哌拉西林(PIP)、头孢他啶(CAZ)、头孢噻肟(CTX)等14种抗菌药物的敏感性,参照CLSI2008标准判读结果,质控菌株为铜绿假单胞菌ATCC27853和大肠埃希菌ATCC25922。
采用平板稀释法测定细菌的最低抑菌浓度(MIC)[5]:将不同药物按要求用无菌水等溶剂稀释后,用冷却至55℃~60℃的M-H培养基(美国DIFCO公司)配制系列倍比浓度的含药平板;将待测菌用无菌生理盐水配制成0.5麦氏单位的菌悬液,取2μL点种于含抗菌药物平板,以不含药的M-H平板为对照,ATCC25922为质控菌株。接种顺序为先接种不含药对照平板,而后依次从低药物浓度到高药物浓度平板,待菌液被琼脂吸收后,把M-H平板倒置放入35℃±2℃孵箱孵育20~24 h,观察结果。采用CLSI2011药敏试验操作标准判定结果,试验平板上无细菌生长的药物浓度为MIC的终点。
1.2.2 PCR扩增整合酶基因及整合子可变区
按照AxyPrep试剂盒提取基因组DNA,用primer 5设计引物(表1)。50μL PCR 反应体系:10×Buffer 5 μL,Mg2+(25 mmol/L)3 μL,dNTP(2.5 mmol/L)3μL,上、下游引物(5μmol/L)各 1μL,模板3μL,TaqDNA聚合酶(5 U/μL,购自 TaKaRa公司)0.2μL,用ddH2O 补足至50μL。扩增整合子循环参数:95℃变性5 min;95℃ 50 s,55℃ 40 s,72℃3 min,35个循环;72℃再延伸10 min。PCR产物经1%琼脂糖凝胶电泳、EB染色后用凝胶成像系统(SynGene公司)拍照分析。
1.2.3 测序
采用Perkin-Elmer公司的ABI PRISM Model 3730型全自动测序仪进行PCR产物、克隆基因等的测序(由上海桑尼生物科技有限公司完成)。
1.2.4 序列的注释
利用ORPHEUS和glimmer程序进行开放阅读框(Open Reading Frames,ORFs)预测。RBSFINDER(www.tigr.org/software)用来确定ORF起始密码子的位置。对于预测的大于30个氨基酸的ORFs的功能注释分别用Blastp搜索非冗余蛋白质数据库nr库、直系同源簇数据库COG和NCBI的CDD(Conserved Domain Database)。注释过程中如发现有移码(shift frame)或者提前出现终止密码子的则通过人工校正或者重新测序来确认。
1.2.5 整合子携带基因的功能分析
根据注释结果,选择含有已知功能基因和未知功能ORF(orf F)的整合子进行克隆表达。用在线软件primer5.0设计相应的引物(表1),将PCR产物连接至pMD18-T载体,PCR和双酶切鉴定后送测序。测序正确的片段用BamHⅠ和XhoⅡ双酶切,纯化后克隆到表达载体pET28a,转化到宿主菌E.coliBL21,在LB平板(含Km)上筛选阳性克隆,具体操作参考分子克隆实验指南[6]进行。提取质粒双酶切验证后,阳性重组子用IPTG进行诱导表达。以E.coliBL21-pET28a为对照,将经IPTG诱导后的重组菌液及对照菌液适当稀释后,进行平板稀释法测定对复方新诺明(含甲氧苄氨嘧啶)、链霉素等药物的最低抑菌浓度(MIC)。
1.2.6 统计学处理
用SPSS 17.0统计软件,Fisher精确概率法,进行Ⅰ类整合子阳性菌株和阴性菌株对14种药物及不同年份等的耐药率进行比较,以P<0.01表示有显著差异。
2 结果与分析
2.1 药敏试验结果
179株不同来源的临床分离肺炎克雷伯菌对14种抗菌药物的敏感性结果见表2和表3。所有菌株均具有耐药性,其中118株(65.9%)表现出对至少两种以上的抗菌药物耐药,有79株(44.1%)对4种以上抗菌药物耐药,主要为β-内酰胺类、氨基糖苷类和喹诺酮类抗菌药物。对氨苄西林的耐药率最高(71.2%),对泰能的耐药率较低(2.4%,表2)。2002~2007年间的回顾性资料分析显示,在2002~2006年间,对于哌拉西林、头孢他啶、庆大霉素、复方新诺明的耐药率升高,但是到2007年略有下降;相反,对妥布霉素、左旋氧氟沙星等耐药性在2007年却有所升高,这可能与近年这些抗菌药物使用量的迅速增高有关,有资料显示,左旋氧氟沙星是喹诺酮类中使用量最大的药物[7]。另外,抗菌药物在农业、畜牧业、水产养殖业中的滥用也可能是细菌耐药性迅速增长的重要原因。

表1 引物序列

表2 179株肺炎克雷伯菌耐药性与Ⅰ类整合子相关性分析
2.2 整合子检测结果及其与耐药性的相关性分析
在179株肺炎克雷伯菌中,共有65株菌检出单条或者双条Ⅰ类整合子基因盒阳性条带(阳性率为36.3%),阳性条带的大小分别约为0.75 kb、1.0 kb、1.5 kb、1.7 kb、2.2 kb、3.0 kb,绝大多数(46/65,70.7%)为1.7 kb条带,8株菌(12.3%)检出2条带,有4种不同条带大小的组合(0.75/1.0 kb、0.75/1.7 kb、1.0/1.7 kb和1.5/2.2 kb)。不同标本来源病原菌的整合子阳性率及对各种药物的耐药率没有显著差异(P>0.01,表3)。对不同年份分离的肺炎克雷伯菌整合子阳性率的分析发现,2002~2005年间整合子阳性率呈上升趋势,2005年最高,2006年和2007年则逐年下降(P<0.01,表4)。整合子阳性组与阴性组的耐药率除5种药物(包括氨基糖苷类的丁胺卡那霉素和庆大霉素、喹诺酮类的环丙沙星和左旋氧氟沙星及复方新诺明)的耐药性存在显著性差异外,其余9种药物差异不显著(表2)。
2.3 基因盒的结构分析
整合子全可变区测序结果显示,整合子携带的耐药性基因主要有二氢叶酸还原酶基因(dfr A1、dfr A5、dfr A12、dfr A14、dfr A15、dfr A17)、氨基糖苷腺苷酰基转移酶基因(aadA、aadA2、aac(6’)、aadA5)、氨基糖苷腺核苷转移酶基因(ant(3’)-I和atn(3’)-Ih)和氯霉素乙酰基转移酶基因(cmlA6),编码细菌对甲氧苄啶类、氨基糖苷类和氯霉素等抗菌药物的耐药性,个别整合子还含有功能未知的orfF,整合子可变区的基因盒包含1~3个耐药性基因(图1,表5)。值得一提的是,在本批肺炎克雷伯菌中,携带2个耐药性基因dfr A17-aadA5基因盒的整合子最为多见,占所有整合子的一半(37/73),该基因组合编码对甲氧苄啶、链霉素和壮观霉素的耐药性。
2.4 整合子携带耐药性基因的功能验证
对一株携带dhfr17-orfF-aadA2基因的整合子基因盒进行了分段克隆和耐药性功能测定。首先将这个基因盒的分段 PCR产物(分别包含d hfr 17、dhfr17-orfF、dhfr17-orfF-aad A2基因)各自克隆入p UC18-T载体,插入片段经测序验证后再克隆入pET28a原核表达载体,得到含有目的基因的重组表达载体pET28a-dhfr17、pET28a-dhfr17-orfF和pET28a-dhfr17-orfF-aadA2;然后将重组表达载体转化至E.coliBL21,得到转化子并进行耐药基因盒相关的耐药基因的药物敏感性测定。MIC实验分析发现,阳性重组菌株与受体菌E.coliBL21相比较,3个重组子对复方新诺明(含甲氧苄氨嘧啶)的抗性均由8µg/mL上升到256µg/mL;重组子pET28a-dhfr17-orfF与重组子pET28a-dhfr17对链霉素的抗性无明显变化(和受体菌E.coliBL2一样均为8µg/mL),而pET28a-dhfr17-orfF-aadA2对链霉素的 MIC值为256 µg/mL(表 6)。

表3 179株不同来源肺炎克雷伯菌整合子阳性率(%)及耐药率(%)
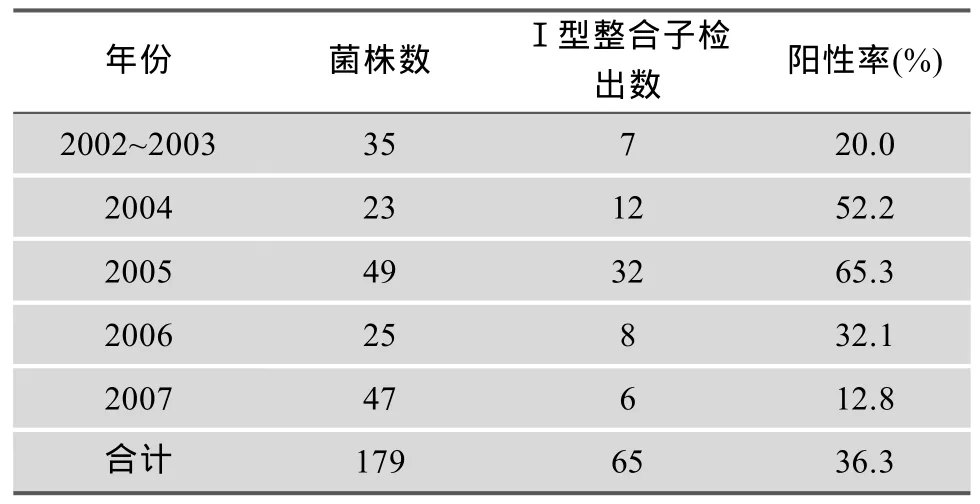
表4 不同年份肺炎克雷伯氏杆菌Ⅰ型可移动整合子的检出率
2.5 一株肺炎克雷伯菌耐药性质粒携带的整合子结构分析
对肺炎克雷伯菌KF3大质粒pKF3-140(NC_013951)的序列分析发现,该质粒共携带8个耐药性基因,其中2个耐药性基因(二氢叶酸还原酶基因dfr和链霉素腺苷酰转移酶基因aadA)同处于一整合子可变区内(图2)。pKF3-140为可接合转移质粒[8],因此,整合子携带的耐药性基因可随质粒的水平转移而导致病原菌的耐药性播散。
3 讨论
整合子是近年来细菌耐药性形成机制研究的热点。整合子是一种可携带多种功能基因可移动的DNA元件,根据整合子5′整合酶基因序列的差异,可将它们分为9类,其中I类整合子在病原菌中最为常见[9]。在整合酶的作用下,整合子可捕获耐药性基因(如耐氨基糖苷类、氯霉素和甲氧苄胺嘧啶等抗菌药物基因),并通过质粒和转座子等可移动DNA元件在同种和不同种属细菌之间进行转移,引起耐药性播散[10]。

图1Ⅰ类整合子15种基因盒的结构示意图
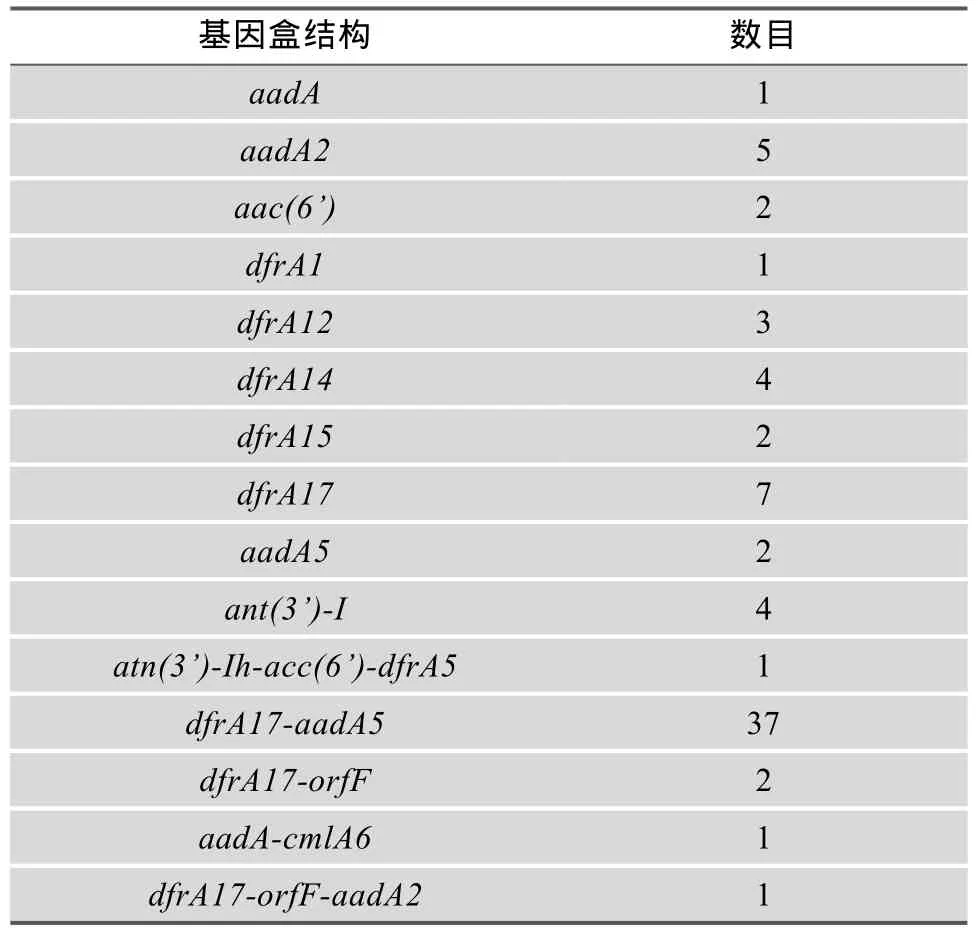
表5Ⅰ类整合子15种不同结构基因盒的检出率
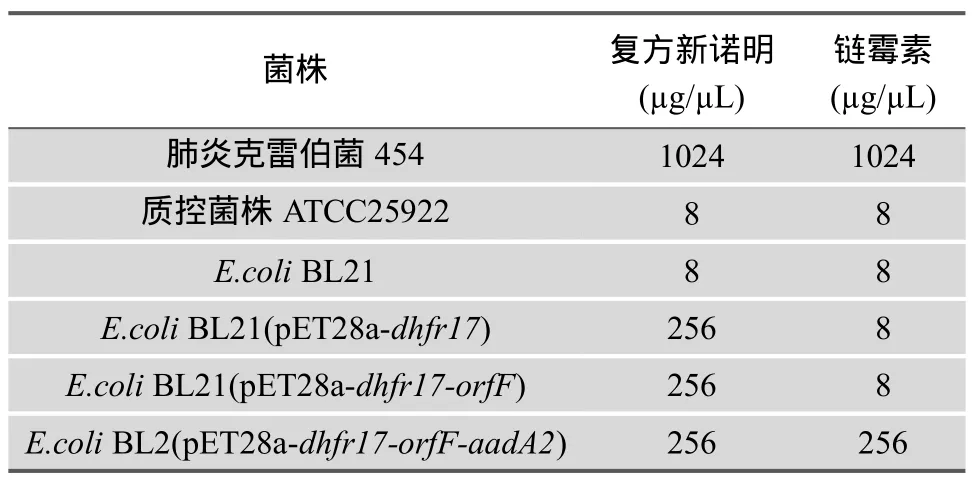
表6 重组菌株MIC测定结果
病原菌的整合子阳性率在不同菌群的阳性率不同[11~13],也有报道显示,虽然在某些病原菌中整合酶基因的检出率可达57%,但是整合子基因盒的检出率只有31.8%[14]。本文在179株临床分离的肺炎克雷伯菌中共有65株菌检出单条或者双条基因盒阳性条带,阳性率为36.3%。造成各地区整合子检出率不同的原因可能与整合子在不同地区的播散速度差异有关;另外,由于检测整合子的引物是根据被扩增的可变区两侧的保守序列而设计的,如果设计引物区域发生了DNA序列的突变或缺失,造成整合子的漏检。本文研究结果还显示,温州地区不同年份临床分离肺炎克雷伯菌整合子阳性率有显著差异,以2005年最高(达65.3%),之后(2006年和2007年)则逐年下降,造成这种现象的原因有待进一步分析。
本文整合子的基因盒共有15种耐药基因构成形式,其中dfr A17-aadA5是本批肺炎克雷伯菌中最为流行的一种基因盒。最早被鉴定的含有dfr A17-aadA5基因盒的整合子定位于一株分离自澳大利亚的大肠埃希菌染色体上[15],后来在台湾发现了编码dfr A17-aadA5同源基因盒整合子的可转移质粒[16]。本文的dfr A17-aadA5基因盒经比较分析发现与表皮葡萄球菌(注册号:AB291061)及肺炎克雷伯菌(注册号:AY994155)的dfrA17-aadA5有较高的同源性(达99.8%)。dfr A1、dfr A12、dfr A14和dfr A15等为编码二氢叶酸还原酶基因,介导对甲氧磺胺嘧啶类的耐药性,这些基因盒曾在沙门氏菌、大肠埃希菌等中检出[17]。cmlA6编码氯霉素外排泵蛋白,介导对氯霉素的耐药性,常与aadA一起出现[18],本文的cmlA6基因也与aadA组合,构成基因盒,该基因盒与大肠埃希菌DQ836058的I类整合子携带的同类基因盒的同源性达99%。aadA、aadA2、ant(3’)-I等均在其他病原菌如大肠埃希菌AJ419169、鲍曼不动杆菌EU977568I中检出,同源性达100%。从以上结果可以看出,同一耐药基因盒可以出现在不同的革兰阴性菌种中,同一菌种中也可以存在多种耐药基因盒。
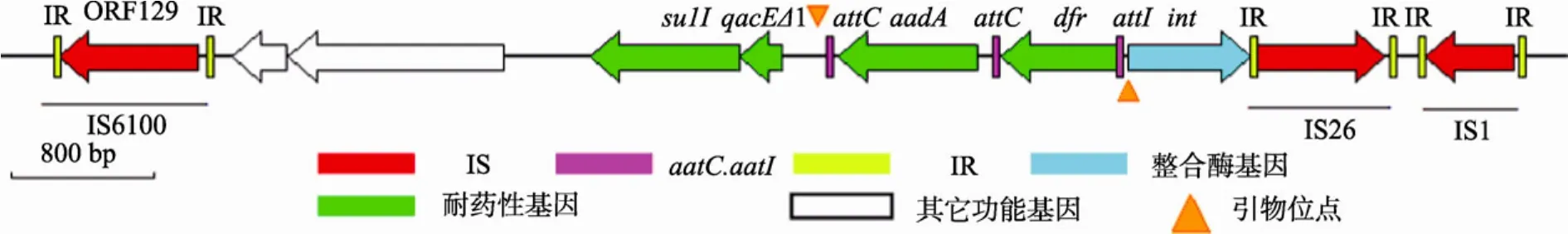
图2 肺炎克雷伯菌KF3质粒p KF3-140的整合子区域结构图
本文I类整合子的基因盒中,氨基糖苷类和甲氧苄啶类的耐药基因盒在数量上占优势,与许多研究报道的结果相一致[19~21],出现此种现象的原因,可能是目前链霉素和壮观霉素在农业、水产养殖业饲养中被广泛使用,人类通过接触动物和食物链而获得携带耐链霉素、壮观霉素抗性基因细菌[22],或是二者抗性基因与其他临床常用药物的抗性基因一起,共同存在于耐药性质粒而得以长期存在[21]。甲氧苄啶耐药基因盒整合子的高检出率,也可能是甲氧苄啶在泌尿道感染的治疗中,作为一线药物被使用[23],造成了泌尿道感染相关耐药性菌株的流行。整合子阳性菌株与阴性菌株对多种抗菌药物的耐药率有显著差异,主要集中在氨基糖苷类、喹诺酮类和磺胺类的耐药,且与整合子基因盒的编码基因类型(如已检测到的氨基糖苷类和磺胺类耐药基因)较为一致,说明整合子参与了肺炎克雷伯菌的耐药性的形成。
另外,我们对整合子携带的已知功能耐药性基因(dhfr17和aadA2)和未知功能的orfF进行了克隆表达,旨在研究已知功能耐药性基因的活性以及orfF与耐药性的相关性。根据3种含不同基因组合的重组子对复方新诺明(含甲氧苄氨嘧啶)和链霉素的MIC值可以看出,含有orfF的重组子与不含orfF的重组子在药物的抗性上并无差异,由此推测orfF与dhfr17或aadA2对抗菌药物的抗性可能没有直接的关系,但其是否具有其他功能仍有待进一步分析鉴定。
整合子是耐药基因播散的重要机制,耐药菌可通过整合子将耐药性相关基因传递给其他细菌,同时还能接受其他细菌的耐药基因[9]。整合子本身是相对比较稳定的结构,但位于质粒上的整合子可随质粒的转移而转移,导致其所携带的耐药性基因在细菌间的相互传播,大大加快了耐药性传播的速度,整合子的水平转移是细菌获得耐药基因的重要来源。通过分析一株多重耐药肺炎克雷伯菌KF3所携带的3个质粒DNA序列,发现其中的可接合转移质粒pKF3-140携带一个I类整合子,该整合子可变区编码了2个耐药性基因(二氢叶酸还原酶基因和链霉素腺苷酰转移酶基因)[8,24],证实了肺炎克雷伯菌的I类整合子可由质粒携带,并随质粒的水平转移而导致耐药性播散。
抗生素在临床、农业、水产养殖业的大量应用,是细菌耐药性形成的最重要原因。对整合子的结构及其携带的耐药性基因的研究,有助于揭示细菌耐药性形成的分子基础,可为制定抗菌药物使用的相关政策、合理使用抗菌药物和控制细菌耐药性发展提供帮助。
[1]Ambe JP,Gasi IS,Mava Y.Review of neonatal infections in University of Maiduguri Teaching Hospital:common bacterial pathogens seen.Niger J Clin Pract,2007,10(4):290–293.
[2]Jurczak A,Kordek A,Grochans E,Giedrys-Kalemba S.Clinical and microbiological characteristics of hospital infections in the neonatal intensive care unit.Adv Med Sci,2007,52(S1):30–33.
[3]Gibreel A,Lawley TD,Tracz DM,Taylor DE.Antibiotic Resistance Plasmids.In:Phillips G,Funnell BE.Plasmid biology.Washington DC:ASM Press,2004:473–492.
[4]郭庆兰.耐药性播散机制进展—整合子-基因匣子系统.国外医学(分子生物学分册),2002,24(4):202–206.
[5]National Committee for Clinical Laboratory Standards.Reference Method for Broth Dilution Antifungal Susceptibility Testing of Yeasts:Approved Standard.NCCLSdocumentM27-A2,Wayne,PA,USA,2002.
[6]Sambrook(萨姆布鲁克)J,Russell(拉塞尔)DW著(黄培堂译).分子克隆实验指南(第3版).北京:科学出版社,2002.
[7]Yun NR,Li SH,Liu XS.Variation of Antibiotic Resistance ofK.pneumoniaefrom 2005 to 2009.Evaluation and Analysis of Drug-Use in Hospitals of China,2010,10(10):898–899.
[8]Bai J,Liu Q,Yang Y,Wang J,Yang Y,Li J,Li P,Li X,Xi Y,Ying J,Ren P,Yang L,Ni L,Wu J,Bao Q,Zhou T.Insights into the evolution of gene organization and multidrug resistance fromKlebsiella pneumonia eplasmid pKF3–140.Gene,2013,1119(13):123–126.
[9]Correia M,Boavida F,Grosso F,Salgado MJ,Lito LM,Cristino JM,Mendo S,Duarte A.Molecular characterization of a new class 3 integron inKlebsiella pneumoniae.Antimicrob Agents Chemother,2003,47(9):2838–2843.
[10]Mazel D.Integrons:agents of bacterial evolution.Nat Rev Microbiol,2006,4(8):608–620.
[11]Chang CY,Chang LL,Chang YH,Lee TM,Chang SF.Characterization of drug resistance gene cassettes associated with class 1 integrons in clinical isolates ofEscherichia colifrom Taiwan.J Med Microbiol,2000,49(12):1097–1102.
[12]Dalsgaard A,Forslund A,Tam NV,Vinh DX,Cam PD.Cholera in vietnam:Changes in genotypes and emergence of class 1 integrons containing aminoglycoside resistance gene cassettes inVibrio cholerae1 strains isolated from 1979 to 1996.JClin Microbiol,1999,37(3):734–741.
[13]Ploy MC,Denis F,Courvalin P,Lambert T.MoIecular characterization of integrons inAcinetobacter baumannii:description of a hybrid class 2 integron.Antimicrob Agents Chemother,2000,44(10):2684–2688.
[14]Wu KH,Wang FP,Sun JJ,Wang Q,Chen Q,Yu SY,Rui YY.Class 1 integron gene cassettes in multidrug-resistant Gram-negative bacteria in southern China.IntJ Antimicrob Agents,2012,40(3):264–267.
[15]White PA,Mciver CJ,Deng YM,Rawlinson WD.Characterization of two new gene cassettes,aadA5anddfrA17.FEMSMicrobiol Lett,2000,182(2):265–269.
[16]Chang CY,Chang LL,Chang YH,Lee TM,Li YH,Chang HF.Two new gene cassettes,dfr17(for trimethoprim resistance)and aadA4(for spectinomycin/streptomycin resistance),inserted in anEscherichia coliclass 1 integron.JAntimicrob Chermother,2000,46(1):87–89.
[17]McIver CJ,White PA,Jones LA,Karagiannis T,Harkness J,Marriott D,Rawlinson WD.Epidemic strains ofShigella sonneibiotype g carrying integrons.J Clin Microbiol,2002,40(4):1538–1540.
[18]Shaheen BW,Oyarzabal OA,Boothe DM.The role of class 1 and 2 integrons in mediating antimicrobial resistance among canine and feline clinicalE.coliisolates from the US.Veterinary Microbiology,2010,144(8):363–370.
[19]Guerra B,Junker E,Sehroeter A.Phenotypic and genotypic characterization of antimicrobials resistance in GermanEseheriehia coliisolates from cattle,swine and poultry.JAntimicrob Chemother,2003,2(3):489–492.
[20]White PA,Mclver CJ,Rawlinson WD.Integrons and gene cassettes in theEnterobacteriaceae.Antimicrob Agents Chemother,2001,45(9):2658–2661.
[21]Ribera A,Vila J,Fernández-Cuenca F,Martínez-Martínez L,Pascual A,Beceiro A,Bou G,Cisneros JM,Pachón J,Rodríguez-Baño J.Type 1 integrons in epidemiologically unrelatedAcinetobaeter banmanniiisolates collected at Spanish hospitals.Antimicrob Agents Chemother,2004,48(l):364–365.
[22]Leverstein-van Hall MA,Box AT,Blok HE,Paauw A,Fluit AC,Verhoef J.Evidence of extensive interspecies transfer of integron-mediated antimicrobial resistance genes among multidrug-resistant Enterobacteriaceae in a clinical setting.J Infect Dis,2002,186(1):49–56.
[23]Maugire AJ,Brown DF,Gray JJ,Desselberger U.Rapid screening technique for class 1 Integrons inEnterobacteriaceaeand nonfermenting gram-negative bacteria and its use in molecular epidemiology.Antimicrob Agents Chemother,2001,45(4):1022–1029.
[24]Yi HG,Xi YL,Liu J,Wang JR,Wu JY,Xu T,Chen W,Chen BB,Lin ML,Wang H,Zhou MM,Li JS,Xu ZY,Jin SG,Bao QY.Sequence analysis of pKF3–70 inKlebsiella pneumoniae:probable origin from R100-like plasmid ofEscherichia coli.PLoSONE,2010,5(1):e8601.

