小麦tae-MIR156前体基因的克隆及其靶基因TaSPL17多态性分析
刘霞, 张斌, 毛新国, 李昂, 孙美荣, 景蕊莲
1. 山西大学生物工程学院, 太原 030006;
2. 中国农业科学院作物科学研究所, 农作物基因资源与基因改良国家重大科学工程, 北京 100081
小麦tae-MIR156前体基因的克隆及其靶基因TaSPL17多态性分析
刘霞1,2, 张斌2, 毛新国2, 李昂2, 孙美荣1, 景蕊莲2
1. 山西大学生物工程学院, 太原 030006;
2. 中国农业科学院作物科学研究所, 农作物基因资源与基因改良国家重大科学工程, 北京 100081
Squamosa-promoter binding protein (SBP)-box基因是植物特有的一类转录因子, 广泛参与植物生长发育,其部分成员受miR156调控。文章克隆了小麦(Triticum aestivum) tae-MIR156前体基因, 转录后能够形成茎环结构。小麦10个SBP-box基因中, 仅TaSPL3和TaSPL17在编码区存在tae-miR156识别位点。SPL17在普通小麦的 A基因组供体种乌拉尔图小麦(Triticum urartu, AA) UR209和 B基因组供体种拟斯卑尔脱山羊草(Aegilops speltoides, BB) Y2001中均为多拷贝(SPL17-A1、SPL17-A2和SPL17-A3; SPL17-B1、SPL17-B2和SPL17-B3), 在D基因组供体种粗山羊草(Aegilops tauschii, DD) Ae38中仅检测到一种序列(SPL17-D); SPL17-A2与SPL17-B2, SPL17-A3与SPL17-B3、SPL17-D两两之间序列的一致性程度均大于99%, 且与普通小麦(中国春、衡观35和双丰收)的 TaSPL17序列具有较高的一致性, 提示它们可能来源于共同的祖先基因, 并且在进化过程中高度保守。靶基因 TaSPL17中的 tae-miR156识别位点非常保守, 在根据单株穗数和基因型多样性挑选的 SubP1和SubP2群体中均未检测到tae-miR156识别位点存在变异碱基。
miR156; SBP-box基因; 多态性分析; 普通小麦
转录因子可以通过 DNA结合域(DNA-binding domains)激活或者抑制基因的表达, 从而调控植物进行正常的生长、分化和代谢[1]。Squamosa-promoter binding protein (SBP)-box基因(SBP-box)也称作SPL (SQUAMOSA promoter-binding protein-like)基因,是植物特有的一类转录因子, 广泛参与植物的生长发育[2], 其部分成员受 miR156的调控。MicroRNA (miRNA)在植物的生长发育过程中具有重要调节作用, 包括开花时序、新陈代谢以及对多种胁迫的应激反应等[3]。miR156是植物中一个高度保守的miRNA基因家族, 在苔藓(Bryophyta)及高等植物中都存在, 其靶向的 SBP-box基因通过调控 miR172,进而调节植物幼年期和成年期之间的转变[4]。Wu等[5]揭示了 miR156通过其靶基因 SPL9/SPL10调控miR172的表达。miR156在苗期表达量最高, 随着植物的生长表达量降低, 其靶向的 SBP-box基因含量则逐渐上升。当靶向 SBP-box基因的表达量达到一定程度时, 则激活下游基因的表达, 从而诱导植物开花[4,5]。
SBP-box基因最早在金鱼草(Antirrhinum majus)中发现(SBP1和SBP2), 能够结合花器官分生组织决定基因SQUAMOSA的启动子[6]。根据对基因组序列进行分析, SBP-box基因在莱茵衣藻(Chlamydomonas reinhardtii)中有 19~22个成员(CrSPL), 小立碗藓(Physcomitrella patens)中有14个成员(PpSBP), 江南卷柏(Selaginella moellendorffii)中有 13个成员(SmSBP), 拟南芥(Arabidopsis thaliana)中有17个成员(AtSPL), 水稻(Oryza sativa)中有19个成员(OsSPL),玉米(Zea mays)中有31个成员(ZmSBP)[1,7,8]。最近研究表明, SBP-box基因除了促进植物不同生育相之间的转变, 在调控花和果实发育方面也具有重要作用,进而影响产量。在水稻中, OsSPL16控制籽粒大小和形状, 进而影响稻米的产量和品质。OsSPL16中存在 miR156的识别位点, 对该位点的多态性分析结果表明:来自伊朗水稻品种Amol3的OsSPL16基因中的miR156识别位点处存在2 bp的插入缺失, 将这种新的等位变异位点导入高产水稻品种HJX74中,可以显著提高稻米品质, 同时产量保持不变; 将该基因导入水稻品种 Basmati中, 在保证优质的基础上, 可以使产量提高14%左右[9]。OsSPL14能够调控水稻的株型进而增加产量, 同时 OsSPL14也受miR156的调控。例如在粳稻品系Shaoniejing和Ri22中发现该识别位点的一个点突变扰乱了 miR156对OsSPL14的调控, 使水稻分蘖减少, 穗粒数和千粒重增加, 同时茎秆变得粗壮, 抗倒伏能力增强, 进而提高产量; 将该变异位点导入粳稻品种秀水11中,可使产量提高约10%[10]。
迄今为止, 关于 tae-miR156与小麦 SPL基因(Triticum aestivum SQUAMOSA promoter-binding protein-like, TaSPL)家族成员的研究报道较少, 两者的靶向关系还不清楚, 尚未见到靶基因中tae-miR156识别位点(miRNA responsive element, MRE)处的多态性研究报道。本研究以本实验室前期克隆的 10个小麦 TaSPL基因为基础, 克隆了tae-MIR156前体基因(pre-miRNA), 研究其与TaSPL基因的靶向关系, 明确具有tae-miR156识别位点的TaSPL基因。同时对小麦前体tae-MIR156和TaSPL17进行基因序列结构分析, 根据单株穗数和基因型的多态性, 从300份普通小麦中挑选出两个小群体(31份和30份材料)检测tae-miR156识别位点处的多态性, 以期为深入研究 TaSPL17的功能、揭示tae-miR156与TaSPL17对小麦生长发育的影响、筛选优异等位基因提供依据。
1 材料和方法
1.1 植物材料
实验材料由中国农业科学院作物科学研究所提供。普通小麦品种偃展4110用于基因克隆。3份普通小麦(AABBDD, 2n=6x=42)中国春、衡观35和双丰收用于检测基因全长序列多态性; 3份普通小麦的二倍体野生近缘种:乌拉尔图小麦 UR209(Triticum urartu, AA, 2n=2x=14)、拟斯卑尔脱山羊草Y2001(Aegilops speltoides, SS, 2n=2x=14)和粗山羊草Ae38(Aegilops tauschii, DD, 2n=2x=14)用于判断普通小麦中目的基因序列的基因组来源。
一套由 300份普通小麦组成的自然群体于2010~2011年同时种植在中国农业科学院作物科学研究所昌平和顺义两个实验基地[11]。在越冬前、拔节期、开花期和灌浆期各灌溉一次, 每次灌水量为75 mm。每份材料种4行小区, 行长2 m, 行距0.3 m,每行点播40粒种子。开花期以后调查单株穗数, 即有效分蘖。在每个小区的中间行中间区段调查 6株的单株穗数, 取平均值。本研究从该群体中挑选两个小群体, 命名为SubP1和SubP2, 通过测序来检测靶基因中tae-miR156靶位点的多态性。SubP1是根据单株穗数调查结果挑选出的31份材料, 其中多穗材料16份, 少穗材料15份。SubP2是根据126个SSR标记位点的多态性信息挑选出的 30份多态性高的材料[11]。SubP1和 SubP2材料的基本情况见表1。
1.2 方法
1.2.1 DNA、RNA的提取及RT-PCR
利用CTAB法提取普通小麦偃展4110叶片中基因组DNA。取偃展4110苗期顶端分生组织、叶片以及拔节期 2 cm 左右的幼穗, 液氮速冻, 采用TRIZOL法提取总RNA, 用DNase I去除DNA, 利用1.0%琼脂糖凝胶电泳检测总RNA的完整性。以总RNA为模板, 用M-MLV Reverse Transcriptase反转录酶(Invitrogen公司)合成cDNA第一链。混合上述取样材料的cDNA, 用于基因克隆。
1.2.2 目的基因的扩增和测序
PCR扩增体系为 15 µL, 其中 TransStart Fast Pfu DNA Polymerase 0.3 U。PCR扩增条件为:96℃5 min; 96℃ 1 min, 52~62℃ 45 s, 72℃ 3 min, 32个循环; 72℃ 10 min; 4℃保存。按照北京百泰克生物技术有限公司 DNA回收试剂盒操作说明回收目标产物。将回收的产物连接到 pEASY-Blunt Simple Cloning Vector, 转化 Trans1-T1 Phage Resistant E. coli 感受态细胞(北京全式金生物技术有限公司),挑取阳性克隆, 根据BigDye Terminator Kit(ABI公司)说明进行测序。
1.2.3 小麦tae-MIR156前体基因与TaSPL17的克隆
根 据 tae-MIR156 前 体 序 列 (miRbase: MI0016450), 参照其所在小麦基因组位点的两侧序列(GenBank登录号:CL902915.1), 分别于前体结构(pre-miRNA)两端外10 bp左右处设计引物MIRF和MIRR。以普通小麦偃展4110的基因组DNA为模板,进行PCR扩增, 长度约为300 bp。在小麦基因组中扩增TaSPL17所用的引物为TaS17F和TaS17R。中间测序引物为 TaSF2、TaSR2和 TaSR3。检测靶基因 TaSPL17中 tae-MIR156识别位点所用引物为miR-TaSF和miR-TaSR。本实验所用引物(表2)均利用 Primer 5.0 设计, 由上海生工生物工程技术服务有限公司合成。
1.2.4 基因结构和序列分析
利用Mfold 和 RNAfold软件预测 tae-MIR156前体基因序列的二级结构[12,13]。通过 psRNATarget进行 tae-miR156靶基因的预测[14], 分析与 10个TaSPL基因[15](TaSPL1、TaSPL3、TaSPL6、TaSPL8、TaSPL15、TaSPL17、TaSPL20-TaSPL23)的靶向关系。利用DNAStar软件包的SeqMan和MegAlign软件进行序列拼接、分析和比对。
1.2.5 TaSPL17基因序列相似性分析
根据普通小麦中TaSPL17的序列从GenBank中搜索到水稻(OsSPL14)、拟南芥(AtSPL9)、垂枝桦
(Betula pendula)、江南卷柏、小立碗藓(SBP13)、莱茵衣藻的SPL基因以及金鱼草SBP1和SBP2基因,登录号依次为GU136674、CAB56591.1、CAD90157.1、XP_002978761.1、ABM67305.1、EDP04027.1、Q38741.1和Q38740.1。将上述搜索到的基因与小麦TaSPL17序列进行相似性分析, 利用SMART软件分析基因的保守结构域; 用 MEGA 4.1软件构建系谱进化树[16,17]。

表1 小麦SubP1和SubP2群体材料名称及其来源

表2 本文所用的引物信息
2 结果与分析
2.1 小麦 tae-MIR156前体基因序列及转录后二级结构预测
以普通小麦偃展 4110的基因组 DNA为模板,扩增 tae-MIR156前体基因(miRbase: MI0016450),对检测到的24个阳性克隆进行测序, 结果表明小麦偃展4110的tae-MIR156前体基因主要有两种序列,分别命名为tae-MIR156-1和tae-MIR156-2, 两种序列的一致性为96.9%。tae-MIR156-1与miRbase数据库中报道的 tae-MIR156核苷酸序列一致性为97.8%; tae-MIR156-2与 tae-MIR156序列一致性为95.5% (图 1)。利用 Mfold 和 RNAfold软件预测tae-MIR156前体基因 3种序列的二级结构, 偃展4110的两种序列经转录后均形成茎环结构(图2)。在tae-MIR156 3种序列的二级结构中, 其茎环结构基本相似, 不过由于三者在中间部位都存在 21 bp的InDel位点, 故 tae-MIR156-1和 tae-MIR156-2比tae-MIR156在中间部位多形成一个由21 bp组成的茎环状突起, 其余结构相似, 3种序列中成熟tae-miR156所在臂的位置基本一致(图2)。
2.2 tae-miR156靶基因预测及TaSPL17基因结构
植物 miRNA与靶基因高度互补, 并引起靶向mRNA的降解, 可以进行靶基因预测[18,19]。在10个小麦 TaSPL家族成员中[15], 仅 TaSPL3和 TaSPL17在编码区存在tae-miR156的靶位点(图3), 表明其受tae-miR156的调控。利用引物TaS17F和TaS17R扩增偃展4110中TaSPL17基因的基因组和cDNA序列,其片段长度分别为4.1 kb和1.2 kb, 该基因结构中包含3个外显子和2个内含子(图4:A、B), tae-miR156的靶位点位于第3个外显子(图4C)。
2.3 TaSPL17基因同源性分析
将TaSPL17的SBP保守结构域在NCBI中进行BLASTP比对, 在氨基酸水平上发现与水稻(OsSPL14)、拟南芥(AtSPL9)、SBP_垂枝桦、石松门(SBP_江南卷柏)、苔藓(SBP13_小立碗藓)和藻类(SBP_莱茵衣藻)的 SPL基因一致性依次为 82.3%、77.2%、65.8%、76.0%、75.9%和 53.8%; 与最早在金鱼草中报道的SBP1和SBP2的一致性为65.8%和67.1% (图5A)。根据上述SPL基因的SBP保守结构域构建系统进化树, 结果表明, TaSPL17与OsSPL14亲缘关系最近, 与SBP_莱茵衣藻中的SPL基因亲缘关系最远(图5B)。另外, SBP结构域中包含两个典型的锌指结构(Cys3His和Cys2HisCys), 其保守位点见图5C, 这一结构在绿色植物中普遍存在。
2.4 TaSPL17蛋白质序列多态性
以小麦偃展4110的cDNA为模板进行PCR扩增和测序, 检测48个阳性单克隆序列。测序结果表明TaSPL17基因主要分为5种序列, 将其蛋白质序列分别命名为YZ4110_cDNA_1、YZ4110_cDNA_2、YZ4110_cDNA_3、YZ4110_cDNA_4和 YZ4110_ cDNA_5(图6)。在SBP结构域和tae-miR156的靶位点处(图6), 5种序列没有差异, 说明结构域和识别位点具有高度保守性。
2.5 普通小麦及其二倍体野生近缘种 SPL17基因组DNA序列多态性

图1 tae-MIR156前体基因3种序列的比对

图2 tae-MIR156前体基因转录后形成的茎环结构A:miRBase中tae-MIR156前体茎环结构; B:偃展4110 tae-MIR156-1转录后的茎环结构; C:偃展4110 tae-MIR156-2转录后的茎环结构。图中黑色方框表示tae-MIR156前体基因序列中成熟tae-miR156的位置。
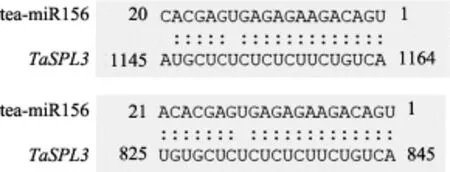
图3 tae-miR156靶基因预测
以3份小麦二倍体野生近缘种及3份普通小麦为材料, 用引物 TaS17扩增 SPL17, 每份材料检测24个单克隆序列。测序结果表明乌拉尔图小麦(UR209)中有3种序列, 命名为SPL17-A1、SPL17-A2和SPL17-A3; 拟斯卑尔脱山羊草(Y2001)中也有到3种序列, 命名为SPL17-B1、SPL17-B2和SPL17-B3;在粗山羊草(Ae38)中仅扩增到 1种序列, 命名为SPL17-D。因此, SPL17在普通小麦的A基因组供体种乌拉尔图小麦(UR209)和 B基因组供体种拟斯卑尔脱山羊草(Y2001)中均为多拷贝, 而在D基因组供体种粗山羊草(Ae38)可能为单拷贝。普通小麦 3个野生近缘种中的 SPL17序列具有高度的一致性, 其中 SPL17-A2与 SPL17-B2的序列一致性为 99.2%, SPL17-A3与SPL17-B3、SPL17-A3与SPL17-D的一致性分别为 99.9%和 99.4%, SPL17-B3与 SPL17-D的序列一致性为99.4%(表3)。

图4 TaSPL17基因cDNA全长及其编码的氨基酸序列A:TaSPL17基因结构图; B:小麦偃展4110 基因组DNA中扩增的TaSPL17产物; C:TaSPL17基因SBP结构域与tae-miR156的靶位点。图C方框中的序列表示TaSPL17基因的SBP结构域; 星号表示TaSPL17基因中的靶位点。

图5 TaSPL17基因与其他物种SBP-box基因的SBP保守结构域序列A:不同物种SBP-box基因的SBP保守结构域序列一致性分析; B:不同物种SBP-box基因的SBP保守结构域系谱进化分析; C:不同物种SBP-box基因的SBP保守结构域序列多重比对。▲表示SBP结构域中两个锌指结构的保守位点, 即:Cys、Cys、Cys、His(Cys3His)和Cys、Cys、His、Cys (Cys2HisCys)。

图6 偃展4110中TaSPL17蛋白质序列比对第一个方框中的序列表示SBP结构域, 第二个方框中的序列表示tae-miR156的靶位点。

表3 小麦二倍体野生近缘种中SPL17基因组DNA序列的一致性分析

图7 普通小麦及其二倍体野生近缘种SPL17的DNA序列系统进化树分析
在普通小麦中国春(CS)和衡观 35(Heng)基因组DNA中均扩增到3种序列, 命名为TaSPL17-CS1、TaSPL17-CS2、TaSPL17-CS3和 TaSPL17-Heng1、TaSPL17-Heng2、TaSPL17-Heng3。在双丰收(Shuang)基因组 DNA中扩增到 2种序列 TaSPL17-Shuang1和 TaSPL17-Shuang2。其中 TaSPL17-CS1、 TaSPL17-CS2、TaSPL17-Heng1、TaSPL17-Heng2、TaSPL17-Shuang1与小麦二倍体野生近缘种的SPL17-A2、SPL17-B2和SPL17-B1聚在同一个进化分枝中(图 7), 而 TaSPL17-CS3、TaSPL17-Heng3、TaSPL17-Shuang2与SPL17-A3、SPL17-B3、SPL17-D聚在一起。SPL17-A1与普通小麦TaSPL17的序列相似性最低。
2.6 靶基因TaSPL17中tae-miR156识别位点的多态性
在本实验室前期克隆的10个小麦TaSPL家族成员中, 仅TaSPL3和TaSPL17受tae-miR156调控。对TaSPL17基因结构及其在普通小麦和二倍体野生近缘种中的序列多态性分析结果表明:TaSPL17在普通小麦的A基因组供体种和B基因组供体种中均为多拷贝, 而在 D基因组供体种中可能为单拷贝,并且其在 3个野生近缘种中存在高度一致的序列,在六倍体普通小麦中也克隆到多种序列。本研究从300份普通小麦中根据单株穗数和基因型的多态性挑选出两个小群体, 即SubP1和SubP2, 重点检测靶基因中 tae-miR156识别位点的多态性, 为筛选优异等位变异提供依据。小群体SubP1的单株穗数如图8所示。利用引物miR-TaS扩增小麦TaSPL17的DNA序列, 扩增产物长度为 700 bp。每份材料至少检测12个单克隆序列。测序结果表明在SubP1的31份材料和 SubP2的 30份材料中均未检测到 TaSPL17中tae-miR156识别位点处存在变异的材料, 这表明TaSPL17中miR156识别位点高度保守。

图8 SubP1群体材料的单株穗数
3 讨 论
miRNA属于小RNA的一种, 由RNA聚合酶II转录生成, 广泛参与植物生长发育过程调控, 可以在不同层次上对靶基因进行调控, 包括转录、转录后和翻译等[20]。miRNA靶基因大多为编码调控蛋白的转录因子, 例如miR156的靶基因为SBP-box基因家族[21]、miR159/319的靶基因为MYB基因家族[22]、miR160的靶基因为ARF基因家族[23]、miR164的靶基因为NAC基因家族[24]、miR172的靶基因为AP2基因家族[25]。研究miRNA所调控的靶基因, 以及两者的相互作用关系对于诠释其功能至关重要。由于植物miRNA与其靶基因的互补程度较高, 也使得预测miRNA的靶基因更加方便可靠[26]。
SBP-box基因家族成员的多样性使其能够调控植物的众多生长发育过程, 比如花的形成和发育[27,28]、叶片形状及表皮性状[21]、对环境信号的应答[29]、育性[30]和果实的成熟和产量[9]等。小麦是世界上最重要的粮食作物之一, 对其 miRNA156及其靶基因的研究具有十分重要的意义。本研究以普通小麦偃展4110为材料, 克隆了小麦tae-MIR156前体基因, 主要分为两种序列, 与 miRbase数据库中报道的序列相似, 经转录后均形成茎环结构, 3种序列中成熟tae-miR156所在臂的位置基本一致(图2)。前人研究表明, 在拟南芥和水稻中各有11个AtSPL和OsSPL家族成员存在miR156识别位点, 大多数位于编码区, 少数位于非编码区(UTR), 如拟南芥中的AtSPL3、AtSPL4和 AtSPL5, 水稻中的 OsSPL4和OsSPL13均位于3′-UTR区域[2,26,31]。然而, 在小麦的 10个 TaSPL基因中[15], 仅检测到两个成员TaSPL3和TaSPL17编码区存在tae-miR156的靶位点(miRNA responsive element, MRE)(图3)。其原因一方面可能是研究中所用的 10个 TaSPL基因[15]并不是小麦中所有的 SBP-box家族成员, 或者这 10个TaSPL基因并未包含全部的5′-UTR和3′-UTR区域,因此需要通过 5′-RACE和 3′-RACE获得全长的UTR区域进一步确定; 另一方面也可能是由于小麦SBP-box基因受tae-miR156调控的模式与拟南芥和水稻中SBP-box基因家族成员不尽相同。
SBP-box基因早在绿藻和陆生植物的祖先分化之前就已经产生, 在其最后一个共同祖先分化之后,陆生植物的SBP-box基因就开始分化[7]。SBP-box靶基因中 miR156的识别位点普遍存在于陆生植物中, miRNA识别位点的保守性也说明了其与靶基因的相互作用对于植物生长发育的重要性。在苔藓和石松门植物中, 仅有少数几个 SBP-box基因受到miR156的调控; 然而被子植物(如拟南芥和水稻)的SBP-box基因家族成员中普遍存在miR156的识别位点[7]。在小麦的10个TaSPL基因中仅检测到两个成员的编码区存在tae-miR156靶位点[15], 这是否暗示小麦SBP-box基因是由古老的SBP-box祖先基因进化而来, 并且在长期的进化过程中, 未获得tae-miR156的识别位点, 有待进一步研究。
植物在长期的进化过程中, 基因或基因组的复制是普遍存在的现象, 这也是基因获得新功能, 进而促使植物进化形成新物种的内在动力[32]。普通小麦是由A、B和D3个染色体组组成的异源六倍体物种, 从原始的二倍体物种进化到现代的六倍体, 普通小麦经历了两次异源多倍化过程。本研究以小麦tae-miR156靶向的 TaSPL17进行多态性分析, 以普通小麦偃展4110的cDNA为模板, 扩增获得5种序列; 以小麦二倍体野生近缘种乌拉尔图小麦(UR209)、拟斯卑尔脱山羊草(Y2001)和粗山羊草(Ae38)基因组DNA进行PCR扩增, 分别获得3、3和1种序列(图6, 表3)。因此, SPL17在普通小麦的A基因组供体种和B基因组供体种中均为多拷贝。并且SPL17-A2与SPL17-B2, SPL17-A3与SPL17-B3, SPL17-A3与SPL17-D和SPL17-B3与SPL17-D之间序列的一致性程度均大于 99%, 据此推测其可能来源于共同的祖先基因, 在进化过程中比较保守, 并且与普通小麦中克隆到的TaSPL17基因序列聚在一起, 属于直系同源。而SPL17-A1与普通小麦中克隆到的TaSPL17基因序列关系最远, 相似性最小(图7)。另外, 本研究根据单株穗数和基因型的多样性共挑选出61份材料, 均未检测到tae-miR156识别位点的变异, 说明六倍体普通小麦 TaSPL17基因tae-miR156的识别位点非常保守。对44个SBP-box基因(包括16个AtSPL、18个OsSPL和10个TaSPL基因)的 SBP保守结构域氨基酸序列构建系谱进化树, 小麦 TaSPL17与水稻的 OsSPL14和 OsSPL17,拟南芥的 AtSPL9和 AtSPL15聚在同一个进化分支上[15]。OsSPL14能够调控水稻的株型, 提高产量; AtSPL9/15也参与调控花器官的形态建成并促进分枝[9,33]。我们的前期研究也表明, TaSPL17主要在茎的顶端分生组织和穗中表达, 并且其表达模式响应穗部发育[15], 这预示着 TaSPL17基因可能通过与tae-miR156的相互作用, 共同参与对小麦分蘖和穗部发育的调控, 相关研究正在进行中。
[1] Riese M, Höhmann S, Saedler H, Münster T, Huijser P. Comparative analysis of the SBP-box gene families in P. patens and seed plants. Gene, 2007, 401(1–2): 28–37.
[2] Chen XB, Zhang ZL, Liu DM, Zhang K, Li AL, Mao L. SQUAMOSA Promoter-Binding Protein-Like Transcription Factors: Star Players for Plant Growth and Development. J Integr Plant Biol, 2010, 52(11): 946–951.
[3] 许振华, 谢传晓. 植物microRNA与逆境响应研究进展.遗传, 2010, 32(10): 1018–1030.
[4] Huijser P, Schmid M. The control of developmental phase transitions in plants. Development, 2011, 138(19): 4117–4129.
[5] Wu G, Park MY, Conway SR, Wang JW, Weigel D, Poethig RS. The sequential action of miR156 and miR172 regulates developmental timing in Arabidopsis. Cell, 2009, 138(4): 750–759.
[6] Klein J, Saedler H, Huijser P. A new family of DNA binding proteins includes putative transcriptional regulators of the Antirrhinum majus floral meristem identity gene SQUAMOSA. Mol Gen Genet, 1996, 250(1): 7–16.
[7] Guo AY, Zhu QH, Gu XC, Ge S, Yang J, Luo JC. Genome-wide identification and evolutionary analysis of the plant specific SBP-box transcription factor family. Gene, 2008, 418(1–2): 1–8.
[8] Hultquist JF, Dorweiler JE. Feminized tassels of maize mop1 and ts1 mutants exhibit altered levels of miR156 and specific SBP-box genes. Planta, 2008, 229(1): 99–113.
[9] Wang SK, Wu K, Yuan QB, Liu XY, Liu ZB, Lin XY, Zeng RZ, Zhu HT, Dong GJ, Qian Q, Zhang GQ, Fu XD. Control of grain size, shape and quality by OsSPL16 in rice. Nat Genet, 2012, 44(8): 950–954.
[10] Jiao YQ, Wang YH, Xue DW, Wang J, Yan MX, Liu GF, Dong GJ, Zeng DL, Lu ZF, Zhu XD, Qian Q, Li JY. Regulation of OsSPL14 by OsmiR156 defines ideal plant architecture in rice. Nat Genet, 2010, 42(6): 541–544.
[11] Chang JZ, Zhang JN, Mao XG, Li A, Jia JZ, Jing RL. Polymorphism of TaSAP1-A1 and its association with agronomic traits in wheat. Planta, 2013, 237(6): 1495–1508.
[12] Zuker M. Mfold web server for nucleic acid folding and hybridization prediction. Nucleic Acids Res, 2003, 31(13): 3406–3415.
[13] Gruber AR, Lorenz R, Bernhart SH, Neuböck R, Hofacker IL. The vienna RNA websuite. Nucleic Acids Res, 2008, 36(S2): W70-W74.
[14] Dai X, Zhao PX. psRNATarget: a plant small RNA target analysis server. Nucleic Acids Res, 2011, 39(S2): W155-W159.
[15] Zhang B, Liu X, Zhao GY, Mao XG, Li A, Jing RL. Molecular characterization and expression analysis of Triticum aestivum SBP-box genes involved in ear development. J Integr Plant Biol, 2013, DOI: 10. 1111/jipb. 12153
[16] Letunic I, Doerks T, Bork P. SMART 7: recent updates to the protein domain annotation resource. Nucleic Acids Res, 2012, 40(D1): D302-D305.
[17] Kumar S, Nei M, Dudley J, Tamura K. MEGA: a biologist-centric software for evolutionary analysis of DNA and protein sequences. Briefings Bioinf, 2008, 9(4): 299–306.
[18] Bartel DP. MicroRNAs: genomics, biogenesis, mechanism, and function. Cell, 2004, 116(2): 281–297.
[19] 吕德康, 葛瑛, 柏锡, 李勇, 朱延明. 生物信息学在植物 miRNA 研究中的应用. 生物信息学, 2009, 7(2): 113–116, 136.
[20] Voinnet O. Origin, biogenesis, and activity of plant microRNAs. Cell, 2009, 136(4): 669–687.
[21] Yu N, Cai WJ, Wang S, Shan CM, Wang LJ, Chen XY. Temporal control of trichome distribution by microRNA156-targeted SPL genes in Arabidopsis thaliana. Plant Cell, 2010, 22(7): 2322–2335.
[22] Reyes JL, Chua NH. ABA induction of miR159 controls transcript levels of two MYB factors during Arabidopsis seed germination. Plant J, 2007, 49(4): 592–606.
[23] Turner M, Nizampatnam NR, Baron M, Coppin S, Damodaran S, Adhikari S, Arunachalam SP, Yu O, Subramanian S. Ectopic expression of miR160 results in auxin hypersensitivity, cytokinin hyposensitivity, and inhibition of symbiotic nodule development in soybean. Plant Physiol, 2013, 162(4): 2042–2055.
[24] Mallory AC, Dugas DV, Bartel DP, Bartel B. MicroRNA regulation of NAC-domain targets is required for proper formation and separation of adjacent embryonic, vegetative, and floral organs. Curr Biol, 2004, 14(12): 1035–1046.
[25] Chen X. A microRNA as a translational repressor of APETALA2 in Arabidopsis flower development. Science, 2004, 303(5666): 2022–2225.
[26] Rhoades MW, Reinhart BJ, Lim LP, Burge CB, Bartel B, Bartel DP. Prediction of plant microRNA targets. Cell, 2002, 110(4): 513–520.
[27] Lännenpää M, Jänönen I, Hölttä-Vuori M, Gardemeister M, Porali I, Sopanen T. A new SBP-box gene BpSPL1 in silver birch (Betula pendula L.). Physiol Plant, 2004, 120(3): 491–500.
[28] Cardon GH, Höhmann S, Nettesheim K, Saedler H, Huijser P. Functional analysis of the Arabidopsis thaliana SBP-box gene SPL3: a novel gene involved in the floral transition. Plant J, 1997, 12(2): 367–377.
[29] Stone JM, Liang X, Nekl ER, Stiers JJ. Arabidopsis AtSPL14, a plant-specific SBP-domain transcription factor, participates in plant development and sensitivity to fumonisin B1. Plant J, 2005, 41(5): 744–754.
[30] Xing S, Salinas M, Höhmann S, Berndtgen R, Huijser P. miR156-targeted and nontargeted SBP-box transcription factors act in concert to secure male fertility in Arabidopsis. Plant Cell, 2010, 22(12): 3935–3950.
[31] Xie K, Wu C, Xiong L. Genomic organization, differential expression, and interaction of SQUAMOSA promoter-binding-like transcription factors and microRNA156 in rice. Plant Physiol, 2006, 142(1): 280–293.
[32] Adams KL, Wendel JF. Polyploidy and genome evolution in plants. Curr Opin Plant Biol, 2005, 8(2): 135–141.
[33] Schwarz S, Grande AV, Bujdoso N, Saedler H, Huijser P. The microRNA regulated SBP-box genes SPL9 and SPL15 control shoot maturation in Arabidopsis. Plant Mol Biol, 2008, 67(1): 183–195.
(责任编委: 夏先春)
Cloning of tae-MIR156 precursor gene and sequence polymorphisms of tae-miR156 targeted TaSPL17
Xia Liu1,2, Bin Zhang2, Xinguo Mao2, Ang Li2, Meirong Sun2, Ruilian Jing2
1. College of Bioengineering, Shanxi University, Taiyuan 030006, China;
2. National Key Facility for Crop Gene Resources and Genetic Improvement, Institute of Crop Science, Chinese Academy of Agricultural Sciences, Beijing 100081, China
Squamosa-promoter binding protein (SBP)-box genes, encoding plant-specific transcription factors, play an important role in plant development. Some members of the SBP-box gene family are regulated by miR156. In this study, we cloned the tae-MIR156 precursor gene, which could form a stem loop after its transcription. Sequence analysis showed thatTaSPL3 and TaSPL17 had putative targets of tae-miR156 among the ten wheat SBP-box genes. The diploid donor species of hexaploid common wheat (Triticum aestivum, genome AABBDD), i.e., Triticum urartu (AA) UR209 and Aegilops speltoides Y2001 (SS, closely related to BB) possessed more than one copy of SPL17 (SPL17-A1, SPL17-A2 and SPL17-A3 from Triticum urartu; SPL17-B1, SPL17-B2 and SPL17-B3 from Aegilops speltoides), while Aegilops tauschii (DD) Ae38 only possessed one (SPL17-D). The identities between nucleotide sequences of SPL17-A2 and SPL17-B2, SPL17-A3 and SPL17-B3 or SPL17-D were higher than 99%. They were highly similar with the sequence of TaSPL17 in common wheat cultivars Chinese Spring, Hengguan 35 and Shuangfengshou. These genes might originate from a common ancestor and were highly conserved in the process of evolution. The target site of tae-miR156 in TaSPL17 was also highly conserved in two subgroups consisted of accessions with diverse spike number per plant and genetic background.
miR156; SBP-box gene; polymorphism analysis; common wheat
2014-01-09;
2014-02-11
国家高技术研究发展计划项目(863计划)(编号:2012AA10A308)资助
刘霞, 硕士研究生, 专业方向:小麦基因资源发掘。E-mail: liuxia1214lx@163.com
景蕊莲, 博士, 研究员, 研究方向:作物抗逆生物学。E-mail: jingruilian@caas.cn
10.3724/SP.J.1005.2014.0592
时间: 2014-5-6 15:13:00
URL: http://www.cnki.net/kcms/detail/11.1913.R.20140506.1513.004.html

