猴B病毒囊膜蛋白gD的B细胞表位预测及鉴定
叶华虎,袁菊芳,刘 欢,范玉筠
(1.军事医学科学院实验动物中心,北京 100071;2.北京城市学院,北京 100094)
表位(epitope)又称抗原决定簇(antigenic determinant, AD),是病毒、疫苗、致敏原等抗原物质刺激机体产生免疫应答的物质基础。现代免疫学研究表明,免疫细胞在与抗原分子的相互作用过程中,细胞表面受体识别的不是整个抗原分子,而是抗原分子上的特定部位(包括线性的,或是空间结构上相互靠近的多个氨基酸残基)[1-2]。可见,由异源蛋白活化B淋巴细胞产生的抗体仅针对抗原上的特异性表位。随着抗体在疾病诊断、预防、治疗等领域的广泛应用,表位鉴定已成为免疫学研究的热点。
到目前为止,表位研究已形成了多种技术体系,如噬菌体展示肽库技术、肽段扫面技术、合成肽技术、生物信息学预测技术。相较于其他技术体系,生物信息学预测技术具有快速、廉价等明显优势[3-5]。本实验以在我国恒河猴体内扩增的B病毒囊膜蛋白gD氨基酸序列为基础,利用生物信息学软件对其二级结构、亲水性、表面可及性、抗原性指数、柔韧性进行分析,筛选可能的表位肽段,再结合合成肽技术,利用B病毒阳性血清通过ELISA进行验证;最后,以20份B病毒标准阳性血清评价肽段的敏感性。本实验旨在为猴B病毒的防控研究奠定基础。
1 材料和方法
1.1 材料
1.1.1 gD蛋白 系从我国实验猕猴体内通过PCR扩增得到的gD基因推导而来,其氨基酸序列如下:该蛋白全长394个氨基酸残基,分子量约为48×103,与E2490标准毒株的同源性为97.5%。
1MGSGIAAVLL SLAVALARVP AGGGEYVPVE RSLTRVNPGR FRGAHLPPLE QKTDPPDVRR VYHVQPFVEN PFQTPSVPVAV YYAVLERAC RSVLLWAPTE AVQVVRGAPE ATRSDARYNL TVAWYRTSDD CAIPILVMEY AECQYDKPLG ACPVRNLPRW SFYDSFSATG DDDLGLLMHA PAFETAGTYV RLVKVNGWVE VTQFIFEHRG KGPCRYTLPL RILPAACLRA PVFEQGVTVD AIGMLPRFIP ENQRIVAVYS LQAAGWHGPK APFTSTLLPP EVVETANVTR PELAPEERGT SRTPGDEPAP AVAAQLPPNW HVPEASDVTI QGPAPAPSGH TGAVVGALAG AGLAAGVVVL AVYLVRRRGR AAGKHVRLPE LLEEAHGPAR RGAPY394
1.1.2 血清和抗体 B病毒阳性血清和B病毒阴性血清,军事医学科学院实验动物中心保存。
1.1.3 ELISA检测试剂 氨基酶标板购于Nunc公司;其他ELISA试剂为湖州英创生物公司产品。
1.1.4 主要仪器 脱色摇床,恒温水浴锅,酶标仪。
1.2 试验方法
1.2.1 B病毒囊膜蛋白gD二级结构预测 利用DNAStar软件,以Chou-Fasman方法、Karplus-Schulz方法和Gamier-Robson方法对预测gD蛋白的二级结构[6]。
1.2.2 猴B病毒囊膜蛋白gD基因B细胞表位预测 利用DNAstar软件,用Jameson-Wolf方法对猴B病毒囊膜蛋白gD基因氨基酸序列抗原性指数区域预测[7];用Emini方法猴B病毒囊膜蛋白gD基因氨基酸序列表面可及性区域预测[8],用Kyte-Doolittle方法对猴B病毒囊膜蛋白gD基因氨基酸序列亲水性预测[8-10]。综合分析将上述几种方法,兼顾各项预测参数对猴B病毒囊膜蛋白gD基因 B细胞表位进行预测,并以吴玉章建立的抗原性指数对预测的表位进行综合评判[11]。
1.2.3 肽段合成 将预测的肽段送南京金斯瑞生物技术有限公司合成。
1.2.4 肽段ELIS条件优化 首先以文献报道的已知表位肽(AVYLVRRRGR)为基础进行ELISA条件优化,过程简述如下:将合成肽段用灭菌的PBS稀释成1 mg/mL,再用碳酸盐缓冲液(pH 9.6)稀释成不同浓度,稀释液用于包被氨基板;将包被过夜的ELISA板用PBS洗涤3次,加100 μL封闭液(博迈德生物技术公司)于37℃封闭30 min;将5份标准阳性血清混合物和3份标准阴性血清混合物分别作5、10、20倍稀释,加入酶标板,在37℃反应35 min;5×3 min洗涤;再加入100 μL二抗反应液(用封闭液稀释HRP标记的兔抗猴IgG到30000倍),37℃反应35 min;5×3 min洗涤;再加入TMB显色液100 μL,37℃反应,8 min;最后加入50 μL的2N H2SO4终止显色;酶标仪上测定450 nm下的OD值。比较不同条件(包被量和血清稀释倍数)下的信噪比(P/N值),确定ELISA的包被量和血清稀释比例。
1.2.5 gD表位鉴定 将7个预测得到表位肽段按优化确定的结果进行稀释和包被,并以上述血清进行ELISA初步鉴定,ELISA结果判定:以阴性血清的OD值的2倍作为阳性判定结果。
1.2.6 不同表位的敏感性和特异性评价 对于初步鉴定为表位的肽段,用20份B病毒标准阳性血清和10份B病毒阴性血清评价其敏感性和特异性。
2 结果
2.1 B病毒囊膜糖蛋白gD基因二级结构预测
利用G Gamier-Robson法和Chou-Fasman法分别对猴B病毒囊膜糖蛋白gD基因的α-螺旋区域、β-折叠区域、转角区域和柔性结构区域进行预测(图1~图4)。综合分析比较两种方法的预测结果,得出最优效果。
2.2 猴B病毒囊膜糖蛋白gD基因B细胞表位预测
利用Jameson-Wolf方法对猴B病毒囊膜糖蛋白gD基因抗原性指数(抗原性指数≥0)进行预测(图5);利用Emini方法对猴B病毒囊膜糖蛋白gD基因表面可及性区域(表面可及性指数≥1)进行预测(图6);利用Kyte-Doolittle方法对猴B病毒囊膜糖蛋白gD基因亲水性区域(亲水性指数≥0)进行预测(图7)。
筛选出具有较好抗原性指数、表面可及性和亲水性且在二级结构上含有易形成不规则卷曲结构和抗原表位转角的区段,这些区段极有可能成为B细胞表位。结果显示猴B病毒囊膜糖蛋白gD基因N端第26-34、46-54、106-116、122-130、203-214、291-306、361-370区段的抗原性指数最高,提示在这些区段是B细胞表位的可能性最大(表1)。
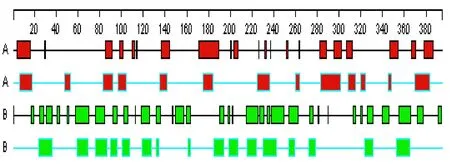
图1 猴B病毒囊膜糖蛋白gD α-螺旋和β-折叠区域预测结果

图2 猴B病毒囊膜糖蛋白gD转角区域预测结果

图3 猴B病毒囊膜糖蛋白gD无规则卷曲区域预测结果

图4 猴B病毒囊膜糖蛋白gD柔性区域预测结果

图5 猴B病毒囊膜糖蛋白gD抗原性指数预测结果

图6 猴B病毒囊膜糖蛋白gD表面可及性预测结果

图7 猴B病毒囊膜糖蛋白gD亲水性预测结果
表1猴B病毒囊膜糖蛋白gD的表位预测结果
Tab.1Prediction of B-cell epitopes on Monkey B virus envelope protein gD

B细胞表位区域Domains of B-cell epitope氨基酸序列Amino acid sequences of epitopes26-34YVPVERSLT46-54LPPLEQKTD106-116RGAPEATRSDA122-130VAWYRTSDD203-214QFIFEHRGKGPC291-306PELAPEERGTSRTPGD361-370AVYLVRRRGR
2.3 合成肽的ELISA检测方法建立
由于合成肽可能与重组蛋白不完全一致。实验先用被证实是B病毒表位肽段的AVYLVRRRGR(位于gD蛋白的361-370)为对象,分别用浓度100、150和300 ng每孔包板,并以5份标准B病毒阳性血清的混合物和3份标准阴性血清进行检测(3次重复)。其中血清稀释倍数分别为1∶5、1∶10和1∶20,兔抗猴IgG-HRP的稀释浓度为1∶30000。ELISA的OD值阈值为3份阴性血清平均OD的2倍。
结果显示:肽段以150 ng每孔包被,血清稀释比例为1∶5的反应条件最好,此时信噪比最高,且结果重复性好(表2)。
2.4 gD表位鉴定
将7个肽段(表1)按照150 ng每孔包板,对5份阳性血清的混合物和3份阴性血清进行ELISA检测,血清稀释比例为1∶5,兔抗猴IgG-HRP工作浓度为1∶30000,OD值阈值为3份阴性样本的平均OD值乘以2 倍。结果显示,gD蛋白上的4个肽段(46LPPLEQKTD54、106RGAPEATRSDA116、291PELAPE ERGTSRTPGD306和361AVYLVRRRGR370)都能与B病毒阳性血清反应反应,且OD值均显示为阳性(表3)。其余肽段均为阴性,即使将这些肽段的包被量提高到300 ng每孔,在其他ELISA参数不变的情况下,结果仍为阴性。表明这些肽段不是B病毒的表位。
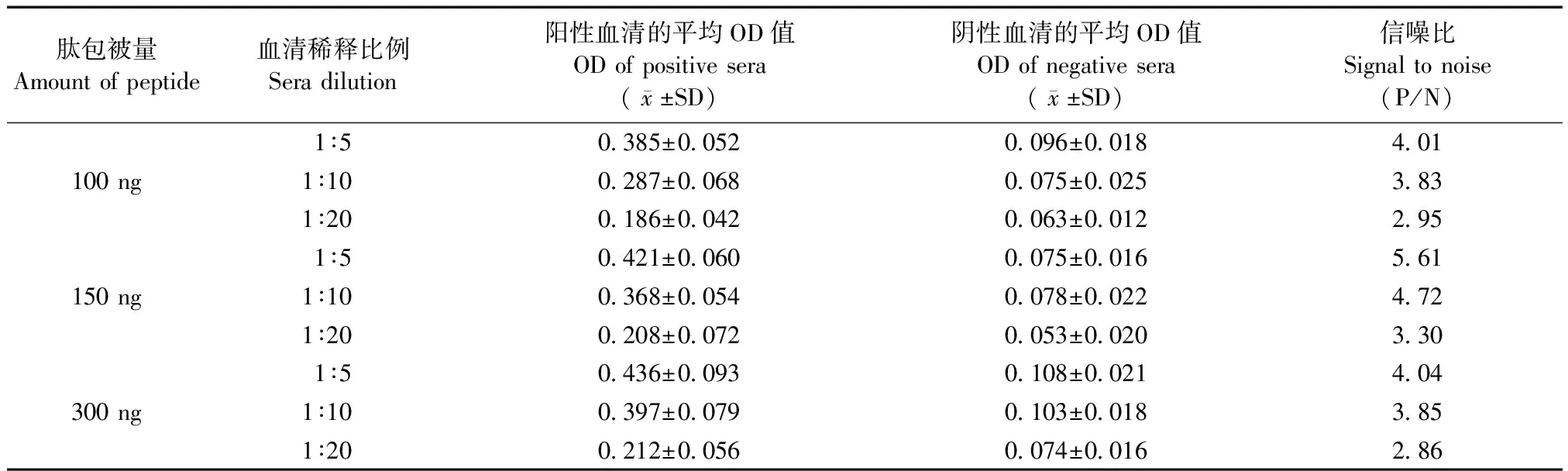
表2 肽段ELISA参数的优化

表3 ELISA方法鉴定的表位肽段

图8 表位肽与20份阳性血清的ELISA检测结果(红色表示阳性,白色表示阴性)
2.5 不同表位的敏感性和特异性评价
将上述表位肽段按150 ng每孔包被,分别用20份B病毒标准阳性血清和10份标准阴性血清鉴定各表位的敏感性和特异性。其中,血清稀释比例为1∶5,兔抗猴IgG-HRP工作浓度为1∶30000,OD值阈值为3份阴性样本的平均OD值乘以2 倍。结果显示,各个肽段都不与阴性血清发生反应,特异性为100%;但就敏感性而言,不同表位肽段各不相同,106RGAPEATRSDA116肽段的敏感性最低,仅能与20份阳性血清的8份(40.0%)发生反应,而361AVYLVRRRGR370)能与20份阳性血清中的14份(70.0%)发生反应,其他肽段的敏感性位于二者之间。
比较7个表位肽段与20份阳性血清的反应性发现,有些表位肽段的阳性样本完全一致,有些表位肽段的阳性样本只是其他表位的一部分,也有一些表位的阳性样本之间有交叉,但也有一些阳性样本在所有表位中均呈阴性表现(图8)。
3 讨论
对于抗原表位的研究策略,人们已建立和发展了多种方法,如噬菌体展示肽库技术、但克隆抗体筛选技术、肽探针扫描技术和基于生物信息学的表位预测技术等[1-5]。其中表位预测因其简单和廉价而越来越受到研究者重视,该方法既克服了肽探针扫描需要大量肽段合成所带来的高成本,也避免了化学和生物学方法以及噬菌体随机肽库展示技术所要求单克隆抗体。同时该方法也是当前广泛采用的,并被证明是行之有效的表位研究技术[3-5]。
表位预测主要依据蛋白质的二级结构、亲水性、表面可及性、抗原性指数以及柔韧性等参数,预测抗原的表位序列。目前开发的分析软件正是基于不同参数组合形成的数据库,因而预测的结果可能有较大差异,且都存在预测失误(包括漏测和错测)等缺陷。有研究者统计,目前建立的表位预测技术,准确率通常在15%~75%之间[12-13]。
我们分别利用了在线预测软件,如ABCpred(ABCpred 采用人工神经网络来预测线性表位,从Bcipep 和SwissProt 数据库中提取非冗余的表位肽和非表位肽作为训练集,采用5- 折交叉验证,预测敏感性约为67%,特异性约为64%[14])、BepiPred(BepiPred 结合氨基酸的性质,如亲水性、柔韧性、可及性、极性、暴露表面、转角和隐形马尔可夫模型来预测线性表位,预测结果表明,同那些仅依赖于氨基酸性质的预测方法相比,BepiPred 预测结果的准确性有一定程度的提高[15])、APP(基于氨基酸通常成对出现在抗原表位的频率要比其出现在非表位肽段的频率高并联合支持向量机[16])等算法,同时结合国内一些学者创建的软件进行预测。综合这些软件的预测结果,在B病毒囊膜gD上得到7个可能的表位肽段。其中,预测得到的gD蛋白上的361AVYLVRRRGR370表位,已被实验所证实,二者的差异仅为1个氨基酸残基,即实验鉴定的表位为的362VYLVRRRGR370[17]。
对7个肽段的鉴定结果显示, 4个肽段都能与B病毒的阳性血清发生反应,表明这4个片段是B病毒的表位。对与B病毒高度同源的HSV-1囊膜蛋白gD功能研究发现,胞外区1-32个氨基酸残基、Y38、D215、R222、F223是受体的结合区,负责与受体结合;胞外区280-302个氨基酸残基为柔性折叠部位,负责维持gD构象;胞外区C端是gB、gL/gH的结合部位,负责膜融合[18-20]。如果B病毒的gD蛋白采用与HSV-1相似的功能机制,那么在本实验中鉴定的3个表位(分别为46LPPLEQKTD54、106RGAPEATRSDA116和291PELAPEERGTSRTPGD306)将与功能区重合或与功能区紧密相连。这为前期文献报道—以gD基因为基础的核酸疫苗能较好保护免疫动物提供了合理解释[21-22]。
实验进一步利用20份B病毒阳性血清对4个表位进行了评价,发现表位肽与血清的反应性不完全一致。其中,106RGAPEATRSDA116肽段的敏感性最低,仅能与20份阳性血清的8份(40.0%)发生反应,而已被证实的表位361AVYLVRRRGR370,敏感性为70%,与其他文献报道的67%相似[22]。将多个具有互补作用的表位串联,形成嵌合性多价表位蛋白/疫苗,作为检测抗原的敏感性和/或作为疫苗的保护作用明显增强,已在其他研究中得到证实[23-24]。将上述表位进行联合或者重组,是否有利于提高检测的敏感性和疫苗保护效果,有待于进一步研究。
总之,本实验利用生物信息学方法在B病毒囊膜蛋白gD上鉴定了4个表位,这些表位的发现,为进一步开展B病毒感染机制研究以及快速检测方法的建立奠定了基础。
参考文献:
[1] Gershoni JM, Roitburd-Berman A, Siman-Tov DD,etal. Epitope mapping: the first step in developing epitope-based vaccines[J]. BioDrugs. 2007;21(3):145-56.
[2] 宋帅,李春玲,贾爱卿,等. 抗原表位研究方法进展[J]. 动物医学进展, 2010, 31( 12) : 87-91
[3] Zhu J, Warren JD, Danishefsky SJ. Synthetic carbohydrate-based anticancer vaccines: the Memorial Sloan-Kettering experience[J]. Expert Rev Vaccines. 2009 , 8(10):1399-413.
[4] Editorial. Immunoinformatics may lead to a reappraisal of the nature of B cell epitopes and of the feasibility of synthetic peptide vaccines[J]. J. Mol. Recognit. 2006; 19: 183-187.
[5] Das P, Grewal JS, Mahajan B,etal. Comparison of cellular and humoral responses to recombinant protein and synthetic peptides of exon2 region of Plasmodium falciparum erythrocyte membrane protein1 (PfEMP1) among malaria patients from an endemic region[J]. Parasitology International , 2007, 56: 51-59.
[6] Karplus P A Schultz G. Prediction of chain flexibility in proteins[J]. Natur wissenschaften,1985,72:212-213.
[7] Jameson B A Wolf H.The antigenic index: a novel algorithm for predicting antigenic determinations[J]. Comput Appl Biosci,1988,4(1):181-186.
[8] Emini E A Hughes J V, Perlow D S,etal. Induction of hepatitis A virus~neutralizing antibody by a virus~specific synthetic peptide[J]. J Virol,1985,55(3):836-839.
[9] Kyte J Doolittle R F.A simple method for displaying the hydropathic character of a protein[J]. J Mol Bio1,1982,157(1):105-132.
[10] Garnier J,Osguthorpe D J,Robson B.Analysis of the accuracy and implications of simple methods for predicting the secondary structure of globular proteins[J]. Journal of molecular biology,1978,120(1):97-120.
[11] 朱锡华,吴玉章. 一种病蛋白B细胞表位预测方法的建立[J]. 科学通报,1994,24(2):2275-2279.
[12] Peters B, Sette A. Integrating epitope data into the emerging web of biomedical knowledge resources[J]. Nat Rev Immunol. 2007 Jun;7(6):485-90.
[13] Wee LJ, Lim SJ, Ng LF, Tong JC. Immunoinformatics: how in silico methods are re-shaping the investigation of peptide immune specificity[J]. Front Biosci (Elite Ed). 2012, 4:311-319.
[14] Saha S, Raghava GP. Prediction of continuous B-cell epitopes in an antigen using recurrent neural net work[J]. Proteins, 2006,65(1): 40-48.
[15] Larsen JE, Lund O, Nielsen M. Improved method for predicting linear B-cell epitopes[J]. Immunnome Res, 2006, 2: 2.
[16] ChenJ, LiuH, YangJ, etal. PredictionoflinearB-cellepitopes using amino acid pair antigenicity scale[J]. Amino Acids, 2007,33(3): 423-428.
[17] Perelygina L, Zurkuhlen H, Patrusheva I,etal. Identification of a herpes B virus-specific glycoprotein D immunodominant epitope recognized by natural and foreign hosts[J]. J Infect Dis, 2002, 186: 453-461.
[18] Giovine PD, Settembre EC, Bhargava AK,etal. Structure of Herpes Simplex Virus Glycoprotein D Bound to the Human Receptor Nectin-1[J]. PLoS Pathogens, 2011, 7(9):489-502.
[19] Karaba AH, Kopp SJ, Longnecker R. Herpesvirus entry mediator and nectin-1 mediate herpes simplex virus 1 infection of the murine cornea[J]. J Virol. 2011 Oct;85(19):10041-10047.
[20] Atanasiu D, Saw WT, Cohen GH,etal. Cascade of events governing cell-cell fusion induced by herpes simplex virus glycoproteins gD, gH/gL, and gB[J]. J Virol. 2010 ,84(23):12292-12299.
[21] Hirano M, Nakamura S, Mitsunaga F,etal. Efficacy of a B virus gD DNA vaccine for induction of humoral and cellular immune responses in Japanese macaques[J]. Vaccine. 2002; 20(19-20):2523-32.
[22] Loomis-Huff JE, Eberle R, Lockridge KM,etal. Immunogenicity of a DNA vaccine against herpes B virus in mice and rhesus macaques[J]. Vaccine. 2001; 19(32):4865-4873.
[23] Liu S, Yu X, Wang C,etal. Quadruple antigenic epitope peptide producing immune protection against classical swine fever virus[J]. Vaccine, 2006, 24 :7175- 7180.
[24] Cairns TM, Shaner MS, ZuoY,etal. Epitope mapping of herpes simplex virus type 2 gH/gL defines distinct antigenic sites, including some associated with biological function[J]. J Virol, 2006, 80(6): 2596-2608.

