血浆中多基因联合甲基化检测在肺癌诊断中的应用*
丁 浩, 沈志高, 李 昊, 邱 宇, 郝晓宁, 祖金池, 钟 理, 2△
(1河北大学生命科学学院生物芯片研究室,河北 保定 071002; 2Western University of Health Science,California 91766,USA; 3河北大学附属医院,河北 保定 071002)
血浆中多基因联合甲基化检测在肺癌诊断中的应用*
丁 浩1, 沈志高1, 李 昊1, 邱 宇1, 郝晓宁1, 祖金池3, 钟 理1, 2△
(1河北大学生命科学学院生物芯片研究室,河北 保定 071002;2Western University of Health Science,California 91766,USA;3河北大学附属医院,河北 保定 071002)
目的: 探究血浆中CDH13、RASSF13A、DLEC13、SEPT13、RUNX13等抑癌基因启动子甲基化及其联合检测在肺癌诊断中的价值。从中选出诊断效能高的组合。方法: 采用巢式甲基化特异性PCR(nest methylation specific PCR,nMSP)法,检测106例健康人血浆样本、106例肺癌组织和癌旁组织以及其对应的106例术前血浆样本、中基因启动子区的甲基化状态。对血浆基因组DNA修饰后进行多重置换扩增 (multiple displacement amplification, MDA),以解决血浆DNA模板不足的问题。结果: 肺癌组织样本中的CDH13、RASSF13A、DLEC13、SEPT13、RUNX13基因启动子甲基化率分别为51.9%、44.3%、54.7%、36.8% 、24.5%。对应的血浆样本中的CDH13、RASSF1A、DLEC1、SEPT9、RUNX3基因启动子甲基化率分别为46.2%、41.5%、50.9%、31.1%、19.8%。Kappa一致性检验结果表明肺癌组织与血浆的甲基化检出率一致。CDH13、DLEC1、RASSF1A、SEPT9组合对肺癌的诊断效能明显高于其它组,准确度ACC为82.08%,Youden指数为0.6415(灵敏度为79.49%,特异度为81.13%)。结论: 血浆多基因联合甲基化检测有望应用于肺癌早期诊断。
肺肿瘤; 血浆; 组织; 甲基化
肺癌是我国发病率和致死率最高的癌症之一[1]。由于诊断手段的限制,大多患者在确诊时已发展到中晚期,取得肺癌组织标本会给患者身体造成很大伤害,难以进行病理检测。因此,亟需一种能实时、有效、无创的检测手段,进行肺癌的诊断。肺癌的发生是各种遗传信息改变和环境因素累积作用的结果。表观遗传学研究表明,肺癌的发生可能与抑癌基因启动子区CpG岛的甲基化相关[2]。CpG岛的异常高甲基化是导致人类抑癌基因的转录失活的机制之一,在所有人的血浆中均存在,由于应激反应所致,其在病人血浆中含量显著高于健康人[3]。肺癌患者血浆中的循环DNA主要来源于肿瘤细胞[4],其分子生物学特征与原发灶相一致[5]。因此,检测血浆中相关抑癌基因的甲基化,有望成为临床上肺癌诊断的有效手段。
本研究采用巢氏甲基化特异性PCR技术检测肺癌患者血浆(术前)、健康人血浆、肺癌组织和癌旁组织中 H-钙黏素 13 (H-cadherin 13,CDH13)、runt相关转录因子(runt-related transcription factor 3,RUNX3)、ras相关区域家族(ras-associated domain family 1A,RASSF1A)、DLEC1 (deleted in lung and esophageal cancer 1)及 septin 9(SEPT9)等抑癌基因启动子的甲基化状况。总结各基因甲基化状态与肺癌临床诊断的关系,比较2种取材检测结果的相关性,探究血浆作为临床取材的可行性,探讨各基因作为肺癌诊断标志物的可能性,综合灵敏度和特异性,选择一组基因甲基化标志物应用于肺癌临床诊断。
材 料 和 方 法
1 临床资料
材料均取自术前未经放化疗,且无其它肿瘤病史的肺癌患者。收集2013年2月~2014年10月保定市第一中心医院和河北大学附属医院106例肺癌患者的肺癌组织、癌旁组织(距癌组织> 3cm)以及术前血液样本,其中男72例,女34例;年龄35~84岁,中位年龄61岁。另采集106份健康人血液样品作对照。
2 样品的收集处理
肺癌组织标本收集后立即放入液氮中,之后放置于超低温冰箱。血液样本用枸橼酸钠蓝色真空采血管收集,采血6 h内4 000 r/min离心10 min分离血浆;取上清,4 ℃条件下,再次以10 000 r/min离心15 min获得血浆,用1.5 mL离心管以每管200 μL分装。
3 主要方法
3.1 基因组DNA的提取 采用DNAiso reagent(TaKaRa)提取组织中的基因组DNA,血浆DNA的提取按照QI Aamp DNA Blood Mini Kit(Qiagen)的血液或体液检测方案进行,用50 μL AE 洗脱 DNA。Nanodrop 2000C及琼脂糖凝胶电泳测浓度纯度,A260/A280值为1.79~1.87。
3.2 基因组DNA的修饰 采用EZ DNA Methylation-Gold Kit(Zymo Research)每次修饰1 μg组织基因组DNA,200 ng血浆基因组DNA,用10 μL M-Elution buffer洗脱。
3.3 血浆DNA多重置换扩增 (multiple displacement amplification,MDA) 使用Epi Tect Whole Bisulfitome Kit扩增试剂盒(Qiagen)对修饰后的血浆DNA进行全基因组扩增。
3.4 巢氏甲基化特异性PCR及电泳检测 运用Methprimer 软件设计nMSP的引物序列。在重亚硫酸盐测序(bisulfite sequencing PCR,BSP)条件下设计外侧引物,在甲基化特异性PCR(methylation-specific PCR,MSP)条件下分别设计甲基化(M)引物和非甲基化(U)引物。引物由苏州金唯智公司合成,引物序列、产物大小和退火温度等见表1。LA Taq with GC buffer(TaKaRa),25 μL反应体系:LA Taq 0.25 μL,2×GC bufferⅡ 12.5 μL,dNTP Mixture 4 μL,修饰后的DNA模板0.5 μL,10 mmol/L的上下游引物各0.75 μL,加入ddH2O定容至25 μL。第1轮PCR的反应条件为95 ℃ 5 min;94 ℃ 30 s,各基因按各自温度 30 s,72 ℃ 2 min,共40个循环;72 ℃ 5 min。第1轮PCR后,取5 μL产物经2%的琼脂糖凝胶电泳,溴化乙锭染色,凝胶成像检测。第2轮PCR反应,取0.5 μL第1轮PCR产物作为模板(稀释50倍),取10 mmol/L的甲基化、非甲基化上下游引物各1 μL,退火温度见表1,其它条件相同扩增30次,电泳检测。以经甲基化转移酶M.SssI(Zymo Research)处理的健康人外周血单核细胞DNA作阳性对照,未经M.SssI处理的健康人外周血单核细胞DNA作阴性对照,以ddH2O代替模板作为空白对照。
4 甲基化结果判断
第1轮PCR出现条带;第2轮PCR甲基化引物出现条带,非甲基化引物未出现条带,该基因启动子区判断为甲基化。甲基化引物、非甲基化引物PCR均出现条带(部分甲基化),亦判断为甲基化。甲基化引物未出现条带,非甲基化引物出现条带,则判断为未甲基化。
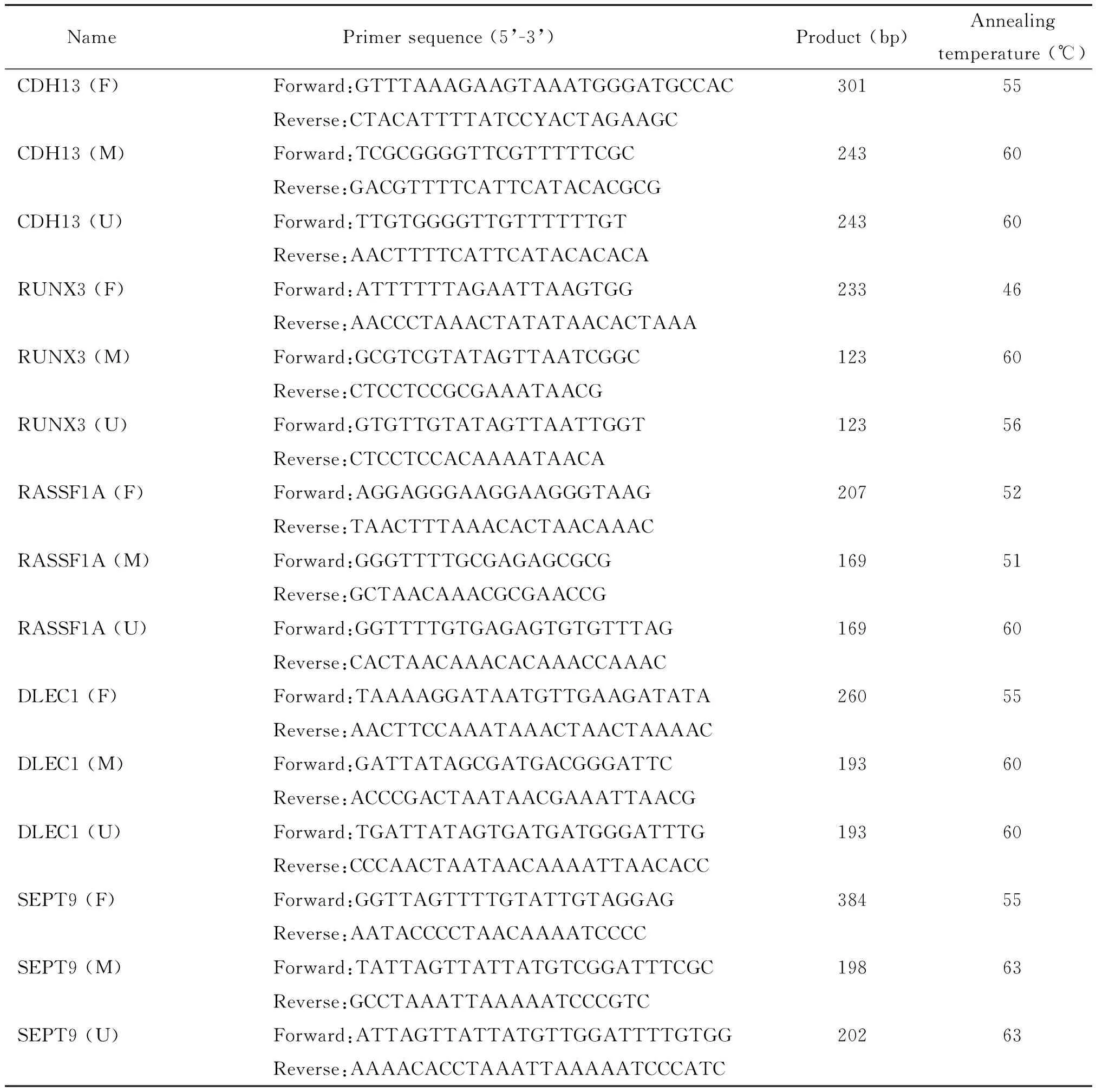
表1 引物序列
5 统计学处理
使用SPSS 17.0软件对各基因甲基化的情况进行计数,采用Fisher精确检验对肺癌样本各基因甲基化状态与临床各种病理因素进行统计分析处理。Fisher精确检验比较肺癌组织和癌旁组织各基因甲基化率的差异性;Kappa一致性检验评价血浆和肺癌组织检验结果的一致性。灵敏度(%)=真阳性人数/(真阳性人数+假阴性人数)×100%;特异度(%)=真阴性人数/(真阴性人数+假阳性人数)×100%。以P<0.05为差异有统计学意义;κ>0.75为两者具有一致性。
结 果
1 各样本DNA甲基化检测结果
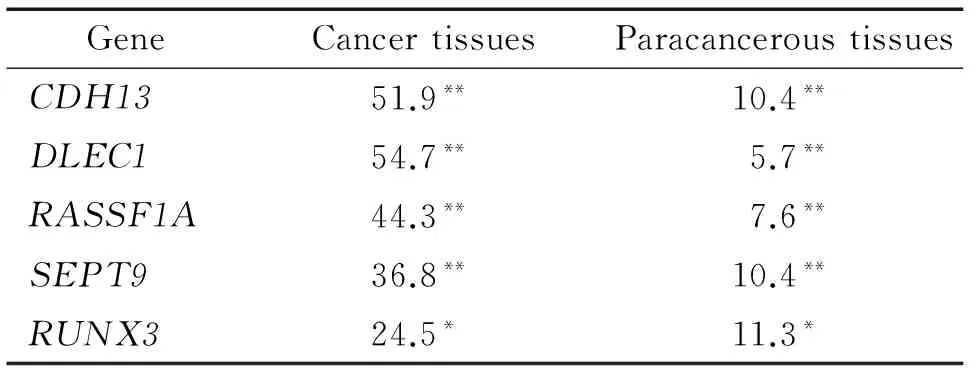
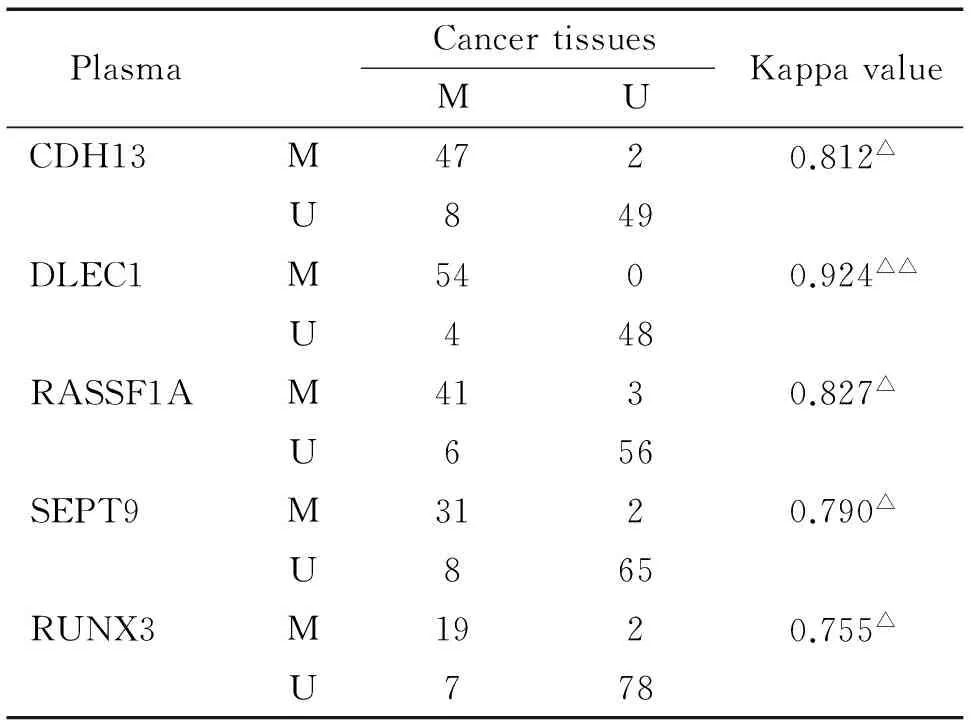
首先用外侧引物分别进行第1轮扩增,结果所有DNA样本均可扩增出相应片段。然后分别用甲基化和非甲基化特异性引物进行第2轮扩增,结果显示106例肺癌组织样本中的CDH13、RASSF1A、DLEC1、SEPT9和RUNX3基因启动子区甲基化分别为51.9%、44.3%、54.7%、36.8%和24.5%。106例癌旁组织样本中的CDH13、RASSF1A、DLEC1、SEPT9和RUNX3基因启动子区甲基化分别为10.4%、7.6%、5.7%、10.4%和11.3%。106例血浆样本中的CDH13、RASSF1A、DLEC1、SEPT9和RUNX3基因启动子区甲基化分别为46.2%、41.5%、50.9%、31.1%和19.8%。阳性对照和阴性对照均有特异性扩增产物。各基因nMSP的凝胶电泳结果见图1。肺癌组织和癌旁组织各基因的甲基化率差异均有统计学意义(P<0.05),其中RUNX3基因在2种组织中的甲基化检出率比较差异有统计学意义(P<0.05);而CDH13、RASSF1A、DLEC1和SEPT9的甲基化检出率在2种组织中的差异亦有统计学意义(P<0.01),见表2。Kappa一致性检验结果表明肺癌组织与血浆的甲基化率一致性明显,见表3。
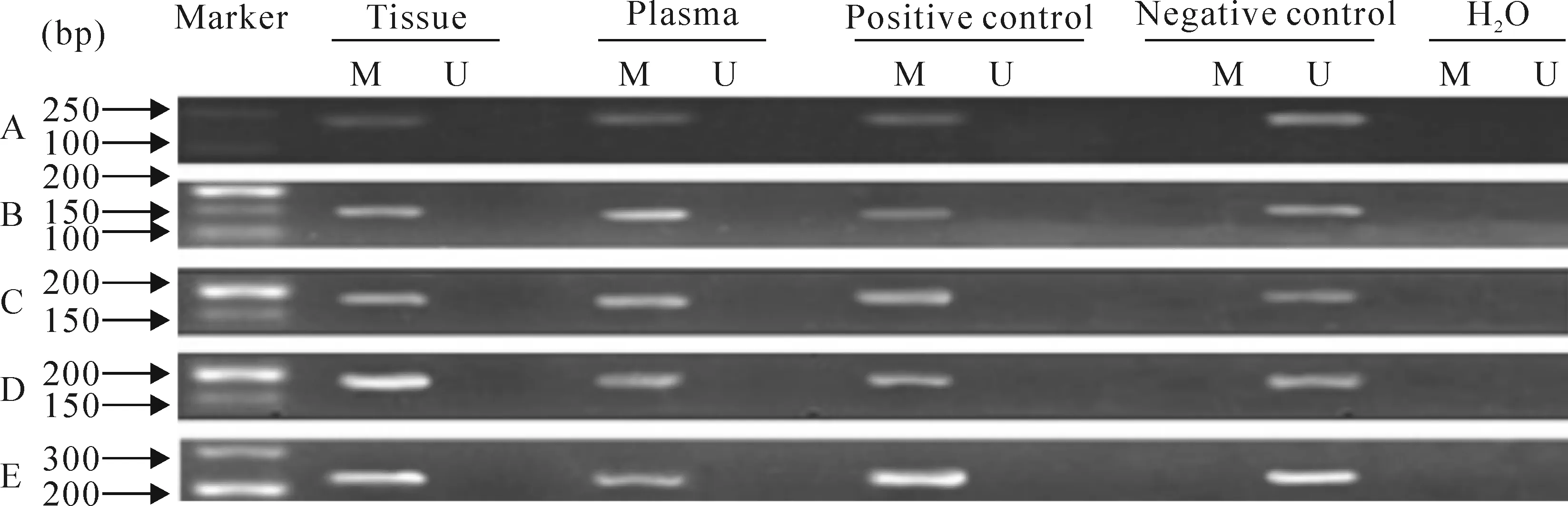
Figure 1.nMSP results ofCDH13 (A),RUNX3 (B),RASSF1A(C),DLEC1 (D),SEPT9 (E).
图1CDH13、RUNX3、RASSF1A、DLEC1、SEPT9 基因的nMSP结果
表2 肺癌和癌旁组织中的基因甲基化情况
Table 2.Methylation frequencies for lung cancer and paracancerous tissues (%.n=106)
GeneCancertissuesParacanceroustissuesCDH1351.9**10.4**DLEC154.7**5.7**RASSF1A44.3**7.6**SEPT936.8**10.4**RUNX324.5*11.3*
TableP<0.05,**P<0.01vsnormal.
表3 肺癌组织与血浆甲基化率的相关性
Table 3.The correlation of methylation detection rates between plasma and the corresponding pathological tissues of the patients with lung cancer
Plasma CancertissuesMUKappavalueCDH13M4720.812△ U849DLEC1M5400.924△△ U448RASSF1AM4130.827△ U656SEPT9M3120.790△ U865RUNX3M1920.755△ U778
△κ>0.75,△△κ>0.90vsplasma.
2 各基因甲基化状态与临床病理特征的关系
本实验发现肺癌组织CDH13甲基化率腺癌组高于其它组,差异有统计学意义(P<0.05);有远处转移的组(M1)其CDH13甲基化率高于没有远处转移的组(M0)(P<0.05)。原发肿瘤大小>T2的患者其RASSF1A甲基化率高于其它组(P<0.05)。DLEC1甲基化率鳞癌组高于其它组,差异有统计学意义(P<0.05);有远处转移的组(M1)其DLEC1甲基化率高于没有远处转移的组(M0)(P<0.05)。各基因甲基化与其它临床病理参数无明显相关(P>0.05),见表4。
3 血浆中多基因联合甲基化检测的灵敏度和特异度
ROC曲线分析显示,DLECI+CDH13+RASSF1A+SEPT9组的诊断准确度和Youden指数最高,分别为82.08%和64.15%。表明本研究中,血浆中DLEC1、CDH13、RASSF1A和SEPT9组合对肺癌的诊断效能明显高于其它组(灵敏度为81.13%,特异度为83.02%),见表5。
肺癌患者的CpG岛甲基化表型 (CpG island methylator phenotype, CIMP)阳性(同一样本存在3个及3个以上基因甲基化)率为57.55%。83.96% 的肺癌血浆样本至少检测到1个基因启动子区甲基化;健康对照组的CIMP阳性率为9.43%;23.58%的健康人血浆样本至少检测到1个基因启动子区甲基化,见表6。

表4 各基因甲基化状态与临床病理特征的相关性
TableP<0.05vsnormal.

表5 血浆中各基因组合甲基化的受试者工作曲线(ROC)分析
表6 肺癌患者和健康人血浆中CpG岛甲基化表型(CIMP)的比较
Table 6.Comparison of CIMP between the plasma of lung cancer patients and normal controls

CIMPLungcancerpatientsNormalcontrols+57.55%(61/106)9.43%(10/106)-42.45%(45/106)90.57%(96/106)
讨 论
肿瘤的发生、发展是一个多因素共同作用的复杂过程。癌症的产生经过2条途径:一是原癌基因的高表达,二是抑癌基因的低表达[6]。后者除与基因突变相关外,主要与表观遗传修饰有关,而DNA甲基化正是表观遗传修饰的主要形式[7]。DNA甲基化在肿瘤发生的早期即出现,在病灶未形成时,肺癌患者的血液及胸腔积液中可发现,且伴随着肿瘤发展的整个过程,DNA甲基化有望成为诊断肺癌的分子标志物。CDH13、RUNX3、DLEC1、RASSF1A、SEPT9基因高甲基化在多种癌症(包括肺癌)均检测到[8-9]。但该5种基因未在同一样本中同时检测过,采用联合检测,可以有效提高标志物的灵敏度和特异度,旨在选出高效能肺癌分子标志物组合。血浆中的游离DNA主要来源于肿瘤细胞本身,本实验评价了循环无细胞DNA(circulating cell-free DNA, cfDNA)与肺癌组织在诊断效能上相关性,证明血浆是一种理想的DNA甲基化检测材料。因为在凝血的过程中白细胞破裂释放的DNA会对cfDNA造成干扰,故在本研究中不使用血清[10]。
MSP是检测基因启动子区甲基化的常用方法,但是其灵敏度低,不适用于游离DNA的甲基化检测[11]。本实验采用 nMSP的方法,先用非特异性外侧引物做1轮扩增;再分别用特异性内侧甲基化引物和非甲基化引物做2轮扩增,能提高检测的灵敏性和特异性。血浆中DNA的含量极少,陈洁等[12]测得正常对照组血浆DNA水平为(14.1±3.8) μg/L,肺癌组血浆DNA水平为15.3 μg/L~1 771.2 μg/L,经过亚硫酸氢盐修饰,模板DNA又会损失一部分。而近年兴起的多重置换扩增技术能通过全基因组扩增解决DNA模板浓度过低的难题[13]。为了保证血浆样本PCR的成功率,我们在血浆基因组硫酸氢盐修饰前都进行全基因扩增,取得了理想的实验效果。
CDH13编码黏附分子,在细胞黏附、细胞侵袭和转移等方面具有重要作用。CDH13表达下调,能降低癌细胞的黏附力,从而促进肿瘤的生长和侵袭转移[14]。本研究发现M1组的甲基化率与M0组比较,差异具有统计学意义,说明CDH13基因甲基化可能是造成肺癌转移的一个重要原因。CDH13基因甲基化还与病理组织类型有关系,我们发现腺癌的甲基化率显著高于其它的病理组织类型。
RASSF1A是常见的抑癌基因,其通过阻断细胞周期蛋白D1的积累,导致蛋白激酶失活,从而阻止细胞分裂周期G1/S的进展,调控细胞周期[15]。本研究采用nMSP法测得血浆RASSF1A甲基化率为41.51%,这与Lee 等[16]用焦磷酸测序法测得的结果44.7%基本一致。但Lee的研究结果显示吸烟患者的RASSF1A甲基化率高于非吸烟患者。而本研究得到的结果与其并不一致,可能与样本数量和来源不同有关。
Levanon等[17]首先于1994年在急性髓性白血病中发现RUNX3基因。RUNX3蛋白是TGF-β信号通路的重要参与者,RUNX3表达下调会限制Smad功能进而影响该细胞信号通路的失调,使细胞增殖受控制,导致肿瘤的发生发展[18]。但该基因在肺癌中研究的并不多,Naoki等[19]用MSP法在NSCLC血浆中测得RUNX3的甲基化率为20%,与本实验结果中19.81%的甲基化率一致。
DLEC1于1999年由Dalgo等[20]首次克隆发现,可能是Ser/Thr蛋白激酶信号通路中某一靶基因。DLEC1在肺癌中的研究很多,DLEC1甲基化是重要的肺癌分子标志物,其甲基化往往引起该基因表达下调[21]。本试验发现DLEC1基因甲基化与病理组织类型、是否转移有关。鳞癌的甲基化率显著高于其它病理组织类型,发生转移的患者DLEC1的甲基化率与未转移的患者相比较,差异具有统计学意义。
Lofton等[22]研究发现SEPT9甲基化是显著的结肠癌标志物,SEPT9基因的表达下调与启动子区域的高度甲基化相关,导致癌细胞增殖和迁移。Powrózek等[23]用实时荧光定量甲基化特异性PCR的方法,测得游离SEPT9作为甲基化标志物在肺癌诊断中的灵敏性为44.3%,特异性为92.0%,说明SEPT9作为抑癌基因对肺癌诊断具有价值。本研究测得血浆游离SEPT9在肺癌诊断中的的灵敏性为31.13%,特异性为90.6%,与前人的结果略有差异,这是由于试验方法的灵敏度和样本来源差异造成的。
本实验在肺癌组织和血浆中CDH13、RASSF1A、DLEC1、SEPT9和RUNX3这5种基因的甲基化状态是基本一致的。且血浆中CDH13、RASSF1A、DLEC1和SEPT9组合的准确度ACC为82.08%,Youden指数为0.6415(灵敏度:79.49%,特异度:81.13%),证明了血浆基因甲基化检测在临床肺癌筛查方面的可行性。而这种检测方法实时微创,能大大减轻患者痛苦。因此,日后的研究重点在于扩大样本量,以提高结果的科学性,并用高通量的手段筛选高灵敏度和特异度的DNA甲基化组合。相信不久的将来,血浆多基因的联合甲基化检测将广泛应用于肺癌的早期诊断中。
[1] Ferlay J, Shin HR, Bray F, et al. Estimates of worldwide burden of cancer in 2008:GLOBOCAN 2008[J]. Int J Cancer, 2010, 12(7):2893-2917.
[2] Santini V, Kantarjian HM, Issa JP. Changes in DNA methylation in neoplasia: pathophysiology and therapeutic implications[J]. Ann Intern Med, 2001,134(7):573-586.
[3] Leon SA, Shapino B, Skaaroff DM, et al. Free DNA in the serum of cancer patients and the effect of therapy[J]. Cancer Res, 1977, 37(3):645-646.
[4] Wroclawski ML, Serpa-Neto A,Fonseca FL, et al. Cell-free plasma DNA as biochemical biomarker for the diagnosis and follow-up of prostate cancer patients[J]. Tumour Biol, 2013, 34(5):2921-2927.
[5] Kim HR, Lee SY, Hyum DS, et al. Detection of EGFR mutations in circulating free DNA by PNA-mediated PCR clamping[J]. Exp Clin Cancer Res, 2013, 32(1):50.
[6] Kwon YJ, Lee SJ, Koh JS, et al. Genome-wide analysis of DNA methylation and the gene expression change in lung cancer[J]. J Thorac Oncol, 2012, 7(1):20-33.
[7] Sharma S, Kelly TK, Jones PA. Epigenetics in cancer[J]. Carcinogenesis, 2010, 31(1):27-36.
[8] Begum S, Brait M, Dasgupta S, et al. An epigenetic marker panel for detection of lung cancer using cell-free serum DNA[J]. Clin Cancer Res, 2011, 17(13):4494-4503.
[9] Ji M, Guan H, Gao C, et al. Highly frequent promoter methylation and PIK3CA amplification in non-small cell lung cancer (NSCLC)[J]. BMC Cancer, 2011, 11:147.
[10]Board RE, Williams VS, Knight L, et al. Isolation and extraction of circulating tumor DNA from patients with small cell lung cancer[J]. Ann N Y Acad Sci, 2008, 1137(8):98-107.
[11]胡灼君, 胡宏波, 刘大鹰, 等.p16基因启动子甲基化联合检测在肺癌早期诊断中的临床意义[J]. 中国病理生理杂志, 2009, 25(10):1941-1945.
[12]陈 洁, 毛雄英, 李春仙, 等. 直接荧光定量法检测肺癌患者血浆游离DNA含量临床意义[J]. 中国卫生检验杂志, 2014,24(6):853-854.
[13]Kalimutho M, Di Cecilia S, Del Vecchio Blanco G, et al. Epigenetically silenced miR-34b/c as a novel faecal-based screening marker for colorectal cancer[J]. Br J Cancer, 2011, 104(11):1770-1778.
[14]Zhang Y, Wang R, Song H, et al. Methylation of multiple genes as a candidate biomarker in non-small cell lung cancer[J]. Cancer Lett, 2011, 303(1): 21-28.
[15]Deng ZH, Wen JF, Li JH, et al. Activator protein-1 involved in growth inhibition byRASSF1Agene in the human gastric carcinoma cell line SGC7901[J]. World J Gastroenterol, 2008, 14(9):1437-1443.
[16]Lee SM, Lee WK, Kim DS, et al. Quantitative promoter hypermethylation analysis of RASSF1A in lung cancer:comparison with methylation-specific PCR technique and clinical significance[J]. Mol Med Rep, 2012, 5(1):239-244.
[17]Levanon D, Negreanu V, Bernstein Y, et al. AML1, AML2, and AML3, the human members of the runt domain gene-family: cDNA structure, expression, and chromosomal localization[J].Genomics,1994,23(2):425-432.
[18]Oki Y, Issa JP. Review: recent clinical trials in epigenetic therapy[J]. Rev Recent Clin Trials, 2006, l (2):169-182.
[19]Yanagawa N, Tamura G, Oizumi H, et al. Promoter hypermethylation of tumor suppressor and tumor-related genes in non-small cell lung cancers[J]. Cancer Sci,2003, 94(7):589-592.
[20]Daigo Y,Nishiwaki T,Kawasoe T,et al.Molecular cloning of a candidate tumor suppressor gene,DLEC1,from chromosome 3p21.3[J]. Cancer Res,1999,59(8):1966-1972.
[21]宋海珠, 易 俊, 张有为, 等. 染色体3p区抑癌基因在非小细胞肺癌中的甲基化状况与临床意义[J]. 中国肺癌杂志, 2011, 3(14):233-238.
[22]Lofton-Day C, Model F, Devos T, et al. DNA methylation biomarkers for blood based colorectal cancer screening [J].Clin Chem, 2008, 54(2):414-423.
[23]Powrózek T, Krawczyk P, Kucharczyk T, et al. Septin 9 promoter region methylation in free circulating DNA-potential role in noninvasive diagnosis of lung cancer: preliminary report[J]. Med Oncol, 2014, 31(4):917-920.
Application of multiple gene methylations in plasma for diagnosis of lung cancer
DING Hao1, SHEN Zhi-gao1, LI Hao1, QIU Yu1, HAO Xiao-ning1, ZU Jin-chi3, ZHONG Li1, 2
(1LaboratoryofBiochipofLifeScience,HebeiUniversity,Baoding071002,China;2WesternUniversityofHealthScience,California91766,USA;3AffiliatedHospitalofHebeiUniversity,Baoding071002,China.E-mail:zhong_leehd@sina.com)
AIM: To determine the aberrant methylation status in the gene promoter regions ofCDH13,RASSF1A,DLEC1,SEPT9 andRUNX3 by detecting the plasma specimens and the value of their combined detection for diagnosis of lung cancers. METHODS: Nest methylation specific PCR (nMSP) was used to detect the promoter methylation status of the 5 genes in the plasma from 106 normal controls, lung cancer tissues, lung benign tissues and the plasma from 106 patients with lung cancers. Multiple displacement amplification (MDA) was used to amplify modified genomic DNA to solve the problem of insufficient of plasma DNA template. RESULTS: The positive rates of promoter methylation ofCDH13,RASSF1A,DLEC1,SEPT9 andRUNX3 in the lung cancer tissues were 51.9%, 44.3%, 54.7%, 36.8%, 24.5%, respectively, and those in the plasma were 46.2%, 41.5%, 50.9%, 31.1%, 19.8%, respectively. The results of the Kappa consistency check showed that the lung cancer tissues and the plasma had obviously coherence in the methylation status of the 5 gene promoter regions. Combination ofDLEC1,CDH13,RASSF1A, andSEPT9 had a higher diagnostic efficiency than the others, as their ACC value was 0.8208 and youden index was 0.6415 (with the sensitivity of 81.13% and the specificity of 83.02%). CONCLUSION: Combination detection of promoter methylation of lung cancer-related genes in the plasma is expected to apply to the early diagnosis of lung cancer.
Lung neoplasms; Plasma; Tissues; Methylation
1000- 4718(2014)12- 2128- 07
2014- 07- 01
2014- 10- 22
国家自然科学基金资助项目(No. 81272444; No. 81472744)
R446.1; R734.2
A
10.3969/j.issn.1000- 4718.2014.12.003
△通讯作者 Tel: 0312-5079365; E-mail: zhong- leehd@sina.com

