羊传染性脓疱病毒湖北分离株CBP二级结构及其细胞表位的预测
黎摄儿,赵苗苗,崔 成,黎满香
(1.湖南农业大学动物医学院,长沙410128;2.甘肃农业大学动物医学院,兰州730070)
羊传染性脓疱病俗称羊口疮(contagious ecthyma),是由羊传染性脓疱病毒(Orf virus,ORFV)引起的以绵羊、山羊为主等多种动物的一种急性、高度接触性皮肤传染病,通过直接接触也可使人感染该病毒。ORFV是痘病毒科、副痘病毒属的代表性成员之一,具有高度的嗜上皮性,感染途径主要是通过损伤的皮肤和黏膜。临床以皮肤形成丘疹、水疱、脓疱和痂皮为特征,如口唇、舌、鼻、乳房以及尾根等部位[1]。该病传染性较强,发病率高,但死亡率低,其中以羔羊等幼畜最为易感[2]。目前,羊传染性脓疮病呈世界性分布,给养羊业带来的经济损失也是不容小觑的。ORFV在免疫学上具有特殊性:初次感染痊愈后它可以再次感染宿主[3]。研究发现,由该病毒编码的几种免疫调节因子可抑制机体的免疫反应从而使宿主发生再感染,羊传染性脓疱病毒编码产生的IL-10、IFNR、VEGF、GIF和CBP等毒力因子和细胞因子主要通过干扰宿主的免疫反应,引起宿主发病,在宿主感染该病毒的致病机制中发挥重要作用[4]。CBP为ORF112的编码产物,在病毒复制的早期表达,能够较强地结合和抑制趋化因子,阻止趋化因子与相应受体相互作用。
为进一步弄清ORFV在宿主体内的感染过程,并能更好的预防该病的发生。本研究以ORFV湖北分离株毒力因子趋化因子结合蛋白(chemokinebinding protein, CBP)为靶抗原,对该毒株的CBP基因进行克隆和表达,运用生物信息学技术预测其二级结构、信号肽、优势的B细胞表位和T细胞表位,了解ORFV CBP基因的分子特性以及其编码蛋白的功能,以及其在宿主机内如何发挥调节作用。
1 材料与方法
1.1 毒株和试剂 ORFV-HB病毒株分离自湖北省某羊场发病山羊,由本实验室分离、保存。LA Taq DNA聚合酶、DNA分子质量标准、DNA提取试剂盒和凝胶回收试剂盒购自大连宝生物工程有限公司。
1.2 羊口疮病毒DNA的提取 从增殖ORFV 的小牛肾(madin-darby bovine kideny, MDBK)细胞病毒液中提取该病毒的DNA。具体方法参照大连宝生物工程有限公司的DNA提取试剂盒说明书进行。
1.3 引物的设计、合成与CBP基因的扩增 根据GenBank中登录的ORFV CBP基因序列设计并合成1对引物,上游引物P1:5'-tgcatagtggacctgcgcaagct-3';下游引物P2:5'-acttccattgttgcggcgttg-3'。引物由上海生工生物工程技术服务有限公司合成。
以ORFV DNA为模板,采用PCR方法扩增CBP全基因,反应条件:94℃预变性2 min;94℃变性30 s,51℃退火45 s,72℃延伸1 min,共30个循环;最后72℃延伸10 min。扩增产物经1%琼脂糖凝胶电泳分析,将纯化回收的PCR产物送大连宝生物工程有限公司测序。使用DNAStar lasergene 7.1程序包的Editseq软件提供的模块将ORFV CBP基因的测序结果转换为氨基酸序列。
1.4 ORFV CBP二级结构的预测 用SOPMA[6]服务器预测ORFV CBP蛋白的二级结构,蛋白二级结构预测网站:http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html。
1.5 ORFV CBP信号肽预测 利用Signal P4.1 Server软件的蛋白质结构神经网络模型(NN)[8],对推导的氨基酸序列进行信号肽预侧。
1.6 ORFV CBPB细胞表位的预测 使用DNAStar lasergene 7.1程序包中的Protean软件提供的模块对ORFV CBP蛋白进行多参数预测,用Kyte-Doolittle法预测蛋白的亲水性;用Emini法预测蛋白的表面可及性;用Karplus-Eisenberg法预测蛋白的柔韧性;用Jameson-Wolf法预测蛋白的抗原指数以及二级结构。通过对ORFV CBP蛋白的二级结构、亲水性指数、柔韧性参数、抗原指数、表面可及性等参数分析,若抗原指数≥1. 031,亲水性指数≥0,氨基酸的抗原表位可及性指数≥1,且其内部或附近又具有柔韧性结构,氨基酸个数≥7,则这一区段为抗原表位的可能性较大。将ORFV CBP蛋白单参数预测结果结合二级结构预测方案进行综合分析。最后将单参数预测综合分析结果采用ABCpred[9]方案进行筛选,并进行验证和评分,确定B细胞抗原优势表位。
1.7 ORFV CBPT细胞表位的预测 应用在线生物信息学网站,分析ORFV CBP T细胞表位。所用生物信息学网站有:细胞毒性T 细胞(CTL)表位预测网站:http://www.imtech.res.in/raghava/ctlpred/;辅助T 细胞(Th)抗原表位的预测网站:http://www.imtech.res.in/raghava/propred/。
2 结果
2.1 ORFV CBP基因的PCR扩增及测序 以增殖ORFV的MDBK细胞上清中提取的该病毒DNA为模板,经PCR扩增,得到一条约843 bp的特异片段,与预期目的片段大小相符(图1)。
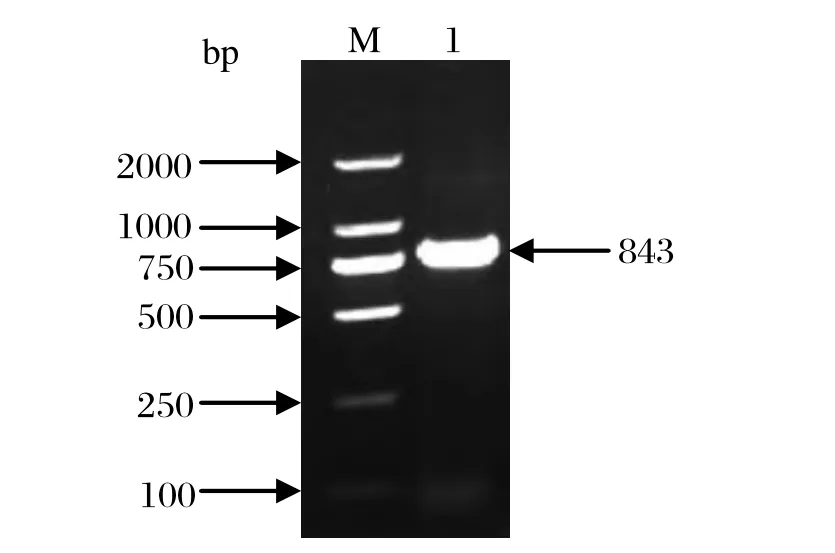
图1 MDBK细胞上清中的ORFV的CBP基因PCR扩增结果Fig.1 PCR amplification of CBP gene from cell cultures of MDBK cell
PCR产物经测序鉴定,确认为ORFV CBP基因。使用DNAStarlasergene 7.1程序包的Editseq软件提供的模块将ORFV CBP基因的测序结果转换为氨基酸序列,发现ORFV CBP蛋白全长共281个氨基酸,序列见图2:

图2 ORFV CBP氨基酸序列Fig.2 ORFV CBP amino acid sequence
2.2 ORFV CBP蛋白二级结构的预测 用SOPMA[6]服务器预测ORFV CBP蛋白的二级结构,结果见图3,ORFV CBP蛋白的二级结构主要包括α-螺旋、β-转角、无规则卷曲和β-片层4种类型:其中α-螺旋占24.64%、β-片层占22.14%、β-转角占3.39%、无规则卷曲占49.29%。整个二级结构中α-螺旋区域和无规则卷曲区域出现得较多,这有利于维持病毒膜蛋白的稳定性。β 转角及无规则卷曲是比较松散的柔性结构,该区域常含有B细胞的优势表位[7]。CBP蛋白的无规则卷曲和β转角即功能区主要位于第15~25、35~38、52~63、69~78、83~84、86~88、96~102、111~117、124~133、139~141、158~214、220~224、241~245、250~255、266~269、275~280位氨基酸(图3)。

图3 ORFV CBP蛋白二级结构的预测Fig.3 Prediction of secondary structure of ORFV CBP protein
2.3 ORFV CBP信号肽的预测 在线软件SignalP4.1的预测结果显示ORFV CBP蛋白没有信号肽(图4)。
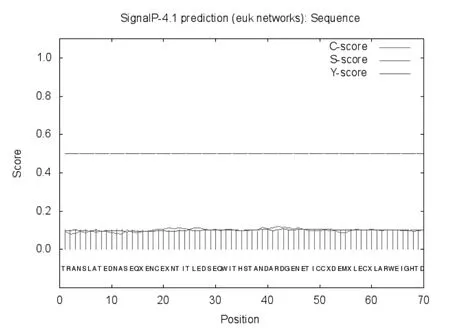
图4 ORFV CBP蛋白信号肽的预测Fig.4 The signal peptide prediction of CBP protein
2.4 ORFV CBPB细胞表位的预测 DNAStar lasergene 7.1程序包中的Protean软件提供的模块对ORFV CBP蛋白进行多参数预测,预测结果见图5。通过对ORF2 蛋白的二级结构、亲水性指数、柔韧性参数、抗原指数、表面可及性等参数分析显示,若抗原指数≥1.031,亲水性指数≥0,氨基酸的抗原表位可及性指数≥1,且其内部或附近又具有柔韧性结构,氨基酸个数≥7,则这一区段为抗原表位的可能性较大。经过筛选,有7个区段可作为B细胞优势抗原表位。将ORFV CBP蛋白单参数预测结果结合二级结构预测方案进行综合分析后发现,同时有3种或以上的参数预测结果显示,表位肽段为19~30、35~42、72~89、131~143、158~175、188~199、203~212、240~247、250~264位氨基酸可能存在B细胞表位,见表1。最后将单参数预测综合分析结果采用ABCpred方案(窗口长度16,阈值0.51)进行筛选,并进行验证和评分,发现ORFV CBP蛋白的第33~48、64~79、128~133、196~211、252~267、80~95、17~32和163~178位等区段可能存在优势B细胞表位,见表2。
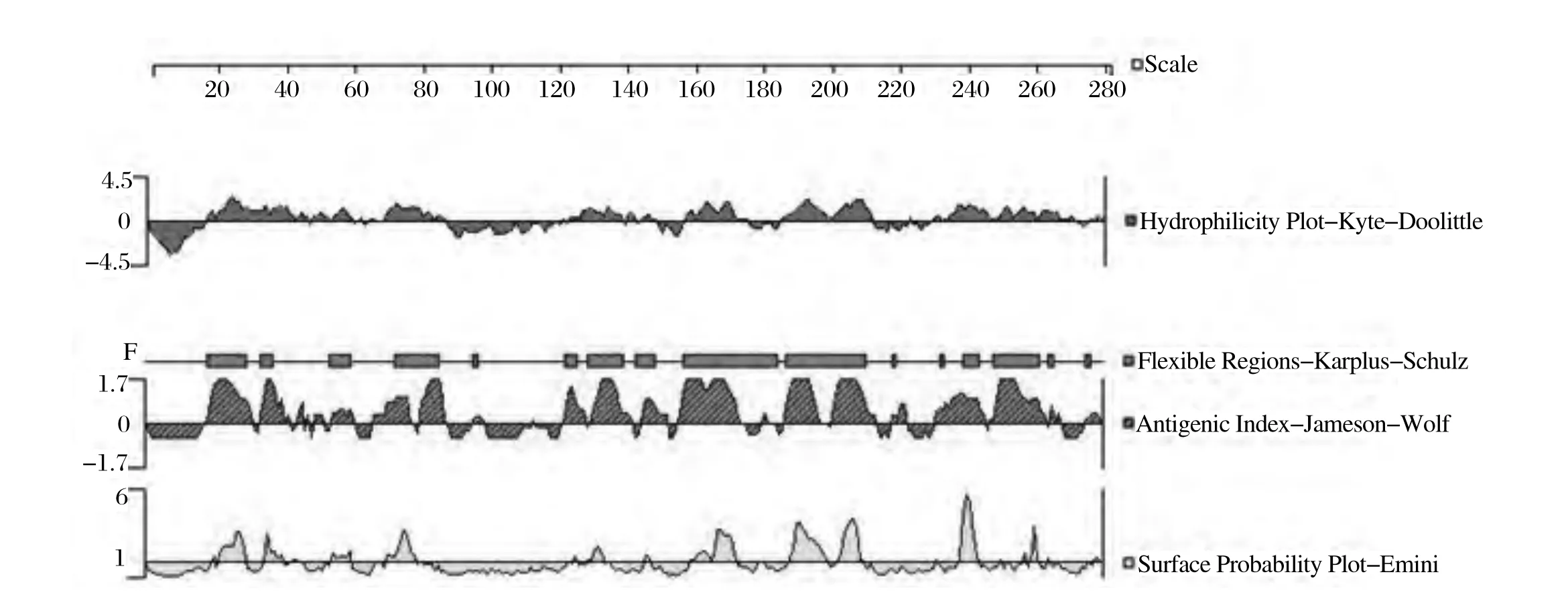
图5 DNAStar软件预测的ORFV CBP细胞表位Fig. 5 Prediction of B cell epitopes of CBP by DNAStar software
2.5 ORFV CBP T细胞表位的预测
2.5.1 ORFV CBP CTL表位的预测 将ORFV CBP蛋白氨基酸序列提交CTL表位预测网站,选择基于人工神经法预测出ORFV CBP蛋白的7段CTL表位,即CSTHQNEVY(33~41位)、YTPSNMCMM(58~66位)、YTSTATGDA(77~85位)、LTDENTDTT(188~196位)、NTDTTPTTT(192~200位)、CVTSVNLHF(223~231位)和CSLNLSPGY(272~280位),见表3。

表1 不同方法预测的ORFV CBP的B细胞表位的肽段位置Table 1 B cell epitopes of ORFV CBP predicted by various methods

表2 ABCpred方案预测初筛肽段的结果Table 2 The score of the peptides obtained by preliminary prediction with ABCpred program

表3 ORFV CBP CTL表位预测Table 3 Prediction for CTL epitopes of ORFV CBP
2.5.2 ORFV CBP蛋白Th表位的预测 在线程序对ORFV CBP蛋白Th表位进行预测,预测出DRB1-0101、DRB1-0102、DRB1-0301 三种不同类型(见表4)。综合分析ORFV CBP的39段3类结合肽,有3段结合肽为3类结合肽等位基因所共有,即VVLLLVLLG(2~10位)、LVLLGALTN(6~14位)和FRLQMRVGV(43~51位),为ORFV CBP蛋白的Th表位。
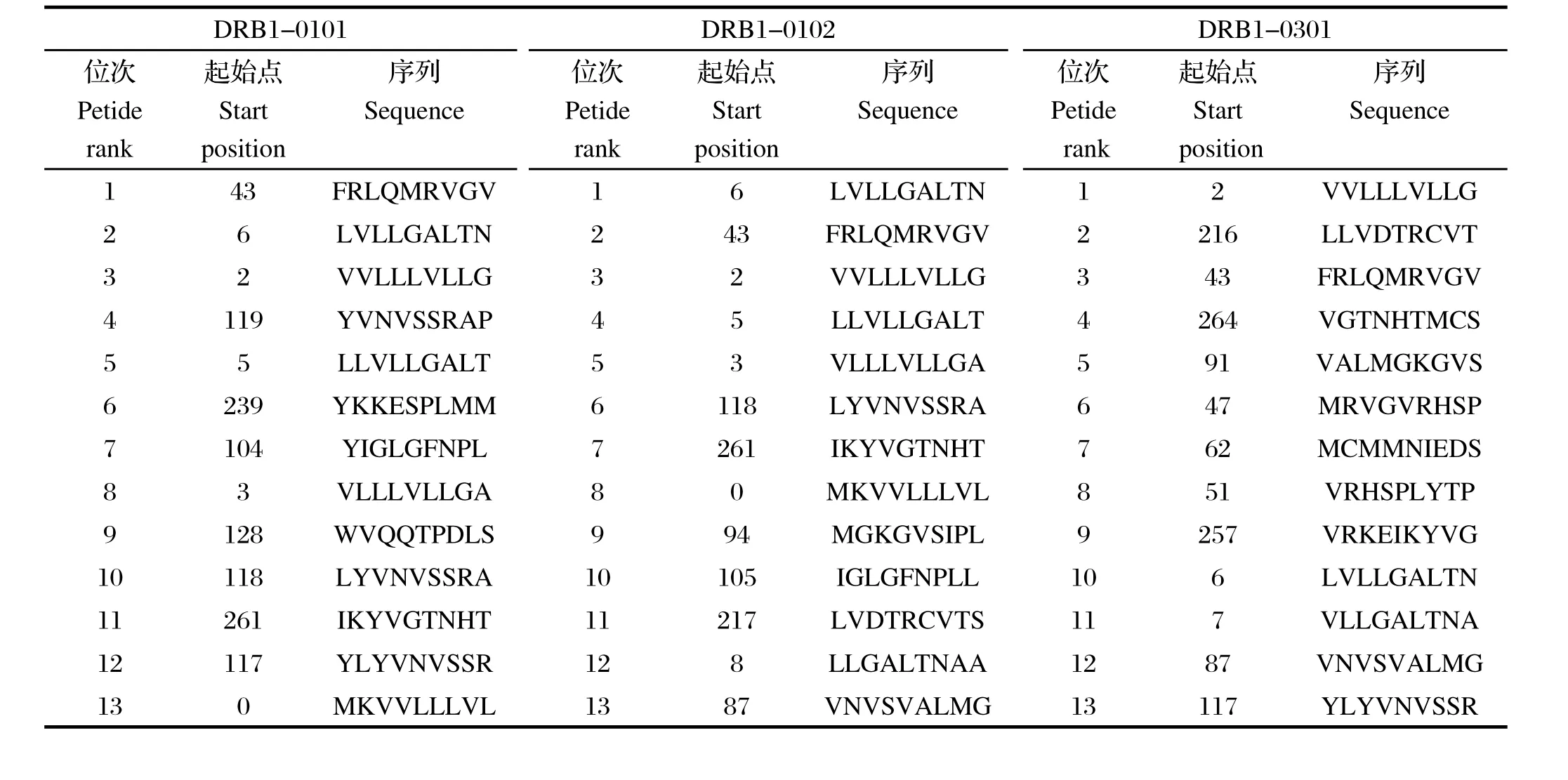
表4 ORFV CBP蛋白Th表位的预测Table 4 Prediction for Th epitopes of ORFV CBP protein
3 讨论
本试验将ORFV湖北分离株的CBP基因编码的氨基酸序列进行了研究分析。分析结果表明,ORFV CBP整个二级结构中α-螺旋区域和无规则卷曲区域出现得较多,这有利于维持病毒蛋白的稳定性。Β-转角及无规则卷曲是比较松散的柔性结构,该区域常含有B细胞的优势表位。蛋白二级结构中的α-螺旋、β折叠的化学键能较高,能较牢固地维持蛋白的高级结构,但很难较好地与抗体嵌合,且经常处于蛋白质内部,因而很少成为抗原表位。但是该蛋白具有较多的β转角和无规卷曲结构单元,这些结构易于扭曲、盘旋并展示在蛋白表面,有利于抗体嵌合,常含有B细胞优势抗原表位[9,10]。
信号肽为分泌蛋白在通过膜时N 末端所含作为信号的氨基酸序列,常由15~25个氨基酸构成,N端含少量的碱性氨基酸,主要含不携带电荷的疏水性氨基酸,在蛋白成熟后信号肽序列将被剪切、去掉。通过预测信号肽可以避免预测的表位落在信号肽区,预测结果显示ORFV CBP蛋白没有信号肽。
当前疫苗学研制新型疫苗的一个研究热点是通过分析病毒的基因组序列来预测病毒的所有抗原信息,然后筛选出合适的候选抗原,进而研制出有效的疫苗,这种研制疫苗的方法策略称为“反向疫苗学”[11]。目前,已有几十种预测B细胞抗原表位的算法,其中亲水性、可及性、柔韧性及抗原性方案的预测效果相对较好[12]。基于人工神经网络算法的ABCpred方案,预测准确率最高可达65.9%[13]。为了减少单参数预测方案的局限性,提高预测的准确性,笔者综合这些方法对ORFV CBP蛋白潜在的B细胞抗原表位进行了预测和分析,推测ORFV CBP蛋白最可能的B细胞优势抗原表位为33~48、64~79、128~133、196~211、252~267、80~95、17~32和163~178。
T 细胞表位由T细胞受体(T cell receptor,TCR)识别,B细胞表位由B细胞受体(B cell receptor,BCR)识别。T细胞表位在细胞内由主要组织相容性复合体(major histocompatibility complex,MHC)I或II分子识别并递呈到细胞表面,然后分别被CD8+细胞毒T细胞的TCR识别和辅助性CD4+T细胞的TCR识别。被CD8+细胞毒T细胞的TCR识别的细胞表位称为细胞毒T细胞(cytotoxic T leukocyge,CTL)表位,后者称为辅助性T细胞(helper T cell,Th)表位。表位疫苗的研究尽管近年来发展迅速,但其应用仍面临一些亟待解决的问题。其中一个问题是:表位研究的不均衡,尤其对于动物病毒。譬如口蹄疫病毒,此前过多的表位研究主要集中于B细胞表位和Th 细胞表位,而缺乏对CTL 细胞表位的研究,导致表位疫苗中表位的组成缺乏必要的CTL 表位。因此,本研究对CTL表位的研究弥补了上述缺陷,使ORFV表位疫苗的理论依据更完善[14]。本研究所测得的CTL表位为33~41、58~66、77~85、188~196、192~200、223~231和272~280。
本研究运用生物信息学网络平台和生物软件分析了ORFV CBP蛋白的二级结构,预测了ORFV CBP蛋白的B细胞表位、CTL抗原表位、Th抗原表位,为宿主如何对抗病毒感染提供新思路,并为进一步探究ORFV的致病机制、免疫研究及表位疫苗的研制提供了理论依据。
[1]Haig D M, Mercer A A. Ovine diseases. Orf[J]. Vet Res,1997, 29(3-4): 311-326.
[2]De La Concha-Bermejillo A, Guo J, Zhang Z, et al.Severe persistent orf in young goats[J]. J Vet Diagn Invest, 2003, 15(5): 423-431.
[3]Haig D M, Thomson J, McInnes C J, et al. A comparison of the anti-inflammatory and immuno-stimulatory activities of orf virus and ovine interleukin-10[J]. Virus Res, 2002, 90(1): 303-316.
[4]Seet B T, McCaughan C A, Handel T M, et al. Analysis of an orf virus chemokine-binding protein: Shifting ligand specificities among a family of poxvirus viroceptors[J].Proc Natl Acad Sci U S A, 2003, 100(25): 15137-15142.
[5]Florea L, Halldorsson B, Kohlbacher O, et al. Epitope prediction algorithms for peptide-based vaccine design[C]//Bioinformatics Conference, 2003. CSB 2003.Proceedings of the 2003 IEEE. IEEE, 2003: 17-26.
[6]Geourjon C, Deleage G. SOPMA: significant improvements in protein secondary structure prediction by consensus prediction from multiple alignments[J].Comput Appl Biosci, 1995, 11(6): 681-684.
[7]Bhasin M, Raghava G P S. Prediction of CTL epitopes using QM, SVM and ANN techniques[J]. Vaccine, 2004,22(23): 3195-3204.
[8]Petersen T N, Brunak S, von Heijne G, et al. SignalP 4.0: discriminating signal peptides from transmembrane regions[J]. Nat Methods, 2011, 8(10): 785-786.
[9]Saha S, Raghava G P S. Prediction of continuous B-cell epitopes in an antigen using recurrent neural network[J].Proteins, 2006, 65(1): 40-48.
[10]Cottone R, B üttner M, Bauer B, et al. Analysis of genomic rearrangement and subsequent gene deletion of the attenuated Orf virus strain D1701[J]. Virus Res, 1998,56(1): 53-67.
[11]Poland G A, Ovsyannikova I G, Johnson K L, et al. The role of mass spectrometry in vaccine development[J].Vaccine, 2001, 19(17): 2692-2700.
[12]刘安定, 杨燕, 李方和, 等. 人宫颈癌基因蛋白 B 细胞表位及其 HLA 限制性细胞毒性 T 细胞表位预测分析[J].第四军医大学学报, 2007, 28(14): 1260-1263.
[13]Sullivan J T, Mercer A A, Fleming S B, et al.Identification and characterization of an orf virus homologue of the vaccinia virus gene encoding the major envelope antigen p37K[J]. Virology, 1994, 202(2): 968-973.
[14]赵德, 冯磊, 张强, 等. 表位疫苗的设计及应用研究进展[J]. 生命科学, 2012, 24(002): 174-180.

