基因及其顺式调控元件在动物表型进化中的作用
孙博渊, 涂剑波, 李英, 杨明耀
四川农业大学动物遗传育种研究所, 成都611130
基因及其顺式调控元件在动物表型进化中的作用
孙博渊, 涂剑波, 李英, 杨明耀
四川农业大学动物遗传育种研究所, 成都611130
顺式调控假说是当前进化发育生物学中重要的理论之一, 该假说认为顺式调控元件的进化是调控外表性状进化的主要遗传机制。然而越来越多的实验结果表明, 仅靠顺式调控假说远不足以解释复杂的进化发育过程, 其他因素也会导致表型的进化, 如:与顺式调控元件相联基因的蛋白序列改变; 基因及染色体组复制; 蛋白结构域与顺式调控元件的灵活性等。文章回顾了近年来顺式调控元件以及与顺式调控元件相联基因的进化发育研究, 探讨了进化发育生物学研究的新方法与新思路。
进化发育生物学; 顺式调控元件; 基因; 外表性状
成年动物外表形态的形成是通过一系列发育基因在胚胎中的表达而实现的,这些基因在时间和空间上精确且有差异的表达, 指导胚胎发育成形态各异的个体。进化发育生物学(Evolutionary developmental biology, 简称evo-devo)是进化生物学、发育生物学以及遗传学相结合的交叉新学科[1,2], 旨在探讨生物进化过程中基因及其调控的变化是怎样通过发育过程而塑造出特殊的形态结构。由于发育基因在时空表达的多样性通常是被编码基因附近的多种多样组织特异性的顺式调控元件(cis-regulatory element, CRE)所调控, 因此通常认为顺式调控元件的进化才是调控外表性状进化的主要遗传机制, 即顺式调控假说[3~5]。
CRE是能够影响与之相联的目的基因表达的一段DNA序列, 包括启动子、增强子、沉默子等。一个CRE能够调控多个基因的表达, 而一个基因也可被多个CREs调控。CRE通过其上的转录因子结合位点(Transcription factor binding site, TFBS)招募转录因子, 从而决定基因在何时、何处表达以及表达量的多少[6,7]。因此, CRE在进化中可以增加或减少蛋白结合位点, 重构转录调控网络, 导致发育相关的多功能基因在表达时发生变化, 从而调控生物的生理功能和外表性状[3,5,8,9]。CRE的研究对于了解基因表达的调控机制具有重要意义。
尽管顺式调控假说认为CRE才是造成物种形态差异的主要原因, 但CRE是如何调控基因表达?尤其是CRE的进化又是如何影响物种形态差异?这些问题仍然尚未解决。本综述回顾了近年来围绕evo-devo的研究, 讨论了在该领域探索中遇到的矛盾及为了解决矛盾而产生的新方法与新思路, 重新思考CRE及其相联基因在evo-devo中的作用。
1 顺式调控元件的进化发育研究
CRE及其在发育功能上的进化一直是evo-devo研究的热点。研究CRE的功能通常是通过调控元件分析(Enhancer assay)来实现的。这是一种将目的CRE与报告基因相连, 在胚胎中观察报告基因在时空的表达模式, 从而研究CRE功能的方法[10,11]。进化发育生物学家通过将多个物种的同源CRE在同一模式生物胚胎中表达, 以研究CRE的进化。随着越来越多物种的基因组被测定, 人们发现各个物种基因组中都含有一些保守的非编码序列, 按其序列同源性分为非编码保守序列(Conserved non-coding elements, CNEs)、超保守元件(Ultra conserved elements, UCEs)以及非保守元件等[12~14]。这些非编码元件有许多是作为CRE来发挥作用的。
1.1 保守元件的功能研究
过去对于一些重要的发育基因及其调控元件的研究表明, 这些基因的编码区非常保守, 与其相邻的调控元件也大多为保守元件, 能够在发育过程中发挥较为保守的功能[15,16]。因此通常认为保守元件能够作为CRE调控基因表达, 并具有相对保守的功能。如Clarke等[17]测试了在脊椎动物与两侧对称动物间均保守的 Bicore1与 Bicore2, 发现人类(Homo sapiens)、斑马鱼(Danio rerio)、海胆(Strongylocentrotus purpuratus)或者蜱(Amblyomma americanum)的 Bicore1/2均可以在斑马鱼胚胎的相同部位表达; Pauls等[18]发现有颚脊椎动物中 Sox21(SRY-box containing gene 21)附近的保守元件能够作为顺式调控元件, 在鸡(Gallus gallus)与斑马鱼中均能调控晶状体的发育。Woolfe等[19]鉴定了超过 1400个在人类与河豚(Takifugu rubripes)中均保守的CNEs, 并选取了sox21、pax6 (paired box 6)、hlxb9 (homeo box HB9)与shh (sonic hedgehog)周围的25个CNEs在斑马鱼上进行调控元件分析, 发现超过 90%的河豚CNEs在斑马鱼胚胎发育中期具有 CRE功能, 且一些CNEs还具有保守的功能。2013年最新的研究发现, 一个保守的CNE能够作为CRE调控雌性黑腹果蝇(Drosophila melanogaster)的色素沉积。这一元件上的细小改变导致了不同种的雌果蝇条纹与色斑的差异[20]。这解释了CNE在调控外表性状中的作用, 以及物种间通过CNE形成外表性状差异的可能的机制。
然而, 也有一些研究发现保守CRE的功能可能并不像其序列那样保守。Punnamoottil等[21]通过调控元件分析, 在斑马鱼胚胎中测试了多个在人类与斑马鱼间保守的Hox4调控元件, 结果却发现一些人类的同源序列虽具有CRE功能, 但它们在斑马鱼胚胎中的表达却不具有组织特异性。Ritter等[22]的实验则更为全面:首先将之前已经在小鼠胚胎中测试过的875个人类CNEs与已经在斑马鱼胚胎中表达过的151个斑马鱼CNEs进行比对, 从中发现了41个人类与斑马鱼同源的 CNEs; 然而, 当将人类同源CNEs在小鼠胚胎中的表达模式(HM)与斑马鱼同源CNEs在斑马鱼胚胎中的表达模式(ZZ)进行比较时,发现只有少数(约30%)同源CNEs能够在斑马鱼及小鼠胚胎中具有保守的活性(表 1, 主要内容来源于Ritter等[22]); 又将41个人类同源CNEs在斑马鱼胚胎中进行了测试(HZ), 发现只有4个hCNEs能够显示出与斑马鱼CNEs相似的表达模式。尽管这种CRE序列的保守性与其功能间的差异可能由于CNE侧翼序列的影响[23], 但也从侧面说明单纯通过传统的序列比对与进化分析来推测CRE功能上的进化可能并不准确。因此, Ritter等[22]又提出通过鉴定CRE上转录因子结合位点的保守性, 能够更为准确地推测CRE的功能。同时, 在果蝇上进行的CRE研究, 也说明高度保守元件中获得或丢失转录因子结合位点会对CRE的功能产生很大的影响[24~26]。
1.2 非保守元件的功能研究
与在CNE中的研究类似, 有关非保守元件的功能进化研究也得到了不同的结果。通常认为在进化上序列越保守的元件, 它们越可能作为CRE而具有一定的功能, 甚至是保守的功能[27]。然而Fisher等[28]在研究人类与斑马鱼的RET(receptor tyrosine kinase)位点的非编码元件时发现, 一些元件尽管没有序列上的保守性, 但仍然可以作为CRE启动相似的表达模式。有意思的是, Fisher与同事也推测这种与序列保守性相矛盾的结果是由于侧翼序列发生改变而CRE上的转录因子结合位点仍然保守导致的。这可能是由于转录因子结合位点往往只有几个保守碱基,当它们埋藏在大段的非保守侧翼序列中时很难通过序列比对进行鉴定。
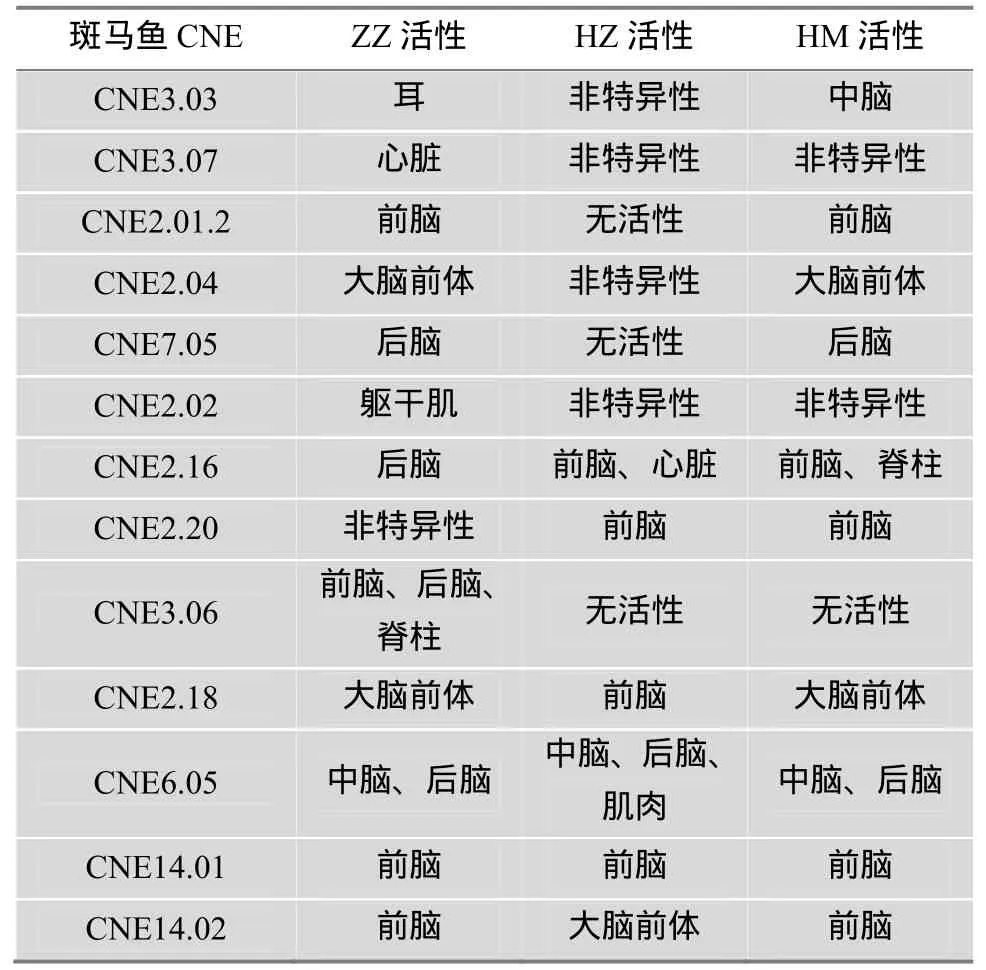
表1 人类与斑马鱼同源CNEs的表达模式比较[22]
Takahashi等[29]于 1999年就发现玻璃海鞘(Ciona intestinalis)与真海鞘(Halocynthia roretzi)中调控brachyury基因表达的CREs不具有序列上的相似性, 却能够产生相似的表达模式。甚至这两个CRE的转录因子结合位点也不相同:C.intestinalis CRE 除了在激活区域具有 SuH结合位点, 还有一个介导负调控的额外区域; 而H.roretzi CRE的激活区域则是一个 T-box结合位点, 并且没有抑制作用的负调控区域。另外, 一项对于球状海胆(Strongylocentrotus purpuratus)中基因 spec调控元件 RSR (Repeat-spacer-repeat)的研究也显示, 尽管RSR在进化过程中获得了4个新的不同的转录因子结合位点,但它们调控的基因表达模式依然十分保守[30]。关于转录因子结合位点保守性与CRE功能间矛盾的研究结果虽然并不多, 但上述研究提示单纯通过比较转录因子结合位点的保守性推测CRE的功能是不完全可靠的。
1.3 顺式调控元件进化研究的新思路
无论从CRE序列本身, 还是从转录因子结合位点入手均无法准确地推测CRE的进化及功能。因此对于CRE进化的研究, 亟需一些新的思路。
首先对于 CRE的定义, 通常认为 CRE大约在100~1000 bp之间[31], 虽然之前有研究发现了富含TFBS与DNaseⅠ超敏位点的大型调控区域, 如IgH增强子(约20 kb)、Th细胞受体(约11.5 kb)、β-球蛋白增强子(约 16 kb)等[32~37], 却没有引起足够的重视。直到2013年, 通过新的算法截取CRE, 并比较转录因子的富集程度发现了超级增强子(Superenhancers), 才重新定义了人们对于 CRE大小的认识。几乎所有有关细胞特性的重要基因都需要超级增强子调控。超级增强子在大小上远大于常规的CRE, 通常为几十 kb, 同时超级增强子上富集了远多于常规调控元件的重要转录因子。超级增强子还与癌症的发生密切相关[38,39]。如果超级增强子确实对细胞的特性具有重要作用, 那么超级增强子的进化必然会影响物种的形态发育。这也许能够为之前CRE进化研究的矛盾提供一个合理的解释与可行的研究方向。关于CRE的进化研究总是局限于常规的CRE大小, 而遗传工具包基因恰恰有可能受到的是更大范围的超级增强子的调控, 那么仅仅对于小范围CRE的序列变化进行研究显然是不准确的, 因为一个超级增强子可能包含多个之前认为的CNE。如果能够对更大范围内的超级增强子进行进化分析,或许能够获得更准确的结果。
其次对于 CRE的鉴定, 过去认为, 当一个细胞完全分化后, 其中维持其细胞特性的CRE将会完全暴露, 以维持该种细胞的特性。然而Ostuni等[40]在2013年发现已分化的巨噬细胞能够对不同的外界刺激产生反应, 使得一些本没有增强子活性的“隐蔽的增强子”(Latent enhancer)暴露在染色体上, 从而使巨噬细胞分化为各种亚型以面对不同的环境。这一发现不仅说明了在进行 ChIP-seq[41~43]与DNaseⅠ超敏位点[44~47]对 CREs进行鉴定时需要考虑不同刺激对于CREs鉴定的影响, 同时也为evo-devo的研究提供了新的方向。众所周知, 胚胎的发育会受到外界环境的影响[48,49], 然而胚胎应答外界刺激的机制并不清楚。将一个物种的CRE在另一个生殖环境迥异的物种胚胎中进行表达, 很有可能会导致其活化位点发生改变, 从而产生矛盾的表达模式。隐蔽增强子的发现为胚胎应答外界刺激提供了一种新的可能性, 有待该领域的学者进行更深入的研究。
最后是CRE进化标记的选择。传统的进化学研究总是以DNA或蛋白质序列的改变为标记, 进而研究其结构和功能的变化。然而现在CRE的鉴定是取决于组蛋白上特定的修饰, 因此决定CRE功能的因素除了DNA序列外, 很大程度取决于其上的组蛋白修饰。通过比较人类、恒河猴(Macaca mulatta)以及小鼠(Mus musculus)中影响胚胎四肢发育的CRE组蛋白 H3K27Ac水平发现, 人类与恒河猴相比, 有13%的启动子和 11%的增强子获得了活性, 这些增加的CRE可能是影响人类四肢进化的重要因素[50,51]。这说明组蛋白修饰与表观遗传对于CRE功能的进化起着关键性的作用。
2 与顺式调控元件相联基因的进化
影响形态发育的因素除了CRE的改变以外, 还包括诸多的方面, 例如编码区的突变、基因乃至染色体组的复制等, 这些变化都可能对形态多样性产生影响[3,52]。但是, 由于顺式调控假说的提出, 大多数进化发育学研究都集中在CRE的进化上。这些研究不但证明了 CRE在形态差异上不可或缺的作用,如scute基因上的DCE增强子的进化导致了不同果蝇后背中部粗刚毛数量的差异[53]、yellow基因上的调控元件变化使得雄性 biarmipes果蝇翅膀上带有斑点[54], 而且在一定程度上解释了CRE进化的机制,如三刺鱼(Gasterosteus aculeatus)背部的棘刺是为了防御大型捕食者, 而这种棘刺的发育需要大量的钙质。淡水三刺鱼背部棘刺数量比咸水三刺鱼有所减少是由于 pitx1(paired-like homeodomain transcription factor 1)邻近的CRE改变引起的[55,56], 这种进化是由于最近一次冰川消退(约 1~2万年前)这些鱼的祖先有一部分留在了当时形成的湖泊中, 随着食物中钙质以及大型捕食者的减少所产生的适应性进化[55,57~59]。但是随着进化发育生物学的发展, 越来越多的研究结果说明形态上的进化是一个更为复杂的过程。
2.1 蛋白编码区的进化
在顺式调控假说出现之前, 人们通常认为蛋白质序列的改变是导致形态多样性以及进化的主要原因, 这些改变能够使蛋白质结构发生变化, 进而产生新的功能[60~62]。然而随后人们发现, 即使经过了10亿年的进化, 许多动物的蛋白序列却进化的十分缓慢, 依然行使着保守的功能。最为著名的两个实验是:调控小鼠眼发育的 Pax-6能够在果蝇中调控昆虫复眼的发育, 与果蝇自身的 Pax-6功能一致[63];水螅中的 achaete-scute同源基因能够诱导果蝇神经系统的发育[64]。基于顺式调控假说的evo-devo认为这种极端的功能保守可能是由于这些蛋白在进化上受到了其功能上的约束。由于蛋白质通常具有多效性, 蛋白质序列的改变常常会影响到多条通路, 因此为了维持某些重要的功能, 蛋白质序列在进化上受到了极大的约束。而顺式调控元件只在蛋白质的时空及表达量水平上进行调控, 受到的选择约束较小, 更可能发生突变, 进而促使进化[2,3]。
但是不少研究却发现, 蛋白质序列的改变在进化与形态多样性上依然扮演着十分重要的角色。灵长类苦味受体TAS2R16(taste receptor type 2 member 16)上的突变, 改变了灵长类对于苦味化学物质的敏感程度, 进而影响了食性[65]; Hemoglobin-B与Duffy抗原基因的突变能够分别抵抗由 P. falciparum 与 P. vivax疟原虫引起的疟疾[66]; 乳糖酶上的突变能够使一些成年人消化乳制品中的乳糖[67]。这些被称为“生理性”[4]的变化, 将最终引起解剖学上的协同进化[68]。现代人类EDAR(Ectodysplasin A receptor)具有遗传多样性[69,70], EDARV370A(EDAR variant 370A)是产生较粗头发与铲型门牙等亚洲人特征的重要因素[71,72], EDARV370A 可能受到了寒冷干燥环境的选择[73]。因此, 在2013年科研人员通过生物信息学手段发现 EDARV370A起源于中国中部, 并将EDARV370A在小鼠模型中表达。研究结果表明, 表达EDARV370A的转基因小鼠显示出强烈的中国汉族人特征, 包括较粗的毛发以及更发达的乳腺与外泌汗腺[74]。这个结果强烈的证明了蛋白质序列受到自然选择而发生进化, 改变了生物的表型。同时也说明尽管 evo-devo认为 CREs在进化上更具优势,但蛋白质序列受到的自然选择还是远远超出了人们的预计。
2.2 工具包基因的复制
自20世纪70年代Ohno提出了进化上的基因复制假说以来, 基因复制在进化上的地位就一直饱受争议。Ohno认为基因复制是物种进化的重要因素,为新基因的出现乃至进化的发生提供了不可或缺的材料。他认为基因复制后的旁系同源基因将会在随后的进化过程中逐渐获得新的功能, 从而使物种进化[75]。而Carroll[2,3,5]则认为基因复制所产生的旁系同源基因总是具有相似的功能。同时, 在一些重要的遗传工具包(Genetic toolkit)基因的进化过程中,工具包中的旁系同源基因也极少获得新功能。因此,在发育过程中新功能与新形态的产生主要依赖于顺式调控元件的进化。如Hox同源框(homeobox), 与原始的 Hox家族相比, 在节肢动物与脊索动物中均没有基因复制事件, 甚至还有不同程度基因丢失(图1),显示基因复制对于新功能的产生与形态的多样性并不是必需的。
尽管工具包基因复制在形态进化上的作用仍有争议, 但是基因复制的另一种形式——染色体组复制, 随着越来越多物种的基因组被测定, 现在被认为对于脊椎动物的起源具有关键性的作用[76~80]。Ohno认为在脊椎动物出现早期, 染色体组曾经经历了两次完整的复制过程, 为脊椎动物的出现提供了重要的物质基础, 被称为2R假说(2 rounds of whole genome duplication, 2R WGD)[75,81]。仍以Hox同源框为例, 节肢动物只有一个 Hox基因座, 而经过两次全染色体组复制之后, 脊索动物的 Hox基因座增加到4个(图1)。最近, 对于这些旁系同源Hox的研究则说明这种基因复制的重要性。Soshnikova等[82]采用全基因座敲除的方法, 分别在转基因小鼠上敲除了 HoxA与 HoxB基因座, 发现尽管旁系同源的Hox基因在表达上有很多相似的地方, 但是如果完全敲除整个旁系同源基因座, 则会对胚胎的发育产生很大的影响。而Goode等[23]的研究将CNE与WGD联系在一起, 发现由WGD得到的大量CNE通过其侧翼序列的改变而获得了新的功能, 继而使复制的旁系同源基因得以在进化中保留。这说明在脊椎动物的起源上, 染色体组的大规模复制对于形成脊椎动物特有的形态特征具有关键性的作用。
2.3 蛋白结构域与顺式调控元件的灵活性
虽然 2R假说证明了基因复制在进化中的重要地位, 为新功能的产生提供了充足的遗传基础[75]。但是它无法解释众多由基因复制产生的同源基因是如何进化, 并在进化中获得新功能而免于被淘汰的。最近, 有关节肢动物fushi tarazu(ftz)基因的研究为同源基因的进化机制提供了证据。节肢动物中ftz作为 Hox基因家族一员调控昆虫的体节发育, 它的表达模式与功能和相邻的Hox基因Antp、Scr重叠[83]。因此ftz中的Hox同源结构域对于它发挥专属功能是不必要的, 这种冗余的功能并不能使它避免被淘汰。但是它在中枢神经系统(Central nervous system, CNS)中的表达却十分的特殊且保守[84]。在随后的研究中发现ftz在CNS发育上的功能依赖于其蛋白上的 Antennapedia(Antp)同源结构域, 敲除 Antp蛋白结构域将导致 CNS发育缺陷, 同时表达全长Antennapedia蛋白无法挽救ftz缺失带来的CNS发育缺陷。由于蛋白结构域与CRE的灵活性, ftz基因获得了一个原神经性的CRE从而获得了独有的功能而在进化中保留了下来[85]。这说明蛋白结构域与CRE是以一种灵活的方式相互组合, 从而在进化中使得原有的蛋白不断获得新的功能。因此单纯研究蛋白序列或CRE对于形态差异的影响无疑是片面的。

图1 Hox家族在节肢动物与脊索动物中演化[3]A:节肢动物Hox基因簇的演化; B:脊索动物Hox基因簇的演化。
无独有偶, 蛋白质结构域与CRE间的灵活组合不但能够使相似的基因获得新的功能, 还可以使分化差异较大的基因保持原始的功能。通过对比抑制果蝇足发育的Hox基因Ultrabithorax(Ubx)、AbdominalA (AbdA)、AbdominalB(AbdB)的功能发现, 这3种Hox基因是通过不同的 DNA结合结构域以及与不同的TALE辅因子结合来行使相同的功能[86]。
然而, 即使是蛋白结构域相似、CRE的表达模式也相似的同一基因在不同物种中也可能发挥不同的作用。在昆虫中Antp基因的表达能够促使胸部足的发育[87~90], 相反Ubx、AbdA/B在腹部的表达会抑制腹部形成足[91]。而 Khadjeh等[92]发现在蜘蛛(Achaearanea tepidariorum)中 Antp却在胸部表达,抑制足的形成。抑制 Antp的表达使蜘蛛形成了 10只脚的极端形态。而将蜘蛛的Antp在果蝇中进行表达, 却发现蜘蛛的Antp在果蝇的胸部依然行使促进足发育的角色。这说明既不是蛋白质序列的改变,也不是 CRE时空表达模式的改变导致了 Antp在Achaearanea tepidariorum中的特殊作用。有意思的是, 尽管Antp在Achaearanea tepidariorum中行使不同的功能, 这种蜘蛛在其形态发育上与其他蜘蛛以及昆虫比较, 腿的发育依然被限制在胸部, 显示了趋同性。这种由不同机制导致的进化趋同性说明了形态发育上的复杂性, 所有参与这一发育网络的辅因子、Hox基因的协作基因或靶基因的进化[92]、甚至蛋白质修饰的改变[93], 都有可能对形态发育产生影响。
3 结语与展望
顺式调控假说的提出使evo-devo得到了迅速的发展, 使人们了解了曾经被称为“垃圾DNA”的无数非编码元件对于物种形态多样性的贡献, 同时也丰富了进化学的理论。但是随着 evo-devo的发展,得到了越来越多不同的研究结果。为了解释这些结果, 必须不断地完善 evo-devo的研究方法与研究思路。未来的evo-devo不应仅局限在CRE的研究, 而应更加全面地研究形态发育网络中的每一个因素,才能更加接近进化发育的真相。
[1] Brakefield PM. Evo–devo and accounting for Darwin's endless forms. Phil Trans R Soc, 2011, 366(1574): 2069–2075.
[2] Carroll SB. Endless forms most beautiful: the new science of evo devo and the making of the animal kingdom. New York: Norton & Company, 2005.
[3] Carroll SB. Evo–devo and an expanding evolutionary synthesis: a genetic theory of morphological evolution. Cell, 2008, 134(1): 25–36.
[4] Ioannidis S. Regulatory Evolution and theoretical arguments in evolutionary biology. Sci Educ, 2013, 22(2): 279–292.
[5] Carroll SB. Endless forms: the evolution of gene regulation and morphological diversity//Philosophy of Biology: An Anthology. Wiley, John & Sons, 2009, 193.
[6] Wittkopp PJ, Kalay G. Cis–regulatory elements: molecular mechanisms and evolutionary processes underlying divergence. Nat Rev Genet, 2012, 13(1): 59–69.
[7] Yanez–Cuna JO, Kvon EZ, Stark A. Deciphering the transcriptional cis–regulatory code. Trends Genet, 2013, 29(1): 11–22.
[8] Simpson P, Ayyar S. Evolution of cis–regulatory sequences in Drosophila. Adv Genet, 2008, 61: 67–106.
[9] Bulger M, Groudine M. Enhancers: The abundance and function of regulatory sequences beyond promoters. Dev Biol, 2010, 339(2): 250–257.
[10] Brasier AR, Tate JE, Habener JF. Optimized use of the firefly luciferase assay as a reporter gene in mammalian cell lines. BioTechniques, 1989, 7(10): 1116–1122.
[11] Haeussler M, Joly JS. When needles look like hay: How to find tissue–specific enhancers in model organism genomes. Dev Biol, 2011, 350(2): 239–254.
[12] Sandelin A, Bailey P, Bruce S, Engström PG, Klos JM, Wasserman WW, Ericson J, Lenhard B. Arrays of ultraconserved non–coding regions span the loci of key developmental genes in vertebrate genomes. BMC Genomics, 2004, 5(1): 99.
[13] Woolfe A, Goodson M, Goode DK, Snell P, Mcewen GK, Vavouri T, Smith SF, North P, Callaway H, Kelly K, Walter K, Abnizova I, Gilks W, Edwards YJ, Cooke JE, Elgar G.. Highly conserved non–coding sequences are associated with vertebrate development. PLoS Biol, 2004, 3(1): E7.
[14] Bejerano G, Pheasant M, Makunin I, Stephen S, Kent WJ, Mattick JS, Haussler D. Ultraconserved elements in the human genome. Science, 2004, 304(5675): 1321–1325.
[15] Elgar G. Pan–vertebrate conserved non–coding sequences associated with developmental regulation. Brief Func Genomic Proteomic, 2009, 8(4): 256–265.
[16] Vavouri T, Lehner B. Conserved noncoding elements and the evolution of animal body plans. Bioessays, 2009, 31(7): 727–735.
[17] Clarke SL, Vandermeer JE, Wenger AM, Schaar BT, Ahituv N, Bejerano G. Human developmental enhancers conserved between deuterostomes and protostomes. PLoS Genet, 2012, 8(8): E1002852.
[18] Pauls S, Smith SF, Elgar G. Lens development depends on a pair of highly conserved Sox21 regulatory elements. Dev Biol, 2012, 365(1): 310–318.
[19] Woolfe A, Goodson M, Goode DK, Snell P, McEwen GK, Vavouri T, Smith SF, North P, Callaway H, Kelly K, Walter K, Abnizova I, Gilks W, Edwards YJK, Cooke JE, Elgar G. Highly conserved non–coding sequences are associated with vertebrate development. PLoS Biol, 2005,3(1): E7.
[20] Rogers WA, Salomone JR, Tacy DJ, Camino EM, Davis KA, Rebeiz M, Williams TM. Recurrent modification of a conserved cis–regulatory element underlies fruit fly pigmentation diversity. PLoS Genet, 2013, 9(8): E1003740.
[21] Punnamoottil B, Herrmann C, Pascual–Anaya J, D'aniello S, Garcia–Fernandez J, Akalin A, Becker TS, Rinkwitz S. Cis–regulatory characterization of sequence conservation surrounding the Hox4 genes. Dev Biol, 2010, 340(2): 269–282.
[22] Ritter DI, Li Q, Kostka D, Pollard KS, Guo S, Chuang JH. The importance of being cis: evolution of orthologous fish and mammalian enhancer activity. Mol Biol Evol, 2010, 27(10): 2322–2332.
[23] Goode DK, Callaway HA, Cerda GA, Lewis KE, Elgar G. Minor change, major difference: divergent functions of highly conserved cis–regulatory elements subsequent to whole genome duplication events. Development, 2011, 138(5): 879–884.
[24] Berman BP, Pfeiffer BD, Laverty TR, Salzberg SL, Rubin GM, Eisen MB, Celniker SE. Computational identification of developmental enhancers: conservation and function of transcription factor binding–site clusters in Drosophila melanogaster and Drosophila pseudoobscura. Genome Biol, 2004, 5(9): R61.
[25] Ludwig MZ, Bergman C, Patel NH, Kreitman M. Evidence for stabilizing selection in a eukaryotic enhancer element. Nature, 2000, 403(6769): 564–567.
[26] Ludwig MZ, Palsson A, Alekseeva E, Bergman CM, Nathan J, Kreitman M. Functional evolution of a cis–regulatory module. PLoS Biol, 2005, 3(4): E93.
[27] Boffelli D, Nobrega MA, Rubin EM. Comparative genomics at the vertebrate extremes. Nat Rev Genet, 2004, 5(6): 456–465.
[28] Fisher S, Grice EA, Vinton RM, Bessling SL, Mccallion AS. Conservation of RET regulatory function from human to zebrafish without sequence similarity. Science, 2006, 312(5771): 276–279.
[29] Takahashi H, Mitani Y, Satoh G, Satoh N. Evolutionary alterations of the minimal promoter for notochord–specific Brachyury expression in ascidian embryos. Development, 1999, 126(17): 3725–3734.
[30] Dayal S, Kiyama T, Villinski JT, Zhang N, Liang SG, Klein WH. Creation of cis–regulatory elements during sea urchin evolution by co–option and optimization of a repetitive sequence adjacent to the spec2a gene. Dev Biol, 2004, 273(2): 436–453.
[31] Davidson EH. The regulatory genome: gene regulatory networks in development and evolution. Access Online via Elsevier, 2010.
[32] Diaz P, Cado D, Winoto A. A locus control region in the T cell receptor alpha/delta locus. Immunity, 1994, 1(3): 207–217.
[33] Forrester WC, Epner E, Driscoll MC, Enver T, Brice M, Papayannopoulou T, Groudine M. A deletion of the human beta–globin locus activation region causes a major alteration in chromatin structure and replication across the entire beta–globin locus. Genes Dev, 1990, 4(10): 1637–1649.
[34] Grosveld F, Van Assendelft GB, Greaves DR, Kollias G. Position–independent, high–level expression of the human beta–globin gene in transgenic mice. Cell, 1987, 51(6): 975–985.
[35] Madisen L, Groudine M. Identification of a locus control region in the immunoglobulin heavy–chain locus that deregulates c–myc expression in plasmacytoma and Burkitt's lymphoma cells. Genes Dev, 1994, 8(18): 2212–2226.
[36] Michaelson JS, Giannini SL, Birshtein BK. Identification of 3α–hs4, a novel Ig heavy chain enhancer element regulated at multiple stages of B cell differentiation. Nucleic Acids Res, 1995, 23(6): 975–981.
[37] Orkin SH. Globin gene regulation and switching: circa 1990. Cell, 1990, 63(4): 665–672.
[38] Whyte WA, Orlando DA, Hnisz D, Abraham BJ, Lin CY, Kagey MH, Rahl PB, Lee TI, Young RA. Master transcription factors and mediator establish super–enhancers at key cell identity genes. Cell, 2013, 153(2): 307–319.
[39] Loven J, Hoke HA, Lin CY, Lau A, Orlando DA, Vakoc CR, Bradner JE, Lee TI, Young RA. Selective inhibition of tumor oncogenes by disruption of super–enhancers. Cell, 2013, 153(2): 320–334.
[40] Ostuni R, Piccolo V, Barozzi I, Polletti S, Termanini A, Bonifacio S, Curina A, Prosperini E, Ghisletti S, Natoli G. Latent enhancers activated by stimulation in differentiated cells. Cell, 2013, 152(1–2): 157–171.
[41] Heintzman ND, Stuart RK, Hon G, Fu Y, Ching CW, Hawkins RD, Barrera LO, Van Calcar S, Qu CX, Ching KA, Wang W, Weng ZP, Green RD, Crawford GE, Ren B. Distinct and predictive chromatin signatures of transcriptional promoters and enhancers in the human genome. Nat Genet, 2007, 39(3): 311–318.
[42] Heintzman ND, Hon GC, Hawkins RD, Kheradpour P, Stark A, Harp LF, Ye Z, Lee LK, Stuart RK, Ching CW, Ching KA, Antosiewicz-Bourget JE, Liu H, Zhang XM, Green RD, Lobanenkov VV, Stewart R, Thomson J A, Crawford GE, Kellis M, Ren B. Histone modifications at human enhancers reflect global cell–type–specific gene expression. Nature, 2009, 459(7243): 108–112.
[43] Creyghton MP, Cheng AW, Welstead GG, Kooistra T, Carey BW, Steine EJ, Hanna J, Lodato MA, Frampton GM, Sharp PA, Boyere LA, Young RA, Jaenisch RA. Histone H3K27ac separates active from poised enhancers and predicts developmental state. Proc Natl Acad Sci USA, 2010, 107(50): 21931–21936.
[44] Boyle AP, Davis S, Shulha HP, Meltzer P, Margulies EH, Weng ZP, Furey TS, Crawford GE. High–resolution mapping and characterization of open chromatin across the genome. Cell, 2008, 132(2): 311–322.
[45] Neph S, Vierstra J, Stergachis AB, Reynolds AP, Haugen E, Vernot B, Thurman RE, John S, Sandstrom R, Johnson AK, Maurano MT, Humbert R, Rynes E, Wang H, Vong S, Lee K, Bates D, Diegel M, Roach V, Dunn D, Neri J, Schafer A, Hansen RS, Kutyavin T, Giste E, Weaver M, Canfield T, Sabo P, Zhang M, Balasundaram G, Byron R, Maccoss MJ, Akey JM, Bender MA, Groudine M, Kaul R, Stamatoyannopoulos JA. An expansive human regulatory lexicon encoded in transcription factor footprints. Nature, 2012, 489(7414): 83–90.
[46] Song LY, Zhang ZC, Grasfeder LL, Boyle AP, Giresi PG, Lee BK, Sheffield NC, Graf S, Huss M, Keefe D, Liu Z, London D, Mcdaniell RM, Shibata Y, Showers KA, Simon JM, Vales T, Wang TY, Winter D, Zhang ZZ, Clarke ND, Birney E, Iyer VR, Crawford GE, Lieb JD, Furey TS. Open chromatin defined by DNaseI and FAIRE identifies regulatory elements that shape cell–type identity. Genome Res, 2011, 21(10): 1757–1767.
[47] Thurman RE, Rynes E, Humbert R, Vierstra J, Maurano MT, Haugen E, Sheffield NC, Stergachis AB, Wang H, Vernot B, Garg K, John S, Sandstrom R, Bates D, Boatman L, Canfield TK, Diegel M, Dunn D, Ebersol AK, Frum T, Giste E, Johnson AK, Johnson EM, Kutyavin T, Lajoie B, Lee BK, Lee K, London D, Lotakis D, Neph S, Neri F, Nguyen ED, Qu HZ, Reynolds AP, Roach V, Safi A, Sanchez ME, Sanyal A, Shafer A, Simon JM, Song LY, Vong S, Weaver M, Yan YQ, Zhang ZC, Zhang ZZ, Lenhard B, Tewari M, Dorschner MO, Hansen RS, Navas PA, Stamatoyannopoulos G, Iyer VR, Lieb JD, Sunyaev SR, Akey JM, Sabo PJ, Kaul R, Furey TS, Dekker J, Crawford GE, Stamatoyannopoulos JA. The accessible chromatin landscape of the human genome. Nature, 2012, 489(7414): 75–82.
[48] Paranjpe DA, Bastiaans E, Patten A, Cooper RD, Sinervo B. Evidence of maternal effects on temperature preference in side–blotched lizards: implications for evolutionary response to climate change. Ecol Evol, 2013, 3(7): 1977–1991.
[49] Rees S, Harding R. Brain development during fetal life: influences of the intra–uterine environment. Neurosci Lett, 2004, 361(1–3): 111–114.
[50] Cotney J, Leng J, Yin J, Reilly SK, Demare LE, Emera D, Ayoub AE, Rakic P, Noonan JP. The evolution of lineage–specific regulatory activities in the human embryonic limb. Cell, 2013, 154(1): 185–196.
[51] Flicek P. Evolutionary biology: The handiwork of tinkering. Nature, 2013, 500(7461): 158–159.
[52] Heffer A, Pick L. Conservation and variation in Hox genes: how insect models pioneered the evo–devo field. Annu Rev Entomol, 2013, 58: 161–179.
[53] Marcellini S, Simpson P. Two or four bristles: functional evolution of an enhancer of scute in Drosophilidae. PLoS Biol, 2006, 4(12): E386.
[54] Gompel N, Prud'homme B, Wittkopp PJ, Kassner VA, Carroll SB. Chance caught on the wing: cis–regulatory evolution and the origin of pigment patterns in Drosophila. Nature, 2005, 433(7025): 481–487.
[55] Shapiro MD, Marks ME, Peichel CL, Blackman BK, Nereng KS, Jónsson B, Schluter D, Kingsley DM. Genetic and developmental basis of evolutionary pelvic reduction in threespine sticklebacks. Nature, 2004, 428(6984): 717–723.
[56] Peichel CL, Nereng KS, Ohgi KA, Cole BLE, Colosimo PF, Buerkle CA, Schluter D, Kingsley DM. The genetic architecture of divergence between threespine stickleback species. Nature, 2001, 414(6866): 901–905.
[57] Lauder GV. The evolutionary biology of the threespine stickleback. Science, 1995, 267:1192.
[58] Cresko WA, Amores A, Wilson C, Murphy J, Currey M, Phillips P, Bell MA, Kimmel CB, Postlethwait JH. Parallel genetic basis for repeated evolution of armor loss in Alaskan threespine stickleback populations. Proc Natl Acad Sci USA, 2004, 101(16): 6050–6055.
[59] Wray GA. The evolutionary significance of cis–regulatory mutations. Nat Rev Genet, 2007, 8(3): 206–216.
[60] Goodman M. Decoding the pattern of protein evolution. Prog Biophys Mol Bio, 1981, 38(2): 105–164.
[61] Zuckerkandl E. Appearance of new structures and functions in proteins during evolution. J Mol Evol, 1975, 7(1): 1–57.
[62] Blake C. Exons and the evolution of proteins. Trends Biochem Sci, 1983, 8(1): 11–13.
[63] Halder G, Callaerts P, Gehring WJ. Induction of ectopic eyes by targeted expression of the eyeless gene in Drosophila. Science, 1995, 267(5205): 1788–1792.
[64] Grens A, Mason E, Marsh JL, Bode HR. Evolutionary conservation of a cell fate specification gene: the Hydra achaete–scute homolog has proneural activity in Drosophila. Development, 1995, 121(12): 4027–4035.
[65] Imai H, Suzuki N, Ishimaru Y, Sakurai T, Yin LJ, Pan WS, Abe K, Misaka T, Hirai H. Functional diversity of bitter taste receptor TAS2R16 in primates. Biol Lett, 2012, 8(4): 652–656.
[66] Kwiatkowski DP. How malaria has affected the human genome and what human genetics can teach us about malaria. Am J Hum Gen, 2005, 77(2): 171–192.
[67] Enattah NS, Sahi T, Savilahti E, Terwilliger JD, Peltonen L, Jarvela I. Identification of a variant associated with adult–type hypolactasia. Nat Genet, 2002, 30(2): 233–237.
[68] Darwin CR. On the origins of species by means of natural selection, or the preservation of favoured races in the struggle for life. London: Murray, 1859.
[69] Sabeti PC, Varilly P, Fry B, Lohmueller J, Hostetter E, Cotsapas C, Xie XH, Byrne EH, Mccarroll SA, Gaudet R, Schaffner SF, Lander ES. Genome–wide detection and characterization of positive selection in human populations. Nature, 2007, 449(7164): 913–918.
[70] Grossman SR, Shylakhter I, Karlsson EK, Byrne EH, Morales S, Frieden G, Hostetter E, Angelino E, Garber M, Zuk O, Lander ES, Schaffner SF, Sabeti PC. A composite of multiple signals distinguishes causal variants in regions of positive selection. Science, 2010, 327(5967): 883–886.
[71] Fujimoto A, Kimura R, Ohashi J, Omi K, Yuliwulandari R, Batubara L, Mustofa MS, Samakkarn U, Settheetham–Ishida W, Ishida T, Morishita Y, Furusawa T, Nakazawa M, Ohtsuka R, Tokunaga K. A scan for genetic determinants of human hair morphology: EDAR is associated with Asian hair thickness. Hum Mol Genet, 2008, 17(6): 835–843.
[72] Fujimoto A, Ohashi J, Nishida N, Miyagawa T, Morishita Y, Tsunoda T, Kimura R, Tokunaga K. A replication study confirmed the EDAR gene to be a major contributor to population differentiation regarding head hair thickness in Asia. Hum Genet, 2008, 124(2): 179–185.
[73] Chang SH, Jobling S, Brennan K, Headon DJ. Enhanced Edar signalling has pleiotropic effects on craniofacial and cutaneous glands. PLoS ONE, 2009, 4(10): E7591.
[74] Kamberov YG, Wang SJ, Tan JZ, Gerbault P, Wark A, Tan LZ, Yang YJ, Li SL, Tang K, Chen H, Powell A, Itan Y, Fuller D, Lohmueller J, Mao J, Schachar A, Paymer M, Hostetter E, Byrne E, Burnett M, Mcmahon AP, Thomas MG, Lieberman DE, Jin L, Tabin CJ, Morgan BA, Sabeti PC. Modeling recent human evolution in mice by expression of a selected EDAR variant. Cell, 2013, 152(4): 691–702.
[75] Ohno S. Evolution by gene duplication. London: George Alien & Unwin Ltd. Berlin, Heidelberg and New York: Springer–Verlag, 1970.
[76] Adams MD, Celniker SE, Holt RA, Evans CA, Gocayne JD, Amanatides PG, Scherer SE, Li PW, Hoskins RA, Galle RF, George RA, Lewis SE, Richards S, Ashburner M, Henderson SN, Sutton GG, Wortman JR, Yandell MD, Zhang Q, Chen LX, Brandon RC, Rogers YH, Blazej RG, Champe M, Pfeiffer BD, Wan KH, Doyle C, Baxter EG, Helt G, Nelson CR, Gabor GL, Abril JF, Agbayani A, An HJ, Andrews-Pfannkoch C, Baldwin D, Ballew RM, Basu A, Baxendale J, Bayraktaroglu L, Beasley EM, Beeson KY, Benos PV, Berman BP, Bhandari D, Bolshakov S, Borkova D, Botchan MR, Bouck J, Brokstein P, Brottier P, Burtis KC, Busam DA, Butler H, Cadieu E, Center A, Chandra I, Cherry JM, Cawley S, Dahlke C, Davenport LB, Davies P, de Pablos B, Delcher A, Deng Z, Mays AD, Dew I, Dietz SM, Dodson K, Doup LE, Downes M, Dugan-Rocha S, Dunkov BC, Dunn P, Durbin KJ, Evangelista CC, Ferraz C, Ferriera S, Fleischmann W, Fosler C, Gabrielian AE, Garg NS, Gelbart WM, Glasser K, Glodek A, Gong F, Gorrell JH, Gu Z, Guan P, Harris M, Harris NL, Harvey D, Heiman TJ, Hernandez JR, Houck J, Hostin D, Houston KA, Howland TJ, Wei MH, Ibegwam C, Jalali M, Kalush F, Karpen GH, Ke Z, Kennison JA, Ketchum KA, Kimmel BE, Kodira CD, Kraft C, Kravitz S, Kulp D, Lai Z, Lasko P, Lei Y, Levitsky AA, Li J, Li Z, Liang Y, Lin X, Liu X, Mattei B, McIntosh TC, McLeod MP, McPherson D, Merkulov G, Milshina NV, Mobarry C, Morris J, Moshrefi A, Mount SM, Moy M, Murphy B, Murphy L, Muzny DM, Nelson DL, Nelson DR, Nelson KA, Nixon K, Nusskern DR, Pacleb JM, Palazzolo M, Pittman GS, Pan S, Pollard J, Puri V, Reese MG, Reinert K, Remington K, Saunders RD, Scheeler F, Shen H, Shue BC, Sidén-Kiamos I, Simpson M, Skupski MP, Smith T, Spier E, Spradling AC, Stapleton M, Strong R, Sun E, Svirskas R, Tector C, Turner R, Venter E, Wang AH, Wang X, Wang ZY, Wassarman DA, Weinstock GM, Weissenbach J, Williams SM, WoodageT, Worley KC, Wu D, Yang S, Yao QA, Ye J, Yeh RF, Zaveri JS, Zhan M, Zhang G, Zhao Q, Zheng L, Zheng XH, Zhong FN, Zhong W, Zhou X, Zhu S, Zhu X, Smith HO, Gibbs RA, Myers EW, Rubin GM, Venter JC. The genome sequence of Drosophila melanogaster. Science, 2000, 287(5461): 2185–2195.
[77] Collins FS, Lander ES, Rogers J, Waterston RH, Conso I. Finishing the euchromatic sequence of the human genome. Nature, 2004, 431(7011): 931–945.
[78] Waterston RH, Lindblad-Toh K, Birney E, Rogers J, Abril JF, Agarwal P, Agarwala R, Ainscough R, Alexandersson M, An P, Antonarakis SE, Attwood J, Baertsch R, Bailey J, Barlow K, Beck S, Berry E, Birren B, Bloom T, Bork P, Botcherby M, Bray N, Brent MR, Brown DG, Brown SD, Bult C, Burton J, Butler J, Campbell RD, Carninci P, Cawley S, Chiaromonte F, Chinwalla AT, Church DM,Clamp M, Clee C, Collins FS, Cook LL, Copley RR, Coulson A, Couronne O, Cuff J, Curwen V, Cutts T, Daly M, David R, Davies J, Delehaunty KD, Deri J, Dermitzakis ET, Dewey C, Dickens NJ, Diekhans M, Dodge S, Dubchak I, Dunn DM, Eddy SR, Elnitski L, Emes RD, Eswara P, Eyras E, Felsenfeld A, Fewell GA, Flicek P, Foley K, Frankel WN, Fulton LA, Fulton RS, Furey TS, Gage D, Gibbs RA, Glusman G, Gnerre S, Goldman N, Goodstadt L, Grafham D, Graves TA, Green ED, Gregory S, Guigó R, Guyer M, Hardison RC, Haussler D, Hayashizaki Y, Hillier LW, Hinrichs A, Hlavina W, Holzer T, Hsu F, Hua A, Hubbard T, Hunt A, Jackson I, Jaffe DB, Johnson LS, Jones M, Jones TA, Joy A, Kamal M, Karlsson EK, Karolchik D, Kasprzyk A, Kawai J, Keibler E, Kells C, Kent WJ, Kirby A, Kolbe DL, Korf I, Kucherlapati RS, Kulbokas EJ, Kulp D, Landers T, Leger JP, Leonard S, Letunic I, Levine R, Li J, Li M, Lloyd C, Lucas S, Ma B, Maglott DR, Mardis ER, Matthews L, Mauceli E, Mayer JH, McCarthy M, McCombie WR, McLaren S, McLay K, McPherson JD, Meldrim J, Meredith B, Mesirov JP, Miller W, Miner TL, Mongin E, Montgomery KT, Morgan M, Mott R, Mullikin JC, Muzny DM, Nash WE, Nelson JO, Nhan MN, Nicol R, Ning Z, Nusbaum C, O'Connor MJ, Okazaki Y, Oliver K, Overton-Larty E, Pachter L, Parra G, Pepin KH, Peterson J, Pevzner P, Plumb R, Pohl CS, Poliakov A, Ponce TC, Ponting CP, Potter S, Quail M, Reymond A, Roe BA, Roskin KM, Rubin EM, Rust AG, Santos R, Sapojnikov V, Schultz B, Schultz J, Schwartz MS, Schwartz S, Scott C, Seaman S, Searle S, Sharpe T, Sheridan A, Shownkeen R, Sims S, Singer JB, Slater G, Smit A, Smith DR, Spencer B, Stabenau A, Stange-Thomann N, Sugnet C, Suyama M, Tesler G, Thompson J, Torrents D, Trevaskis E, Tromp J, Ucla C, Ureta-Vidal A, Vinson JP, Von Niederhausern AC, Wade CM, Wall M, Weber RJ, Weiss RB, Wendl MC, West AP, Wetterstrand K, Wheeler R, Whelan S, Wierzbowski J, Willey D, Williams S, Wilson RK, Winter E, Worley KC, Wyman D, Yang S, Yang SP, Zdobnov EM, Zody MC, Lander ES. Initial sequencing and comparative analysis of the mouse genome. Nature, 2002, 420(6915): 520–562.
[79] Nakatani Y, Takeda H, Kohara Y, Morishita S. Reconstruction of the vertebrate ancestral genome reveals dynamic genome reorganization in early vertebrates. Genome Res, 2007, 17(9): 1254–1265.
[80] Van De Peer Y, Maere S, Meyer A. The evolutionary significance of ancient genome duplications. Nat Rev Genet, 2009, 10(10): 725–732.
[81] Kasahara M. The 2R hypothesis: an update. Curr Opin Immunol, 2007, 19(5): 547–552.
[82] Soshnikova N, Dewaele R, Janvier P, Krumlauf R, Duboule D. Duplications of hox gene clusters and the emergence of vertebrates. Dev Biol, 2013, 378(2): 194–199.
[83] Löhr U, Pick L. Cofactor–interaction motifs and the cooption of a homeotic Hox protein into the segmentation pathway of Drosophila melanogaster. Curr Biol, 2005, 15(7): 643–649.
[84] Löhr U, Yussa M, Pick L. Drosophila fushi tarazu: a gene on the border of homeotic function. Curr Biol, 2001, 11(18): 1403–1412.
[85] Heffer A, Xiang J, Pick L. Variation and constraint in Hox gene evolution. Proc Natl Acad Sci USA, 2013, 110(6): 2211–2216.
[86] Sambrani N, Hudry B, Maurel–Zaffran C, Zouaz A, Mishra R, Merabet S, Graba Y. Distinct molecular strategies for Hox–mediated limb suppression in Drosophila: from cooperativity to dispensability/antagonism in TALE partnership. PLoS Genet, 2013, 9(3): E1003307.
[87] Struhl G. Genes controlling segmental specification in the Drosophila thorax. Proc Natl Acad Sci USA, 1982, 79(23): 7380–7384.
[88] Levine M, Hafen E, Garber RL, Gehring WJ. Spatial distribution of Antennapedia transcripts during Drosophila development. EMBO J, 1983, 2(11): 2037–2046.
[89] Carroll SB, Laymon RA, McCutcheon MA, Riley PD, Scott MP. The localization and regulation of Antennapedia protein expression in Drosophila embryos. Cell, 1986, 47(1): 113–122.
[90] Gibson G, Gehring WJ. Head and thoracic transformations caused by ectopic expression of Antennapedia during Drosophila development. Development, 1988, 102: 657–675.
[91] Vachon G, Cohen B, Pfeifle C, Mcguffin ME, Botas J, Cohen SM. Homeotic genes of the Bithorax complex repress limb development in the abdomen of the Drosophila embryo through the target gene Distal–less. Cell, 1992, 71(3): 437–450.
[92] Khadjeh S, Turetzek N, Pechmann M, Schwager EE, Wimmer EA, Damen WG, Prpic NM. Divergent role of the Hox gene Antennapedia in spiders is responsible for the convergent evolution of abdominal limb repression. Proc Natl Acad Sci USA, 2012, 109(13): 4921–4926.
[93] Yang MY, Hatton–Ellis E, Simpson P. The kinase Sgg modulates temporal development of macrochaetes in Drosophila by phosphorylation of Scute and Pannier. Development, 2012, 139(2): 325–334
(责任编委: 王义权)
Role of genes and their cis-regulatory elements during animal morphological evolution
Boyuan Sun, Jianbo Tu, Ying Li, Mingyao Yang
Institute of Animal Genetics and Breeding, Sichuan Agricultural University, Chengdu 611130, China
Cis-regulatory hypothesis is one of the most important theories in evolutionary developmental biology (evo-devo), which claims that evolution of cis-regulatory elements (CREs) plays a key role during evolution of morphology. However, an increasing number of experimental results show that cis-regulatory hypothesis alone is not far enough to explain the complexity of evo-devo processes. Other modifications, including mutations of protein coding, gene and genome duplications, and flexibility of homeodomains and CREs, also cause the morphological changes in animals. In this review, we retrospect the recent results of evolution of CREs and genes associated with CREs and discuss new methods and trends for research in evo-devo.
evo-devo; cis-regulatory element; gene; morphology
2013-10-07;
2013-12-06
四川农业大学“双支”计划基金(编号2920200)和四川省教育厅“科研创新团队”基金资助
孙博渊, 在读硕士研究生, 专业方向:动物遗传与进化。E-mail: sunboy0415@foxmail.com
杨明耀, 博士, 教授, 博士生导师, 研究方向:动物遗传与进化。E-mail: yangmingyao@sicau.edu.cn
10.3724/SP.J.1005.2014.0525
时间: 2014-5-20 11:05:57
URL: http://www.cnki.net/kcms/doi/10.3724/SP.J.1005.2014..html

