石油输入对河口芦苇湿地根际微生物的影响
田伟君,王勇梅,孙会梅,赵阳国,2,白 洁,2,吴承林 (.中国海洋大学环境科学与工程学院,山东 青岛26600;2.海洋环境与生态教育部重点实验室,山东 青岛 26600)
河口湿地是陆地和海洋生态系统之间重要的过渡带,由陆源产生的营养物质和有机有毒性物质在通过河口湿地进入海洋时经历了生物、地质及水文等因子的共同作用,经过各种物理、化学过程得到净化.无论是对地表水和海水,它都起到一定的滞留作用,是保护海洋生态系统免受来自陆源污染的最后一道屏障[1-2].但作为陆海相互作用的集中地带,河口湿地生态系统也相对敏感脆弱.近年来,外源污染物不断输入,对湿地生态系统的稳定已经形成潜在危害.尤其是我国河口湿地分布着许多大型油田(如辽河油田,胜利油田等),油田开发强度大、井喷油管破裂、原油泄漏、油井钻探等使河口湿地普遍存在不同程度的石油污染,严重威胁到湿地生态系统的健康发展[3].
土壤微生物群落是湿地生态系统的重要组成部分,在湿地净污过程中发挥重要的作用[4-6].绝大多数有机物的降解、氮磷硫物质的生物地球化学循环等都是微生物在群落水平,通过各种单一功能的种群依次梯级作用实现的[7-8],某种或某类微生物的数量或活性的消长都会直接影响到群落功能的完成,最终导致生态系统的失衡甚至破坏.因此,已有一些学者研究石油污染物的输入对微生物群落的影响,但主要是分析石油污染物对农业、林地和草地土壤及植物根际微生物群落的影响[9-16],而对河口湿地微生物群落影响的研究则较少,主要原因一方面是输入河口湿地的污染物种类繁多,模拟分析设备环境条件控制困难,且在短时间内揭示漫长的污染驯化过程难度很高;另一方面受传统微生物分析方法的限制.因此,本文以辽东湾双台子河河口湿地为对象,针对该地区油井密布的实际情况,以石油污染物为主要污染物,采用湿地模拟实验装置,通过常规监测和PCR-DGGE技术分析微生物群落长期暴露于污染物中时逐渐演替、更叠的真实情况,为开发其中的微生物资源和保护河口湿地提供科学依据.
1 材料与方法
1.1 湿地模拟及实验用水
针对双台子河河口湿地进水的实际情况,结合湿地土壤及湿地植物根系的生长状况,利用PVC及有机玻璃等材料研制6组带配水装置(以满足模拟湿地进水的可控性)的湿地模拟试验成套装置,将高约 50cm 的双台河河口湿地土壤及湿地植物原位移入湿地模拟装置.

表1 采样条件及实验用水情况Table 1 Characterization of river water for drip-irrigation and diesel oils addition
根据芦苇湿地实际进水调查结果,芦苇湿地主要进水分别为每年的5月初和6月底,其余时间均为适量补水.因此,分别于 2011年 5月和 6月采集主要进水河流和引水泵站的水样,测定的石油类污染物浓度为 0.27~1.3mg/L.提水站水样中石油类污染物的浓度明显高于河流中的,说明柴油提水泵的使用增加了水中石油类的浓度,由于芦苇湿地用水主要依赖提水泵站的供应,因此本研究实验用水用柴油与河水进行配水,且实验期间保持进水中石油类污染物浓度为1.3 mg/L.具体实验期实验用油量及环境条件如表1所示.
1.2 样品采集与处理
采用抖根法分别取对照组和实验组-10,-20,-30,-40cm 处芦苇根际土壤样品,用无菌封口袋装好带回实验室,土壤首先通过孔径 2mm 筛,去除较大植物组织与石块,然后在冰上手工剔除肉眼可见的细根.土样分成3份,第1份保存在4℃用于土壤环境参数和种群数量计数,第 2份保存在-20℃用于变性梯度凝胶电泳(DGGE )研究,第3份低温避光条件下自然风干,粉碎研磨后过100目筛,用于正构烷烃和多环芳烃分析.所有样品处理在取样后6h内完成.
1.3 正构烷烃和多环芳烃的测定
称 10.0g土壤样品,加入少量无水硫酸钠后用滤纸包好放入索氏抽提器中,瓶底放2.0g铜片去除硫的干扰,加入回收率指示物.150mL正己烷/二氯甲烷(V:V/1:1)索氏提取 24h(水浴锅温度80℃).将提取液旋转蒸发浓缩至 5mL,加入 5mL正己烷再次旋转蒸发浓缩至 5mL.浓缩液过氧化铝(3cm)、硅胶(6cm)和无水硫酸钠(0.5 cm)层析柱,分别用正己烷、二氯甲烷/正己烷(V:V/3:7)混合液洗脱样品得到正构烷烃、多环芳烃.最后将淋洗液旋转蒸发至1mL转移至2mL棕色进样瓶中,用柔和的氮气吹至近干,然后用正己烷定容至1mL,待上机测定.
采用Agilent气相色谱-质谱联用仪HP6890 GC-HP5975MSD对样品中的烷烃和多环芳烃进行分析.对样品的定性分析主要依据其特征离子和色谱保留时间,同时与标准物质的质谱图和质谱库内标准质谱图进行对照.烷烃和多环芳烃定量采用外标法.
1.4 土壤微生物种群数量测定方法
细菌、放线菌、真菌数量采用稀释平板法计数,其所用培养基分别为:细菌-牛肉膏蛋白胨培养基(牛肉膏 3.0g,蛋白胨 10.0g,NaCl 5.0g,琼脂18.0g蒸馏水 1000mL,pH7.4~7.6);放线菌-高氏一号培养基(可溶性淀粉20.0g, NaCl 0.5g, KNO31.0g,H2O 0.5g, M0.5g,7H2O 0.01g,琼脂 18.0g,水 1000mL, pH7.4~7.6);真菌-链霉素-马丁孟加拉红培养基(蛋白胨 5g,葡萄糖 10.0g, KH2PO41.0g, MgSO4·7H2O 0.5g,琼脂 18.0g,1/3000孟加拉红溶液 100mL,蒸馏水1000mL,链霉素0.03g,pH 7.4~7.6).
石油降解菌数量测定采用最大或然数(MPN)法测定[17-18],所用的培养基为改良的 Bushnel-Haas 培 养 基 (MgSO4·7H2O 0.2g,CaCl20.02g,KH2PO41.0g,(NH4)2HPO4,1.0g KNO31.0g,FeCl30.05g,柴油1.0mL,蒸馏水1000mL,pH7.6~7.8).
1.5 土壤微生物群落结构分析
1.5.1 DNA的提取 所有土壤样品DNA的提取均采用PowerSoil DNA Isolation Kit (MOBIO Laboratories)试剂盒,称取0.25g土壤样品,按试剂盒提供的操作步骤进行土壤微生物总DNA提取.在1%琼脂糖(1×TAE缓冲液)凝胶(内含5%的EB染料)中对提取的DNA进行检测.
1.5.2 PCR 扩增 所用引物为细菌 16S rDNA V3区通用引物,F341和BA534-GC[19].扩增反应体系为 40μL 体系:包括 10×PCR buffer 4μL;dNTP(2.5mmol/μL) 3.2μL;每种引物 1.2μL;模板2μL;Taq DNA聚合酶0.4μL;加ddH2O至终体积40μL.反应程序为:预变性 94℃ 5min 30 个循环包括 94℃变性 40s,50℃退火 30s,72℃延伸 90s,最后 72℃延伸 10min.PCR完成后取反应产物5µL用1%琼脂糖凝胶电泳检测,并与定量Maker DL 2000进行对比.
1.5.3 变性梯度凝胶电泳(DGGE)分析 采用Bio-Rad公司DcodeTM的基因突变检测系统对PCR产物进DGGE分析[20].制备8%的丙烯酰胺溶液(丙烯酰胺与甲叉双丙烯酰胺的比例为37.5:1),使用梯度混合装置,制备 8%聚丙烯酰胺梯度凝胶,变性剂尿素和甲酞胺的浓度从 30%到60%,变性剂浓度从胶的上方到下方递增.促凝胶催化剂TEMED和过硫酸氨(APS)的终浓度分别为 0.09%(V/V)和 0.05%(V/V).将凝固后的胶板放入加有 l×TAE缓冲液的电泳槽中,取 PCR产物20µL 和 4µL 6×Loading Buffer混合后加入上样孔中,60℃,150V 电泳 9h.电泳结束后,将凝胶用EB (l:1000的稀释液)进行染色 30min,然后去离子水浸洗 2min.染色之后,在紫外凝胶成像系统下观察凝胶的条带,并拍摄图像.
1.5.4 DGGE条带的切割、克隆与测序 切割亮度较明显的条带,编号放入1.5mL灭菌的离心管中,并加入20µL灭菌水磨碎,用引物F341和BA534R重新进行PCR扩增产生长度为200bp的片段,其他步骤同上述 PCR扩增.电泳切取亮带放入收集管中待纯化.产物纯化按spin胶纯化试剂盒说明书进行.将已纯化的 PCR产物连接到载体 PMD19-T Vector上,导入DH5α感受态细胞, 加入LB液体培养基培养.以M13为引物进行菌落PCR,检测产物是否为阳性.测序由南京金斯瑞公司执行.
1.6 数据处理与统计
实验数据统计及相关性分析采用SPSS 19.0.测序后其结果在GenBank数据库中用BLAST进行检索并进行同源性比较.15条优势带的基因片段序列与GenBank中细菌序列相似性分析,其测序结果可以确定出各条带所代表的菌群分类地位.应用QuantityOne软件对DGGE图谱中的条带数目和亮度进行分析.
2 结果与讨论
2.1 正构烷烃和多环芳烃在芦苇根际的分布
芦苇根区土壤中正构烷烃各组分的浓度及分布情况见图 1.湿地芦苇根区土壤中定量检出的正构烷烃系列主要集中在C10~C33之间.含油河水灌溉的实验组芦苇根区土壤正构烷烃的分布与对照组明显不同,实验组浓度较高且主要集中在C13~C26之间,其中C20左右的浓度最高,达到了 3.33μg/g.而对照组各碳数的分布较为平均且浓度均较低,最高值仅为 0.052μg/g,近于实验组的百分之一.从芦苇整个生长期来看,实验组各月份正构烷烃的分布特征较为相似,均为单峰型分布,显示了石油烃的输入特征[21].从纵向看,对照组-10cm 处正构烷烃总浓度(∑n-alkanes)为0.313~0.395μg/g,-40cm 处∑n-alkanes 浓度为0.233~0.292μg/g,在纵向上基本符合-10cm>-20cm>-30cm>-40cm,但随土层深度的变化幅度较小,分布较为均匀;实验组-10cm 处∑n-alkanes浓度在 23.69~34.84μg/g之间,且随深度变化较大,-40cm 处∑n-alkanes浓度为 1.19~2.38μg/g,仅为-10cm的 1/20,说明添加的正构烷烃主要集中于湿地土壤的表层.
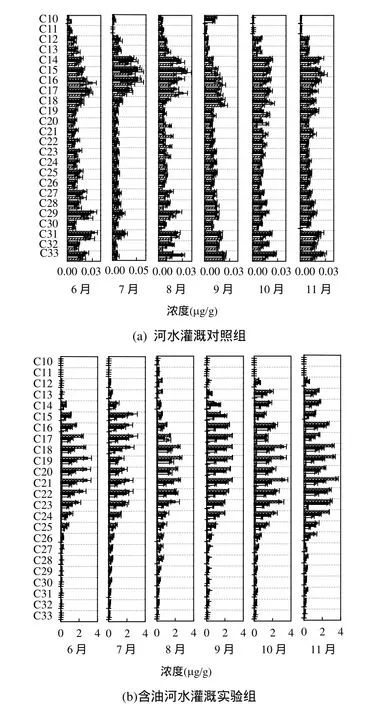
图1 湿地植物根区土壤正构烷烃各组分的浓度及分布Fig.1 Distributions of n-alkanes in reed rhizosphere soils drip-irrigated with/without river water added diesel oils
芦苇根区土壤中多环芳烃(PAHs)各组分的浓度及分布见图2.实验组和对照组芦苇根区土壤中均检测出 14种 PAHs,对照组中高分子量(4~6环)、难生物降解的PAHs占∑PAHs的近50%,且分布在芦苇整个生长季内无明显变化,土著微生物对PAHs的降解能力较弱.实验组PAHs各组分分布与对照组相似,其中萘、芴、菲、芘占∑PAHs比重较高,约为 37.0~74.3%.浓度最高的主要集中于-10cm 土层中,随着深度的增加迅速减小.实验组∑PAHs浓度在-10cm处的平均值为963.25ng/g约为-40cm处平均值385.89ng/g的2.5倍.

图2 湿地植物根区土壤多环芳烃各组分的浓度及分布Fig.2 Distributions of PAHs in reed rhizosphere soils drip-irrigated with/without river water added diesel oils

2.2 石油污染对芦苇根际微生物数量的影响
细菌、放线菌和真菌是土壤微生物群落的3种重要组成部分,对有机物的矿化起着重要作用.实验初期芦苇根际的这 3种微生物群落数量明显受到石油污染物连续输入的影响(表 2).石油污染物的输入减少芦苇根际细菌、放线菌和真菌的数量,但增加了根际土壤的有机质和微量元素的含量,促进了石油烃降解菌的生长和繁殖[22-23].但经过 6个月灌溉后,除添加石油污染物的细菌数量约为未添加的1/4外,实验组的放线菌和真菌的数量均与未添加的相近,说明经过长时间的石油输入,放线菌和真菌已适应了石油污染的环境.石油烃降解菌的数量则增加了55~215倍.此外,皮尔森相关分析也表明芦苇根际微生物种群数量与正构烷烃和多环芳烃的浓度有明显关系(表 3),即污染物浓度越高,微生物种群数量越小.其中,细菌对石油最为敏感,根际土壤每一层的细菌数量均与氧化还原电位(ORP),正构烷烃和多环芳烃浓度呈显著的负相关性.而放线菌和真菌只是在表层土壤受 ORP,正构烷烃和多环芳烃浓度的影响比较明显.石油降解菌的数量与氧化还原电位,正构烷烃和多环芳烃的浓度呈显著正相关性(R=0.886~0.978,P<0.01).含水量对芦苇根际种群数量的影响并不明显.
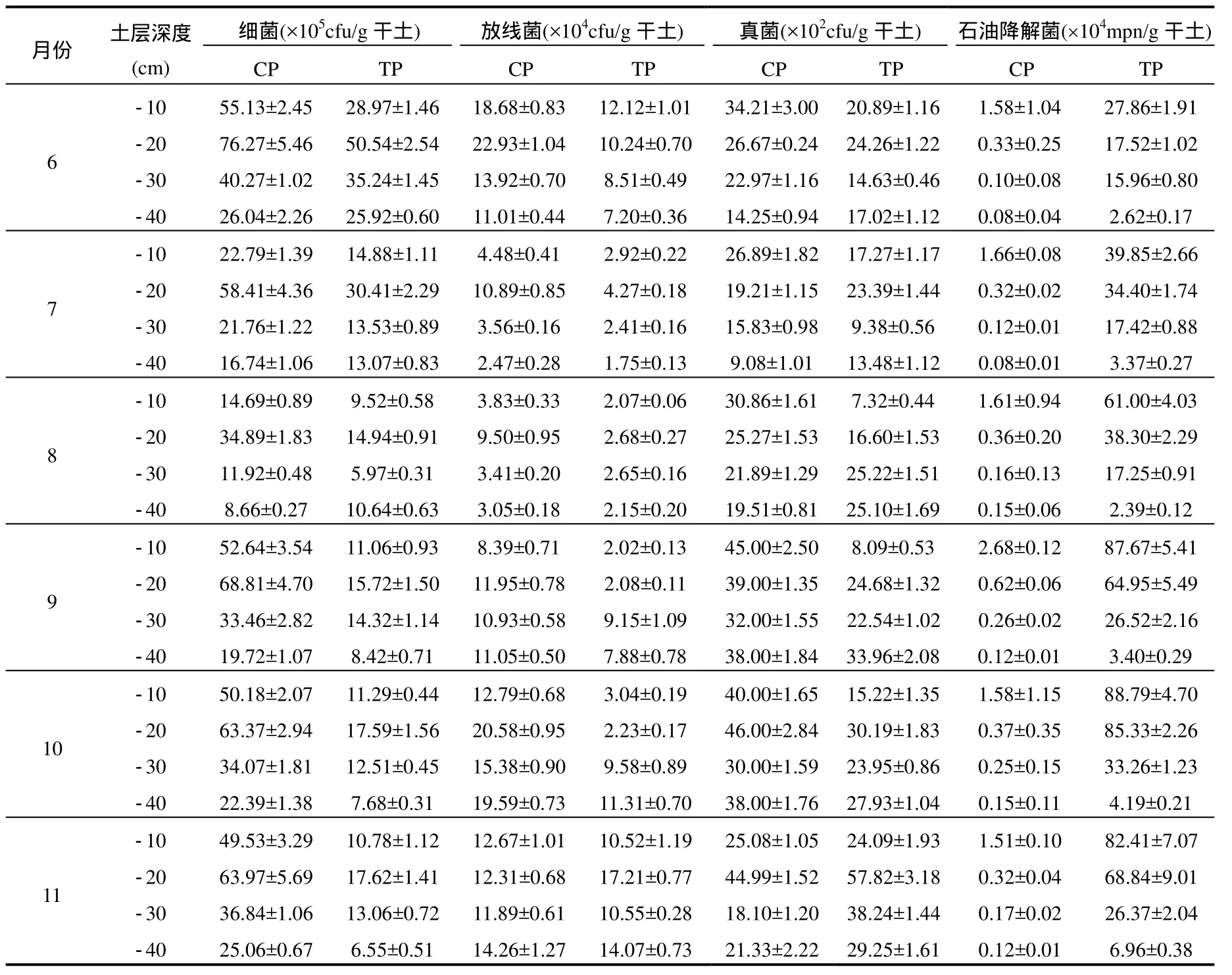
表2 灌溉期内芦苇根区微生物种群数量变化Table 2 Microbial abundance of reed rhizosphere soils drip-irrigated with/without river water added diesel oils in six-month irrigation process
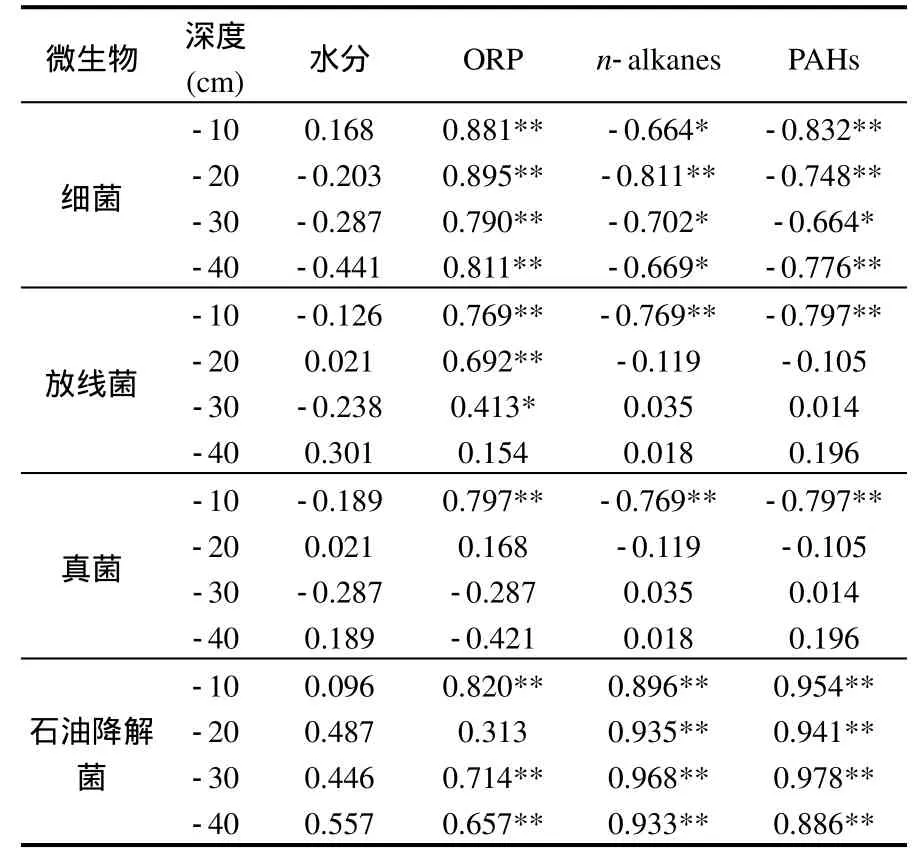
表3 微生物种群数量与土壤参数及有机质相关性分析Table 3 Coefficients of Pearson’s correlations between microbial abundances and the soil parameters and organic matters
2.3 石油污染对芦苇根际细菌群落结构的影响
对含油河水灌溉组和对照组各月份-10cm土壤样品进行 DGGE分析,各土壤样品的变性梯度凝胶电泳(DGGE)谱图如 3(a)所示.由指纹图谱可以看出,S1~S12各样品的条带数目,位置和亮度均存在一定的差异性.S1~S6样品中条带2均有出现且较亮,而在S7~S12中并不明显,说明条带2是石油污染影响下的优势种群.S4样品中出现了有异于其他样品的条带,且条带数目较多,说明经过长时间的石油输入,微生物适应了石油污染的环境,以烃类作为碳源和能源的石油降解菌菌群数量增加[24-25],与 9月份石油降解菌菌群数量最高相符.其中条带1在S1出现后,在S3和S4都均存在,说明该菌群为能降解石油的菌群.S7~S12样品条带数目较 S1~S6多,表明石油污染一定程度上降低了土壤微生物群落多样性.细菌图谱的聚类分析结果如图3(b)所示,群落结构最为相似的是S10和S11,其中S6和S9,S7和S8,S1和S3的群落结构分别较相似,S1~S5与S6~S12最后聚在一起,相似性差异较大,进一步说明石油污染对土壤细菌群落结构产生了影响.根据芦苇根际微生物群落DGGE图谱中出现的优势和特殊条带进行割胶回收测序,测序后其结果在 GenBank数据库中用BLAST进行检索并进行同源性比较.15条优势带的基因片段序列与 GenBank中细菌序列相似性如表 4.结果显示,经过 6个月的含油河水灌溉后,形成了以变形菌门(Proteobacteriumspp.)和拟杆菌门(Bacteroidetesspp.)为主要优势菌的群落结构,而厚壁菌门(Firmicutesspp.),放线菌门(Actinobacteriaspp.)和蓝藻门(Cyanobacteriaspp.)则受到不同程度的抑制.

图3 芦苇湿地-10cm深度土壤细菌群落的DGGE分离图谱及聚类分析树状图谱Fig.3 DGGE profiles of microbial communities of reed rhizosphere soils (-10cm depth) and cluster analysis
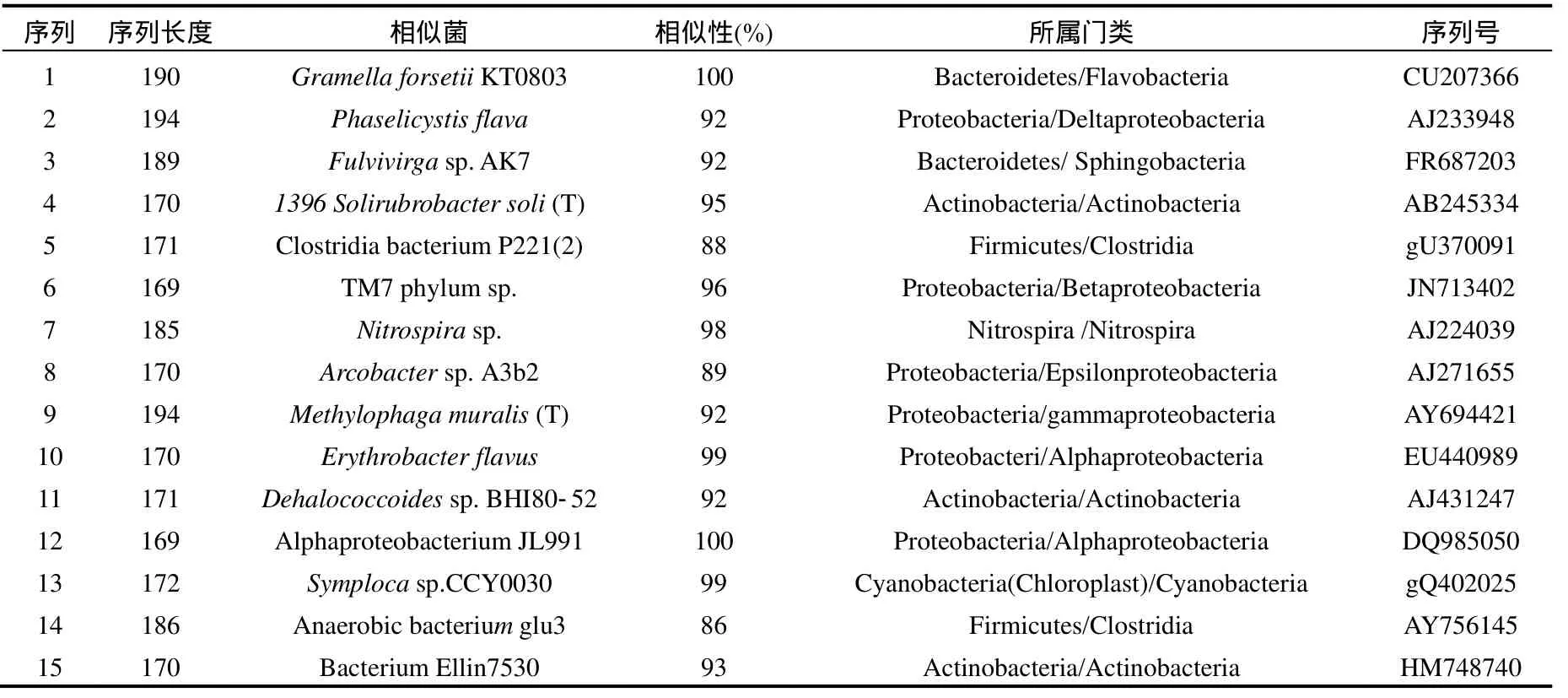
表4 测序比对结果Table 4 Sequence similarities to closest relatives and phylogenetic affiliations of DNA recovered from DGGE gel
3 结论
3.1 湿地植物根区主要检测到 C10~33的正构烷烃,含油组正构烷烃均为单峰型分布且波峰主要集中在 C13~C26,奇偶优势不明显且主峰碳数较低,显示了石油烃的输入特征;而未添加柴油对照组各碳数的分布较为平均且浓度均较低,最高值仅为0.052μg/g.说明微生物降解尤其针对低碳数的作用明显.
3.2 含油河水灌溉的总多环芳烃(∑PAHs)含量是未添加柴油对照组的近10倍,萘、芴、菲、芘在 16种多环芳烃中所占的比重较大.多环芳烃(PAHs)的分布特征除浓度增加外变化不明显,说明湿地植物根区的多环芳烃也来源于石油污染,且土著微生物对PAHs的降解能力较弱.
3.3 石油污染物的输入减少芦苇根际细菌的数量,但促进了石油烃降解菌的生长和繁殖.经过 6个月的含油河水灌溉后,添加石油污染物的细菌的数量约为未添加的 1/4.而石油烃降解菌的数量则增加了55~215倍.皮尔森相关分析也表明芦苇根际细菌数量与根区土壤中的 ORP,正构烷烃和多环芳烃的浓度有明显关系.
3.4 运用PCR-DGGE技术对细菌群落结构进行分析时也发现石油污染物质连续输入的初期阶段降低了微生物的数量,但经过 6个月的自我调节,微生物数量在连续输入的末期逐渐又恢复到初期的程度,但微生物群落结构发生了变化,形成了以变形菌门(Proteobacteriumspp.)和拟杆菌门(Bacteroidetesspp.)为主要优势菌群的群落结构.
[1]张晓龙,李培英,李 萍,等.中国滨海湿地研究现状与展望 [J].海洋科学进展, 2005,23(1):87-94.
[2]Ji G D, Sun T H, Ni J R, et al. Impact of heavy oil-polluted soils on reed wetlands [J]. Ecological Engineering, 2007,29:272-279.
[3]李丽锋,惠淑荣,宋红丽,等.盘锦双台河口湿地生态系统服务功能能值价值评价 [J]. 中国环境科学, 2013,33(8):1454-1458.
[4]Kennedy A C, Smith K L. Soil microbial diversity and the sustainability of agrocultural soils [J]. Plants Soil, 1995,170:75-86.
[5]Finlay B J, Maberly S C, Cooper J I. Microbial diversity and ecological function [J]. Oikos, 1997,80:209-213.
[6]Groffman P M, Bohlen P J. Soil and sediment biodiversitycross-system comparisons and large-scale effects [J]. BioScience,1999,49:130-148.
[7]Bagwell C E, Piceno Y M,Ashburne-Lucas A,et al. Physiological diversity of the rhizosphere diazotroph assemblages of selected salt marsh grasses [J]. Applied and Environmental Microbiology,1998,64:4276-4282.
[8]Cunha M A, Pedro R, Almeida M A, et al. Activity and growth efficiency of heterotrophic bacteria in a salt marsh (Ria de Aveiro,Portugal) [J]. Microbiology Research, 2005,160:279-290.
[9]姜睿玲,杨统一,唐玉斌,等.多环芳烃污染对桑园土壤微生物结构及种群多样性的影响 [J]. 中国环境科学, 2012,32(9):1655-1661.
[10]Waldrop M P,Balser T C,Firestone M K. Linking microbial community composition to function in a tropical soil [J]. Soil Biology and Biochemistry, 2000,32:1837-1846.
[11]Yao H, He Z, Wilson M J, et al. Microbial biomass and community structure in a sequence of soils with increasing fertility and changing land use [J]. Microbial Ecology, 2000,40:223-237.
[12]Grayston S J, Wang S,Campbell C D, et al. Selective influence of plant species on microbial diversity in the rhizosphere [J]. Soil Biology and Biochemistry, 1998,30:369-378.
[13]Ibekwe A M, Kennedy A C. Fatty acid methyl ester (FAME)profiles as a tool to investigate community structure of two agricultural soils [J]. Plant Soil, 1999,206:151-161.
[14]Miethling R, Wieland G, Backhaus H, et al. Variation of microbial rhizosphere communities in response to crop species,soil origin, and inoculation with Sinorhizobium meliloti L33 [J].Microbial Ecology,2000,41:43-56.
[15]Marschner P, Yang C H, Lieberei R, et al.Soil and plant specific effects on bacterial community composition in the plant rhizosphere [J]. Soil Biology and Biochemistry, 2001,33:1437-1445.
[16]Su Y H, Yang X Y. Interactions between selected PAHs and the microbial community in rhizosphere of a paddy soil [J]. Science of the Total Environment, 2009,407:1027-1034.
[17]中国科学院土壤研究所微生物室.土壤微生物的研究方法 [M].北京:科学出版社, 1985:1-209.
[18]俞疏馨,吴国庆,孟宪庭. 环境工程微生物检验手册 [M]. 北京:中国环境科学出版社, 1990:138-142.
[19]赵先丽,周广胜,周 莉,等. 盘锦芦苇湿地土壤微生物特征分析[J]. 气象与环境学报, 2006,22(4):64-66.
[20]聂 明.芦苇及其根周围微生物对石油污染响应的生态系统生态学研究 [D]. 上海:复旦大学生命科学学院, 2010.
[21]韩 雪,马启敏,周 华,等.渤海典型海域表层沉积物正构烷烃特征比较 [J]. 环境化学, 2012,31:1316-1319.
[22]Stapleton R D, Sayler G S, Boggs J M, et al. Changes in subsurface catabolic gene frequencies during natural attenuation of petroleum hydrocarbons [J]. Environmental Science and Technology,2000,34:1991-1999.
[23]Nie M, Zhang X D, Wang J Q, et al. Rhizosphere effects on soil bacterial abundance and diversity in the Yellow River Deltaic ecosystem as influenced by petroleum contamination and soil salinization [J]. Soil Biology and Biochemistry, 2009,41:2535-2542.
[24]Bento F M, Oliveira Camargo F A, Okeke B C,et al. Diversity of biosurfactant producing microorganisms isolated from soils contaminated with diesel oil [J]. Microbiology Research, 2005,160:249-255.
[25]Li H, Zhang Y, Zhang C G, et al. Effect of petroleum-containing wastewater irrigation on bacterial diversities and enzymatic activities in a paddy soil irrigation area [J]. Journal of Environmental Quality, 2005,34:1073-1080.

