基于T-RFLP和因子分析的香蒲根际细菌群落研究
马栋山,熊 薇,张琼琼,郭羿宏,赵文吉,郭逍宇* (1.首都师范大学资源环境与旅游学院,北京100048;2.北京市城市环境过程与数字模拟重点实验室-省部共建国家重点实验室培育基地,北京 100048)
T-RFLP分析较DGGE、SSCP或ARDRA等方法具有分析精细、分析能力强、易于自动化等优势,另外通过ABI自动测序仪对任何一末端限制性片段的测序结果均可与 Internet数据库(http://www.cme.msu.edu)的数据进行比较分析[1].已有学者成功应用T-RFLP技术进行菌种的鉴定、湿地、农田、水体等多生态环境中微生物群落多样性及其动态变化研究[2].但对环境样品进行T-RFLP分析时,可产生大量的酶切片段,其中 80%~90%的酶切片段(T-RFs)属于非培养片段或数据库信息不全而无法比对到种属特征,因而无法对该片段进行深入分析.多元统计特征分析因其在海量数据中挖掘数据的内在规律方面有较大的优势,因而得到了人们的重视.已有学者初步尝试借用统计学方法对T-RFLP分析中酶切片段进行分析[3].
再生水作为北京市稳定的第二新型水源,在维持河湖景观、恢复河湖生态等方面发挥了重要作用,而再生水水质特性决定了其城市河湖景观补水存在多种负面生态环境效应[4].以氨氮为河道水体富营养化主控因素的再生水补水河湖湿地水污染防治成为湿地研究的热点问题之一[5].人工湿地具有低投资、低能耗、低处理成本和脱氮除磷功能,是目前再生水厂河湖景观补水之前普遍采用的水质净化方式.湿地再生水处理机理不仅体现在植物通过吸收作用去除氮、磷等污染物,更体现在植物通过根际分泌物影响系统中微生物特性进而影响系统处理效果[6].湿地植物根际微生物群落结构与功能随湿地植物种类、季节及湿地类型等因素的变化而变化[7],湿地补水水源水质的好坏同样影响植物根际微生物功能和生物过程[8].大量文献分析了氨氧化细菌在水体、底泥、农田等诸多环境中群落结构及其多样性特性[9-10].但对水生植物根际氨氧化细菌群落组成及多样性的研究尚不多见.基于此本文拟借助于因子分析(FA)、去除势典范对应分析(CCA)等多元统计分析方法,结合T-RFLP技术产生的大量T-RFs片段构建的多样性指数和PAT比对结果等常规分析方法对王平湿地再生水补水口、远离补水口香蒲根际细菌和氨氧化细菌群落多样性及其结构特征进行对比研究,为再生水补水湿地植物根际微生物群落净化机理提供科学依据.
1 材料与方法
1.1 研究区概况
王平湿地(39°58.580′E,115°58.930′N)西起王平沟口,东至南涧沟口,全长 1050m,属门头沟永定河段.王平湿地系统内水生植物主要包括香蒲、芦苇、浮萍等,其中,香蒲的覆盖度可达70%~90%.采样点位于门头沟王平村左岸附近.再生水主要来源于王坪村电厂和周边的居民生活区,再生水排放量约为0.9万t/a.
1.2 样品采集及理化指标分析
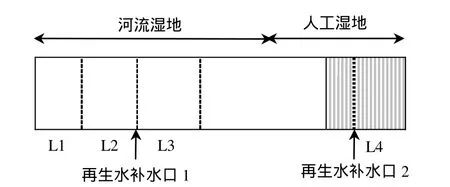
图1 北京市王平湿地采样点分布示意Fig.1 Distribution of sampling sites in Wangping Wetland in Beijing
选取王平湿地不同处理区植物香蒲为研究对象,分别取河流湿地再生水补水口的上游点位L1(补水口上游 200m),再生水补水口 L2以及补水口下游300m处的L3;人工湿地再生水补水口位点 L4(距离 L2位点 1000m)(图 1).采样时间2012年9月,去除植物根系上的动植物残骸及石块等非底质杂物,收集附着在根系上的底泥作为样本.每个点位设5个平行.采集的样品装在干净密封的聚四氟乙烯塑料袋中,
于 4℃下保存并及时送回实验室,每个位点的 5个平行样等量混合后最终成为一个样点,共计四个样点,分别记为 L1、L2、L3、L4[11].样品分两部分处理:一部分进行理化指标分析,总氮(TN)采用凯氏定氮法,总磷(TP)采用碱熔—钼锑抗分光光度法,总有机碳(TOC)采用重铬酸钾氧化—分光光度法测定,铵态氮采用2mol/L KCl浸提—靛酚蓝比色法测,氧化还原电位采用ORP仪直接测量,重金属采用原子吸收光谱法,具体测定结果见表 1.剩余根际土壤样品于-20℃下保存,用于微生物群落结构分析.
1.3 基于T-RFLP的微生物群落结构分析
1.3.1 DNA提取 采用土壤样品提取试剂盒(天根生化科技北京有限公司)提取根际样品总DNA,操作步骤按照使用说明书进行.提取总DNA经 0.8%(m/V)琼脂糖凝胶电泳检测鉴定,提取的DNA放置于-20℃条件下保存备用.
1.3.2 PCR扩增 PCR技术采用史青应用T-RFLP 技术分析滇池污染水体的细菌群落技术[12]进行扩增.
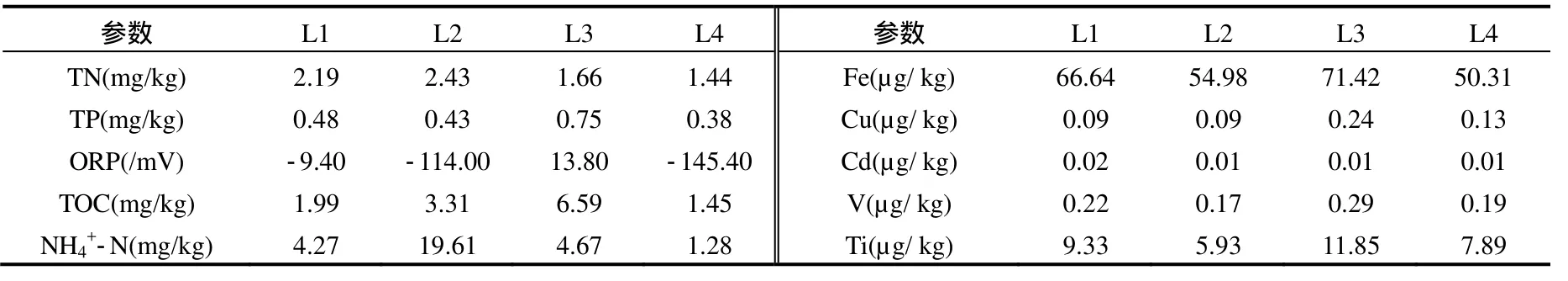
表1 不同样点香蒲根际土壤的理化性质Table 1 Physical and chemical properties of cattail rhizospheric soil in different plots
1.3.3 末端限制性片段长度多态性(T-RFLP)分析 分别采用MspⅠ、HhaⅠ、HaeⅢ对荧光PCR产物进行酶切.在 37℃下放置 3~5h.然后在 65℃条件下温浴15min使酶失活.随后将酶切产物送至天根生物技术有限公司进行基因扫描(GeneScan),得到T-RFLP图谱.
1.3.4 氨氧化细菌克隆文库的建立和测序 采用王亚男等[13]的直接法克隆和测序分析,取总DNA,进行PCR扩增、克隆、测序.
1.4 数据处理与分析
用Peak Scanner进行分析.舍去小于50bp和大于500bp的片段.对于细菌,由于相对数量过小的限制性末端片段(T-RFs)不会对群落的特性产生明显的影响[14-16],故在本分析中舍去了相对数量<1%的 T-RFs,然后分别计算图谱中每一个峰的峰面积与所有峰总面积的比值,将每个 T-RF所占的百分比作为权重导入Primer软件,聚类方法选择组间平均距离法,距离选择平方欧氏距离,做出聚类分析图.综合采用 Peak Scanner、CanoDraw for Windows、primer和 Excel2007 软件对数据进行分析,得出聚类图、优势菌种、多样性指数以及CCA排序.
2 结果与讨论
2.1 基于T-RFLP谱图的聚类分析
把采集样品基于 T-RFLP微生物群落结构分析的谱图进行预处理之后,采用Primer分析结果如图2(a)所示.以相似性50%为标准,可以将细菌各样点分为三类:细菌类群 BⅠ由位于再生水补水口上游位置的L1样点组成,类群BⅡ由再生水补水口L2与L4样点组成,类群BⅢ是位于再生水补水口下游位置L3样点构成;如图2(b)所示,以相似性 32%为标准,可以将氨氧化细菌各样点分为两类:氨氧化细菌类群AOBⅠ包含远离再生水补水口L1和L3样点;氨氧化细菌类群AOBⅡ则包含再生水补水口L2和L4两样点.
从聚类结果可知,细菌类群以受再生水干扰程度的强弱而进行分群并类,不同湿地类型 L2与L4因受再生水补水口水质影响同样归并为一类.与氨氧化细菌群落的分类结果相比,细菌群落对环境条件的变化较氨氧化细菌群落结构更为敏感,可能与氨氧化细菌群落主要由环境条件中不同形态氮素浓度梯度决定,细菌群落变异可能是环境条件变异的综合反应[17].

图2 各样点细菌群落和氨氧化细菌的等级聚类分析Fig.2 Dendrogram of hierarchical cluster analysis of the bacterial and AOB from different sampling plots
2.2 再生水补水对不同样点细菌和 AOB群落结构多样性的影响
酶切图谱和多样性指数结果均显示,MspI酶切样品微生物多样性略高于HhaⅠ、HaeⅢ酶切,故后续分析采用MspI酶切结果[18].以MspⅠ为酶切的不同类群香蒲根际细菌和AOB群落多样性指数见表 2.细菌各类群多样性指数均随再生水干扰强度的增加依次表现 BⅠ>BⅢ>BⅡ的变化趋势,而氨氧化细菌群落则与细菌群落呈现相反的变化趋势,即再生水补水口氨氧化细菌各群落多样性指数均明显高于远离补水口氨氧化细菌群落.再生水补水不仅降低了细菌群落结构中物种的丰富度,同时为应对再生水中丰富氮磷等水质特性,细菌群落结构中与再生水水质特性密切联系的优势类群在群落结构中占有主导优势,群落均匀度降低,进而群落综合多样性指数降低.对于 AOB而言,再生水中高浓度氮含量是导致再生水补水口氨氧化细菌群落各多样性指数显著升高的直接原因[20].
2.3 再生水补水对不同样点细菌和 AOB群落结构的影响
以MspⅠ酶切结果计算各类群中 T-RFs片段的丰度,T-RFs片段丰度值>4%的类型为优势菌群[20],而片段丰度值<1%的类型为偶见菌群,其余为非优势菌群[21],以MspⅠ为酶切的香蒲根际细菌和AOB群落结构的空间差异见表3.随再生水干扰强度增强细菌类群 T-RFs片段数呈明显降低趋势;细菌类群 BⅠ因其较高丰度的非优势菌群和偶见菌群,从而群落均匀度较高,而细菌类群 BⅡ则因较低丰度的非优势菌群及不具偶见菌群从而群落均匀度较低;远离再生水补水口氨氧化细菌群落AOBⅠ较补水口AOBⅡ具较少的T-RFs片段数,且其优势氨氧化菌群相对丰度值远高于AOBⅡ,表明在再生水作用下氨氧化细菌优势片段数量的增加、非优势片段数量的减少和偶见片段数量的消失是导致 AOBⅠ群落丰富度和均匀度降低的主要原因.
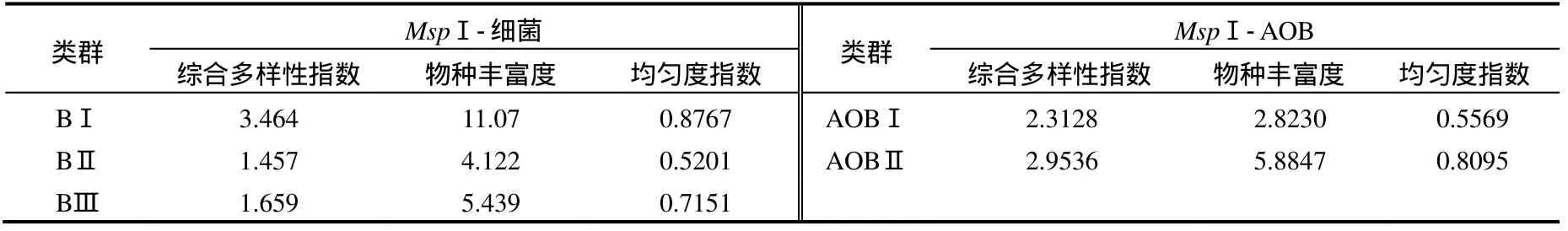
表2 以MspⅠ为酶切的不同类群香蒲根际细菌和氨氧化细菌群落多样性指数Table 2 Diversity indexes of cattail rhizospheric soils bacterial and AOB community in different groups with Msp I enzyme
2.4 基于传统T-RFs片段与多元统计相结合分析
2.4.1 MiCA 比对结果 将HaeⅢ、MspⅠ、Hha3Ⅰ种内切酶消化的T-RFLP图谱属性数据上传到 Phylogenetic Assignment Tool(PAT,https://secure.limnology.wisc.edu/trflp/newuser.jsp)网站[22-23],并结合 MiCA (http:// MiC A.ibest.uidaho.edu/pat.php通过 Virtual Digest(ISPaR)模块产生的基础数据库对起主要作用T-RFs类型的系统发育分类进行推测.之后通过计算其细菌 T-RFs比例,以占据整个 T-RFs的4%以上为优势菌种;而 AOB通过电子酶切(MspⅠ、HhaI、Hae)Ⅲ得出的片段和克隆得出的片段相比对,比对可能种属见表4.

表3 MspⅠ酶切细菌和氨氧化细菌群落结构空间差异Table 3 The spatial differences of cattail rhizospheric soils bacterial and AOB community structures with Msp I enzyme
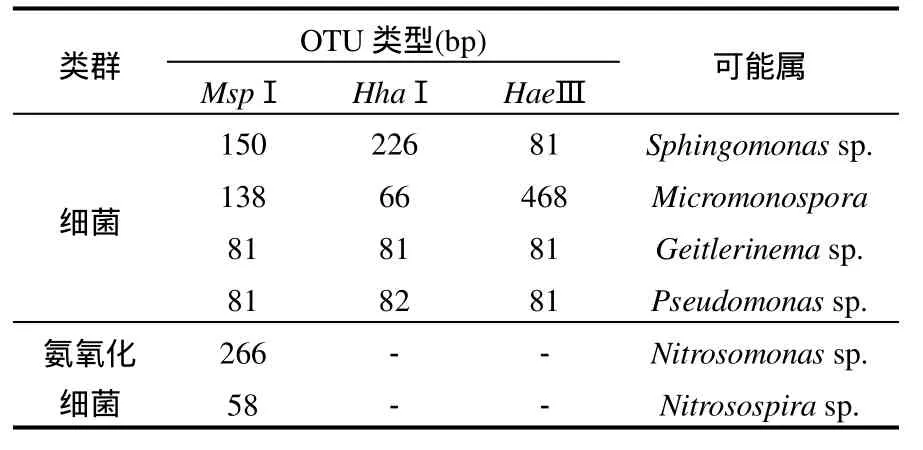
表4 MspI酶切香蒲根际细菌和AOB类群可能种属Table 4 The corresponding species and features of cattail rhizospheric soils bacterial and AOB communities with Msp I enzyme
2.4.2 因子分析 以基于细菌和氨氧化细菌群落MspⅠ酶切,结果显示细菌群落筛选出的73个T-RFs片段反应的信息累计方差达到 92.3%,而氨氧化细菌群落筛选出的56个T-RFs片段反应的信息累计方差达到 95.4%,表明两个原始数据矩阵均可以较好地反应基于片段的源解析[24],统计结果见表5.细菌群落因子分析将73个T-RFs片段降为三个维度,其中 PCA1提取了 33个T-RFs片段,除81bp片段之外,其余32个非培养或数据库中无匹配.根据因子分析原理可比对的81bp片段可能与不可比对的32个T-RFs片段具有共同或相近的源,即81bp片段可能与不可比对的32个T-RFs片段共同受水质生境特征中某一相同环境因子的制约和影响,或与水质生境特征中某一环境因子的生物化学循环过程具有密切的关系.同样 PCA2和 PCA3中提取27个和11个非培养或数据库中无匹配的 T-RFs片段可能与其对应维度中可比对的150bp和138bp片段具有共同或相近的源.基于氨氧化细菌群落的因子分析将56个T-RFs片段降为两个维度,对于氨氧化细菌群落中PCA4中提取的39个T-RFs片段可能与Nitrosospirasp.具有共同或相近的源,而PCA5代表的 17个 T-RFs片段可能与是Nitrosomonassp.共同或相近的源.
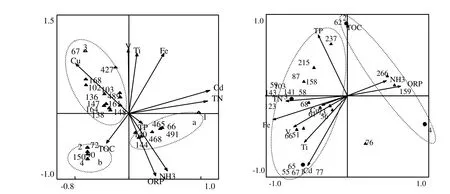
2.4.3 典范对应分析 为验证基于MiCA比对结合因子分析结果见图3.排序结果表明CCA排序图第一轴AX1和第二轴AX2的特征值累计占总特征值的 71.23%,排序图包含了大部分的信息,排序效果良好.对于细菌群落而言,再生水补水口样点2和样点4在排序图中具有共同的分布格局与范围,同时与样点2和样点4具共同分布格局的T-RFs片段主要包括了以150bp为代表的PCA2中所有T-RFs片段,这些片段占到样点2和样点4所代表的BⅡ群落总丰度的55.6%.环境因子中TOC与样点2和样点4具有共同分布格局与范围的结果则进一步证明再生水补水口以 150bp为代表的占群落总丰度55.6%菌群均与水中TOC的生物化学循环过程具有密切关系. 而且过比对发现 150bp片段对应的种属为鞘氨醇单胞菌属,Onruthai研究表明鞘氨醇单胞菌是环境中比较独特的一类降解菌,它们的降解范围非常宽,从单环、多环芳烃到其它有机污染物都可以作为它们生长的碳源[25].PCA2是通过聚类而得,表明在PCA2类群当中,除去不能确定的非培养的细菌类群外,很有可能其余的片段大部分与碳循环是相关的.
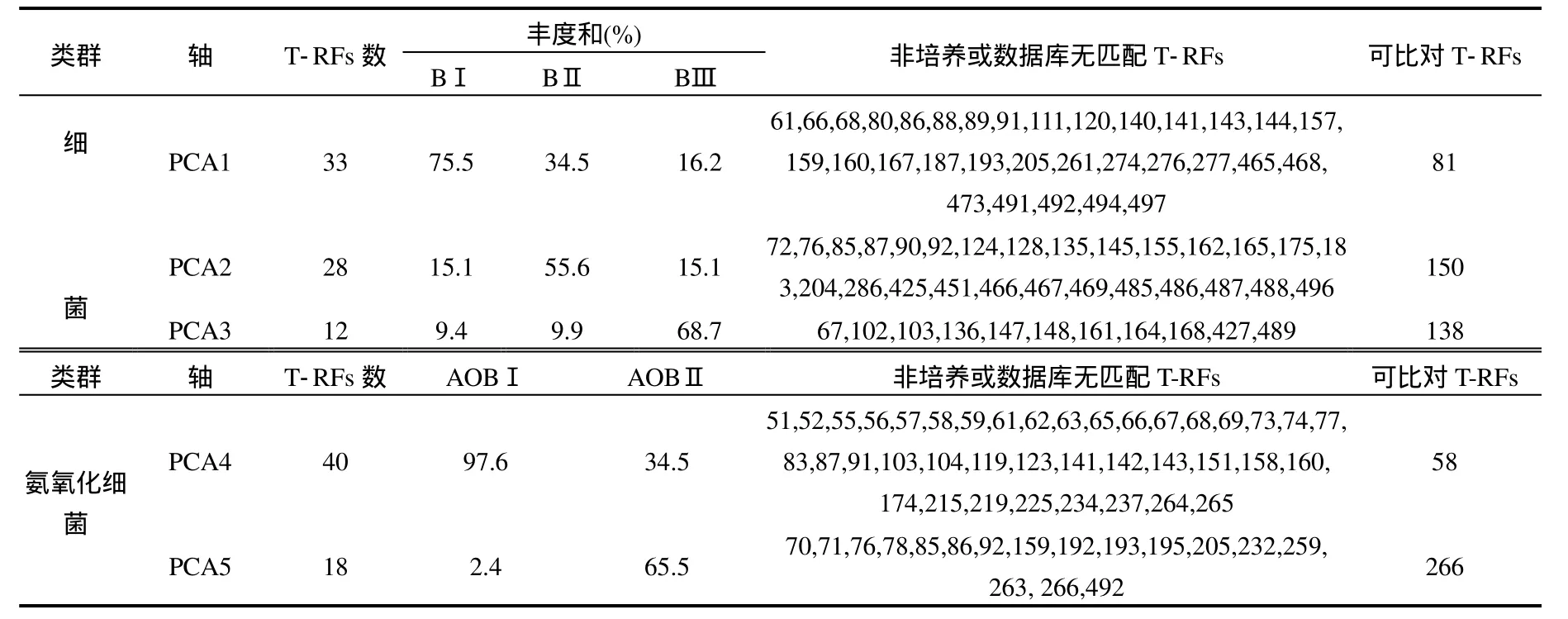
表5 以MspI为酶切的香蒲根际细菌和AOB类群的PCA聚类片段和比例Fig.5 Clustering fragments and proportion of cattail rhizospheric soils bacterial and AOB communities with Msp I enzyme
左图:细菌a:61,68,80,81,86,88,89,91,111,120,141,143,144,157,159,160,167,187,193,205,261,274,276,277,473,492,494,497 b:76,85,87,92,124,128,135,145,155,162,165,175,183,204,286,425,451,466,467,469,485,486,487,488,496右图:AOB a:70,71,78,85,86,92,192,193,195,205,232,259,263,492 b:52,57,63,69,74,83,104,119,142,151,160,174,219,225,234,237,264,265, 491

图3 以MspI为酶切的香蒲根际细菌和氨氧化细菌样方、物种与环境因子的典范对应分析(CCA)Fig.3 Canonical correspondence analysis biplot of the cattail rhizospheric soils bacterial and AOB communities in relation to the species and environmental factors
再生水补水口上游样点 1在排序图中的T-RFs片段主要包括了以81bp为代表的PCA1中所有bp片段,它们的相对丰度和占到整个BⅡ群落总丰度的 75.5%.环境因子中 TN、TP和铵态氮与样点 1具有共同分布格局的结果则进一步证明再生水补水口上游以81bp为代表的菌群与水质生境特征中TN、TP和铵态氮的生物化学循环过程具有密切关系. 经过比对发现 81bp对应的是假单胞菌属和Geitlerinemasp.属.假单胞菌属是一种在有机污染中普遍存在的菌属,可以利用包括单碳在内的许多有机物作为自身的能量和碳源,以有机氮或无机氮为氮源进行化能营养生活[26],而Geitlerinemasp.是专性属于底栖生物环境,与水环境中氮的循环具有密切关系已得到普遍的认同[27],这与 CCA分析的结果中与环境因子氮循环具有密切关系的结果保持一致.再生水补水口下游样点3在排序图中的T-RFs片段主要包括了以 138bp为代表的 PCA3中所有bp片段数,它们的相对丰度和到整个BⅢ群落总丰度的68.7%.环境因子重金属Cu、V和Ti与样点 3具有共同分布格局与范围的结果则进一步证明再生水补水口以 138bp为代表的相似菌群均与水质生境特征重金属Cu、V和Ti具有密切关系.
对于AOB而言,与样点2和样点4具共同分布格局的T-RFs片段主要包括了以266bp为代表的PCA5中所有T-RFs片段,这些片段占到样点 2和样点 4所代表的 AOBⅡ群落总丰度的65.5%.环境因子中NH3-N与样点2和样点4具有共同分布格局的结果进一步证明再生水补水口以 266bp代表的亚硝化单胞菌属(Nitrosomonassp.)受到再生水铵态氮的影响较大.以58bp为代表的PCA4中所有T-RFs片段,这些片段占到样点1和样点3所代表的AOBⅠ群落总丰度的97.6%.环境因子中TOC与样点1和样点 3具有共同分布格局与范围的结果则进一步证明再生水补水口以58bp为代表的亚硝化螺菌属菌(Nitrosospirasp.)受到铵态氮的影响较小.AOBⅡ中包含两个再生水补水口的铵态氮的含量远远大于AOB,Ⅰ AOBⅡ中数量丰度最多的亚硝化单胞菌属是在富含氨氮环境中具有竞争优势,而偏好相对低氨环境的亚硝化螺菌属则数量极少.
3 结论
3.1 细菌类群以受再生水干扰程度的强弱而进行分群并类,不同湿地类型因受再生水补水口水质影响归并为一类.
3.2 再生水补水口细菌群落以有机碳循环为主,补水口上游主要以氮循环为主,远离补水口细菌类群主要体现重金属的生物化学循环.
3.3 266bp为代表的Nitrosomonassp.具相似功能的菌群适合在高氨环境中生长;58bp所代表的
Nitrosospirasp.具相似功能特性的菌群适合在相对低氨环境中生长.
[1]Maidak B L, Cole J B, Lilburn T G. The RDP Ribosomal Data-base Project continues [J]. Nucleic Acids Res., 2000,28:173-174.
[2]Siripong Slil, Rittmann Bruce E. Diversity study of nitrifying bacteria in full-scale municipal wastewater treatment plants [J].Water Research, 2007,41(5):1110−1120.
[3]Tao Ding, Michael W Palmer and Ulrich Melcher Community terminal restriction fragment length polymorphisms reveal insights into the diversity and dynamics of leaf entophytic bacteria BMC [J]. Microbiology, 2013,13:1-11.
[4]孟庆义,吴晓辉,赵立新,等.再生水回用于北京景观水体引起的水质变化及其改善措施 [J]. 水资源保护, 2011,27(1):51-54.
[5]曾 薇,李 磊,杨莹莹,等.A2O 工艺处理生活污水短程硝化反硝化的研究 [J]. 中国环境科学, 2010,30(5):625-632.
[6]Grove J K, Ste in O R. Polar Organic Solvent Rem oval in Microcosm Constructed Wet lands [J]. Water Research, 2005,39:4040-4050.
[7]McBride M B. Environmental Chemistry in Soils [M]. Oxford:Oxford University. Press, 1994:406-407.
[8]陈卫平,张炜铃,潘 能,等.再生水灌溉利用的生态风险研究进展 [J]. 环境科学, 2012,33(12):4070-4080.
[9]Teske A, Alm E, Regan J, et al. Evolutionary relationships among Ammonia and nitrite deoxidizing bacteria. [J]. Bacterial, 1994,176:6623-6630.
[10]Bock E, Koops H P. The genus nitrobacteria and related genera;Oxidation of inorganic nitrogen compounds as energy source;The prokaryotes: A handbook on the biology of bacteria.Ecophysiology, isolation, identification, applications [M]. New York: Springer-Verlag, 1992.
[11]王 薇,俞 燕,王世和.人工湿地污水处理工艺与设计 [J]. 城市环境与城市生态, 2001,14(1):59-62.
[12]史 青,柏耀辉,李宗逊,等.应用 T-RFLP技术分析滇池污染水体的细菌群落 [J]. 环境科学, 2011,32(6):1788-1792.
[13]王亚男,曾希柏,俄胜哲,等.施肥对设施菜地氨氧化细菌群落和丰度的影响 [J]农业环境科学学报, 2012,31(12):2425-2432.
[14]Liu W T, Marsh T L, Cheng H, et al. Characterization of microbial diversity by determining terminal restriction fragment length polymorphisms of genes encoding 16S rRNA [J].Canadian Metallurgical Quarterly, 1997,63(11):4516-4522.
[15]Mayrand P E, Corcoran K P, Ziegle J S, et al. The use of fluorescence detection and internal lane standards to size PCR products automatically [J]. Applied and Theoretical Electrophoresis, 1992,3(1):1-11.
[16]Ziegle J S, Su Ying, Corcoran K P, et al.Application of automated DNA sizing technology for genotyping microsatellite loci [J].Genomics, 1992,14(4):1026-1031.
[17]Horswill A R, Stoodley P, Stewart P S, et al. The effect of the cheMiCAl, biological, and physical environment on quorum sensing in structured microbial communities [J]. Anal. Bioanal.Chem., 2007,387:371–380.
[18]牟文婷,张 涛,孙 建,等.新疆特殊生境岩石内生细菌末端限制性片段长度多态性技术分析 [J]. 微生物学报, 2012,52(3):381-388.
[19]Daims H, Nielsen P H, Nielsen J, et al. Novel Nitrospira-like bacteria as dominant nitrite-oxidizers in biof i lms from wastewater treatment plants: diversity and in situ physiology [J].Water Sci. Technol., 2000,41(4/5):85–90.
[20]Ringelberg D B, Foley K L, Reynolds C M. Electrogenic capacity and community composition of anodic biofilms in soil-based bioelectroche MiCAl systems [J]. Applied Microbiology and Biotechnology, 2011,90(5):1805-1815.
[21]Zhang Rui, Thiyagarajan Ve, Qian Pei-Yuan. Evaluation of terminal-restriction fragment length polymorphism analysis in contrasting marine environments [J]. FEMS Microbiology Ecology, 2008,65(1):169–178.
[22]Sch ü tte Ursel M E, Abdo Z, Bent St J,et al. Advances in the use of terminal restriction fragment length polymorphism (T-RFLP)analysis of 16S rRNA genes to characterize microbial communities [J]. Applied Microbiology and Biotechnology, 2008,80(3):365-380.
[23]Kent Angela D, Smith Dan J, Benson Barbara J, et al. Web-based phylogenetic assignment tool for analysis of terminal restriction fragment length polymorphism profiles of microbial communities[J]. Applied and Environmental Microbiology, 2003,69(11):6768-6776.
[24]李惠民.水稻土细菌和氨氧化细菌的 RFLP分析 [D]. 杨凌:西北农林科技大学, 2009.
[25]Pinyakong On, Habe H, Yoshida T, et al. Identification of three novel salicylate 1-hydroxylases involved in the phenanthrene degradation ofSphingobiumsp. strain P2 [J]. Biotechnology Research Center, 2003,301(2):350-357.
[26]Moore E R B, Tindall B J,V A P, Martins,et al. Palleroni.No-nmedical: Pseudomonas [J]. The Prokaryotes, 2006,6:646-703.
[27]Eric H Andrianasolo1, Douglas Goeger, William H, et al.Mitsoamide: A cytotoxic linear lipopeptide from the Madagas-car marine cyanobacteriumGeitlerinemasp.[J]. Pure and Applied Chemistry, 2007,79(4):593-602.

