赖氨酸胍基化对蛋白质组分析的影响
郑 昊, 毛家维, 潘彦博, 刘忠山,刘哲益, 叶明亮, 邹汉法
(1.中国科学院分离分析化学重点实验室,国家色谱研究分析中心,中国科学院大连化学物理研究所,辽宁大连116023;2.中国科学院大学北京100049)
质谱技术由于其高度的自动化水平和快速分析能力被广泛用于复杂生物样品的定性和定量分析中[1]。复杂生物样品用胰蛋白酶酶解后产生C端是精氨酸或赖氨酸的肽段,通过液相色谱-串联质谱分析[2],经数据库检索,可实现蛋白质的定性或定量分析。1967年Kimmel[3]提出胍基化选择性地发生在赖氨酸侧链氨基上,N端氨基或其他侧链基团不发生此反应。近年来,这一现象在蛋白质定性和定量领域受到了广泛的关注。用胍基化同位素试剂(13C、15N2标记的甲氧脲和12C、14N2标记的甲氧脲)平行处理HeLa细胞蛋白质胰蛋白酶酶解产物,不仅能增加碎片离子信息,而且还可以识别蛋白质C 端[4,5],利于蛋白质鉴定。另外,利用胍基化保护赖氨酸侧链,再用二甲基化同位素试剂标记肽段N端,利于识别低丰度蛋白质,提高鉴定的可信度[6]。
根据质子转移理论[7]和电荷诱导碎裂原则[8],肽段含有氨基酸的碱性越高,其捕获质子的能力和保持离子结构稳定的能力越强,就能产生更多的碎裂信息,有利于提高质谱鉴定的灵敏度。由于空间位阻的原因,非N端赖氨酸侧链的氨基选择性地发生胍基化反应,而N端氨基或其他侧链基团则不发生该反应[9]。赖氨酸发生反应后转化为高精氨酸,使其碱性增强,进而可提高含有赖氨酸的肽段的序列覆盖率和鉴定可信度。本文对比了以下3种HeLa细胞蛋白质胰蛋白酶酶解产物中的肽段胍基化前后的质谱响应情况:(1)仅在C端有赖氨酸的肽段;(2)中间位置含有赖氨酸的肽段;(3)不含有赖氨酸的肽段。发现鉴定到的第1类肽段占总肽段的比例由51.7%上升为57.3%,并且其二级质谱图中y离子的比重有所上升;而第3类肽段没有明显的变化。说明胍基化能改变质谱的选择性,并提高了第1类肽段的质谱鉴定灵敏度。这些结果对于深入了解胍基化对蛋白质分析的影响以及实现其广泛应用具有重要的意义。
1 实验部分
1.1 仪器与试剂
子宫颈癌细胞(HeLa)来源于中国医学科学院血液病研究所;RPMI(Roswell Park Memorial Institute)1640培养基和青霉素/链霉素购自美国Gibco Invitrogen公司;新生牛血清购自德国 Biochrom AG公司;氢氧化钠、盐酸、丙酮、胰蛋白酶、碳酸氢铵(NH4HCO3)、二硫苏糖醇(dithiothreitol,DTT)、碘代乙酰胺(iodoacetamide,IAA)、乙二胺四乙酸(EDTA)、蛋白酶抑制剂(protease inhibitor cocktail)和甲氧脲(O-methylisourea)购自美国Sigma公司;苯甲基磺酰氟(phenylmethanesulfonylfluoride,PMSF)购自美国Ameresco公司;聚乙二醇单辛基苯基醚(Triton-X 100)购自美国Bio Basic公司;C18AQ反相色谱填料(5 μm,12 nm)购自日本DAISO Chemical公司,75 μm 和 150 μm 内径毛细管购自美国Polymicro公司;乙腈为色谱纯,购自美国Merck公司。其余试剂均为国产分析纯。实验用水为Milli-Q超纯水。
Surveyor HPLC液相色谱系统(配有脱气机和四元液相泵)及LTQ Velos双压线性离子阱质谱系统(配有纳升电喷雾离子源和六通阀)购自美国Thermo公司。
1.2 细胞培养与蛋白质的提取、酶解
HeLa细胞用RPMI 1640培养基(含10%(v/v)新生牛血清,青霉素和链霉素的终浓度分别为1 unit/mL和1 μg/mL)在37℃和5%CO2条件下培养。待细胞生长至对数生长期后用胰蛋白酶消化,于1 000 g离心力下离心3 min,收集细胞。用4℃预冷的磷酸盐缓冲液(PBS)清洗3次后加入细胞裂解液(8 mol/L 尿素、1%(v/v)Triton-X 100、65 mmol/L DTT、1 mmol/L EDTA、1 mmol/L PMSF、2%(v/v)cocktail、40 mmol/L pH 7.4 的 Tris-HCl)[10],用超声波细胞碎裂仪碎裂细胞。于4℃25 000 g离心力下离心60 min,弃去上清液,用4℃丙酮洗涤2次,在通风橱中干燥后存于-80℃备用。
将提取的蛋白质复溶于1 mL含8 mol/L尿素和0.1 mol/L NH4HCO3的变性溶液中;再加入DTT,在56℃水浴环境中还原1 h;加入IAA,在避光条件下对打开的二硫键处理 40 min;用 0.1 mol/L NH4HCO3将溶液稀释到1 mol/L,加入适量胰蛋白酶,在37℃的水浴环境中过夜。
采用实验室自制的C18固相萃取柱对得到的酶解产物除盐纯化,真空干燥后存于-20℃待用。
1.3 胍基化标记
将O-甲氧脲试剂用3~4 mol/L的NaOH溶液配制成浓度为0.8 mol/L的溶液,调节 pH值至11.0。取干燥的蛋白质酶解产物500 μg,加入制备好的 O-甲氧脲溶液 1 000 μL,用 0.1 mol/L pH 11.0 的 NaHCO3调节 pH 值至10.0 ~11.0。在65℃的水浴环境下反应10 min后除盐,干燥待用。
1.4 分析条件及数据库检索
将长15 cm、内径75 μm的毛细管柱在高温下拉成内径约为3 μm的尖头,用气压法将C18AQ填料压入柱中制成C18毛细管柱,用于上样。用HPLC系统分离处理过的HeLa细胞蛋白质酶解产物溶液。样品以5 μL/min的流速进样2 min到富集柱上。以250 nL/min的流速对富集的肽段进行洗脱。流动相A是0.1%(v/v)的甲酸水溶液,流动相B是0.1%(v/v)甲酸乙腈溶液。洗脱梯度为:0~2 min,0%B~3%B;2~92 min,3%B~35%B;92~95 min,35%B~80%B;80%B保持10 min;105~110 min,80%B~100%A;100%A保持10 min。
采用LTQ Velos质谱仪的正离子模式进行肽段检测,喷雾电压为1.8 kV。采用数据依赖模式进行采集,包含一次MS全扫描(m/z 425~2 000),分辨率为60 000,对其中丰度前10的峰进行二级MS/MS扫描分析。
实验所得的MS2数据用Maxquant(version 1.4.1.2)软件转化成“* .mgf”文件,再通过 Mascot(version 2.3.0)在 人 源 蛋 白 质 数 据 库 (IPI human v.3.52.fasta,包含 55 303 个条目)进行检索。选择胰蛋白酶酶切,允许最多两个漏切位点,固定修饰设定为半胱氨酸碘代酰胺烷基化(57 Da),可变修饰为甲硫氨酸氧化(16 Da)及赖氨酸侧链和肽链N端胍基化(47 Da),母离子的质量容忍偏差为10 ppm,碎片离子为0.05 Da。导出肽段时通过设置score>25、P<0.01控制假阳性率(FDR)<1%。
2 结果与讨论
2.1 胍基化的标记效率和选择性
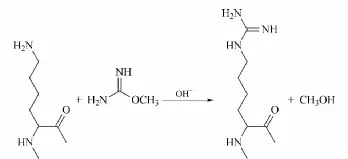
图1 胍基化反应原理图Fig.1 Schematic diagram of the guanidination
如图1所示,经胍基化反应后,多肽侧链上的赖氨酸转化为高精氨酸。取按实验部分所述方法处理得到的胍基化肽段25 μg,复溶于0.1%(v/v)甲酸水溶液中,利LC-MS/MS进行分析,通过检索数据库鉴定肽段。为了考察标记效率,需要将标记的和未标记的肽段分别鉴定出来,在检索时将赖氨酸侧链和肽链N端胍基化(47 Da)设定为可变修饰。得到的结果见表1,其中用于统计标记效率的赖氨酸包括位于C端的赖氨酸(如IGAEVYHNLK)和由于漏切产生的位于肽段中间的赖氨酸(如LKEIVTNFLAGFEA)。测得胍基化效率为96.8%,比文献报道的平均标记效率(95%)高[11],说明该反应比较完全。肽链N端被标记比例只占6.8%,说明胍基化具有很好的选择性。由于标记效率较高、选择性好,在进行数据库检索时,我们将赖氨酸侧链胍基化(47 Da)设为固定修饰。
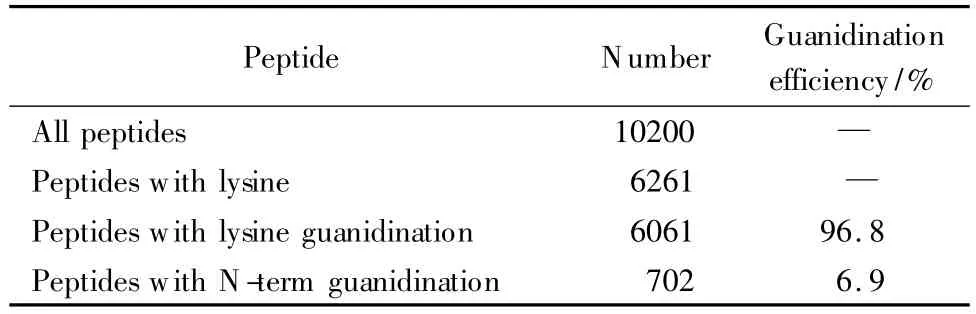
表1 HeLa细胞蛋白质酶解肽段胍基化反应的标记效率和选择性Table 1 Labeling efficiency and selectivity of the guanidination of peptides in HeLa cell digest
2.2 胍基化对肽段质谱鉴定的影响
为了考察胍基化对肽段质谱鉴定的影响,将提取的HeLa细胞蛋白质用胰蛋白酶酶解后平行分为两份,其中实验组进行胍基化处理,对照组不做处理。两份样品分别用LC-MS/MS分析,通过数据库检索鉴定肽段。由于胍基化仅发生在赖氨酸的侧链上,我们将所鉴定的肽段分成3类:(1)仅在C端有赖氨酸的肽段,如IGAEVYHNLK;(2)由于漏切产生的中间位置含有赖氨酸的肽段,如LKEIVTNFLAGFEA;(3)不含赖氨酸的肽段,如YNQLLR。如表2所示,实验组与对照组相比,胍基化后鉴定到的第1类肽段占总肽段的比例由51.7%增加到57.3%,说明胍基化能在一定程度上提高这一类肽段的鉴定灵敏度。
进一步比较实验组与对照组所鉴定到的以上3类肽段的互补性。胍基化前后分别鉴定到肽段3 633条和3 490条,有1 884(35.9%)条肽段在两组实验中均被鉴定到,1 749(33.4%)条肽段仅在对照组被鉴定到,1 606(30.7%)条肽段仅在实验组被鉴定到(见图2a)。两组实验分别鉴定到仅在C端有赖氨酸的肽段为1 877条和1 998条,有 983(34.0%)条肽段在两组实验中均被鉴定到,894(30.9%)条肽段仅在对照组被鉴定到,1 015(35.1%)条肽段仅在实验组被鉴定到(见图2b)。两组实验分别鉴定到中间位置含赖氨酸的肽段为410条和310条,有117(19.4%)条肽段在两组实验中均被鉴定到,293(48.6%)条肽段仅在对照组被鉴定到,193(32.0%)条肽段仅在实验组被鉴定到(见图2c)。两组实验分别鉴定到不含赖氨酸的肽段为1 345条和1 183条,有728(40.4%)条肽段在两组实验中均被鉴定到,617(34.3%)条肽段仅在对照组被鉴定到,455(25.3%)条肽段仅在实验组被鉴定到(见图2d)。通过对比可以看出,包含赖氨酸的肽段(第1和2类)比不包含赖氨酸的肽段(第3类肽段)中共同鉴定到的肽段比例少。这可能是由于胍基化将赖氨酸转化为高精氨酸后,使肽段的性质发生了变化,从而改变了其在二级质谱中的碎裂行为,因此在实验组鉴定到很多新的肽段(1 015条,占35.1%)。而在对照组中,由于胍基化只选择性地发生在赖氨酸的侧链上,因此对于不包含赖氨酸的肽段,其质谱鉴定结果前后差异较小,在对照组和实验组中共同鉴定到的肽段数目较多(728条,占40.4%)。可见,胍基化改变了带赖氨酸残基的肽段性质,由此可以提高鉴定的互补性。
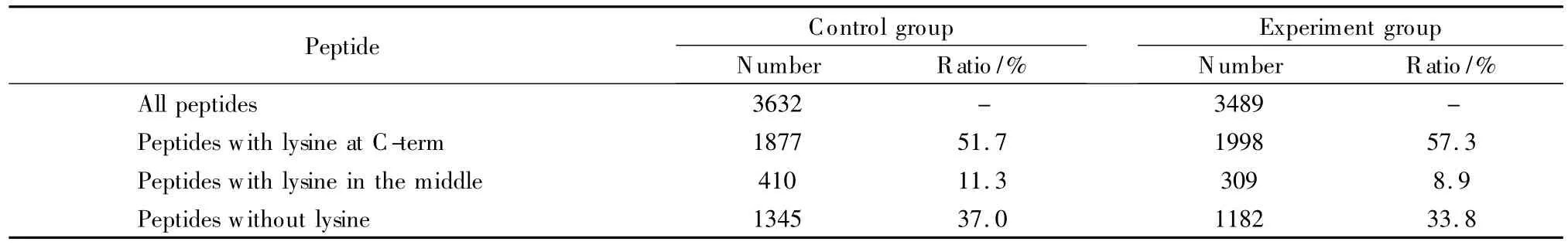
表2 HeLa细胞蛋白质酶解肽段胍基化前后肽段鉴定数量的比较Table 2 Comparison of peptides identified in HeLa cell digest before and after the guanidination
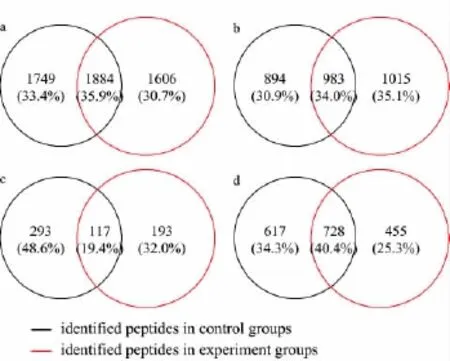
图2 HeLa细胞蛋白质酶解肽段在胍基化前后质谱鉴定结果中(a)肽段的互补性、(b)C端有赖氨酸的肽段的互补性、(c)中间含赖氨酸的肽段的互补性及(d)不含赖氨酸的肽段的互补性Fig.2 Overlap of(a)identified peptides,(b)peptides with lysine at C-term,(c)peptides with lysine in the middle and(d)peptides without lysine in HeLa cell digest before and after the guanidination
2.3 胍基化对肽段在二级质谱中碎裂的影响
为了进一步探究上述胍基化影响肽段质谱响应的原因,对对照组和实验组二级质谱图的离子碎裂情况进行了统计学分析。质谱检测时,质子化的肽段沿着肽链骨架发生断裂,因发生断裂的位置不同而产生6种类型的离子:N端产生a、b、c离子,C端产生相应的 x、y、z离子[12]。在本实验条件下,产生的主要为b、y离子。运用本实验室开发的程序,我们可以统计出每条肽段二级质谱图鉴定到的b、y离子的数目。如肽段VNQIGSVTESLQACK,平均鉴定到的b、y离子数目分别是13和15。按照b/(y+b)计算其b离子所占的比例为46.4%,按照y/(y+b)计算y离子所占的比例为53.6%。对所有鉴定到的肽段的b、y离子比例进行概率统计,结果见图3。
C端为赖氨酸的肽段在胍基化后,y离子比例大于40%的肽段在所有肽段中所占的比重都相应提高,如图3a所示,其总比重由原来的38.8%上升至50.0%。而b离子占主体的肽段比原来均有所减少。说明胍基化产生了更多的y离子信息。这可能是由于赖氨酸的胍基化发生在肽段C端,其碱性更强且容易捕获离子化质子,在正离子检测模式下,这类肽段骨架断裂主要发生在C端,产生了更多的y离子。由于这部分新增的离子碎裂信息,提高了这类肽段的质谱鉴定灵敏度,和对照组相比,鉴定结果中出现了许多新的肽段信息。
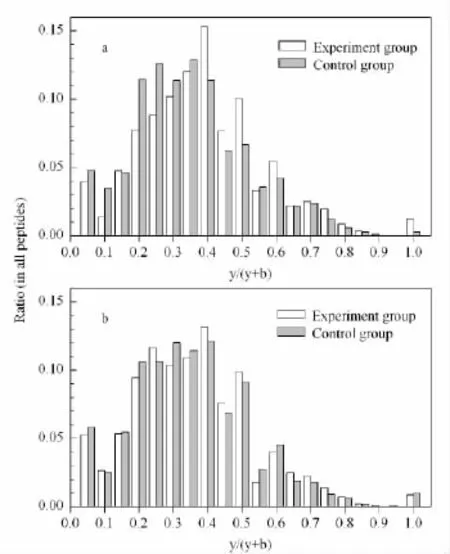
图3 (a)C端为赖氨酸的肽段和(b)不含赖氨酸的肽段胍基化前后y离子的比例分布图Fig.3 R atio distributions of y ions of(a)peptides with lysine at C-term and(b)peptides without lysine before and after the guanidination
我们进一步对不含有赖氨酸的肽段进行分析,其y离子比例大于40%的肽段在所有肽段中所占的比例在胍基化前后分别为29.5%和31.1%,没有明显变化,如图3b所示,说明胍基化对这类肽段的质谱检测基本没有影响。这可能是由于胍基化只选择性地发生在赖氨酸的侧链,因此不含赖氨酸的肽段的离子碎裂过程不受影响。
3 结论
本文探讨了胍基化对复杂生物样品蛋白质组分析的影响,发现胍基化反应可以选择性地在赖氨酸侧链上发生,使C端为赖氨酸的肽段在总肽段中所占的比例从51.7%上升到57.3%,在一定程度上提高了质谱鉴定的灵敏度和互补性。对鉴定到肽段的b、y离子比例进行统计学分析,发现胍基化后产生了更多的y离子碎裂信息,这可能是由于胍基化后赖氨酸碱性提高造成的。
[1] Zhang Y,Yang P Y,Lu H J.Chinese Journal of Chromatography(张莹,杨芃原,陆豪杰.色谱),2013,31(6):503
[2] Zhou Y,Shan Y C,Zhang L H,et al.Chinese Journal of Chromatography(周愿,单亦初,张丽华,等.色谱),2013,31(6):496
[3] Kimmel J R.Methods Enzymol,1967,11:584
[4] Keough T,Lacey M P,Youngquist R S.Rapid Commun Mass Spectrom,2000,14(24):2348
[5] Brancia F L,Montgomery H,Tanaka K,et al.Anal Chem,2004,76(10):2748
[6] Ye M L.Chinese Journal of Chromatography(叶明亮.色谱),2013,31(1):1
[7] Dongré A R,Jones J L,Somogyi Á,et al.Chem Soc,1996,118(8):8365
[8] Hogan J M,McLuckey S A.J Mass Spectrom,2003,38(3):245
[9] Marcotte E M.Nat Biotech,2007,25(7):755
[10] Zhang M Y,Wang C L,Bian Y Y,et al.Chinese Journal of Chromatography(张曼玉,王春丽,边阳阳,等.色谱),2011,29(8):706
[11] Wang X Y,Zhang Y J,Wang X,et al.Journal of Chinese Mass Spectrometry Society(王雪颖,张养军,王昕,等.质谱学报),2011,32(5):257
[12] Hennrich M L,Boersema P J,van den Toorn H,et al.Anal Chem,2009,81(18):7814

