一株弗氏链霉菌噬菌体的分离及其分子特性研究
白宇 徐春燕 苏建宇
(宁夏大学生命科学学院 西部特色生物资源保护与利用教育部重点实验室,银川 750021)
一株弗氏链霉菌噬菌体的分离及其分子特性研究
白宇 徐春燕 苏建宇
(宁夏大学生命科学学院 西部特色生物资源保护与利用教育部重点实验室,银川 750021)
鉴定一株从泰乐菌素异常发酵液中分离出的烈性噬菌体SF1并研究其分子特性。采用双层平板法分离纯化弗氏链霉菌的噬菌体SF1,通过负染法电镜观察噬菌体SF1的大小和形态;测定噬菌体SF1对氯仿和异丙醇的敏感性分析其包膜结构;通过SDS-PAGE分析噬菌体的结构蛋白;提取噬菌体基因组DNA进行限制性片段长度多样性分析。噬菌体SF1的噬菌斑直径约11-15 mm,边缘整齐透明,为烈性噬菌体;SF1为长尾型,头部为多面体,直径约24-30 nm,尾长约52-64 nm,尾宽约为3-5 nm;对氯仿和异丙醇不敏感,无包膜;基因组为双链DNA,大小约为35 kb;至少含有14种相对分子量为14.8-59.5 kD的结构蛋白。
弗氏链霉菌 噬菌体 泰乐菌素
作为高效的动物疾病防治药物,泰乐菌素是一种十分重要的16元大环内酯类畜禽用抗生素,是国家规划重点发展的兽药产品[1],其工业生产菌株是弗氏链霉菌[2]。链霉菌发酵生产过程中易感染噬菌体,严重影响菌体的生长和产物的合成,导致发酵异常,造成严重损失。因此,分离弗式链霉菌噬菌体并考察其生物学特性对泰乐菌素发酵生产中预防和控制噬菌体感染具有非常重要的意义。但是,目前发酵工业生产中针对噬菌体感染的报道主要集中在谷氨酸、乳酸、丙酮、丁醇等产品[3,4],国内外对泰乐菌素发酵生产过程中噬菌体感染与防治的研究甚少,未见相关报道。
本研究针对从泰乐菌素异常发酵液中分离得到的一株烈性噬菌体,对其分子特性进行研究,旨在
为弗氏链霉菌噬菌体的防治提供理论参考。
1 材料与方法
1.1 材料
1.1.1 噬菌体来源 弗式链霉菌噬菌体SF1由本实验室分离自泰乐菌素异常发酵液。
1.1.2 培养基和主要试剂 双层平板培养基:上层高氏一号培养基(含0.7%琼脂),下层高氏一号培养基(含2%琼脂),液体培养基:菌丝体培养基[5],SM缓冲液[6];限制性核酸内切酶Xho I、BamH I、Bgl II、Hind III、EcoR I和Xba I购自Fermentas公司;DNase I、RNase A、蛋白酶K和饱和平衡酚购自Sigma公司。DL15 000 DNA marker、λ DNA/Hind III marker等核酸分子量标准购自TaKaRa公司。
1.2 方法
1.2.1 噬菌体分离 将泰乐菌素异常发酵液以12 000 r/min离心5 min去除菌体,取上清液经0.22 μm微孔滤膜过滤,滤液即为噬菌体原液,置于4℃保存。
将宿主弗氏链霉菌与噬菌体原液混合后利用双层平板法[6]培养48 h后,挑取直径较大的噬菌斑与已在液体培养基中培养至对数期的宿主菌液混合,于28℃,220 r/min摇床中培养48 h后,12 000 r/min离心5 min去除菌体,取上清液经0.22 μm微孔滤膜过滤,再用双层平板法观察噬斑的形态。
1.2.2 噬菌体效价测定 纯化后的噬菌体悬液按10倍稀释法进行稀释,取适当稀释倍数的噬菌体液100 μL与弗氏链霉菌孢子悬液100 μL混匀,铺双层平板,28℃培养48 h后计数噬菌斑。效价(pfu/mL)=噬菌斑数×稀释倍数×10。
1.2.3 噬菌体电镜形态学观察 噬菌体悬液经浓缩、密度梯度离心和透析后制成高浓度噬菌体颗粒悬液[6],将该悬浮液制片后采用磷钨酸染色,自然干燥后在透射电子显微镜(日立H-7650)下观察噬菌体形态。
1.2.4 噬菌体包膜分析 将噬菌体悬液100 μL加入到900 μL氯仿中,摇匀后于28℃分别作用0.5、1.5、3 h,静置分层后取上层水相部分测定噬菌体效价,以未加氯仿的噬菌体悬液为对照。
将噬菌体悬液100 μL置于900 μL不同浓度的异丙醇(50%、75%、100%)中,摇匀后于28℃分别作用0.5、1.5和3 h,测定噬菌体效价,以未加异丙醇的噬菌体悬液为对照。
1.2.5 噬菌体蛋白SDS-PAGE分析 将浓缩纯化的噬菌体颗粒充分加热变性,于室温冷却后进行SDSPAGE电泳,分离胶和浓缩胶的浓度分别为12%和4%。电压设定90 V,待样品电泳至分离胶界面时,调电压至110 V,继续电泳至溴酚蓝指示剂达凝胶下缘,停止电泳。凝胶以考马斯亮蓝染色4 h,转入脱色液脱色过夜后置于双蒸水中保存并摄像。
1.2.6 噬菌体核酸提取和定性 噬菌体核酸提取方法参照文献[7]进行,略有改动。为测定核酸单双链,按照文献[8]的方法将提取的噬菌体核酸置于无菌EP管中,水浴加热,每升高10℃,取1 μL核酸检测其在260 nm处的吸光值,试验重复3次,并绘制吸光值和温度的曲线图。
噬菌体核酸分别用DNaseⅠ、RNase A在37℃消化1 h后进行电泳观察结果。噬菌体核酸分别用6种限制性内切酶Xho I、BamHⅠ、Bgl Ⅱ、Hind Ⅲ、EcoRⅠ和XbaⅠ于37℃酶切5 h,根据酶切图谱鉴定核酸近似大小。
2 结果
2.1 噬菌体SF1的噬菌斑特性
SF1在双层平板上形成的噬菌斑(28℃倒置培养48 h)(图1)显示,噬菌斑形态、大小基本一致,直径约11-15 mm,边缘整齐透明,为无晕环透明圆斑,呈现典型的烈性噬菌体的噬菌斑特征。
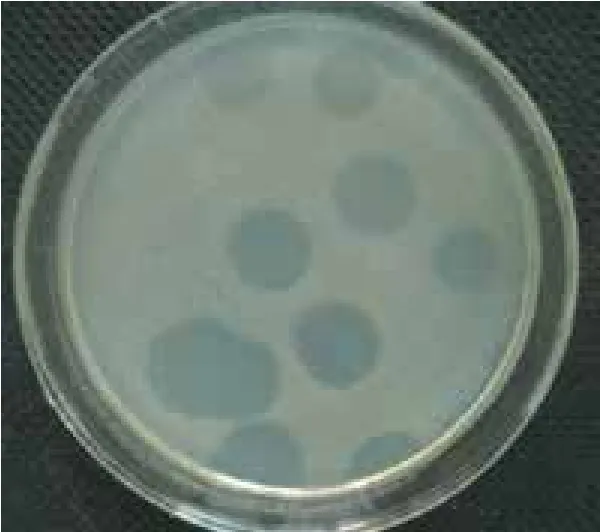
图1 弗氏链霉菌噬菌体SF1的噬菌斑
2.2 噬菌体SF1的电镜观察
纯化后的噬菌体SF1颗粒经负染后在透射电
镜下观察其形态,结果(图2)显示,噬菌体SF1与大多数链霉菌噬菌体形态相似,为蝌蚪状噬菌体,由多面体的头部和细长的尾部组成。头部直径约为24-30 nm,尾部相对细长,尾长约为52-64 nm,尾宽约为3-5 nm。按照国际病毒分类委员会(International Committee on Taxonomy of Viruses,简称ICTV)第8次报告的最新病毒分类系统的标准[9],噬菌体SF1从形态上分类属于长尾噬菌体科(Siphoviridae)。

图2 噬菌体颗粒电镜(30 000×)
2.3 噬菌体SF1的包膜分析
有些噬菌体在核衣壳之外具有包膜,包膜来源于宿主细胞膜,由脂类和蛋白质组成,对氯仿等有机溶剂敏感,因此可以通过检测噬菌体对氯仿的敏感性研究噬菌体是否具有包膜。用高比例氯仿处理噬菌体SF1,一段时间后测定噬菌体效价,结果(图3)显示,与对照相比,用氯仿处理噬菌体0.5-3 h后,噬菌体的效价均处在同一数量级内。将试验组与对照组数据两两进行显著性差异分析,结果表明试验组和对照组的噬菌体效价相比无显著性差异(α=0.05),说明噬菌体SF1对氯仿不敏感,推测噬菌体SF1的核衣壳外无脂质包膜。

图3 氯仿对噬菌体SF1效价的影响
通常异丙醇对亲脂性病毒灭活效果好,而对亲水性病毒效果较差。噬菌体SF1对异丙醇的抗性如图4所示,噬菌体SF1经异丙醇处理不同时间后下降1-4个数量级,说明噬菌体SF1对异丙醇有一定的敏感性,但并不能使噬菌体SF1完全灭活。推测噬菌体SF1的重要组成部分可能不含脂质,符合细菌病毒dsDNA病毒、尾病毒目噬菌体的特点。

图4 异丙醇对噬菌体SF1效价的影响
2.4 噬菌体SF1结构蛋白分析
将高浓度噬菌体颗粒进行SDS-PAGE电泳分析,结果(图5)显示,在凝胶上出现了14条蛋白质条带。根据文献报道,纯化的噬菌体颗粒SDS-PAGE电泳图谱上清晰可见的往往是噬菌体的结构蛋白[10]。经Quantity One软件分析,蛋白的相对分子量在14.8-59.5 kD之间,其中14.8、16.0和34.6 kD对应的3条带最明显,提示14.8、16.0和34.6 kD所对应的3种蛋白含量较高,可能是噬菌体SF1的主要衣壳蛋白。
2.5 噬菌体SF1的核酸类型分析
核酸酶对核酸的水解特征可用来判断核酸类型。提取噬菌体SF1的基因组并分别用DNaseⅠ和RNase A消化,结果表明其核酸可被DNaseⅠ消化,而不能被RNase A消化(图6),说明噬菌体SF1的核酸类型为DNA。这与目前已在GenBank上登录的另外14株链霉菌噬菌体核酸类型一致。
双链DNA加热变性时双螺旋解链会使其在260 nm处吸光值增高,称之为DNA链的“增色效应”(Hyperchromic effect)[8]。噬菌体SF1的基因组在
260 nm的吸光值随温度的变化如图7所示,当温度的升高到80℃-90℃时,噬菌体DNA溶液在260 nm处吸光值显著增高,说明噬菌体SF1的基因组类型为dsDNA。具备dsDNA病毒中的尾病毒目长尾病毒科的特征,并与按照形态初步分类的结果相同。
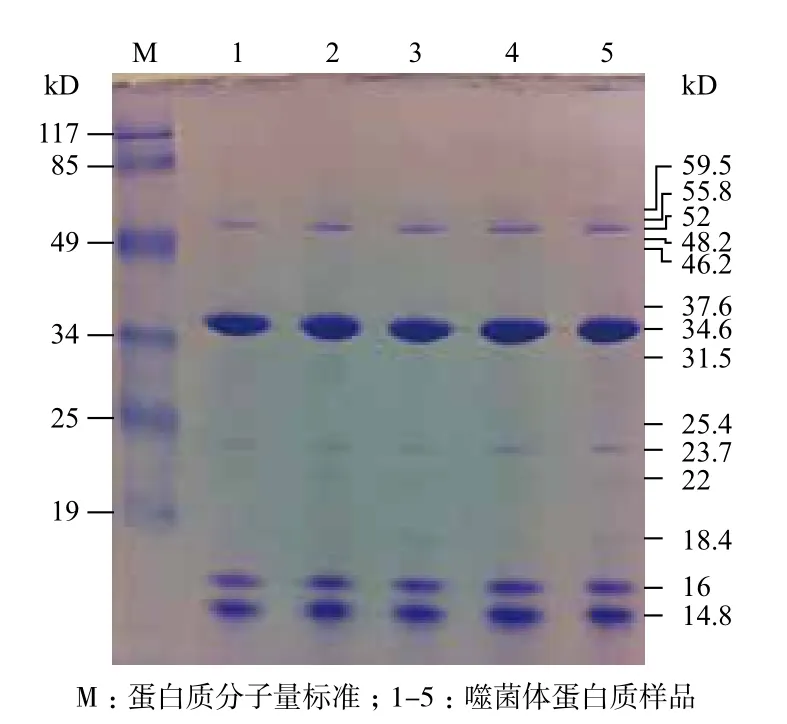
图5 噬菌体SF1结构蛋白的SDS-PAGE分析

图6 噬菌体基因组经核酸酶消化电泳图

图7 不同温度下噬菌体基因组在260 nm的吸光值
噬菌体SF1基因组经XhoⅠ、BamHⅠ、Bgl Ⅱ、Hind Ⅲ、EcoR I和Xba I六种限制性内切酶酶切后经琼脂糖凝胶电泳分析表明,噬菌体SF1基因组具有XhoⅠ、BamHⅠ、Bgl Ⅱ和Hind Ⅲ的多个酶切位点,但不具有EcoRⅠ和XbaⅠ的酶切位点(图8)。噬菌体SF1的基因组能够被XhoⅠ切割成3个片段;被BamHⅠ切割成4个片段;被Bgl Ⅱ切割成4个片段;被Hind Ⅲ切割成至少4个片段。由于这4种限制性内切酶能够识别并切割双链DNA分子特定的核苷酸序列,所以进一步证明了噬菌体SF1核酸类型为双链DNA,与迄今所报道的链霉菌噬菌体情况一致。噬菌体SF1的基因组酶切图谱与已发表的其它链霉菌噬菌体DNA的酶切图谱类型有较大差异[11-15],酶切图谱显示噬菌体SF1很有可能是一株未报道的新噬菌体。在已知的链霉菌噬菌体中,其基因组大多具有EcoR I酶切位点而没有XhoⅠ酶切位点,且其他3种限制性内切酶的酶切图谱与噬菌体SF1的酶切结果也有很大差异;只有金色链霉菌噬菌体μ1/6的基因组有XhoⅠ酶切位点,但酶切片段大小与噬菌体SF1的不相同,而且噬菌体μ1/6的基因组不能被Hind Ⅲ酶切,噬菌体SF1的基因组则可以。
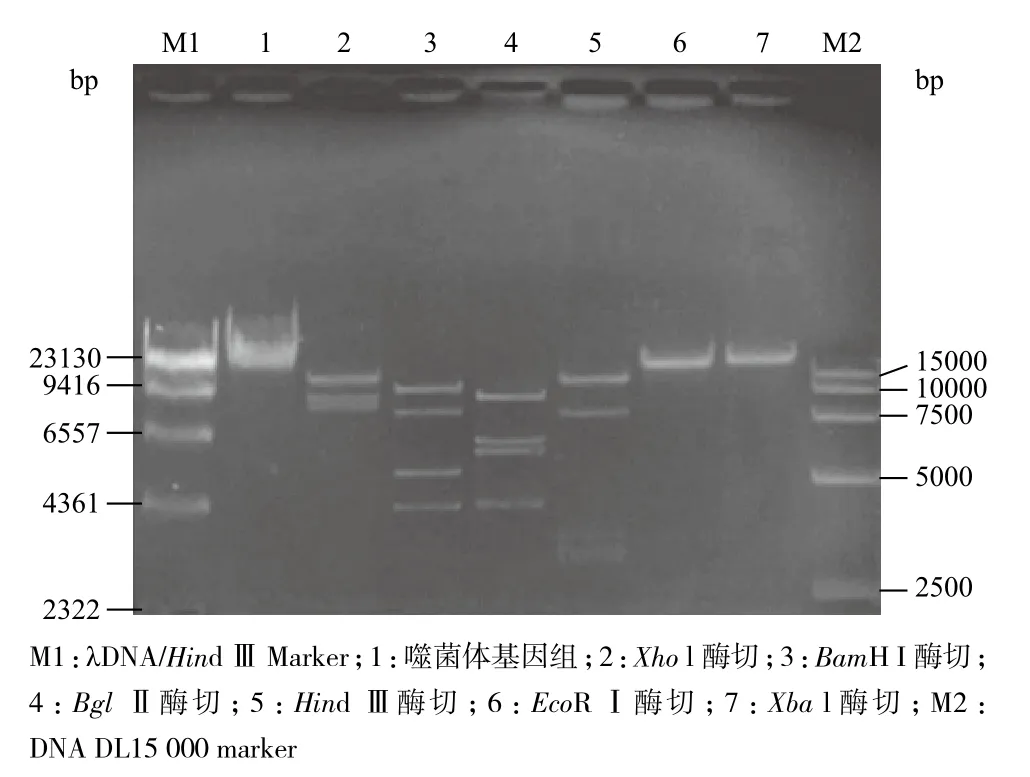
图8 噬菌体SF1基因组限制性酶切图谱
用λ DNA/Hind Ⅲ酶切片段作为分子量标准,经XhoⅠ、BamHⅠ、Bgl Ⅱ和Hind Ⅲ酶切后得到不同片段的分子量相加,初步计算噬菌体SF1基因组分子量约为35 kb。
3 讨论
近年来,随着人们对链霉菌噬菌体研究工作的日益重视,研究内容已由早期的分离鉴定逐渐发展为对其基因组学和功能基因组学的深入研究。目前在GenBank中登录基因组信息的链霉菌噬菌体有14株,其寄主分别为阿维菌素链霉菌、变铅青链霉菌、金色链霉菌、天蓝色链霉菌、委内瑞拉链霉菌等,核酸类型全部为双链DNA。与以λ噬菌体为代表的大肠杆菌噬菌体、乳酸菌噬菌体以及分支杆菌噬菌体的研究相比,目前对链霉菌噬菌体,尤其是工业链霉菌噬菌体的研究仍远远落后。因此,任何新的链霉菌噬菌体的分子特性研究都具有重要的学术意义。新的链霉菌噬菌体基因组信息也将有助于我们研究噬菌体宿主的相互交叉作用、开发链霉菌属遗传交换分析的分子工具以及开展辅助链霉菌属的分类分群工作[16]。
通过对分离出的弗氏链霉菌噬菌体分子特性的研究并查阅相关文献[11-15],发现噬菌体SF1与已发现的链霉菌噬菌体在形态特征和核酸酶切特性上均有所差异,因此噬菌体SF1很可能是一株未经报道的新噬菌体。为全面认识弗氏链霉菌噬菌体SF1的遗传背景、基因调控方式以及与其宿主菌之间的相互关系,全基因组测序工作非常必要,相关研究正在开展。
4 结论
本研究首次分离出一株以弗氏链霉菌为宿主的噬菌体SF1,经鉴定其为烈性噬菌体,属于dsDNA病毒中的尾病毒目长尾病毒科。SF1的基因组大小约为35 kb,至少含有14种结构蛋白,与已发现的链霉菌噬菌体在形态特征和核酸酶切特性上均有一定的差异。
[1]Bate N, Butler AR, Smith IP, et al. The mycarose-biosynthetic Genes of Steptomyces fradiae producer of tylosin[J]. Microbiology, 2000, 146(1):139-146.
[2]赵东峰, 任翔, 朱丽. 泰乐菌素及其衍生物研究进展[J]. 医药产业资讯, 2006, 3(15):46-48.
[3]Proux C, Sinderen DV, Suarez J, et al. The dilemma of phage taxonomy illustrated by comparative genomics of Sfi21-like Siphoviridae in lactic acid bacteria[J]. Bacteriology, 2002, 184(21):6026-6036.
[4]南南, 曹放, 沈俊涛, 等. 一株产1, 3-丙二醇的克雷伯氏菌的噬菌体生物学特性[J]. 微生物学报, 2013, 53(9):943-949.
[5]李晓华, 周秀芬, 邓子新. 小单孢菌40027菌株噬菌体的分离及其生物学特性的研究[J]. 微生物学报, 2007, 47(3):482-485.
[6]萨姆布鲁克 J, 拉塞尔 DW. 分子克隆实验指南[M]. 第3版.北京:科学出版社, 2002:170-195.
[7]张琳, 乐率, 卢曙光, 等. 铜绿假单胞菌噬菌体PaP4的分离与鉴定[J]. 微生物学通报, 2013, 40(4):609-616.
[8]周爱儒, 查锡良, 于秉治. 生物化学[M]. 北京:人民卫生出版社, 2001:45-46.
[9]洪健, 周雪平. ICTV第八次报告的最新病毒分类系统[J]. 中国病毒学, 2006, 21(1):84-96.
[10]Zhu JM, Rao XC, Tan YL, et al. Identification of lytic bacteriophage MmP1, assigned to a new member of T7-like phages infecting Morganella morganii[J]. Genomics, 2010, 3(96):167-172.
[11]Smith MCM, Burns RN, Stuart E, et al. The complete genome sequence of the Streptomyces temperate phage φC31:evolutionary relationships to other viruses[J]. Nucleic Acids Research, 1999, 27(10):2145-2155.
[12]Dessela WV, Mellaerta LV, Liesegang H, et al. Complete genomic nucleotide sequence and analysis of the temperate bacteriophage VWB[J]. Virology, 2005, 331(2):325-337.
[13]Farkasovska J, Klucar L, Vlcek C, et al. Complete genome sequence and analysis of the streptomyces aureofaciens phage μ1/6[J]. Folia Microbiol, 2007, 52(4):347-358.
[14]Wang SW, Qiao XW, Liu XX, et al. Complete genomic sequence analysis of the temperate bacteriophage phiSASD1 of Streptomyces avermitilis[J]. Virology, 2010, 403(1):78-84.
[15]Monson R, Salmond GPC. Genome sequence of a new streptomyces coelicolor generalized transducing bacteriophage, Φcam[J]. Virology, 2012, 86(24):13860.
[16]彭云霞, 姜怡, 段淑蓉, 等. 稀有放线菌的选择性分离方法[J].云南大学学报, 2007, 29(1):86-89.
(责任编辑 李楠)
Identification and Molecular Characterization of a Phage from Streptomyces fradiae
Bai Yu Xu Chunyan Su Jianyu
(Key Laboratory of Ministry of Education for Conservation and Utilization of Special Biological Resources in the Western,School of Life Science,Ningxia University,Yinchuan 750021)
It was to identify and explore the molecular characterization of a phage SF1, which was isolated from Streptomyces fradiae fermentation broth during the failed industrial production of tylosin. Phage SF1 was characterized by observing the shape of plaque, titering on double-layer agar culture plate. The morphology of SF1 was observed by the electron microscopic observation after negative staining. The sensitivity of the phage to chloroform and isopropyl alcohol was tested to detect its envelope. The structural proteins analysis of phage SF1 was carried out by SDS-PAGE. Restriction fragment length polymorphism(RFLP)analysis of genomic DNA was further performed to characterize the phage. Results showed that the plaque(11-15 mm in diameter)is neat and transparent. SF1 has a polyhedral head(about 24-30 nm in diameter)and a long tail(about 52-64 nm long and 3-5 nm wide). It is insensitive to chloroform and isopropyl alcohol. The genome of SF1 consists of double-stranded DNA and is approximately 35 kb. Capsid protein analysis shows a higher level of 14 major protein bands ranging from 14.8-59.5 kD, which is presumed to be the structural proteins of SF1.
Streptomyces fradiae Phage Tylosin
10.13560/j.cnki.biotech.bull.1985.2014.12.028
2014-04-25
白宇,女,硕士研究生,研究方向:微生物与基因工程;E-mail:baiyu1119@163.com
苏建宇,男,博士,教授,研究方向:工业微生物;E-mail:su_jy@nxu.edu.cn

