黏细菌:天然的制药厂
赵婷峰 龚国利
(陕西科技大学生命科学与工程学院,西安 710021)
黏细菌:天然的制药厂
赵婷峰 龚国利
(陕西科技大学生命科学与工程学院,西安 710021)
黏细菌及其抗性代谢产物的药用开发已经成为近年来科研工作者研究的热点之一。黏细菌是一类重要的天然产物生产者,它产生的次级代谢产物无论是在化学结构还是生物活性上都具有丰富的多样性。代谢产物的多样性及广谱活性,在开发成药物方面具有巨大的潜力。主要综述了黏细菌独特的细胞行为,产生次级代谢产物的杰出能力,并且阐述了黏细菌药物埃博霉素的研发进展,最后对黏细菌次级代谢产物开发应用潜力进行了展望。
黏细菌 次级代谢产物 药物研究 埃博霉素
一个多世纪以来,从植物和微生物中发现的天然产物在药物研究中发挥了举足轻重的作用[1-3]。近年来,黏细菌(Myxobacteria)作为一类重要天然产物的生产者,可产生异常丰富的次级代谢产物[4]。黏细菌的抗生素产生菌的比例高于目前已知的产生抗生素最多的放线菌(Actinomycetes),如纤维堆囊菌(Sorangium cellulosum)中能产生抗生素的菌株,几乎可以高达百分之百,目前所发现的生物活性物质的数量已达400种以上[5]。更重要的是,研究调查表明大部分黏细菌次级代谢产物可以预防人类疾病如癌症,以及细菌和病毒感染等疾病[6-8],因此受到越来越多的科技工作者的高度重视,正在成为专家学者们的研究热点。
1 黏细菌的复杂细胞行为
黏细菌是一类高等的原核生物,是一类能够滑动运动的革兰氏阴性杆菌,在系统分类上属于多变细菌(Proteobacteria)[9]。黏细菌基因组庞大,约为9 454-10 010 kb,GC含量在64%-75%之间,它具有复杂的多细胞行为和形态发生,在细胞分化、发育和生物进化中占有重要地位,主要生存在富有微生物和有机物的地方如土壤、腐烂的植物,草食动物的粪便,活树或死树的树皮上等[10,11]。
这种社会性细菌主要通过轴突细胞的运动来移动,即所谓的滑翔[12,13],虽然细胞的生长是独立的,
但是当营养缺乏时[14],它们可以成群地移动来捕食,细胞大量聚集并简单分化后能形成一种群体细胞结构称为子实体。在共同捕食时,单个多细胞组织如波浪中的一个涟漪进行运动[13,15]。随着细胞的波浪碰撞,它们聚集生长形成子实体大小的土墩,它能够携带大约105个体,这些结构中的细胞变成黏孢子。当营养物质是可用的,细胞通过表面接触来传递信号[12]。另外,黏细菌已经进化出了复杂的包括胞内和胞外信号(包括多种蛋白质和小代谢物)的机制来控制这一过程[16]。
黏细菌子实体是黏细菌增强生存能力的方式之一:一方面子实体给抗性休眠细胞和黏孢子提供生存空间,同时细胞群体生活在共同的黏液层下以群体的力量抵抗恶劣的环境和小生境中的竞争者;另一方面子实体可以保证黏细菌群体新的生命循环是从细胞集体开始而不是以单个细胞的形式开始细胞的聚集,这种方式可以高效地产生一些胞外酶,并且与个体细胞相比群体细胞对细胞的溶解损失不太敏感。
2 黏细菌—天然的制药厂
自1977年从黏细菌中发现了第一个确定化合物结构的生物活性物质安布鲁星(Ambruticin)以来,人类已发现黏细菌中100多种次级代谢产物的基本结构和600多种结构类似物。黏细菌的代谢物无论是在其化学空间结构的多样性还是在其生物活性上都是罕见的[5,17,18]。据不完全统计,黏细菌次级代谢产物中约1/3为聚酮类(Polyketide,PK)或非核糖体肽类化合物(Non-ribosomal peptide,NRP)。实际上,大约40%的黏细菌次级代谢产物都表现出新颖的化学结构[10]。此外,与放线菌产生的代谢产物相比,黏细菌产生的大多数小分子是没有糖基化的,并且这些小分子物质的作用靶点和其他微生物所产生的一些代谢产物的作用靶点往往不同[19]。例如,线粒体呼吸抑制剂、真核细胞的蛋白质合成抑制剂、羧化酶和聚合酶抑制剂以及影响微管装配的分子。
黏细菌产生的生物活性物质种类繁多,图1中列举了部分黏细菌次级代谢产物的结构。这些次级代谢产物作用水平在多个层次上,作用机理也多种多样,根据作用机理的不同,可将其分为以下几类[18]:抑制线粒体呼吸的如黏噻唑(myxothiazol);干扰蛋白质磷酸化系统、稳定微管及抑制蛋白合成的如埃博霉素(Epothilone)、Tartrolon;干扰金属离子运输、影响脂类代谢的如Soraphen;抑制糖类代谢的如Ambruticin;抑制细胞壁合成的如Chivosazol;抑制核酸合成的如Disorazol、Ratjadon,影响细胞膜的透性和合成的如Sorangiolid,Jerangolid等。尤其是黏细菌中发现的生物活性物质有许多是抗肿瘤、抗真菌、抗病毒的活性物质,这也是黏细菌不同于其他生物活性物质产生菌的独特之处。

图1 几种来源于黏细菌的化合物的结构
虽然对于黏细菌的次级代谢产物有如此多种类的原因没有很好的阐明,但已经证明,这些代谢产物可以赋予黏细菌在土壤环境中具有一定的竞争优势,可以用来调节群体中细胞与细胞之间的相互作用,可以使它们在竞争环境中处于适当的位置,还可以用作捕猎的武器[20]。代谢产物如此复杂的化学功能需要一个同样复杂的调控网络,因此提高了其个体和群体的生存和竞争力[11]。这也体现在由黏细菌产生次级代谢产物的遗传空间上。迄今为止,报道的最大的细菌基因组是黏细菌纤维堆囊菌的基因组,这个基因组大约有20个甚至更多的次级代谢产物的编码基因[16]。黏细菌中黄色黏球菌(Myxococcus xanthus)的深入研究表明,大约18种次级代谢物的基因簇占其整个基因组的9%左右[21],这种比例超过了放线菌次级代谢产物的基因簇占其整个基因组的大约6%[22,23]。由于基因组很大,因此在不同种类的黏细菌以及同种黏细菌中不同的菌株之间都表现出较大的差异性,这似乎为黏细菌药物的勘探和开采提供了一个巨大的空间。
3 埃博霉素的研究
埃博霉素是黏细菌最有价值的次级代谢产物,最初是从纤维堆囊菌中分离得到,它是天然存在的一类十六元环的大环内酯化合物,图2是埃博霉素类化合物的结构。至今已发现由纤维堆囊菌产生的天然埃博霉素家族化合物已逾40种。埃博霉素结构中共有7个手性中心和一个大环,包括含有噻唑的侧链与一个环氧化合物。
3.1 埃博霉素的作用机理与药物特性
埃博霉素类在结构上与紫杉烷类化合物是不同的,但两者都可以促进微管的聚合和稳定,并且能结合到β-微管蛋白共同的地区[24,25],促进微管蛋白聚合,装配成微管,抑制因其解聚致使形成纺锤体的微管束排列异常,失去正常功能,导致染色体分离受阻,细胞核不能分裂,细胞有丝分裂终止在G2-M期,最终引起细胞毒性并致细胞死亡。然而这两类化合物与β-微管蛋白上氨基酸的结合位点是完全不同的[26],这就使得它们对微管蛋白的聚合以及产生细胞毒性的方式也是不同的[27,28]。
当埃博霉素被发现可以有效地对抗耐紫杉类药物的癌细胞之后,就被选为抗癌候选药物[29]。埃博霉素的几个属性对于它们作为药物制剂的发展是十分有利的。与紫彬醇相比较,埃博霉素水溶性好[30,31],注射、口服均可;结构简单,有利于化学合成及衍生化[32];在P-糖蛋白表达型的多药耐药性(MDR)细胞中也维持很大的细胞毒性。
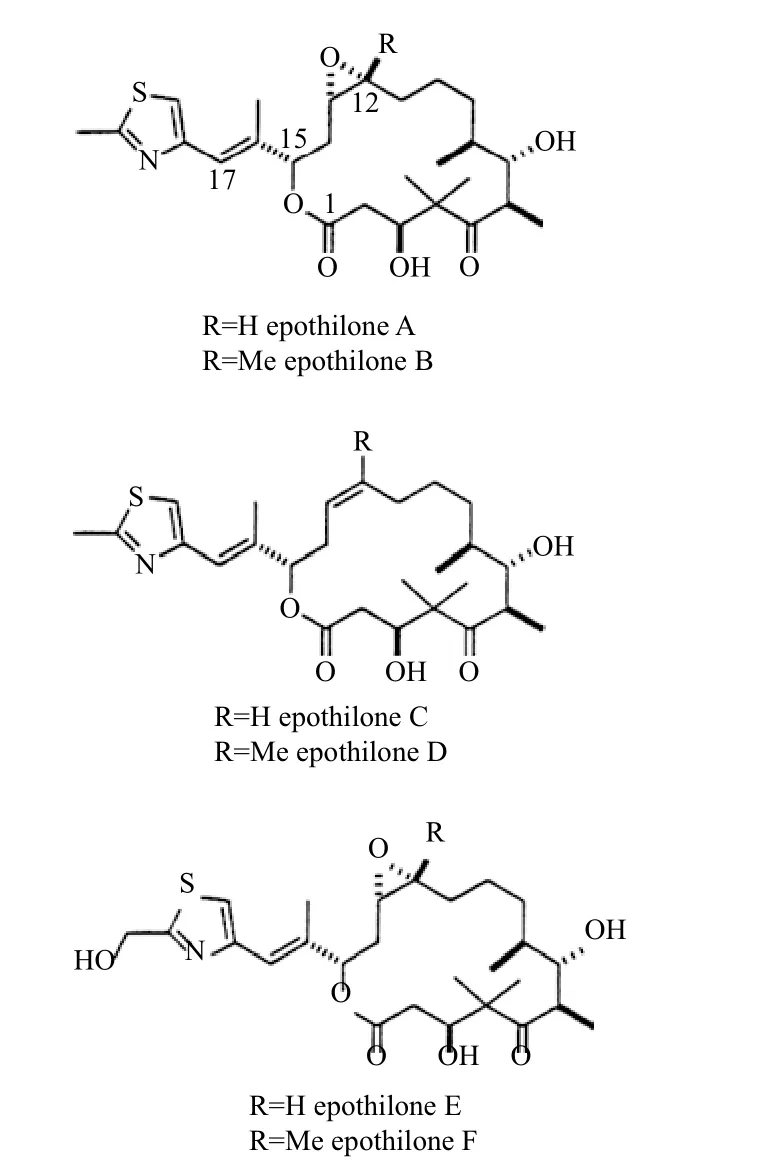
图2 埃博霉素类化合物的结构
3.2 埃博霉素及其衍生物的研究
埃博霉素中,目前已经进入临床实验的是埃博霉素B与D及其类似物。帕土匹龙(Patupilone)是一种化学合成物,与天然存在的埃博霉素B具有相同的结构。埃博霉素B的衍生物中有3种已经进入临床试验,分别是伊沙匹隆(Ixabepilone,BMS-247550,Ixempra,BMS-247550)、沙戈匹隆(Sagopilone,ZK-EPO)和BMS-310705。这些化合物的结构都是在埃博霉素B的基础上合成的化合物,与亲本结构相比,这些化合物添置或替换了部分基团。KOS862、KOS1584和KOS1903都含有埃博霉素D的结构骨架。其中,KOS862是埃博霉素D,KOS1584和KOS1903是埃博霉素D的衍生物。
埃博霉素B的衍生物已经在临床上得到了广
泛的应用。其中,Ixabepilone是利用耐紫杉醇肿瘤异种移植物开发成功的第一个抗肿瘤埃博霉素药物[33],它也是迄今为止批准上市的唯一一个埃博霉素药物。另一个埃博霉素B类似物,BMS-310705,尽管具有很好的水溶性,但是由于在前期临床试验中出现了几例严重的毒性反应[34],因而已经放弃了对其药用开发。
总之,埃博霉素及其衍生物是一种有价值的候选的抗肿瘤药物。它们具有较强的抑制微管解聚的功能,其表现出的强效抗癌活性,让人们在克服癌症的道路上再一次看到了希望及新药研发的潜力。
3.3 埃博霉素的生产研究
与大量药理及临床研究报道相比,对关于埃博霉素生产方面的报道较少。目前,埃博霉素的制备方法主要有化学合成法和生物合成法两类。就像大部分小分子药物一样,埃博霉素的化学合成法制备首先成为研究热点[35-40]。先后报道的合成路线有十几种,仅埃博霉素A的合成路线就报道过6种,埃博霉素B的合成路线报道过11种,埃博霉素C和D的全合成路线分别报道过1和2种。Danishfsky研究小组的科学家们首次成功完成了埃博霉素A、B、C、D 的全合成研究工作。另外,Nicolaou[41]研究小组在埃博霉素及其类似物的全合成研究工作中也取得了许多具有代表性的研究成果。但是由于埃博霉素化学全合成步骤繁多,得率较低,与生物发酵法相比不具备成本优势。
生物合成法制备埃博霉素成为埃博霉素生产制备技术研究的主流方向[42,43]。目前对于埃博霉素生物合成的各种尝试,所要解决的核心问题均在于如何提高产量,降低成本,实现工业化的大规模生物合成。
研究人员在克隆了埃博霉素合成基因簇后首先实现了在天蓝色链霉菌(Streptomyces coelicolor)中的异源生产,但由于埃博霉素对于宿主的细胞毒性而结果不理想,仅获得50-100 μg/L的产量[44]。2002年,Julien和Shah[45]又将埃博霉素的合成基因簇转移到黄色黏球菌(Myxococcus xanthus)中进行表达产生埃博霉素D,异源表达菌株的初始埃博霉素产量很低,只有120 μg/L,Kosan公司随后对其进行了发酵条件优化,根据Lau和Frykman等[46]的最新报道,该基因工程菌M. xanthus K111-40-1发酵合成埃博霉素D的产量为:分批发酵14 d最高达30 mg/L,连续发酵30 d最高产量达80 mg/L,半连续发酵22 d最高产量为85 mg/L[45-48]。利用原始产生菌—黏细菌纤维堆囊菌生产埃博霉素的研究国外主要由德国GBF、瑞士Norvatis和美国BMS几家实验室进行,只有Gerth等在1996年报道So ce90发酵产生埃博霉素A的检测产量为22 mg/L,埃博霉素B的检测产量为11 mg/L[42],并通过同位素标记的底物进行发酵检测,阐述了埃博霉素的碳骨架的组成[49]。国内所涉及的天然产生菌有来自于美国Emporia州立大学Peterson的纤维堆囊菌SMP44菌株[50]和来自于德国国家生物技术研究中心GBF的纤维堆囊菌So ce90菌株[42]。国内山东大学微生物技术国家重点实验室李越中教授的团队在建立我国唯一的黏细菌资源菌库的基础上,筛选到了多株能产生埃博霉素的纤维堆囊菌(如So0157-2,So0007-3,So0003-3等),为发酵制备埃博霉素提供了更加多样化的选择[51-54]。另外,本实验室在埃博霉素的发酵生产方面也开展了一些工作,已经利用基因组重组技术选育获得了埃博霉素B高产菌株[55-58],并用响应面法等统计学方法对纤维堆囊菌SoF5-76产埃博霉素B发酵培养基进行了优化[59,60],研究了多孔陶瓷吸附固定纤维堆囊菌发酵制备埃博霉素技术[61,62],最终埃博霉素B的发酵产量达到90.2 mg/L[55],这是目前国内外报道的埃博霉素的较高产量水平。目前正在开展发酵动力学模型构建和发酵过程放大研究。
4 讨论与展望
黏细菌次级代谢产物中抑制土壤细菌(Soil bacteria)的小分子占29%,抑制真菌(Fungi)的小分子占54%,并且黏细菌和其他微生物不同,它在指数生长期能具有相对较高的次级代谢产物的生产速率,这进一步使人们坚信其产生的次级代谢产物将具有非常广泛的应用[14,18]。任何掠夺性的微生物都得益于它们产生的丰富次级代谢产物,但为什么大量的这些代谢物可有效地对抗人类疾病和病原体呢?一个有吸引力的解释是,它们的许多代谢产
物具有多种活性功能,可以和多个靶点目标发生相互作用[63,64]。例如,哺乳动物细胞中的LSm1-7蛋白质复合物对于抑制丙型肝炎病毒(HCV)的转录和翻译是必要的[64]。雀麦草花叶病毒(BMV)是一种植物病毒,它可以利用酵母细胞中的同系物在酵母细胞中进行复制[65]。同样地,噬菌体Qβ与HCV和BMV一样是一个正链的RNA病毒,需要一个与LSm1-7是同系物的Hfq。因此,这些代谢产物对抗病毒药物的开发具有重要的指导意义。目前,科研工作者对黏细菌有关生物活性化合物的活性谱进行了大量研究,但是,将其代谢物制成一种适用的药物还不是一件并非易事,尤其是考虑到天然产物化学成分的复杂性、副作用以及生物利用度较差等因素。因此,为了更好地利用天然的药物工厂及新技术,如将微生物通过化学手段或生物工程合成复杂的分子结构,用生物芯片来预测药物靶点和候选药物分子存在的可能副作用等等,将在未来发挥越来越大的作用。
[1]Davies J, Ryan KS. Introducing the parvome:bioactive compounds in the microbial world[J]. ACS Chem Biol, 2012, 7(2):252-259.
[2]Mishra BB, Tiwari VK. Natural products:an evolving role in future drug discovery[J]. Eur J Med Chem, 2011, 46(10):4769-4807.
[3]Newman DJ, Cragg GM. Natural products as sources of new drugs over 147 the 30 years from 1981 to 2010[J]. J Nat Prod, 2012, 75(3):311-335.
[4]Gerth K, Pradella S, Perlova O, et al. Myxobacteria:proficient producers of novel natural products with various biological activities -past and future biotechnological aspects with the focus on the genus Sorangium[J]. J. Biotechnol, 2003(106):233-253.
[5]Bode HB, Muller R. Analysis of myxobacterial secondary metabolism goes molecular[J]. J Ind Microbiol Biotechnol, 2006, 33(7):577-588.
[6]Weissman KJ, Muller R. Myxobacterial secondary metabolites:bioactivities and modes-of-action[J]. Nat Prod Rep, 2010, 27(9):1276-1295.
[7]Gentzsch J, Hinkelmann B, Kaderali L, et al. Hepatitis C virus complete life cycle screen for identification of small molecules with pro- or antiviral activity[J]. Antiviral Res, 2011, 89(2):136-148.
[8]Nickeleit I, Zender S, Sasse F, et al. Argyrin a reveals a critical role for the tumor suppressor protein p27(kip1)in mediating antitumor activities in response to proteasome inhibition[J]. Cancer Cell, 2008, 14(1):23-35.
[9]Juana Diez1, Javier PM, Jordi M, et al. Myxobacteria:natural pharmaceutical factories[J]. Microbial Cell Factories, 2012, 11(52):1-3.
[10]Reichenbach H. Myxobacteria, producers of novel bioactive substances[J]. J Ind Microbiol Biotechnol, 2001, 27(3):149-156.
[11]Velicer GJ, Vos M. Sociobiology of the myxobacteria[J]. Annu Rev Microbiol, 2009(6):599-623.
[12]Kaiser D. Coupling cell movement to multicellular development in myxobacteria[J]. Nat Rev, 2003, 1(1):45-54.
[13]Nan B, Chen J, Neu JC, et al. Myxobacteria gliding motility requires cytoskeleton rotation powered by proton motive force[J]. Proc Natl Acad Sci USA, 2011, 108(6):2498-2503.
[14]Xiao Y, Wei X, Ebright R, et al. Antibiotic production by myxobacteria plays a role in predation[J]. J Bacteriol, 2011, 193(18):4626-4633.
[15]Berleman JE, Kirby JR. Deciphering the hunting strategy of a bacterial wolfpack[J]. FEMS Microbiol Rev, 2009, 33(5):942-957.
[16]Schneiker S, Perlova O, Kaiser O, et al. Complete genome sequence of the myxobacterium Sorangium cellulosum[J]. Nat Biotechnol, 2007, 25(11):1281-1289.
[17]Bon RS, Waldmann H. Bioactivity-guided navigation of chemical space[J]. Acc Chem Res, 2010, 43(8):1103-1114.
[18]Weissman KJ, Muller R. A brief tour of myxobacterial secondary metabolism[J]. Bioorg Med Chem, 2009, 17(6):2121-2136.
[19]Rix U, Fischer C, Remsing LL, et al. Modification of post-PKS tailoring steps through combinatorial biosynthesis[J]. Nat Prod Rep, 2002, 19(5):542-580.
[20]Davies J, Spiegelman GB, Yim G. The world of subinhibitory antibiotic concentrations[J]. Curr Opin Microbiol, 2006, 9(5):445-453.
[21]Bode HB, Muller R. The impact of bacterial genomics on natural
product research[J]. Angew Chem Int Ed, 2005, 44(42):6828-6846.
[22]Bentley SD, Chater KF, Cerdeno-Tarraga AM, et al. Complete genome sequence of the model actinomycete Streptomyces coelicolor[J]. Nature, 2002, 417(6885):141-147.
[23]Ikeda H, Ishikawa J, Hanamoto A, et al. Complete genome sequence and comparative analysis of the industrial microorganism Streptomyces avermitilis[J]. Nat Biotechnol, 2003, 21(5):526-531.
[24]Giannakakou P, Gussio R, Nogales E, et al. A common pharmacophore for epothilone and taxanes:molecular basis for drug resistance conferred by tubulin mutations in human cancer cells[J]. Proc Natl Acad Sci USA, 2000(97):2904-2909.
[25]Nettles JH, Li H, Cornett B, et al. The binding mode of epothilone A on alpha-, beta-tubulin by electron crystallography[J]. Science, 2004(5):866-869.
[26]Verrills NM, Flemming CL, Liu M, et al. Microtubule alterations and mutations induced by desoxyepothilone B:implications for drug-target interaction[J]. Chem Biol, 2003(10):597-607.
[27]Chou TC, O’Connor OA, Tong WP, et al. The synthesis, discovery, and development of a highly promising class of microtubule stabilization agents:curative effects of desoxyepothilones B and F against human tumour xenografts in nude mice[J]. Proc Natl Acad Sci USA, 2001(98):8113-8118.
[28]Bode CJ, Gupta ML, Reiff EA, et al. Epothilone and paclitaxel:unexpected differences in promoting the assembly and stabilization of yeast microtubules[J]. Biochemistry, 2002(41):3870-3874.
[29]Lee FY, Smykla R, Johnston K, et al. Preclinical efficacy spectrum and pharmacokinetics of ixabepilone[J]. Cancer Chemother Pharmacol, 2009, (63):201-212.
[30]Mekhail T, Chung C, Holden S, et al. Phase I trial of novel epothilone B analog BMS-310705 IV q 21 days[J]. Proc Am Soc Clin Oncol, 2003(22):129(abstract 515).
[31]Sessa C, Perotti A, Malossi A, et al. Phase I and pharmacokinetic(PK)study of the novel epothilone BMS-310705 in patients(pts)with advanced solid cancer[J]. Proc Am Soc Clin Oncol, 2003(2):130(abstract 519).
[32]Wartmann M, Altmann KH. The biology and medicinal chemistry of epothilones[J]. Curr Med Chem Anticancer Agents, 2002(2):123-148.
[33]Lee FYF, Borzilleri R, Fairchild CR, et al. BMS-247550:a novel epothilone analog with a mode of action similar to paclitaxel but possessing superior antitumor activity[J]. Clin Cancer Res, 2001, (7):1429-1437.
[34]Sessa C, Perotti A, Lladò A, et al. Phase I clinical study of the novel epothilone B analogue BMS-310705 given on a weekly schedule[J]. Ann Oncol, 2007(18):1548-1553.
[35]Nicolaou KC, Winssinger N, Pastor J, et al. Synthesis of epothilones A and B in solid and solution phase[J]. Nature, 1997(387):268-272.
[36]Nicolaou K, Roschangar F, Vourloumis D. Chemistry and biology of epothilone[J]. Angew Chem, 1998(110):2120-2153.
[37]Nicolaou KC, King NP, Finlay MR, et al. Total synthesis of epothilone E and related side chain modified analogs via a Stille coupling based strategy[J]. Bioorg Med Chem, 1999, 7:665-697.
[38]Su DS, Horwitz SB, et al. Total synthesis of(3)-epothilone B:an extension of the Suzuki coupling method and insights into structure activity relationships of the epothilones[J]. Angew Chem Int Ed Engl, 1999, 36:757-759.
[39]Yang Z, He Y, Vourloumis D, et al. Total synthesis of epothilone A:the ole¢n metathesis approach[J]. Angew Chem Int Ed Engl, 1997, 36:166-168.
[40]Sawada D, Shibasaki M. Enantioselective total synthesis of epothilone A using multifunctional asymmetric catalyses[J]. Angew Chem Int Ed, 2000, 39:209-213.
[41]Nicolaou KC, He Y, Roschangar F, et al. Total synthesis of epothilone E and analogues with modified side chains through the Stille coupling reaction[J]. Angew Chem Int Ed Engl, 1998, 37:84-87.
[42]Gerth K, Bedorf N, Höfle G, et al. Epothilons A and B:antifungal and cytotoxic compounds from Sorangium cellulosum(myxobacteria)-production, physico-chemical and biological properties[J]. J Antibiot, 1996, 49:560-564.
[43]Gerth K, Steinrich H, Hofle G, et al. Studies on the biosynthesis of epothilones:the biosynthetic origin of the carbon skeleton[J]. J Antibiotics, 2000, 53:1373-1377.
[44]Tang L, Shah S, Chung L, et al. Cloning and heterologous expression of the epothilone gene cluster[J]. Science, 2000(287):640-642.
[45]Julien B, Shah S. Heterogonous expression of epothilone biosynthetic genes in Myxococcus xanthus[J]. Antimicrob Agents Chemother, 2002, 46:2772-2778.
[46]Lau J, Frykman S, Regentin R, et al. Optimizing the heterologous production of Epothilone D in Myxococcus xanthus[J]. Biotechnol Bioeng, 2002, 78:280-288.
[47]Arslanian RL, Tang L, Blough S, et al. A new cytotoxic epothilone from modified polyketide synthases heterologously expressed in Myxococcus xanthus[J]. J Nat Prod, 2002, 65:1061-1064.
[48]Gerth K, Washausen P, Höfle G, et al. The jerangolids:A family of new antifungal compounds from Sorangium cellulosum(myxobacteria). production, physico-chemical and biological properties of jerangolid A[J]. J Antibiot, 1996, 49:71-75.
[49]Mahmud T, Bode HB, Silakowski B, et al. A novel biosynthetic pathway providing precursors for fatty acid biosynthesis and secondary metabolite formation in myxobacteria[J]. J Biol Chem, 2002, (277), 23768-32774.
[50]Bollag DM, McQueney PA, Zhu J, et al. Epothilones, a new class of microtubule-stabilizing agents with a taxol-like mechanism of action[J]. Cancer Res, 1995, 55:2325-2333.
[51]Wang JD, Zhang H, Ying LP, et al. Five new epothilone metabolites from Sorangium cellulosum strain So0157-2[J]. The Journal of Antibiotics, 2009(62):483-487.
[52]龚国利.黏细菌的Genome shuffling育种技术及其抗癌药物埃博霉素的高产改造[D].济南:山东大学, 2007.
[53]Gong GL, Sun X, Liu XL, et al. Mutation of Sorangium cellulosum and a high-throughput screening method for improving the production of Epothilones[J]. J Ind Microbio Biot, 2007, 34:615-623.
[54]龚国利, 孙欣, 刘新利, 李越中. Genome shuffling提高黏细菌产生埃博霉素的能力[C], 中国微生物学会全国会员代表大会及学术年会论文摘要集, 2006:78.
[55]龚国利, 陈松, 李慧, 等.基因组重组技术选育埃博霉素B高产菌株[J]. 中国抗生素杂志, 2013, 38(2):106-110.
[56]龚国利, 陈松, 李慧, 曾桥.改良Genome shuffling技术选育埃博霉素B高产菌株[J]. 中国酿造, 2012, 31(11):42-45.
[57]Gong GL, Jia L, Li H.Preparation and adsorption properties of mixed-templates molecularly imprinted polymers of epothilone B[J]. J ChemPharm Res, 2014, 6(3):1421-1427.
[58]龚国利, 陈松, 李慧. 一种埃博霉素B的发酵生产工艺:中国, ZL 201110346091.0[P]. 2012-3-14.
[59]龚国利, 王娜, 刘丽丽.响应面法优化纤维堆囊菌SoF5-76产埃博霉素B发酵培养基[J].生物技术通报, 2014(1):171-176.
[60]龚国利, 贾琳, 黄菲菲, 许重要.微生物合成抗癌药物埃博霉素的研究进展[J].中国新药杂志, 2009, 18(16):1515-1520.
[61]龚国利, 刘丽丽. 多孔陶瓷吸附固定纤维堆囊菌发酵制备埃博霉素[J].中国生物工程杂志, 2014, 34(3):109-114.
[62]龚国利, 刘丽丽, 王娜, 用于吸附固定纤维堆囊菌的硅藻土基多孔陶瓷制备[J].现代化工, 2013, 33(11):66-70.
[63]Hong J. Role of natural product diversity in chemical biology[J]. Curr Opin Chem Biol, 2011, 15(3):350-354.
[64]Scheller N, Mina LB, Galao RP, et al. Translation and replication of hepatitis C virus genomic RNA depends on ancient cellular proteins that control mRNA fates[J]. Proc Natl Acad Sci USA, 2009, 106(32):13517-13522.
[65]Noueiry AO, Diez J, Falk SP, et al. Yeast Lsm1p-7p/Pat1p deadenylation-dependent mRNA-decapping factors are required for brome mosaic virus genomic RNA translation[J]. Mol Cell Biol, 2003, 23(12):4094-4106.
(责任编辑 狄艳红)
Myxobacteria:Natural Pharmaceutical Factories
Zhao Tingfeng Gong Guoli
(College of Life Science and Engineering,Shaanxi University of Science & Technology,Xi’an 710021)
In recent years, drug development of myxobacteria and its resistant metabolic products has become one of the hot topics of researches study. Myxobacteria is an kind of important natural products producer. There is rich diversity in both the chemical structure and biological activity of these secondary metabolites. The diversity and broad-spectrum activity of metabolites have great potential in drug development field. This review describes the unique cell behavior in myxobacteria and its outstanding ability of producing secondary metabolites, and also expounds the progress of epothilone drug development. Finally, we discuss the potential of developing myxobacterial secondary metabolites into new drugs.
Myxobacteria Secondary metabolites Epothiones Drug discovery
10.13560/j.cnki.biotech.bull.1985.2014.12.007
2014-05-12
国家自然科学基金项目(20906058),陕西科技大学学术骨干培育项目(XSG2010009)
赵婷峰,女,硕士研究生,研究方向:发酵工程;E-mail:zhaotingfeng0211@163.com
龚国利,男,博士,教授,研究方向:应用微生物技术;E-mail:gongguoli@sust.edu.cn

