黄萎病菌诱导海岛棉酵母双杂交cDNA文库构建及评价
杨君张艳王伟巧荣伟王省芬马峙英
(1. 河北农业大学农学院 教育部华北作物种质资源研究与利用重点实验室 河北省作物种质资源重点实验室,保定 071001;2. 河北农业大学作物学博士后科研流动站,保定 071001)
黄萎病菌诱导海岛棉酵母双杂交cDNA文库构建及评价
杨君1,2张艳1王伟巧1荣伟1王省芬1马峙英1,2
(1. 河北农业大学农学院 教育部华北作物种质资源研究与利用重点实验室 河北省作物种质资源重点实验室,保定 071001;2. 河北农业大学作物学博士后科研流动站,保定 071001)
采用SMARTTMcDNA合成技术,通过同源重组方法构建了海岛棉Pima90-53经强致病力黄萎病菌诱导的酵母双杂交cDNA文库。经测定,文库容量为2.82×106,滴度为5.02×108cfu/mL。菌落PCR显示,重组到pGADT7-Rec载体上的cDNA片段大小集中在500-1 500 bp之间,重组率93.33%。该文库完全可用于下一步互作蛋白筛选、信号转导组件确定及抗病蛋白结构和功能分析。
黄萎病菌 海岛棉 酵母双杂交 cDNA文库
棉花是全球最重要的经济作物之一。棉花黄萎病是一种毁灭性的病害,广泛分布于我国乃至世界的各产棉区,严重威胁棉花产量及纤维品质,是当前影响棉花生产的主要病害之一[1]。近年来,棉花基因组研究取得长足进展,CottonGen(http://www. cottongen.org)富集了来源于不同棉种的基因数据[2];中国农业科学院棉花研究所完成了棉花二倍体D基因组雷蒙德氏棉(Gossypium raimondii L.)的测序
和注释[3];转录组测序揭示了不同棉花品种在多种因子诱导下的基因表达模式[4,5]。这些进展为解析棉花纤维发育、抗病虫、耐涝旱等分子机理奠定了基础。
然而,蛋白质作为基因功能的体现者和执行者,有其自身特有的表达规律和功能模式,基因序列和转录水平上的表达信息并不足以揭示其在生物体内的确切功能[6]。生命活动的过程实质上是蛋白在特定时空下的互作,大多数蛋白是通过与其它蛋白或配体进行结合形成复合物来发挥功能,进而调控生物学过程。植物受病原菌侵染后的抗病或感病反应往往伴随细胞内转录重编程,即一些蛋白会出现表达量、构象、修饰等的变化,导致蛋白间的互作状况发生改变,从而影响植物的免疫信号转导[7]。酵母双杂交系统(Yeast two-hybrid system)被认为是研究蛋白质互作的一种非常有效的手段[8],研究人员利用此技术完成了第一个植物蛋白互作网络图谱,即拟南芥(Arabidopsis thaliana)互作图谱[9]。目前,近80%的蛋白质互作研究都是利用酵母双杂交体系完成的[10]。
抗黄萎病遗传改良和抗病机制研究一直是棉花遗传育种领域的重要科学问题。构建黄萎病菌(Verticillium dahliae)诱导的海岛棉(Gossypium barbadense L.)酵母双杂交cDNA文库,应用于筛选与已知抗病蛋白或黄萎病菌毒素(效应子)的互作蛋白,不仅有助于已知抗病蛋白的功能注释和受体识别蛋白的发现,而且还可为解析棉花抗黄萎病网络和阐明抗病机制奠定理论基础。本研究采用SMARTTMcDNA合成技术,通过同源重组方法构建海岛棉Pima90-53经强致病力黄萎病菌诱导的酵母双杂交cDNA文库,旨在为帮助棉花基因功能注释,并推动抗病基因的发掘和抗病机制的解析奠定基础。
1 材料与方法
1.1 材料
海岛棉品种Pima90-53由河北农业大学棉花遗传育种研究室保存[11]。绿色荧光蛋白标记的黄萎病菌Vd-gfp77由中国农业科学院戴小枫研究员馈赠,该病菌致病能力与Vd991相当[12]。黄萎病菌临西2-1株系为本实验室分离鉴定,为强致病力菌系[13]。Plus植物总RNA提取试剂盒购自天根生化科技(北京)有限公司;PolyATtract®mRNA Isolation System IV购自普洛麦格(北京)生物技术有限公司;Make Your Own “Mate & PlateTM” Library System和YeastmakerTMYeast Transformation System 2购自Clontech公司;其他培养基及生化试剂等购自宝生物工程(大连)有限公司。引物合成及测序由生工生物工程(上海)股份有限公司完成。
1.2 方法
1.2.1 棉苗培育与菌系孢子制备 据参考文献[4],将棉苗培育于MS培养基中,培养条件为:光照强度6 000 Lux,光照时间12 h,昼温28℃,夜温25℃,湿度40%;将4℃保存的黄萎病菌单孢菌系接种到PDA固体培养基上活化,再转接到Czapek液体培养基震荡培养(25℃,150 r/min)10 d,通过纱布过滤去除菌丝收集孢子。
1.2.2 接菌与组织取样 利用血球计数板,将黄萎病菌孢子液浓度调至107个孢子/mL,对刚出现第一片真叶的棉苗进行蘸根接菌。分别在接菌后1、2、4、6、8、12、24、36、48、72、96和120 h取棉苗幼根,无菌水清洗后用液氮速冻,置于-80℃保存备用。
1.2.3 组织显微观察与病菌分离 取黄萎病菌Vdgfp77侵染后的棉苗茎进行徒手切片,于荧光显微镜下观察维管束中病菌侵染进程。按照常规组织分离法,将临西2-1侵染的棉苗茎切割成长约0.3 cm的小段,置于PDA培养基,25℃黑暗培养3 d后观察病菌出现情况。
1.2.4 RNA提取与mRNA分离 在液氮中对保存的样品进行充分研磨,按照“Plus植物总RNA提取试剂盒”说明书进行RNA提取操作。将提取的12个时间点样品总RNA等量混合(每个时间点30 μg),用PolyATtract®mRNA Isolation System IV分离纯化mRNA,采用紫外分光光度计测定RNA的浓度和纯度,1.5%琼脂糖凝胶电泳检测RNA的质量。
1.2.5 cDNA合成与纯化 mRNA作为模板,参照Make Your Own “Mate & PlateTM” Library System说明书合成第一链cDNA及 ds cDNA。利用CHROMA SPINTMTE-400柱对dscDNA进行分级分离纯化。
1.2.6 酵母感受态细胞制备 应用PEG(polyethylene glycol)/LiAc(醋酸锂)法制备酵母菌Y187感受态细胞。具体操作按照 YeastmakerTMYeast Transformation System 2的说明进行。
1.2.7 酵母双杂交cDNA文库构建 采用Make Your Own “Mate & PlateTM” Library System试剂盒进行cDNA文库的构建。共转化体系如下:20 μL(4 μg)dscDNA、6 μL(3 μg)线性化pGADT7-Rec、20 μL(200 μg)变性YeastmakerTMCarrier DNA及600 μL Y187感受态细胞。转化后的酵母细胞液(15 mL)涂布于直径150 mm的SD/-Leu平板上,每板涂布菌液150 μL(共100个平板),30℃倒置培养,3-5 d后用Freezing Medium收集转化子,按照每管1 mL分装,保存于-80℃。
1.2.8 文库容量、滴度、重组率和多态性分析 将转化后的酵母细胞液稀释100倍后涂布于直径100 mm的SD/-Leu平皿,每皿50 μL,共涂布3皿,30℃倒置培养3-4 d,统计平皿上的菌落数,从而计算文库总的克隆数=每皿平均克隆数/涂布体积×稀释倍数×转化体积(15 mL),即代表文库容量。取收集后分装的文库菌液稀释10 000倍后涂布于直径100 mm的SD/-Leu平皿,每皿50 μL,共涂布3皿,30℃倒置培养3-4 d,统计平皿上的菌落数,从而计算文库滴度(cfu/mL)=每皿平均菌落数/涂布体积×稀释倍数。挑取平皿上的菌落进行小量培养,用1.5 μL酵母菌液作模板,pGADT7-Rec载体通用筛选引物(5' primer:5'-CTATTCGATGATGAAGATACCCCACCAAACCC-3'和3' primer:5'-GTGAACTTGCGGGGTTTTTCAGTATCTACGAT-3',可扩增载体序列约300 bp)进行菌落 PCR,根据扩增结果计算文库重组率(%)=阳性菌落数/检测菌落总数×100%,并分析文库多态性。
2 结果
2.1 海岛棉建库材料准备
棉苗在温、光、湿可控的条件下生长健壮,整齐一致,根系发达,且棉苗在无菌条件下免受其它杂菌和虫害的影响,最大程度减少了环境因素对试验的影响(图1-A)。为了能够对取样时间进行确定,以黄萎病菌Vd-gfp77处理的棉苗为对照,结果显示,接菌72 h后,棉苗根变为浅褐色(图1-B),下胚轴基部维管束中出现明亮的绿色荧光(图1-C和图1-D),并且棉苗均能分离出接种的病菌(图1-E)。这说明接菌72 h足以保证病菌侵入棉苗维管束,即棉苗与黄萎病菌建立侵染关系。因此,本研究对棉苗取样时,以接菌后72 h作为时间节点,外加96 h和120 h,以此共计12个时间点的材料用于后续文库构建。

图1 海岛棉材料准备

图2 棉苗根组织总RNA电泳检测
2.2 总RNA提取和mRNA纯化
提取黄萎病菌处理后12个时间点的棉苗幼根总RNA,如图2所示,28S与18S rRNA条带清楚,比值约为2,表明RNA完整度好,基本无降解;A260/A280在1.9-2.1之间,浓度为2-3 μg/μL,说明RNA无蛋白和DNA污染,浓度较高。将12份材料的等量RNA混合进行mRNA的分离和纯化,电泳检测mRNA大小分布范围在400-3 000 bp(图3),浓度为320 ng/μL,A260/A280=1.92,表明mRNA的质
量和纯度较高,满足建库的要求。

图3 mRNA纯化
2.3 dscDNA合成及分级分离
应用CDSIII(Oligo-dT Primer)和SMART III-modified oligo引物合成cDNA第一链,以之为模板采用LD-PCR(Long distance PCR),对扩增循环数进行优化后,最终经过21个PCR循环获得dscDNA。CHROMA SPINTMTE-400柱截留纯化,最终收集4个泳道的dscDNA(图4泳道6-9),混合后检测片段长度集中于500-3 000 bp(图5)。

图4 cDNA分级分离

图5 dscDNA电泳检测
2.4 酵母双杂交cDNA文库构建和质量评估
根据同源重组的原理,将合成的棉苗dscDNA和pGADT7-Rec载体共转化酵母Y187感受态细胞,经过SD/-Leu平板筛选,4 d后获得阳性重组子(图6-A)。统计酵母转化液稀释100倍后涂布获得的重组子数目(图6-B),计算文库容量= 94(每皿平均克隆数)/ 50 μL×100×15×103μL = 2.82 × 106。取文库菌液稀释10 000倍后涂布(图6-C),根据出现的重组子数目计算文库滴度= 2 510(每皿平均菌落数)/0.05 mL×104= 5.02×108cfu/mL。随机挑取60个单菌落进行PCR检测(部分结果见图7),显示重组到pGADT7-Rec载体上的cDNA片段大小集中在400-1 500 bp之间;阳性菌落数为56个,重组率= 93.33%。

图6 文库构建与质量评估
3 讨论
生产上的棉花品种主要是陆地棉(Gossypium hirsutum L.),其对黄萎病基本表现为感病或耐病,还没有高抗黄萎病的陆地棉品种。陆地棉中多为微效多基因抗病位点,受环境影响大,难以进行基因聚合育种。众多学者认为海岛棉和部分野生棉种中存在对黄萎病高抗或免疫的材料,并且抗性表现为单基因或少数主效基因控制[11,14,15]。此外,大量的研究表明受黄萎病菌胁迫后,在防御酶类、抗病及参与次生代谢合成相关基因的表达速率或表达量方面,抗病海岛棉显著高于感病陆地棉[4,16,17]。因此,本研究以高抗黄萎病的海岛棉Pima90-53作为建库材料。
棉花黄萎病是由黄萎病菌引起的土传真菌维管束病害。在适宜条件下,黄萎病菌的微菌核或分生孢子萌发产生菌丝侵入棉花根部,经过皮层进入导管并繁殖产生大量的菌丝和分生孢子,分生孢子随导管中的液流上升而扩散到整个植株。因而,根作为黄萎病菌侵入棉花的首要组织器官对于研究棉花
抗病机理更具有意义。虽然棉花抗黄萎病侵染机制还存在争议,但可以肯定的是其抗病过程包括免疫识别、信号转导和抗病基因表达等[1]。为此,本研究以多达12个时间点的棉花根组织作为建库材料,保证了文库包含的抗病相关基因数量的最大化。
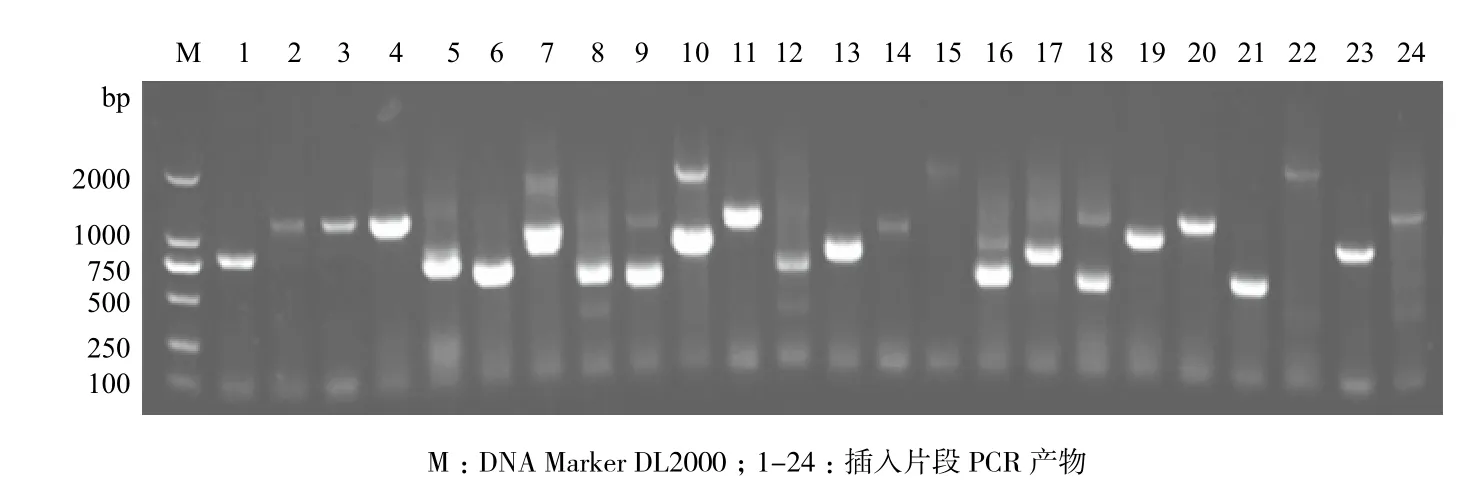
图7 PCR检测文库插入片段
在过去的一年中,生物基因组测序呈现爆炸式增长,研究和注释这些基因功能也就成了后续重要而紧迫的任务[18]。蛋白质是基因功能的执行者和体现者,研究已知功能蛋白质和未知功能蛋白质相互作用是揭示未知蛋白功能的重要手段之一。酵母双杂交cDNA文库技术具有研究成本较低、可以检测到一些微弱的蛋白质互作、在酵母细胞内验证互作而无需纯化蛋白质等优点,成为当前高通量筛选蛋白质互作的主要方法[19]。目前,二倍体棉花——雷蒙德氏棉全基因组图已绘制完成,预测出40 976个蛋白质编码基因[3]。本研究所在课题组前期已构建黄萎病菌诱导的海岛棉Pima90-53全长cDNA文库,获得了23 126个Unigenes。虽然这些基因已通过KEGG(Kyoto Encyclopedia of Genes and Genomes)定位于各种信号转导和代谢通路,但其在棉花细胞内的实际功能还有待验证和发掘[4]。因此,本研究构建的黄萎病菌诱导下的棉花酵母双杂交cDNA文库,不仅有助于棉花基因功能注释,还将推动抗病基因的发掘和抗病机制的解析。
4 结论
本研究构建了海岛棉Pima90-53经强致病力黄萎病菌诱导的酵母双杂交cDNA文库。文库容量为2.82×106,滴度为5.02×108cfu/mL,重组cDNA片段大小集中在500-1 500 bp之间,重组率93.33%。该文库完全可用于下一步互作蛋白筛选、信号转导组件确定及抗病蛋白结构和功能分析。
[1]徐理, 朱龙付, 张献龙.棉花抗黄萎病机制研究进展[J].作物学报, 2012, 38(9):1553-1560.
[2]Yu J, Jung S, Cheng CH, et al. CottonGen:a genomics, genetics and breeding database for cotton research[J]. Nucleic Acids Research, 2014, 42(1):1229-1236.
[3]Wang K, Wang Z, Li F, et al. The draft genome of a diploid cotton Gossypium raimondii[J]. Nature Genetics, 2012, 44(10):1098-1103.
[4]Zhang Y, Wang XF, Ding ZG, et al. Transcriptome profiling of Gossypium barbadense inoculated with Verticillium dahliae provides a resource for cotton improvement[J]. BMC Genomics, 2013, 14:637.
[5]Bowman MJ, Park W, Bauer PJ, et al. RNA-Seq transcriptome profiling of upland cotton(Gossypium hirsutum L.)root tissue under water-deficit stress[J]. PloS One, 2013, 8(12):e82634.
[6]Li J, Assmann SM. Mass spectrometry. An essential tool in proteome analysis[J]. Plant Physiology, 2000, 123(3):807-809.
[7]Spoel SH, Dong X. How do plants achieve immunity? Defence without specialized immune cells[J]. Nature Reviews Immunology, 2012, 12(2):89-100.
[8]Braun P, Aubourg S, Van Leene J, et al. Plant protein interactomes[J]. Annual Review of Plant Biology, 2013, 64:161-187.
[9]Arabidopsis Interactome Mapping Consortium. Evidence for network evolution in an Arabidopsis interactome map[J]. Science, 2011, 333(6042):601-607.
[10]Oender K, Niedermayr P, Hintner H, et al. Relative quantitation of protein-protein interaction strength within the yeast two-hybrid system via fluorescence beta-galactosidase activity detection in a high-throughput and low-cost manner[J]. Assay and Drug Development Technologies, 2006, 4(6):709-719.
[11]马峙英, 王省芬, 张桂寅, 等.不同来源海岛棉品种黄萎病抗性遗传研究[J].作物学报, 2000, 26(3):315-321.
[12]徐明, 桂月晶, 祁伟彦, 等.绿色荧光蛋白基因标记棉花黄萎病菌[J].植物保护, 2013, 39(5):128-133.
[13]王国宁, 赵贵元, 岳晓伟, 等.河北省棉花黄萎病菌致病性与ISSR遗传分化[J].棉花学报, 2012, 24(4):348-357.
[14]孙济中, 陈布圣.棉作学[M].北京:中国农业出版社, 1999:327-358.
[15]潘家驹, 张天真, 蒯本科, 等.棉花黄萎病抗性遗传研究[J].南京农业大学学报, 1994, 17(3):8-18.
[16]Xu L, Zhu L, Tu L, et al. Lignin metabolism has a central role in the resistance of cotton to the wilt fungus Verticillium dahliae as revealed by RNA-Seq-dependent transcriptional analysis and histochemistry[J]. Journal of Experimental Botany, 2011, 62(15):5607-5621.
[17]Gao W, Long L, Zhu LF, et al. Proteomic and virus-induced gene silencing(VIGS)analyses reveal that gossypol, brassinosteroids, and jasmonic acid contribute to the resistance of cotton to Verticillium dahliae[J]. Molecular & Cellular Proteomics, 2013, 12(12):3690-3703.
[18]Ellegren H. Genome sequencing and population genomics in nonmodel organisms[J]. Trends in Ecology & Evolution, 2014, 29(1):51-63.
[19]Auerbach D, Stagljar I. Yeast two-hybrid protein-protein interaction networks[M]// Proteomics and protein-protein interactions:biology, chemistry, bioinformatics, and drug design. Singapore:Springer Science & Business Media, Inc, 2005:19-31.
(责任编辑 马鑫)
Construction and Characterization of Yeast Two-Hybrid cDNA Library Derived from Roots of Gossypium barbadense Inoculated with Verticillium dahliae
Yang Jun1,2Zhang Yan1Wang Weiqiao1Rong Wei1Wang Xingfen1Ma Zhiying1,2
(1. North China Key Laboratory for Crop Germplasm Resources of Education Ministry,Key Laboratory for Crop Germplasm Resources of Hebei,Department of Agriculture,Hebei Agricultural University,Baoding 071001;2. Postdoctoral Research Station of Crop Sciences,Hebei Agricultural University,Baoding 071001)
A yeast two-hybrid cDNA library derived from roots of Gossypium barbadense cv. Pima90-53 inoculated with severe virulence Verticillium dahliae was constructed based on SMARTTMtechnique and homologous recombination reaction. Detection of the library indicated that its capacity and titer were 2.82×106and 5.02×108cfu/mL, respectively. The result from colony PCR showed that the length of insert cDNA fragments ranged from 500 to 1 500 bp on average and the recombination rate was 93.33%. These results demonstrate that the library meets the demands of the standard cDNA library, which will be useful for screening interaction proteins, determining signal transduction components and analyzing the structure and function of disease-resistant proteins in the future.
Verticillium dahliae Gossypium barbadense Yeast two-hybrid cDNA library
10.13560/j.cnki.biotech.bull.1985.2014.12.017
2014-04-28
国家自然科学基金项目(31301370),国家自然科学基金项目(31301371),河北省博士后科研项目(冀人社字[2013]303号)
杨君,男,博士后,研究方向:棉花遗传育种;E-mail:yang22181@163.com;张艳为并列第一作者
王省芬,博士,教授,研究方向:棉花遗传育种;E-mail:cotton@hebau.edu.cn

