植物JAZ蛋白的功能概述
孙程 周晓今 陈茹梅 范云六 王磊
(中国农业科学院生物技术研究所,北京 100081)
植物JAZ蛋白的功能概述
孙程 周晓今 陈茹梅 范云六 王磊
(中国农业科学院生物技术研究所,北京 100081)
茉莉酸作为重要的植物激素,在植物生长、繁殖和抗逆等诸多方面起重要的作用。茉莉酸途径的抑制因子JAZ(Jasmonate ZIM-domain,JAZ)蛋白(JAZs)是调节茉莉酸(JA)激素应答的关键因子,在没有JA存在时,JAZs抑制DNA-结合转录因子活性,从而调控JA应答基因转录;当有JA存在时,JAZs和冠菌素不敏感1(Coronatine insensitive 1,COI1)依赖JA分子发生互作,复合体被SCFCOI1识别并进入26S蛋白酶解途径降解,释放的转录因子启动JA应答基因转录。随着研究的深入,发现JAZs可以与许多转录因子互作,不仅调控了JA信号响应,还参与了其他激素信号通路。对JAZs的互作机制进行描述,阐述JAZs在植物激素调控网络中的作用。
JAZs TIFY 茉莉酸
茉莉酸途径的抑制因子JAZ(Jasmonate ZIM-domain,JAZ)蛋白(JAZs)是一类参与多个信号通路的阻遏蛋白,它可以响应茉莉酸(JA)的刺激,释放所结合MYC2转录因子,从而启动JA应答基因的转录。JAZs可以同其他转录因子以及共阻遏蛋白相互结合,把茉莉酸信号通路与其他信号通路相关联,已有研究把JA介导的JAZs转录水平变化与植物生长素(AUX/IAA)[1]、乙烯(ET)[2-4]、赤霉素(GA)[5,6]和油菜素内酯(BR)[7]等激素信号的调节相联系起来。这些研究表明JAZ蛋白是转录抑制子,它的功能不仅仅能抑制茉莉酸信号传递应答的转录,还通过与其他通路调控因子的互作,影响诸多信号通路和植物生长于代谢进程,JAZs在广阔的激素调节网络中起中心枢纽的作用。
1 JAZ蛋白简介
1.1 JAZs的发现
茉莉酸类化合物是植物内源激素,(3R,7S)-JA-Ile是植物体内茉莉酸活性分子,冠菌素(Coronatine,COR)空间结构与JA-Ile相似,它们在植物体内均起到激素刺激的作用。
用冠菌素筛选拟南芥突变体,获得冠菌素不敏感突变体Coronatine insensitive 1(coi1)[8]。coi1表现为雄性不育并丧失大部分JA应答反应。COI1属F-box蛋白,形成的SCFCOI1复合体属E3泛素连接酶的一种[9-11]。上述发现说明JA应答的起始,需要某些关键蛋白被降解。
opr3(12-Oxophytodienoate reductase 3)是JA合成通路突变体[12],用JA处理opr3,经转录谱分析得知1 296个基因的表达受JA调控[13]。在JA处理30 min时共有31个响应基因,其中有7个未知基因含有28个氨基酸的ZIM基序,还有一个30 min后响应JA的基因,也含有ZIM基序,这8个基因最初被认为是JA应答转录因子[14]。通过高通量测序又得到4个含有ZIM结构域的基因,但它们不受JA诱导,因为这12个蛋白都含有ZIM结构域,并与茉莉酸信号传导相关,所以将这12个成员命名为拟南芥JAZ蛋白。
1.2 JAZ蛋白家族
JAZs属于TIFY家族,TIFY家族是植物特有的家族,在最早的绿藻中并没有TIFY基因[15],而在所有的陆生植物中都含有TIFY家族,所以推测TIFY家族是在陆生植物进化时产生的。苔藓是最早出现的陆生植物,在其中鉴定出两个TIFY成员,这两个成员被分成两个亚群,亚群I含有C2C2-GATA结构域,亚群II则不含。在拟南芥中已鉴定出18个TIFY成员,同样也分为两个亚群。说明在植物进化过程中TIFY家族也不断的发展,并赋予植物多样化的生理功能和发育机制。
TIFY家族包括JAZs、ZIM和PPD亚类[16,17]。ZIM和ZLM(ZIM-LIKE)是推测的转录因子,含有GATA-锌指结构;JAZs是阻遏蛋白不含DNA结合结构域,并必须依赖蛋白间的互作行使功能[16]。
1.3 JAZs的结构
JAZs成员都拥有NT、ZIM和Jas三个保守结构域。蛋白的N末端包含一个弱保守的NT结构域,此结构域可与DELLA蛋白互作[18]。ZIM结构域由36个氨基酸组成,其中包含保守的TIFY基序(TIF[F/Y]XG),JAZs形成同源或异源二聚物依赖TIFY基序[19-21]。JAZ家族C端的Jas结构域十分保守,由12-29个氨基酸组成,在拟南芥的12个JAZ成员中Jas结构域基本一致或有保守替换序列[22],Jas结构域可以同很多蛋白互作,在有JA-Ile情况下负责JAZs的降解[3,6,23]。Jas结构域中存在细胞核定位信号,使JAZs具有核定位特性,如图1所示[24]。
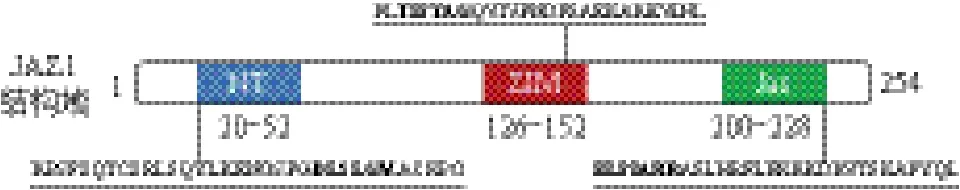
图1 JAZs结构域
2 JAZs与茉莉酸信号通路
2.1 JA信号传导模型
MYC2为JA应答基因的起始转录因子[25,26],而植物体的JA感应由COI1控制,JAZs作为它们之间的联系物于2007年被鉴定出[22,27,28],自此JA信号传导模型被揭示:植物在正常生长条件下,体内JA-Ile水平很低,JAZs结合MYC2等转录因子并吸引TPL和NINJA共阻遏物抑制JA应答基因转录,从而阻止植物体进行JA应答反应。
当植物体处于某发育过程或受到外界胁迫时,体内JA-Ile的水平增高,JAZ阻遏蛋白被COI1结合并通过SCFCOI1/26S蛋白酶通路降解,MYC2等转录因子被释放,从而启动相应JA应答基因的转录[29,30]。当发生JA应答时JAZs也被诱导,诱导表达的JAZs再次抑制MYC2转录因子活性,从而封闭JA响应,这种调节过程类似于反馈调节,使植物体不会产生过于猛烈的JA应答反应,避免植物体能量的过度消耗[31,32],如图2所示。
2.2 JAZs与COI1互作
JAZs与COI1依赖于茉莉酸类激素(JAs),并通过Jas结构域发生互作。植物体为了减弱JA应答造成的强大消耗,在JAZs的Jas区域发生可变剪切。拟南芥JAZ 12个成员中有9个都含有一段保守内含子序列,当JAZs发生可变剪切保留内含子时转录被提前终止,Jas保留前20个氨基酸,并缺少保守的X5PY序列,这些可变剪接体被称为ΔPYJAZs,增加
了JAZs的调控机制[33-36]。
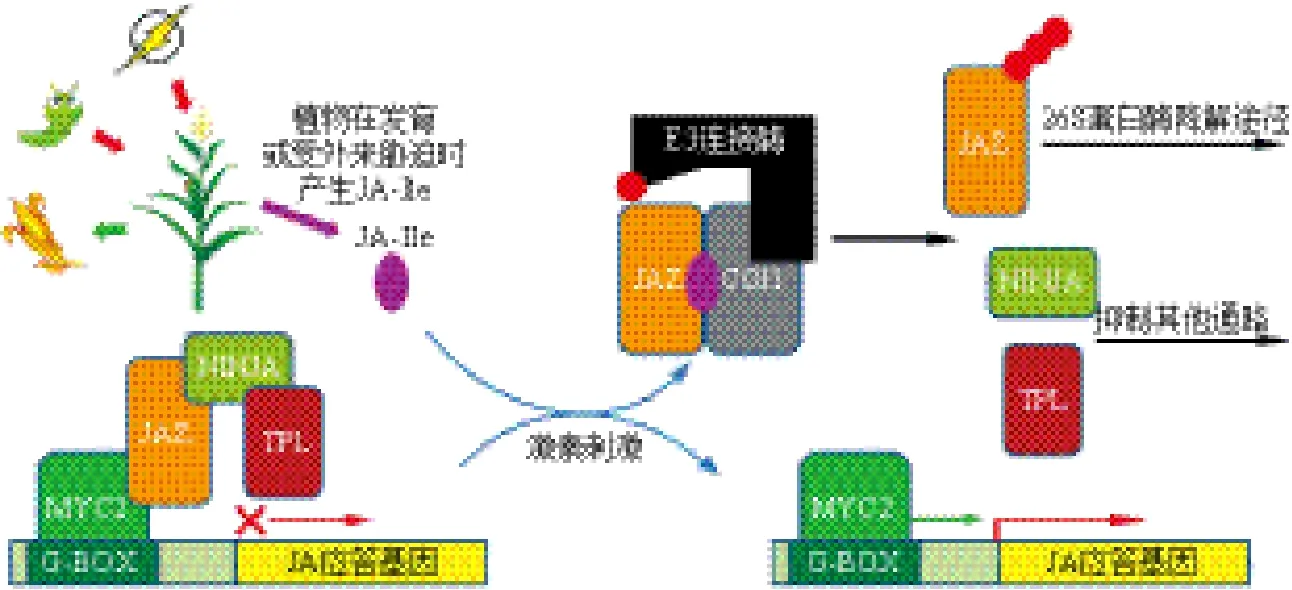
图2 JA 信号传导模型
JAZ1的Jas肽段、COI1和JA-Ile的复合晶体结构揭示了JAs分子机制[37]。在晶体结构中F-box肽段的11个氨基酸构成口袋区,通过3个氨基酸结合(3R,7S)JA-Ile。JAs的酮基和Ile的羧基暴露在外并结合Jas结构域。
1,2,4,5,6-五磷酸肌醇(IP5)在分子组装方面起到重要的作用[38]。IP5在Jas/COI1/JA-Ile复合晶体中紧靠着JA-Ile分子,结合在COI1的口袋处,位于COI1的3个精氨酸和Jas肽段的R206上。如果通过透析法去除IP5会使得阻遏蛋白复合体失活,而失活的阻遏蛋白复合体在添加IP5后会恢复活性。
2.3 JAZs与bHLH/MYC转录因子互作
MYC2、MYC3和MYC4 属于Basic helix-loophelix(bHLH)家族,与大多数的JAZs都互作[39]。MYC2和MYC3中有一段序列可与11个JAZs成员互作,被称为JID(JAZ-interaction domain)。MYC2、MYC3和MYC4都可结合G-box(CACGTG)结构,说明MYC成员间的功能存在冗余性。而三者的突变体表型略有不同,说明MYC2、MYC3和MYC4转录因子对于完全启动JA应答都是必要的。
3 JAZ互作的转录因子
3.1 JAZs结合WD40/bHLH/MYB复合体调控花青素积累和表皮毛发育
GL3、EGL3和TT8属于bHLH家族IIIf亚群,含有JID结构域,除了三者可互相形成复合体外,还可以和8个JAZs互作。GL3/EGL3/TT8复合体可以一边结合WD40蛋白,一边结合不同的MYB蛋白,如此GL3/EGL3/TT8复合体调控诸多生命进程。例如,根毛和表皮毛发育、黄酮类化合物的合成、下胚轴上的气孔发育和种皮上的黏液产生等[40]。
JAZs还能直接结合R2R3 MYB转录因子PAP1和GL1,在PAP1、GL3和EGL3过表达植株中,JAZs产生的抑制作用被抵消,转化体积累更多的花青素并且毛状体发育受限程度低。WD40/bHLH/MYB复合体的形成是通过PAP1与TT8和GL1与GL3之间的互作形成的[41]。虽然bHLH转录因子可以通过JID结构域互作,但目前为止还没有R2R3 MYB蛋白与MYC互作的报道。
3.2 JAZs结合MYB21和MYB24调控植物体雄性不育
MYB21和MYB24属于R2R3 MYB转录因子,可以通过R2R3结构域与JAZs的Jas和NT结构域与发生互作。MYB21和MYB24与JA介导的雄性不育相关,在拟南芥花器发育时合成 JA-Ile,促使JA应答降解JAZs,MYB转录因子被释放调节雄蕊的正常发育[42]。
myb21突变体生育能力减弱,基本的生殖进程受到影响,如雄蕊伸长、花粉活力和花药破裂等。myb24虽然没有表型,但是会使myb21的上述表型更明显,说明两个蛋白功能冗余[13,43]。在coi1中过表达MYB21可以恢复植株不育的表型,但获得的
种子很少。过表达MYB21并没有恢复coi1中与JA应答相关的表型,如对JA介导的根伸长抑制不敏感,花青素积累或是对蕈蚊(Bradysia impatiens)敏感等。这些结果说明MYB21起到调节生育的作用,并且和其他JA介导的生长和防御应答不太相关。
3.3 JAZs结合EIN3/EIL1调控JA/ET信号通路
ET是重要的植物激素,可与JAs共同调节植物体防御,抵抗植物腐生病原菌并控制植物体的发育进程[44]。ET与其受体结合使其下游的CTR1(Constitutive ethylene response1)激酶失活。这个反应会促进一系列的Et响应转录因子的激活,并调节ET响应基因的表达[45]。
Ethylene insensitive 3(EIN3)和EIN3-like1(EIL1)转录因子负责促进ET传感的下游基因转录。JA与ET具有协同和依赖作用,这是通过彼此调控蛋白间的互作实现的。相关研究证明JAZ1、JAZ3和JAZ9都可以同EIN3和EIL1互作,并且互作是通过JAZs的Jas结构域实现的[3]。
ET存在时EIN3和EIL1可激活一系列的病原菌抵抗因子和植物发育调节因子,ET效应基因在这时都保持稳定表达状态。当JAZs结合EIN3和EIL1时会部分的抑制它们的活性,这样ET转录因子EIN3和EIL1通过与JAZs的联系使得ET调控分级进行。当有JA存在时,JAZ被降解,使得EIN3和EIL1全部激活,因此JA信号通路和ET信号通路在抵抗植物腐蚀病原菌方面有协同作用。
4 JAZs互作的阻遏蛋白
4.1 JAZs结合DELLAs调控植物的生长与防御
GA是一种萜类激素,是植物种子萌发、茎和子叶下胚轴伸长、花的发育等生长过程中所必须的。DELLA蛋白(DELLAs)是GA信号传递的重要调节因子,在没有GA时DELLAs与PIF3转录因子结合,抑制GA诱导的基因表达。当植物体合成GA时,会被GA受体(GID1)识别,接着相继与4个DELLA成员依次结合并被E3连接酶识别,形成SCFSLY1复合体后,DELLAs被泛素化降解,DELLAs结合的转录因子被释放,激活GA应答基因的转录,促进植物体的光合生长[46]。
DELLAs主要作用是抑制GA信号传递,同时还能促进JA应答基因的转录。DELLAs成员RGA可以同JAZ1、JAZ3和JAZ9互作,互作区域分别为JAZ的NT结构域和RGA C末端的DELLA结构域[47]。RGA与MYC2竞争性的结合JAZ1,并且不能形成三者的复合体,说明DELLAs可以隔绝JAZ与MYC2的互作,在抑制植物体进行光合生长的同时,还促进植物体进入防御应答状态。
JAZs主要作用是抑制茉莉酸信号传递,同时还能促进GA应答基因的转录。JAZs与DELLAs结合,干扰DELLAs与PIF3转录因子的互作。因为JAZs隔绝了DELLAs与PIF3的互作,所以抵消了DELLAs产生的生长抑制,促进了生物体进行光合发育。
GA起到促进植物体生长的目的,JA起到促进植物体防御的机制,但是最有趣的是它们行使功能却是更倾向于抵消对方的转录应答效应。通过对JAZs与DELLAs功能的研究,清楚的阐明了植物体内GA与JA相互协同的精细调控关系,从而使植物体具有协调生长与抵抗逆境的能力[48]。
4.2 JAZs与共阻遏物NINJA、TPL和HDA6互作
NINJA(Novel interactor of JAZ)能与大多数JAZs互作,同时NINJA能与Groucho/Tup1家族共阻遏物TPL(Topless)和TPL-like等发生互作。此机制说明JAZs抑制MYC转录因子的转录活性是通过结合NINJA和TPL阻遏蛋白实现的[49,50]。
JAZ1和NINJA通过ZIM结构域互作,对TIFY基序进行缺失会造成JAZ1和NINJA的互作消失。拟南芥JAZs中只有JAZ7和JAZ8不能与NINJA互作,因为它们在TIFYXG基序的第5位氨基酸有一个天冬酰胺(图3-A),推测是它抑制了JAZs与NINJA的互作,但是并没有直接的试验证据。
JAZs含有EAR和EAR-like基序(图3-B),此基序为TPL或TPR的结合位点,在理论上JAZs可以直接同TPL蛋白结合,但试验证明只有JAZ5和JAZ8可以直接结合TPL。TPL与JAZs互作直接抑制JA应答基因转录,相似而不同于JAZs互作于NINJA再结合TPL的机制。
JAZ1可以与染色质修饰酶HDA6(Histone deacetylase 6)互作抑制JA介导的转录应答[3]。JAZ3和JAZ9也可以与HDA6互作,JAZ1与HDA6
互作区域位于NT或ZIM结构域,目前还没有相关试验说明。
对hda6进行JAs诱导发现,ERF1位点表达量增高,同样对HDA6过表达植株诱导发现,ERF1位点表达量减少,说明组蛋白去乙酰化在JA应答基因的转录调控中起到了关键的作用,特别是作为协同阻遏物来调控ERF1位点。不同,因此鉴定JAZs功能的关键就是找到它的互作因子。研究发现,JAZ家族成员间可以形成同源或异源二聚体,而且JAZs行使功能大多都以二聚体形式执行,但是这种二聚体的存在形式有何作用还没有得到清晰解释[65]。

图3 JAZs的NT结构域及ZIM结构域保守序列
随着越来越多的作物基因组测序工作的完成,使得系统分析TIFY家族成员逐渐变得相对容易,JAZs研究的工作重点将从全家族基因的预测与鉴定转向每个JAZs成员的功能鉴定。在研究各物种JAZs功能的过程中,经验证明,鉴定JAZs的功能需要找到其调控的互作因子,而对于JAZs的研究不能只停留于表面的单基因功能鉴定,还需要总结归纳JAZs家族在各代谢过程中起到的作用,将JAZs所调控的网络串联起来,解析在庞大的生物进程中JAZs参与调控的信号途径,及其对植物体生长、发育和抗逆的贡献。
5 展望
目前,对JAZs的研究取得了许多成果,这也使得人们对JA信号通路的研究进展迅速。阐明在细胞核中JAZs发生互作的机制,有助于深入了解JA信号模型的本质。JAZ阻遏蛋白起到调控枢纽的作用,它把JA信号通路与其他信号通路相互联系在一起,深入对JAZs的了解将有助于诠释植物的激素调控网络。在植物激素介导的信号传递过程中,JAZs作为枢纽蛋白起着重要的调控作用,在今后的研究中诠释JAZs的精确功能将成为研究重点[32,50-55]。
近两年对经济作物中JAZs的研究进展迅速。目前,已经有相关报道的经济作物包括水稻、大豆、葡萄和烟草[56-59]。但是,在上述几种作物中过表达JAZs所产生的表型却不尽相同:在水稻中过表达TIFY11b可以增加籽粒大小[60];在大豆中过表达GsJAZ2可以增加植株对盐碱的敏感性[61];在烟草中过表达NaJAZd和NaJAZh分别可以抑制花蕾脱落和促进尼古丁的合成[62-64]。JAZ家族各成员间的功能有差异,这是由于每个JAZ成员所能互作的因子
[1] Tiryaki I, Staswick PE. AnArabidopsismutant defective in jasmonate response is allelic to the auxin-signaling mutant axr1[J]. Plant Physiology, 2002, 130:887-894.
[2] Pré M, Atallah M, Champion A, et al. The AP2/ERF domain transcription factor ORA59 integrates jasmonic acid and ethylene signals in plant defense[J]. Plant Physiology, 2008, 147:1347-1357.
[3] Zhu Z, An F, Feng Y, et al. Derepression of ethylene-stabilized transcription factors(EIN3/EIL1)mediates jasmonate and ethylene signaling synergy inArabidopsis[J]. Proc Natl Acad Sci USA, 2011, 108:12539-12544.
[4] Adams E, Turner J. COI1, a jasmonate receptor, is involved in ethylene-induced inhibition ofArabidopsisroot growth in the light[J]. Journal of Experimental Botany, 2010, 61:4373-4386.
[5] Navarro L, Bari R, Achard P, et al. DELLAs control plant immune responses by modulating the balance of jasmonic acid and salicylic acid signaling[J]. Curr Biol, 2008, 18:650-655.
[6] Hou X, Lee LYC, Xia K, et al. DELLAs modulate jasmonate signaling via competitive binding to JAZs[J]. Developmental Cell, 2010, 19:884-894.
[7] Ren C, Han C, Peng W, et al. A leaky mutation in DWARF4 reveals an antagonistic role of brassinosteroid in the inhibition of root growth
by jasmonate inArabidopsis[J]. Plant Physiology, 2009, 151:1412-1420.
[8] Feys BJ, Benedetti CE, Penfold CN, Turner JG.Arabidopsismutants selected for resistance to the phytotoxin coronatine are male sterile, insensitive to methyl jasmonate, and resistant to a bacterial pathogen[J]. The Plant Cell Online, 1994, 6:751-759.
[9] Xie DX, Feys BF, James S, et al. COI1:anArabidopsisgene required for jasmonate-regulated defense and fertility[J]. Science, 1998, 280:1091-1094.
[10] Turner JG, Ellis C, Devoto A. The jasmonate signal pathway[J]. The Plant Cell Online, 2002, 14:S153-S164.
[11] Xu L, Liu F, Lechner E, et al. The SCFCOI1 ubiquitin-ligase complexes are required for jasmonate response inArabidopsis[J]. The Plant Cell Online, 2002, 14:1919-1935.
[12] Stintzi A. TheArabidopsismale-sterile mutant, opr3, lacks the 12-oxophytodienoic acid reductase required for jasmonate synthesis[J]. Proceedings of the National Academy of Sciences, 2000, 97:10625-10630.
[13] Mandaokar A, Thines B, Shin B, et al. Transcriptional regulators of stamen development inArabidopsisidentified by transcriptional profiling[J]. Plant J, 2006, 46:984-1008.
[14] Shikata M, Matsuda Y, Ando K, et al. Characterization ofArabidopsisZIM, a member of a novel plant-specific GATA factor gene family[J]. Journal of Experimental Botany, 2004, 55:631-639.
[15] Derelle E, Ferraz C, Rombauts S, et al. Genome analysis of the smallest free-living eukaryoteOstreococcus tauriunveils many unique features[J]. Proc Natl Acad Sci USA, 2006, 103:11647-11652.
[16] Vanholme B, Grunewald W, Bateman A, et al. The tify family previously known as ZIM[J]. Trends Plant Sci, 2007, 12:239-244.
[17] Bai Y, Meng Y, Huang D, et al. Origin and evolutionary analysis of the plant-specific TIFY transcription factor family[J]. Genomics, 2011, 98:128-136.
[18] Kazan K, Manners JM. JAZ repressors and the orchestration of phytohormone crosstalk[J]. Trends Plant Sci, 2012, 17:22-31.
[19] Chini A, Fonseca S, Chico JM, et al. The ZIM domain mediates homo-and heteromeric interactions betweenArabidopsisJAZ proteins[J]. The Plant Journal, 2009, 59:77-87.
[20] Chung HS, Howe GA. A critical role for the TIFY motif in repression of jasmonate signaling by a stabilized splice variant of the JASMONATE ZIM-domain protein JAZ10 inArabidopsis[J]. The Plant Cell Online, 2009, 21:131-145.
[21] Pauwels L, Barbero GF, Geerinck J, et al. NINJA connects the corepressor TOPLESS to jasmonate signalling[J]. Nature, 2010, 464:788-791.
[22] Thines B, Katsir L, Melotto M, et al. JAZ repressor proteins are targets of the SCFCOI1 complex during jasmonate signalling[J]. Nature, 2007, 448:661-665.
[23] Song S, Qi T, Huang H, et al. The jasmonate-ZIM domain proteins interact with the R2R3-MYB transcription factors MYB21 and MYB24 to affect jasmonate-regulated stamen development inArabidopsis[J]. The Plant Cell Online, 2011, 23:1000-1013.
[24] Grunewald W, Vanholme B, Pauwels L, et al. Expression of theArabidopsisjasmonate signalling repressor JAZ1/TIFY10A is stimulated by auxin[J]. EMBO Reports, 2009, 10:923-928.
[25] Berger S, Bell E, Mullet JE. Two methyl jasmonate-insensitive mutants show altered expression of AtVsp in response to methyl jasmonate and wounding[J]. Plant Physiology, 1996, 111:525-531.
[26] Lorenzo O, Chico JM, Sánchez-Serrano JJ, Solano R. JASMONATEINSENSITIVE1 encodes a MYC transcription factor essential to discriminate between different jasmonate-regulated defense responses inArabidopsis[J]. The Plant Cell Online, 2004, 16:1938-1950.
[27] Chini A, Fonseca S, Fernandez G, et al. The JAZ family of repressors is the missing link in jasmonate signalling[J]. Nature, 2007, 448:666-671.
[28] Yan Y, Stolz S, Chételat A, et al. A downstream mediator in the growth repression limb of the jasmonate pathway[J]. Science Signaling, 2007, 19:2470.
[29] Chico JM, Chini A, Fonseca S, Solano R. JAZ repressors set the rhythm in jasmonate signaling[J]. Current Opinion in Plant Biology, 2008, 3008, 11:486-494.
[30] Browse J. Jasmonate passes muster:a receptor and targets for the defense hormone[J]. Annual Review of Plant Biology, 2009, 60:183-205.
[31] Figueroa P. TheArabidopsisJAZ2 promoter contains a G-box and thymidine-rich module that are necessary and sufficient for
jasmonate-dependent activation by MYC transcription factors and repression by JAZ proteins[J]. Plant and Cell Physiology, 2012, 53:330-343.
[32] Leon J. Role of plant peroxisomes in the production of jasmonic acid-based signals[J]. Subcell Biochem,(2013,69:299-313.
[33] Chung HS, Cooke TF, DePew CL, et al. Alternative splicing expands the repertoire of dominant JAZ repressors of jasmonate signaling[J]. Plant J, 2010, 63:613-622.
[34] Jiang S,Yao J,Ma KW,et al. Bacterial effector activates jasmonate signaling by directly targeting JAZ transcriptional repressors[J]. PLoS Pathog,2013,9:e1003715.
[35] Zhou W,Yao R,Li H,et al. New perspective on the stabilization and degradation of the F-box protein COI1 inArabidopsis[J]. Plant Signal Behav,2013(8). pii:e24973. doi:10. 4161/psb. 24973.
[36] Gimenez-Ibanez S,Boter M,Fernandez-Barbero G,et al. The bacterial effector HopX1 targets JAZ transcriptional repressors to activate jasmonate signaling and promote infection inArabidopsis[J]. PLoS Biol,2014,12:e1001792.
[37] Sheard LB, Tan X, Mao H, et al. Jasmonate perception by inositolphosphate-potentiated COI1-JAZ co-receptor[J]. Nature, 2010, 468:400-405.
[38] Mosblech A, Thurow C, Gatz C, et al. Jasmonic acid perception by COI1 involves inositol polyphosphates inArabidopsis thaliana[J]. Plant J, 2011, 65:949-957.
[39] Cheng Z, Sun L, Qi T, et al. The bHLH transcription factor MYC3 interacts with the jasmonate ZIM-domain proteins to mediate jasmonate response inArabidopsis[J]. Molecular Plant, 2011, 4:279-288.
[40] Qi T,Huang H,Wu D,et al.ArabidopsisDELLA and JAZ proteins bind the WD-repeat/bHLH/MYB complex to modulate gibberellin and jasmonate signaling synergy[J]. Plant Cell,2014,26:1118-1133.
[41] Traw MB, Bergelson J. Interactive effects of jasmonic acid, salicylic acid, and gibberellin on induction of trichomes inArabidopsis[J]. Plant Physiology, 2003, 133:1367-1375.
[42] Wang W,Liu G,Niu H,et al. The F-box protein COI1 functions upstream of MYB305 to regulate primary carbohydrate metabolism in tobacco(Nicotiana tabacumL. cv. TN90)[J]. J Exp Bot, 2014,65:2147-2160.
[43] Mandaokar A, Kumar VD, Amway M, Browse J. Microarray and differential display identify genes involved in jasmonate-dependent anther development[J]. Plant Mol Biol, 2003, 52:775-786.
[44] Wang KLC, Li H, Ecker JR. Ethylene biosynthesis and signaling networks[J]. The Plant Cell Online, 2002, 14:S131-S151.
[45] Guo H, Ecker JR. The ethylene signaling pathway:new insights[J]. Current Opinion in Plant Biology, 2004, 7(1):40-49.
[46] Gao XH, Xiao SL, Yao QF, et al. An updated GA signaling ‘relief of repression’ regulatory model[J]. Molecular Plant, 2011, 4:601-606.
[47] Yang DL, Yao J, Mei CS, et al. Plant hormone jasmonate prioritizes defense over growth by interfering with gibberellin signaling cascade[J]. Proc Natl Acad Sci USA, 2012, 109:E1192-1200.
[48] Hou X,Ding L,Yu H . Crosstalk between GA and JA signaling mediates plant growth and defense[J]. Plant Cell Rep,2013,32:1067-1074.
[49] Acosta IF,Gasperini D,Chetelat A,et al. Role of NINJA in root jasmonate signaling[J]. Proc Natl Acad Sci USA,2013,110:15473-15478.
[50] Perez AC,Goossens A. Jasmonate signalling:a copycat of auxin signalling?[J]. Plant Cell Environ,2013,36:2071-2084.
[51] Moreno JE,Shyu C,Campos ML,et al. Negative feedback control of jasmonate signaling by an alternative splice variant of JAZ10[j]. Plant Physiol,2013,162:1006-1017.
[52] Zhu X,Zhu JK. Double repression in jasmonate-mediated plant defense[j]. Mol Cell,2013,50:459-460.
[53] Ballare CL. Light regulation of plant defense[J]. Annu Rev Plant Biol,2014,65:335-363.
[54] Sasaki-Sekimoto Y,Saito H,Masuda S,et al. Comprehensive analysis of protein interactions between JAZ proteins and bHLH transcription factors that negatively regulate jasmonate signaling[J]. Plant Signal Behav,2014,9(1). pii:e27639. [Epub ahead of print].
[55] Wasternack C. Perception,signaling and cross-talk of jasmonates and the seminal contributions of the Daoxin Xie's lab and the Chuanyou Li's lab[J]. Plant Cell Rep,2014,33:707-718.
[56] Rustioni L,Rocchi L,Guffanti E,et al Characterization of grape(Vitis viniferaL. )berry sunburn symptoms by reflectance[J]. J
Agric Food Chem,2014,[Epub ahead of print].
[57] Ishiga Y,Ishiga T,Uppalapati SR,Mysore KS. Jasmonate ZIM-domain(JAZ)protein regulates host and nonhost pathogeninduced cell death in tomato andNicotiana benthamiana[J]. PLoS One,2013,8:e75728.
[58] Toda Y,Tanaka M,Ogawa D,et al. RICE SALT SENSITIVE3 forms a ternary complex with JAZ and class-C bHLH factors and regulates jasmonate-induced gene expression and root cell elongation[J]. Plant Cell,2013,25:1709-1725.
[59] Toda Y,Yoshida M,Hattori T,Takeda S. RICE SALT SENSITIVE3 binding to bHLH and JAZ factors mediates control of cell wall plasticity in the root apex[J]. Plant Signal Behav,2013,8:e26256.
[60] Hakata M, Kuroda M, Ohsumi A, et al. Overexpression of a rice TIFY gene increases grain size through enhanced accumulation of carbohydrates in the stem[J]. Biosci Biotechnol Biochem, 2012, 76:2129-2134.
[61] Zhu D, Cai H, Luo X, et al. Over-expression of a novel JAZ family gene fromGlycine soja, increases salt and alkali stress tolerance[J].Biochemical and Biophysical Research Communications, 2012, 426(2):273-279.
[62] Oh Y, Baldwin IT, Gális I. NaJAZh regulates a subset of defense responses against herbivores and spontaneous leaf necrosis inNicotiana attenuataplants[J].Plant Physiology, 2012, 159:769-788.
[63] Oh Y, Baldwin IT, Galis I. A jasmonate ZIM-domain protein NaJAZd regulates floral jasmonic acid levels and counteracts flower abscission inNicotiana attenuataplants[J]. PloS One, 2013, 8:e57868.
[64] Dewey RE,Xie J. Molecular genetics of alkaloid biosynthesis inNicotiana tabacum[J]. Phytochemistry,2013,94:10-27.
[65] Memelink J. Regulation of gene expression by jasmonate hormones[J]. Phytochemistry, 2009, 70:1560-1570.
(责任编辑 狄艳红)
Comprehensive Overview of JAZ Proteins in Plants
Sun Cheng Zhou Xiaojin Chen Rumei Fan Yunliu Wang Lei
(Biotechnology Research Institute of Chinese Academy of Agricultural Sciences,Beijing 100081)
Jasmonates(JA)as an important phytohormone regulates many aspects of plant development, reproduction, and defence. Within the JA signaling cascades, jasmonate zim-domain(JAZ)proteins that are triggered by jasmonates play a central role. JAZ proteins regulate JA-responsive gene transcription by inhibiting DNA-binding transcription factors(TFs)in the absence of JA. However, in the presence of JA, JAZ proteins interact in a hormone-dependent manner with Coronatine Insensitive 1(COI1). After recognition component of the E3 ubiquitin ligase SCFCOI1, as a result, JAZ proteins are ubiquitinated and subsequently degraded in the 26S proteasome, releasing TFs from inhibition and activating JA-responsive gene transcription. This conclusion indicate that JAZ proteins, which function as transcriptional repressors of the JA signaling response, are not merely regulators of the JA signaling pathway, but, through interaction with other proteins, also serve as signaling hubs in the wider hormone regulatory network, plays important roles in plants..
JAZs TIFY Jasmonates
2013-05-21
转基因专项(2010ZX08010-002)
孙程,男,硕士研究生,研究方向:植物分子生物学与基因工程;E-mail:caas_sc@163.com
王磊,男,研究员,博士生导师,研究方向:植物基因工程;E-mail:wanglei01@caas.cn

