极端嗜盐古菌TRM 51T的多相分类研究
任 敏 歹明辉 夏占峰
(新疆生产建设兵团塔里木盆地生物资源保护利用重点实验室,新疆 阿拉尔 843300)
极端嗜盐古菌(Extremely halophilic archaea)是一类生活在高盐环境的微生物类群,其独特的遗传特质、生理生化特性以及进化世系使其具有重要的理论研究意义和应用研究价值。目前,极端嗜盐古菌资源与分类学研究主要采用多相分类学的理论与技术集中在三个层次特征:表型特征、化学特征(主要是极性脂的组分)、基因型特征(16S rDNA 和DNA–DNA 杂交、G+C 含量mol%)[6]的研究。到目前为止,嗜盐菌科包括36 个属,129 个种[7]。
新疆拥有丰富的盐湖资源,不同类型的盐湖蕴藏着各不相同的嗜盐古菌资源。为丰富嗜盐古菌物种多样性,并为进一步的理论与应用研究提供菌种资源。本研究采集新疆阿图什市境内的硝尔库勒湖盐湖土壤样品[1],利用纯培养技术分离其土壤样品得到多株极端嗜盐古菌,通过对分离菌株的16S rDNA 基因扩增测序后,对潜在新物种TRM51T进行形态鉴定、革兰氏染色、生长盐浓度试验、Mg2+浓度试验、生长pH 范围、生理生化鉴定、极性脂组成、16S rDNA 基因序列等分析,确定TRM 51T属于极端嗜盐古菌科中的新属类群。
1 材料与方法
1.1 菌株
菌株TRM 51T从新疆硝尔库勒湖(76°53'E,40°10'N)中分离得到纯培养物(GenBank/EMBL/DDBJ登录号为 JN166404);标准菌株 Haloterrigena turkmenica CGMCC 1.2364T、Haloarcula vallismortis CGMCC1.2048T、Halopiger xanaduenensis CGMCC 1.6379T(=Halopiger xanaduensis JCM 14033T)、Halopiger aswanensis JCM 11628T等购自中国微生物菌种保藏管理委员会普通微生物中心(China General Microbiological Culture Collection Center,CGMCC)和日本菌种保藏中心(Japan Collection of Microorganisms,JCM)。所有菌株均在CM 培养基上进行多相分类鉴定[3]。
1.2 实验方法
1.2.1 形态学特征 细胞形态及运动性按照文献[3]进行。
1.2.2 生理生化鉴定 生长所需盐浓度范围及最适NaCl、MgCl2浓度、生长pH 及温度范围依照极端嗜盐古菌新分类单元描述标准中的方法[5-6]。
1.2.3 生长条件实验 硝酸盐、精氨酸和二甲基亚砜的厌氧实验,硝酸盐、亚硝酸盐还原,硝酸盐产气,H2S 产气,碳氮源利用及抗生素敏感实验均按照文献[3]进行。
1.2.4 基因型分类特征
1.2.4.1 菌株16S rDNA 基因序列分析
根据文献[2]提取菌体DNA,沉淀溶于灭菌的超纯水中;嗜盐古菌16S rDNA 基因序列的PCR 扩增使用特异性引物F1:5'-TTCCGGTTGATCCTGCC-3';R1::5'-AAGGAGGTGATCCAGCC-3'[3]。PCR 反应体系:10 ×Taq Buffer 2.5 μL,dNTP Mixture(2.5 mM each)3μL,F1(10 μM)0.5 μL,R1(10 μM)0.5 μL,Taq 酶(5 U/μL)0.3 μL,模板DNA 0.5 μL,ddH2O 补至25 μL。PCR 反应条件:95℃5 min ;95℃1 min ;51℃1 min ;72℃1.5 min ;30 个循环;72℃10 min。
取3 μL PCR 扩增产物与一定比例的上样缓冲液混合后进行琼脂糖凝胶电泳(1%琼脂糖凝胶,1 ×TAE 电泳缓冲液,100 V 电泳30 min),EB(0.5 μg/mL)染色后在凝胶成像系统用紫外光透射观察结果,拍摄电泳图,PCR 产物为约1.5 kb,PCR 产物胶回收后与pMD18-T 载体连接,转化入DH5α 大肠杆菌中,菌落PCR 验证后,选取阳性克隆12 个,送由上海生物工程有限公司测序。
1.2.4.2 基于16S rDNA 基因的系统发育学分析
测序结果用DNAStar 软件包中的SeqMan 软件剪辑,所得序列通过Eztaxon server 2.1 在线分析,确定其潜在的分类地位,下载相关序列,并采用MEGA5.0[9]软件包中的Clustal W 进行多序列比对,运用Kimura 2-Parameter Distance 模型及Neighbor-Joining 法,设定Bootstrap 值为1000 次来构建系统发育树[9]。
1.2.4.3 DNA G+C mol%含量的测定
基因组DNA的提取方法参见Marmur (1961),G+C mol%含量通过反相HPLC 法(Mesbah et al,1989)测定。根据样品色谱图中各种脱氧核糖核苷酸的峰面积对DNA G+C mol %进行计算。
DNA G+C mol %含量的计算公式:
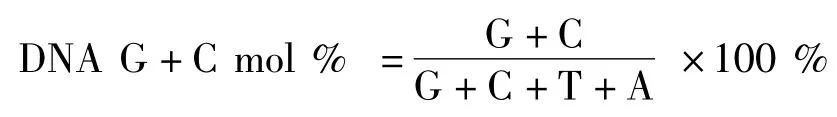
1.2.5 化学分类特征
根据崔恒林的方法[3],对培养好的200 ml 菌液(OD600为1.0 以上)提取极性脂,取6 uL 在10 cm×10 cm的硅胶板(Merk 公司,25TLC aluminium sheets 20 ×20 cmSilica gel 60F254)进行单向TLC 检测菌体的磷脂。同时取等量上述极性脂在10 cm×20 cm的硅胶板双向TLC,检测糖脂组份。
2 结果
2.1 形态与生长特征
菌株TRM 51T为革兰氏阴性菌(图2),牛奶盐平板上培养至7d 时菌落呈圆形,淡粉色,表面光滑湿润,有黏性,边缘较整齐,菌落直径约为1~2 mm(图1)。细胞呈杆状,菌株大小2.73~18.81 μm,无鞭毛,能运动(图3)。

图1 TRM 51T 菌落形态
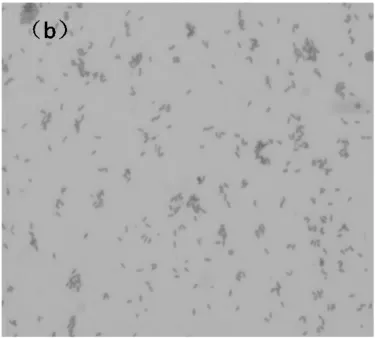
图2 TRM 5T 革兰氏染色

图3 TRM 51T 电镜照片
2.2 营养特性
菌株TRM 51T生长的NaCl 浓度为2.1~4.3 M,最适生长NaCl 浓度为3.1 M,pH 生长范围6.0~9.0(最适pH 8.0),温度范围28~50℃(最适生长温度 37℃),而相近菌株 Halopiger xanaduenensis CGMCC 1.6379T和 Haloterrigena turkmenica CGMCC 1.2364T的NaCl 浓度分别为2.5~5.0 M、2.2~4.0 M,它们的最适生长NaCl 浓度分别为4.3 M、3.1 M,其pH 范围6.0~11.0(最适pH7.5~8.0)、6.5~9.0(最适pH7.0),其生长温度范围28~45℃(最适生长温度37℃)、35~58℃(最适生长温度50℃)。
2.3 生化特征
菌株TRM 51T进行生理生化特性表现为:水解淀粉,液化明胶,不生成H2S,不水解吐温80,吲哚实验阴性,氧化酶、过氧化氢酶阳性,硝酸盐还原实验阳性,不能利用硝酸盐、精氨酸和DMSO。
TRM51T利用葡萄糖、果糖、D- 核糖、阿拉伯糖、麦芽糖、蔗糖、淀粉、甘油、甘露醇、D-山梨醇、醋酸盐、DL-乳酸盐、琥珀酸盐、柠檬酸盐、L-鸟氨酸、L-脯氨酸、L-谷氨酸、盐酪氨酸、精氨酸等作为唯一碳源、能源生长;不能利用甘露糖、半乳糖、L-山梨糖、D-木糖、L-天冬氨酸、L-谷氨酸、L-赖氨酸、L-丝氨酸、L-亮氨酸等;TRM51T与相近物 种 Halopiger xanaduenensis CGMCC 1.6379T、Haloterrigena turkmenica CGMCC 1.2364T在碳、氮源的利用上存在差异(如表1)。

表1 TRM 51T 与相近种的生化特征
2.4 抗生素敏感
TRM51T对杆菌肽、新生霉素、甲氧苄氨嘧啶敏感;对红霉素、新霉素、利福平、氯霉素、氨苄青霉素、诺氟沙星、链霉素、卡那霉素、四环素、万古霉素、制霉菌素、呋喃妥因、萘啶酸等不敏感。而Halopiger xanaduenensis CGMCC 1.6379T对甲氧苄氨嘧啶不敏感和对利福平敏感,与 TRM51T不同;Haloterrigena turkmenica CGMCC 1.2364T对 利 福平、呋喃妥因敏感,与TRM51T不同。
2.5 基因型特征
2.5.1 16S rDNA 基因扩增
采用古菌通用引物,对TRM51T所提取的基因组DNA 进行16SrDNA 基因序列进行扩增并测序,得到几乎全长的16S rDNA 基因序列,经过电泳检测,条带大小约为1.5kb(如图4)。序列提交NCBI并获得GenBank 登录号(JN166404)。
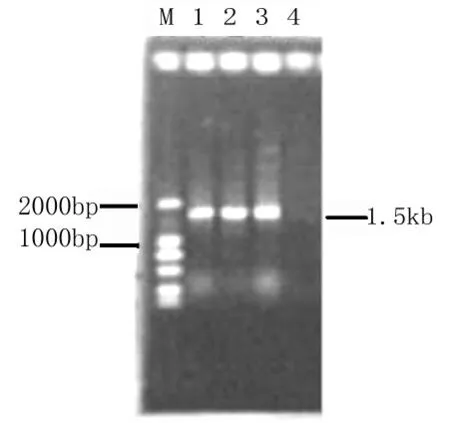
图4 TRM 51T 16S rDNA的PCR 琼脂糖凝胶电泳检测
2.5.2 16S rDNA 基因序列系统发育树分析
克隆测序后,所得序列进行拼接,TRM 51T的16S rDNA 基因序列为1472 bp,与有效发表的参比菌株Halopiger 同源性处于94.2%~95.3%之间,从各菌株以16S rDNA 基因序列为基础构成的系统发育树(见图5),可以看出相比较于Haloterrigena、Halopiger、Natrinema 等属,TRM 51T形成一独立分支,将TRM 51T归入新的属级分类单元。(采用邻接法构建的系统进化树,表明菌株TRM51T在嗜盐菌科的分类地位。Methanospirillum hungatei JF-1(NR 042789)作为外群。自举值选取1000(1000 次重复取样,大于50%的数值被标出)。标尺0.02 代表200 个核苷酸。
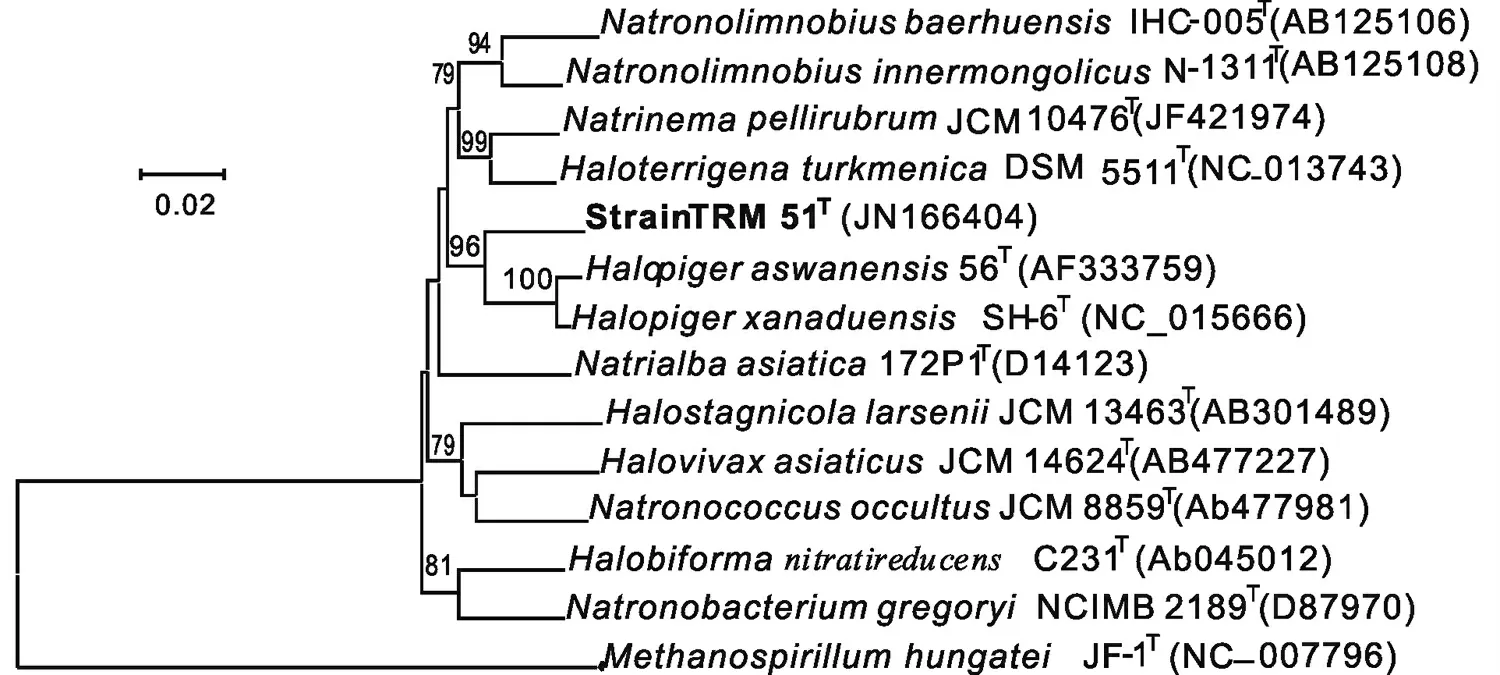
图5 基于16S rDNA 基因序列构建的极端嗜盐古菌系统发育N-J 树)
2.5.3 基因组DNA的G+C mol%含量
菌株TRM 51T、Halopiger xanaduenensis CGMCC 1.6379T、Haloterrigena turkmenica CGMCC 1.2364T基因组DNA G+C mol% 含量分别为63.2%(Tm)、63.1%(Tm)、61.9%(Tm),其值均大于60,属于高G+C的革兰氏阴性菌。
2.6 嗜盐古菌细胞极性脂组分
TRM 51T单向TLC 条带与参比菌株的条带相比,TRM 51T含有TGD-2(如图6);菌株TRM51T细胞中主要的磷脂组分为PG(磷脂酰甘油)、PGPMe(磷脂酰甘油磷酸甲基酯)、TGD-2(葡糖苷甘露糖苷葡糖二醚),明显与相近菌株Halopiger aswanensis JCM 11628T双向TLC 不同(如图7)。
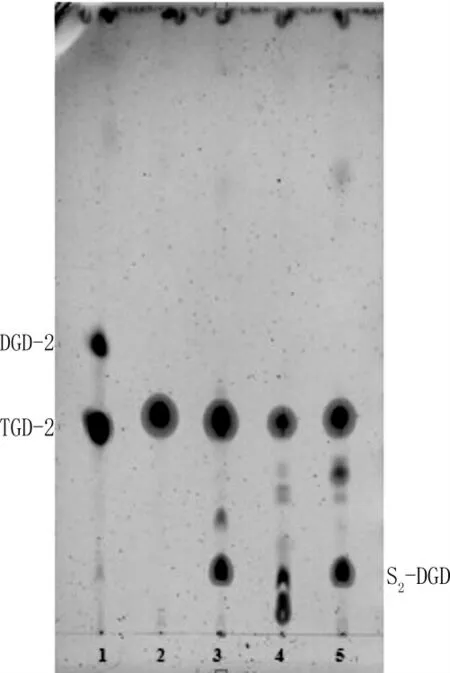
图6 嗜盐古菌菌株TRM51T 糖脂TLC 图谱

图7 嗜盐古菌菌株TRM51T(a)和Halopiger aswanensis JCM 11628T(b)磷脂TLC 图谱
3 讨论
极端嗜盐古菌属于嗜盐菌科,属于生命三域中的古菌域[6,8]。本研究表明TRM51T革兰氏阴性,可运动,菌株呈现粉红色/白色,对数生长期细胞呈现长短不一的杆状,NaCl 浓度为2.1~4.3 M,最适生长NaCl 浓度为3.1 M,不需要镁离子即可生长,最适Mg2+浓度0.3 M,生长pH 6.0~7.0;根据16 S rDNA 系统发育分析TRM51T与Halopiger 属[7]的同源性最高,达95.3%,确认为属于嗜盐菌科的一个新属。
硝尔库勒湖总盐浓度接近于饱和状态,前期的土样分离工作中,得到了盐碱球菌属(Natronococcus)、盐无色菌属(Natrialba)、钠白菌属(Natrinema)、盐东方菌属(Halorientalis)、盐陆生菌属 (Haloterrigena)、盐碱单胞菌属(Natronomonas)、盐池栖菌属(Halostagnicola)、盐惰菌属(Halopiger)、盐粒菌属(Halogranum)7 个属,34 株极端嗜盐古菌,其中盐碱球菌属(Natronococcus)、盐陆生菌属(Haloterrigena)占的数量最多,菌落颜色呈现黄色、白色、红色、橙色等,多数菌株有淀粉酶活性,少数有纤维素酶活性(文章另待发表),有16 株潜在新种,菌株编号TRM51T就是一株。所有的分离的极端嗜盐古菌的理化特征,更多酶活特点还在进行中。
本研究首次对该地区盐湖的极端嗜盐古菌进行了多相分类鉴定,是对该地区盐湖微生物物种的资源的开发。该菌种资源可进一步作为微生物遗传、生态和分类学等研究的材料,有一定的理论和应用价值。
[1]关统伟,吴晋元,职晓阳,等.硝尔库勒湖沉积物中非培养放线菌多样性[J].微生物学报,2008,48(7):851-856.
[2]夏占峰.艾丁湖沉积物放线菌多样性研究[D].塔里木大学硕士学位论文,2011,27-28.
[3]崔恒林,杨勇,迪丽拜尔·托乎提.新疆两盐湖可培养嗜盐古菌多样性研究[J].微生物学报.2006,46(2):171-176.
[4]Elshahed M S,Najar F Z,Roe B A,et al.Survey of archaeal diversity reveals an abundance of halophilic Archaea in a low- salt,sulfide- and sulfur- rich spring[J].Appl Environ Microbiol,70:2230-2239.
[5]Gibbons N E.Family V.Halobacteriaceae fam.nov.In Bergey’s Manual of Determinative Bacteriology,8th,1974:269-273.
[6]Oren A,Arahal D R,Ventosa A.Emended descriptions of genera of the family Halobacteriaceae[J].Int J Syst Evol Microbiol,2009,59,637-642.
[7]Aharon Oren.Taxonomy of the family Halobacteriaceae:a paradigm for changing concepts in prokaryote Systematics[J].Int J Syst Evol Microbiol,2012,62,263–271.
[8]Grant W D,Kamekura M,McGenity T J,et al,Class III.Halobacteria class.nov.In:Boone DR,Castenholz RW.Bergey,s manual of systematic bacteriology.vol.1.2nd eds.New York:Springer,2001:294-334.
[9]Koichiro Tamura,Deniel Peterson,Nrcholas Peterson,et al.MEGA5:Molecular Evolutionary Genetics Analysis Using Maximum Likelihood,Evolutionary Distance,and Maximum Parsimony Methods.Mol.Biol.Evol.2011,28 (10):2731-2739.

