几种江蓠属海藻3个分子序列的系统学分析
赵小波 , 逄少军 刘 峰
(1. 中国科学院 海洋研究所, 山东 青岛266071; 2. 中国科学院大学, 北京 100049)
江蓠属(Gracilaria)海藻在自然分类上隶属于红藻门(Rhodophyta), 江蓠目(Gracilariales), 江蓠科(Gracilariaceae), 广泛分布于热带、亚热带和温带海域, 是世界范围内十分重要的经济海藻[1]。江蓠属中栽培的物种主要是龙须菜、细基江蓠、脆江蓠, 还有智利的智利江蓠。龙须菜栽培主要在我国广东、福建、山东沿海, 其主要用途是提取琼胶、鲍鱼饵料、食用等。江蓠属内其他物种也是重要的食品以及工业原料。因此, 江蓠属海藻产业市场发展潜力大。目前, 江蓠属海藻的研究还存在一些问题: 由于缺乏足够的证据, 这些海藻之间复杂的分类关系尚未完全厘清[2], 例如传统分类学家将龙须菜归入江蓠属[3],但是许多分子生物学证据认为龙须菜并不隶属于江蓠属[2,4-5]; 虽然分子生物学手段越来越多的被应用于江蓠属系统分类研究之中, 但大多数研究局限于ITS以及rbcL序列, 且研究结果存在差异[4-9]; 此外,虽然分子系统学研究进展迅速, 但是一个可信度较高的系统分类结果对于系统分类学家来说仍然是困难的。单纯凭借一个DNA序列进行分析, 往往会由于进化速率不同等因素导致在不同的研究中得出完全不同甚至是错误的结论。为了更清楚的了解中国科学院海藻种质库活体保存的不同江蓠属海藻物种在分类进化上的关系, 作者利用核基因编码的18S rRNA基因、位于线粒体的cox2-3间隔区、以及位于叶绿体的RUBISCO间隔区研究了来自不同地区的5个物种, 并与GenBank中已知相关序列进行比对分析, 力图从分子水平上阐明江蓠属系统进化关系,为江蓠属的系统进化、种质鉴定等提供新的佐证。
1 材料与方法
1.1 实验材料
本实验所用江蓠属、Gracilariopsis属样品采集自我国不同海区(表1)。所有样品首先依照形态学特征进行分类鉴定[3,10-11], 在提取DNA之前, 将藻体用消毒海水清洗, 活体保存于中国科学院海洋研究所海藻种质库。
1.2 总DNA提取与PCR扩增
总DNA提取参考Goff[12]的方法, 并对部分细节进行了改动。将藻体用消毒海水清洗干净之后, 取0.1g于液氮中充分研磨后, 加入大约2mL提取缓冲液[4% SDS, 0. 05 mol/ LTris. HCl, 0. 1 mol/ L EDTA,0.2 mol/ L NaCl], 加蛋白酶K至终质量浓度为100 μg/mL,然后震荡3~4h (50 ℃ ), 12000 r/min 离心10 min, 取上清后加等体积的酚抽提, 12000r/min离心10min取上清, 再以酚/氯仿/异戊醇(25∶24∶1)及氯仿抽提,12000r/min离心10min取上清。上清液加入2倍的无水乙醇和1/10体积的醋酸钾离心, 70%乙醇洗涤2次, 干燥后, 溶解在50μL无菌三蒸水储存备用。采所提取DNA用1%琼脂糖凝胶进行电泳检测。使用PCR技术[13]扩增18S rRNA、cox2-3间隔区、RUBISCO间隔区序列。用于18S rRNA序列扩增的引物为: 18SF 5-CAACCTG GTTGATCCTGCCAGT-3; 18SR 5-TGATCCTTCTGCAGGTTCACCTAC-3。扩增程序为: 94℃预处理5 min后, 94 ℃变性1min, 60℃复性2min, 72℃延伸4min, 总35个循环, 最后在72℃延伸7min, 4℃保存[2]。cox2-3间隔区扩增引物为: coxF 5-GTACCWTCTTTDRGRRK DAAAT GTGATGC-3;coxR 5-GGATCTACWAGATGRAAWGGATGTC-3。扩增程序为: 94℃预处理4 min后, 93 ℃变性1min,45℃复性1min, 72℃延伸1min, 总5个循环, 然后93℃变性30s, 55℃复性30s, 72℃延伸30s, 总30个循环。最后在72℃延伸5min, 4℃保存[14]。RUBISCO间隔区序列扩增引物为: rbcF 5-TATACTTCTACAGACACAGCTGA-3; rbcR 5-ATGTCAAATAATGGTAGT CCCCA-3。扩增程序为: 94℃预处理4 min后, 93 ℃变性1min, 45℃复性1min, 72℃延伸1min, 总5个循环, 然后93℃变性1min, 55℃复性1min, 72℃延伸1min, 总30个循环。最后在72℃延伸5min, 4℃保存[15]。
PCR反应体系20μL包括: 10×PCR反应缓冲液,MgCl2(25mmolL-1)1.5μL, dNTPs (2.5 mmolL-1)2μL,Taq (5UμL-1)0.2μL, 引物 (10 mmolL-1)0.5μL,ddH2O 12.3μL。所有PCR试剂均购自大连宝生物工程有限公司(Takara, Dalian)。
PCR产物用UNIQ-10 PCR Purification Kit试剂盒(上海生工)胶回收纯化。纯化产品与pMD19-T载体(大连宝生物工程有限公司)连接后转化至感受态E. coliTOP 10(北京全是金生化科技有限公司)中, 经氨苄青霉素(AMP)抗性筛选后, 随机挑取5个阳性克隆进行扩大培养, 菌液PCR扩增预检测插入片段大小, 委托上海生工进行双向测序。所得PCR产品序列上传至GenBank。
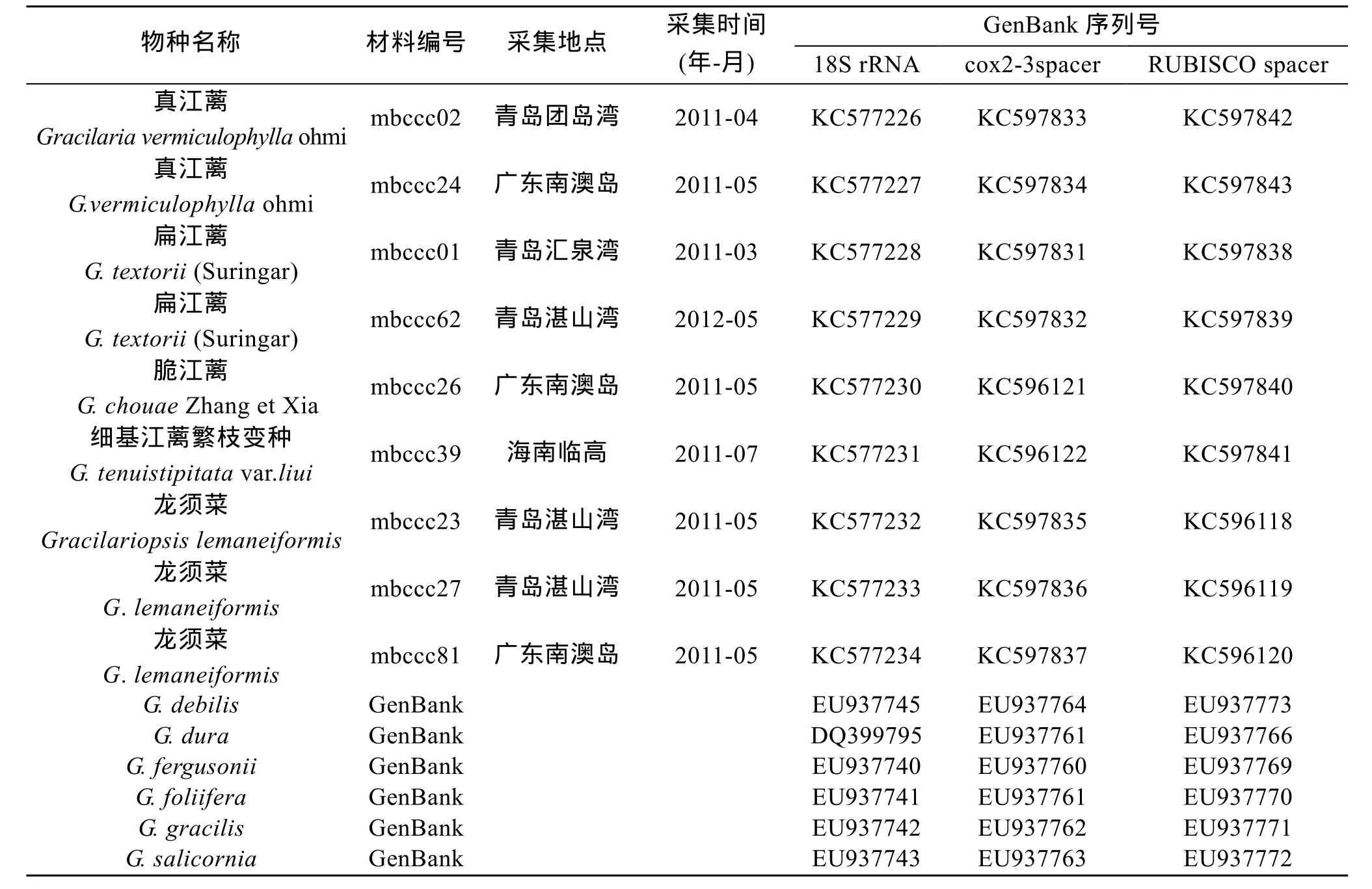
表1 样品采集信息Tab.1 Information of algal samples
1.3 数据分析
序列数据首先利用BLAST与GenBank中已有序列信息进行比对。然后利用ClustalW软件对测序数据进行人工对齐排序。用MEGA 5.0计算其碱基含量和变异位点, 并选择Kimura 2-parameter公式[16]计算遗传距离。并在MEGA5.0中采用最大简约法(Maximum parsimony, MP)构建系统树, 自举值Bootstrap为1000次重复。保留大于60的Bootstrap数值。选择线形紫菜(Porphyra linearisGreville)作为外群。
2 结果
2.1 18S rRNA序列分析结果
所有样品扩增条带去除引物后, 序列长度一致,均为1722bp。江蓠属海藻不同样品碱基频率一致, 均为A: 25.1%; T: 25.9%; G: 28.3%; C: 20.7%。龙须菜碱基频率与江蓠属略有不同A: 25.1%; T: 25.5%; G:28.5%; C: 20.9%。江蓠属样品转换/颠换比例(R)为1.42。江蓠属种间遗传距离为0.005~0.017, 共计61个变异位点; 江蓠属与龙须菜物种间的遗传距离在0.019~ 0.028 (表2), 共计90个变异位点。采自不同地区的龙须菜种内遗传距离为0.000, 变异位点为0个。与GenBank下载的数据人工比对之后序列长度为1699bp, 可分为2大分支, 包括龙须菜分支, 江蓠属分支。其中, 脆江蓠Gracilaria chouae与扁江蓠G.textorii聚合, 形成分支Ⅰ; 真江蓠Gracilaria vermiculophylla形成分支Ⅱ;G. debilis,G. fergusonii, G.salicornia与G. foliifera聚合组成分支Ⅲ(图1)。
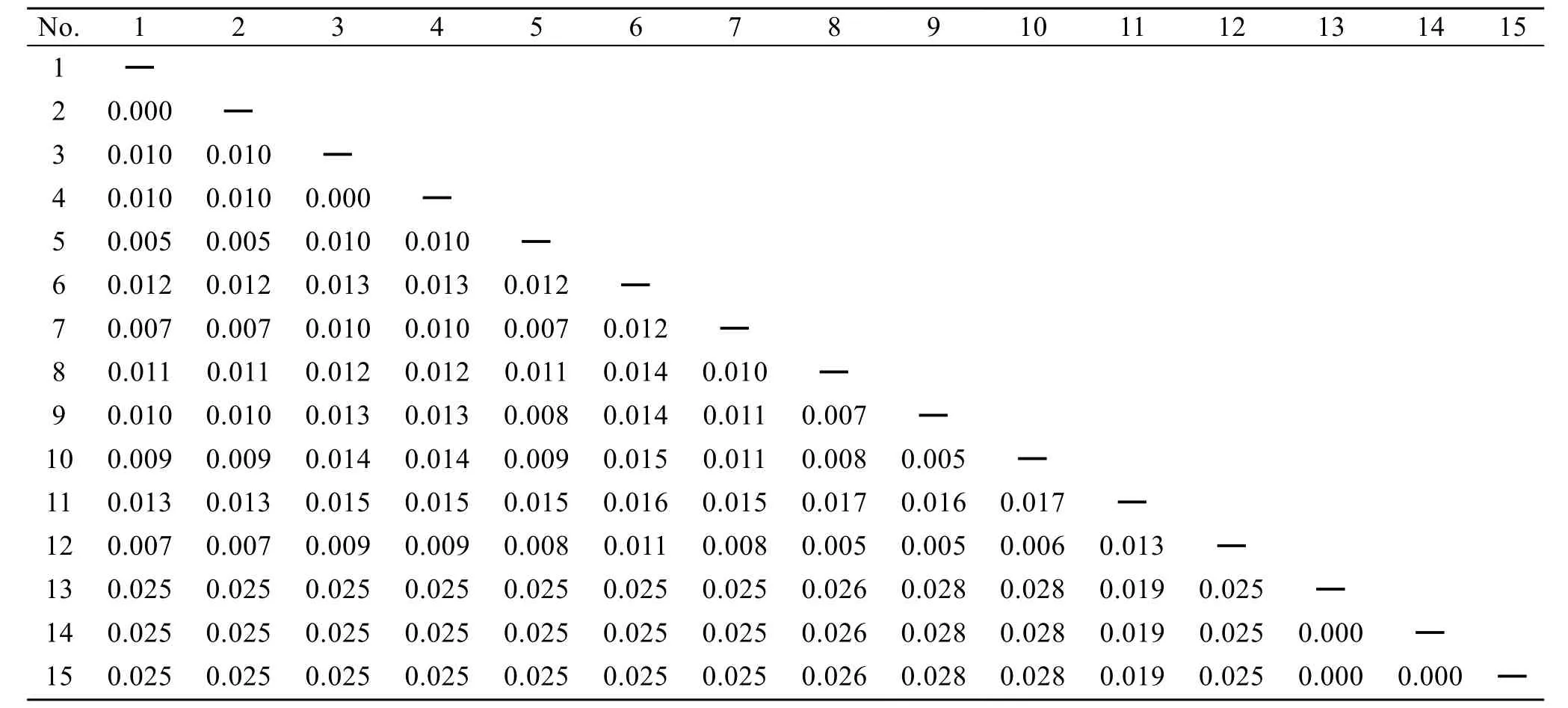
表2 基于18S rRNA序列的样品间遗传距离Tab.2 Genetic distance calculated using Kimura 2-parameter model for 18S rRNA sequences of samples

图1 样品18S rRNA序列系统进化树Fig.1 Maximum-parsimony (MP)phylogeny estimated using 18S rRNA sequence
2.2 COX2-3序列分析结果
目的片断去除扩增引物之后, 江蓠属海藻大小为343bp, 片段碱基频率略有不一, 平均GC含量为24.43%。龙须菜片段大小略长, 为347bp, GC含量为22.2%。江蓠属样品转换/颠换比例(R)为 1.32。江蓠属海藻种间遗传距离为 0.105~0.221, 共计 102个变异位点, 江蓠属与龙须菜属间的遗传距离为0.196~0.244, 变异位点共计112个, 龙须菜群体内遗传距离为 0.000(表 3), 变异位点 0个。与 GenBank下载的数据人工比对之后长度为310bp。江蓠属与龙须菜在系统树上分为两大分支, 龙须菜位于系统树的最基部。江蓠属中, 扁江蓠与脆江蓠聚成分支Ⅰ;真江蓠与细基江蓠繁殖变种,G. foliifera聚成分支Ⅱ;G. dura与G. debilis分别聚成分支Ⅲ(图2)。

表3 基于cox2-3间隔区序列的样品间遗传距离Tab.3 Genetic distance calculated using Kimura 2-parameter model for cox2-3 intergenic spacer sequences of samples
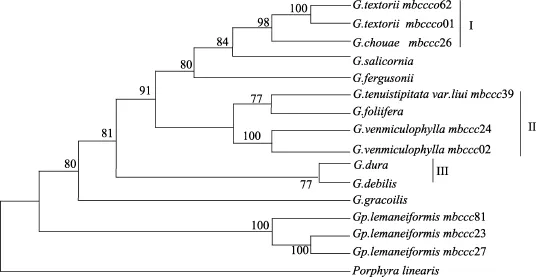
图2 样品cox2-3间隔区序列系统进化树Fig.2 Maximum-parsimony (MP)phylogeny estimated using cox2-3 intergenic spacer sequence
2.3 RUBISCO spacer序列分析结果
目的片段去除扩增引物后, 江蓠属海藻大小不一为 246~289bp, 平均 GC 含量为 49.7%; 龙须菜RUBISCO spacer序列长度为 289bp, GC含量为47.4%。江蓠属样品转换/颠换比例(R)为 1.41。江蓠属海藻种间遗传距离为0.048~0.215, 共有78个变异位点; 江蓠属与龙须菜之间的遗传距离为0.190~0.259, 共计86个变异位点。龙须菜群体遗传距离为0.000(表4), 变异位点0个。样品与GenBank下载的数据人工比对后长度为237bp, 系统进化树有2大分支, 江蓠属与龙须菜。江蓠属分支由4个小分支组成。其中, 扁江蓠与脆江蓠聚成分支Ⅰ,G.salicornia与G. fergusonii分支Ⅱ、G. dura与G. debilis聚成分支Ⅲ, 真江蓠与细基江蓠繁殖变种聚成分支Ⅳ。G. gracilis处于江蓠属分支的基部。龙须菜处于整个进化树的基部(图3)。
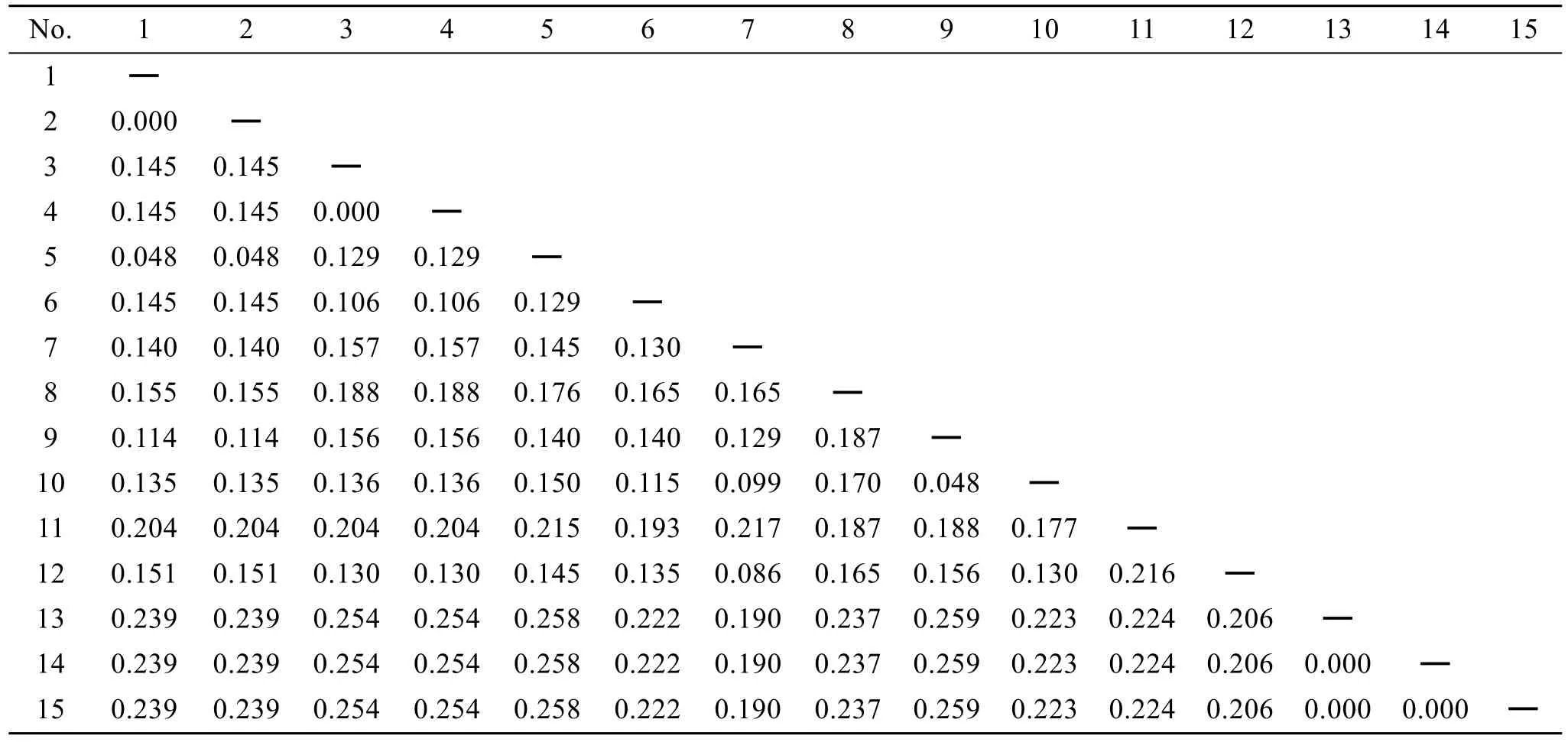
表4 基于RUBISCO间隔区序列的样品间遗传距离Tab.4 Genetic distance calculated using Kimura 2-parameter model for RUBISCO spacer sequences of samples
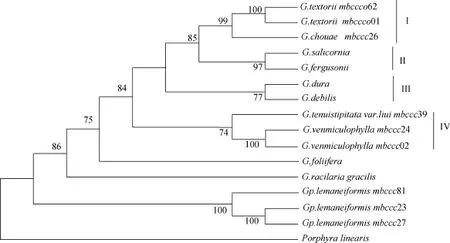
图3 样品RUBISCO间隔区序列系统进化树Fig.3 Maximum-parsimony (MP)phylogeny estimated using RUBISCO spacer sequence
3 讨论
历史上, 关于龙须菜的分类地位一直存有争议[17]。Papenfuss[18]认为龙须菜隶属江蓠属, 我国2007年出版的《中国黄渤海海藻》一书仍然将龙须菜归于江蓠属[11]。但近10年来, 越来越多的分子生物学研究结果表明龙须菜并不隶属于江蓠属。在本研究中, 位于基因组不同位点的18S rRNA、cox2-3间隔区、RUBISCO间隔区3个序列进行系统进化分析的结果表明: 龙须菜位于进化树的基部, 单独成支, 说明其与江蓠属相比, 其分化较早, 相对原始; 并且龙须菜与江蓠属海藻具有较远的遗传距离。这些结果进一步印证了Gurgel等人基于rbcL序列的研究结果[19]。他们认为, 江蓠科的物种在进化上是多起源的, 而龙须菜所在Gracilariopsis属较早出现了分化, 逐步进化成独立于另外2个江蓠属的单独一支, 因此龙须菜不应隶属于江蓠属。在本研究中, 扁江蓠与脆江蓠,G.dura与G. debilis在三个系统树中均聚合成支, 显示了它们之间具有较近的亲缘关系。Pareek等[20]研究了印度沿海地区江蓠属海藻系统进化关系, 他们发现G.foliifera与G. corticata的两个变种(var. corticata和cylindrica)具有很近的亲缘关系;G. salicornia和G.fergusonii可能是由共同祖先进化而来; 而G. gracilis与其他江蓠属海藻的亲缘关系较远。这一点, 在作者的结果中得到了证实,G. gracilis与其他样品之间也具有明显较远的亲缘关系。G. gracilis处于江蓠属分支的最底部, 说明其分化较早, 属于江蓠属内较为原始的物种。
本研究结果显示基于cox2-3间隔区、RUBISCO间隔区的进化树有相似的关系, 但是与基于18S rRNA的进化树又略有不同。原因在于相对于cox2-3间隔区、RUBISCO间隔区序列, 18S rRNA序列则更加保守。早在上世纪90年代, Bird[21-22]就发现18S rRNA序列不能在种水平区分江蓠科海藻, Bellorin[2]以及Iyer[4]均取得了类似结论。Medlin指出使用18S rRNA序列研究系统进化具有一些缺陷: 对于亲缘关系较近的物种的分辨度不足[23]。在作者的研究中也发现cox2-3间隔区、RUBISCO间隔区的保守性要弱于18S rRNA序列, 能更好的反应江蓠属海藻的系统进化关系。
本实验使用了18S rRNA、cox2-3间隔区、RUBISCO间隔区这3个序列, 而没有采用ITS序列。原因在于: 第一, 之前已经有相当多的工作使用ITS序列研究江蓠科的海藻[5,9]; 第二, Saunders发现ITS序列在红藻中具有一定的多态性问题[24]。虽然李敏等[9]认为来自同一居群龙须菜个体的ITS序列相同,但是在作者之前的研究中扩增江蓠属真江蓠的ITS序列时确实遇到了这一问题[25], 因此如果使用ITS序列研究整个江蓠属海藻, 有可能会对后续的工作造成不同程度的干扰。同时, 18S rRNA、cox2-3间隔区、RUBISCO间隔区这3个序列尚未发现多态性问题,这也是作者选择这三个序列的原因之一。
随着分子生物学的不断发展, 相关生物学数据的不断积累, 利用DNA序列研究物种系统进化, 已经成为生物学研究十分有利的工具。但目前来看, 还存在相当多的问题。例如, 不同种群同一DNA序列,同一种群不同DNA序列的进化速率都有可能存在差异。因此, 利用基因组不同位点的基因系统地研究海藻的基因进化关系就显得更为重要和迫切, 这样得到的结果相对单纯凭借一个DNA序列进行分析, 会更加客观, 全面。在本研究中, 作者使用核基因编码的18S rRNA基因、位于线粒体的cox2-3间隔区、以及位于叶绿体RUBISCO间隔区这三个序列研究我国的江蓠属海藻与龙须菜的系统进化, 研究结果进一步证明龙须菜并不隶属于江蓠属, 且相对原始。同时也表明: 扁江蓠与脆江蓠,G. dura与G. debilis具有较近的亲缘关系;G. gracilis分化较早, 是江蓠属内较为原始的物种; cox2-3间隔区、RUBISCO间隔区比起18S rRNA更适用于研究江蓠属海藻的系统进化关系。
致谢: 感谢中国科学院海洋研究所夏邦美老师在物种分类鉴定方面的帮助与指导。本项目得到了中国科学院野生生物资源库运补费项目的资助。
[1]Troell M, Halling C, Neori A, et al. Integrated mariculture: asking the right questions [J]. Aquaculture,2003, 226, 69-90.
[2]Bellorin A M, Oliveira M C, Oliveira E C. Phylogeny and systematics of themarine algal family Gracilariaceae (Gracilariales, Rhodophyta)based on small subunit rDNA and ITS sequences of Atlantic and Pacific species [J]. J. Phycol, 2002, 38: 551-563.
[3]夏邦美, 张峻甫. 中国海藻志[M]. 第二卷, 红藻门,第五册. 北京: 科学出版社, 1999: 55-58.
[4]Iyer R, Tronchin E M, Boliton J J, et al. Molecular sysytematics of the Gracilariaceae (Gracilariales)with emphasis on southern Africa[J]. J Phycol, 2005, 41:672-684.
[5]李婷婷, 陈斌, 陈省平, 等.江蓠属和龙须菜属5种海藻ITS序列分子系统学分析[J]. 中山大学学报, 2012,51(4): 97-105.
[6]Guillemin M L, Akkisa, Givernaud T, et al.Molecular characterisation and development of rapid molecular methods to identify species of Gracilariaceae from the Atlantic coast of Morocco[J]. Aquatic Botany, 2008, 89:324-330.
[7]Byrne K, Zuccarello G C, West J, et al. Gracilaria species(Gracilariaceae, Rhodophyta) from southeastern Australia, including a new species,Gracilaria perplexasp. Nov. : Morphology, molecular relationships and agar content[J]. Phycological Research, 2002, 50:295-311.
[8]Weinberger F, Guillemin M L, Destombe E C, et al.Defense evolution in the Gracilariaceae ( Rhodophyta):substrate-regulated oxidation of agar oligosaccharides is a more ancient than the oligoagar-activated oxidative burst[J]. J Phycol, 2010, 46: 958-968.
[9]李敏, 隋正红, 易恒, 等. 龙须菜5.8SrRNA和ITS区的克隆与系统学分析[J]. 中国海洋大学学报, 2009,39(1): 77-83.
[10]Tseng C K. Common Seaweeds of China[M]. Beijing:Science Press, 1983: 102-110.
[11]曾呈奎, 夏邦美, 周显铜.中国黄渤海海藻[M]. 北京:科学出版社, 2009: 179-185.
[12]Goff L J, Coleman A W. The use of plastid DNA restriction endonuclease patterns in delineating red algal species and populations[J]. J Phycol, 1988, 24(3):357-368.
[13]Saiki R K, Gelfand D H, Stoffel S, et al,Primer-directed enzymatic amplification of DNA with a thermo stable DNA polymerase[J]. Science, 1988, 239:487-491.
[14]Zuccarello G C, Burger G, West J A, et al. A mitochondrial marker for red algal intraspecific relationships[J]. Mol. Ecol, 1999a, 8: 1443-1447.
[15]Zuccarello G C, West J A, Kamiya M, et al. A rapid method to score plastid haplotypes in red seaweeds and its use in determining parental inheritance of plastids in the red alga Bostrychia (Ceramiales)[J]. Hydrobiologia,1999b, 401: 207-214.
[16]Kimura M. A simple method for estimating evolutionary rate of base substitutions through comparative studies of nucleotide sequences[J]. J Mol Evol, 1980, 16: 111-120.
[17]隋正红, 张学成. 委内瑞拉产地龙须菜藻红蛋白基因的克隆及其系统学研究[J]. 青岛海洋大学学报(自然科学版), 2003, 33(3): 384-390.
[18]Papenfuss G F. Notes on nomenclature V. Various Chlorophyceae and Rhodophyceae [J]. Phykos, 1966, 5:95-105.
[19]Gurgel C F D, Liao L M, Fredericq M. Systematics of gracilariopsis (Gracilariales, Rhodophyta)based onrbcL sequence analyses and morphological evidence [J].J Phycol, 2003, 39: 1-19.
[20]Pareek M, Mishra A, Jha B. Molecular phylogeny ofGracilariaspecies inferred from molecular markers belong to three different genomes [J]. J Phycol, 2010,46, 1322-1328 .
[21]Bird C J, Rice E L, Murphy C, et al. Phylogenetic relationships in the Gracilariales (Rhodophyta)as determined by 18S rDNA sequences [J]. Phycologia,1992, 31: 510-522.
[22]Bird C J, Ragan M A, Critchley A T, et al. Molecular relationships among the Gracilariaceae (Rhodophyta):further observation on some undetermined species [J].Eur J Phycol, 1994, 29: 195-202.
[23]Medlin L K, Metfies K, John U. Algal molecular systematics: a review of the past and prospects for the future. [C]// Brodie J, Lewis J. Unravelling the Algae:The Past, Present, and Future of Algal Systematics.Systematic Association Special Volume. London: CRC Press, 2007: 341-353.
[24]Saunders G W. Applying DNA barcoding to red macroalgae: a preliminary appraisal holds promise for future applications. Philos[J]. Trans R Soc B Biol Sci,2005, 360: 1879-1888.
[25]Zhao X B, Pang S J, Shan T F , et al. Applications of Three DNA barcodes in assorting intertidal red macroalgal flora in Qingdao, China[J]. J Ocean Univ China, 2013, 12(1): 139-145.

