中国裸胸鳝属10种鱼类分子系统发育关系的16S rDNA分析
杜 民, 尹绍武, 刘艳红, 牛宝珍, 齐兴柱, 张 本, 廖经球, 霍 蕊
(1.红河学院 云南省高校农作物优质高效栽培与安全控制重点实验室, 云南 蒙自 661199; 2.南京师范大学生命科学学院, 江苏 南京 210023; 3.海南大学 海洋学院 热带生物资源教育部重点实验室, 海南 海口570228)
裸胸鳝属于硬骨鱼纲(Osteichthyes), 鳗鲡总目(Anguillomorpha)鳗鲡目(Anguilliformes)海鳝科(Muraenidae)[1], 中国裸胸鳝属(Gymnothorax)共有20个物种[2]。裸胸鳝为暖水性鱼类, 在中国广泛分布于西沙群岛、海南岛、台湾以及广东、福建沿海等海域。裸胸鳝的传统分类主要依据体表的颜色、斑点、条纹等。但裸胸鳝鱼类在不同的成长阶段, 不同的生活环境及不同的应激状态下, 其外部斑纹都会有一定的变化, 此外可能存在种间杂交等因素也对裸胸鳝鱼的系统分类造成很大困难。
通过现代分子生物学技术, 获得物种特定遗传标记的大量数据, 构建系统进化树是对传统分类的重要佐证以及补充或修正。线粒体 DNA结构简单,严格母系遗传, 是研究物种起源进化及分析种间亲缘关系的理想标记, 其中16S rDNA基因进化速度较为适中, 常用于种以上水平的系统进化研究, 是探讨种间和种内生物进化和系统发育最常用的分子标记之一, 它的一个基因片段包含了从种内到种间乃至科间的进化遗传信息[3-4], 在系统进化和分类研究上有较强的适用性[5-6], 该基因已被广泛应用到鱼类属和种间的系统发育关系中, 解决了一些分类和系统演化关系的问题[1,7-8], 齐兴柱等[9]利用细胞色素氧化酶亚基Ⅰ(mtDNA-COⅠ)基因片段序列对6种裸胸鳝属鱼类进行了分析, 又利用细胞色素氧化酶亚基Ⅱ(mtDNA-COⅡ)的基因片段序列对另外6种裸胸鳝属的分类情况进行了聚类研究[10], 杜民等[11]利用细胞色素b基因的片段序列对6种裸胸鳝的分类情况进行了分析。
本研究以分布于中国南海裸胸鳝属常见的细斑裸胸鳝(G. fimbriatus)、黄纹裸胸鳝(G. pseudothyrsoideus)、网纹裸胸鳝(G. reticularis)、宽带裸胸鳝(G.rueppeliae)、斑第氏裸胸鳝(G.berndti)、斑颈裸胸鳝(G. margaritophorus)、布雷顿氏裸胸鳝(G. breedeni)、黑点裸胸鳝(G. melanospilus)、褐祼胸鳝(G. hepaticus)等 9种鱼类[12-13]为对象, 采用聚合酶链式反应将PCR扩增产物克隆到T载体后进行16S rDNA基因序列测定, 结合GenBank中蠕纹裸胸鳝(G. kidako)的16S rDNA基因序列, 并用从GenBank中下载的花鳗鲡(Anguilla marmorata)的 16S rDNA基因序列作为外群进行比较分析和构建分子系统树, 初步探讨裸胸鳝属鱼类的系统发育关系, 为进一步完善裸胸鳝属鱼类分类系统演化关系提供基础资料。
1 材料与方法
1.1 样品来源
样品从中国海南附近海域及西沙群岛采集(表1),共有9个种类, 每种采集3~6尾, 实验的裸胸鳝属鱼类根据成庆泰等[1]编著的《中国鱼类系统检索》和沈世杰[12]编著的《台湾鱼类检索》上报道的标准进行种类鉴定。除了蠕纹裸胸鳝、外群花鳗鲡(Anguilla marmorata)以外, 其他的裸胸鳝均来自中国南海海域, 其来源见表1。

表1 研究动物和DNA序列数据来源Tab. 1 Sources of DNA sequence data and tissue samples of studied animals
1.2 总DNA的提取
样品取背部肌肉于 95%乙醇中保存(–20℃冻存)。基因组DNA的提取参考Sambrook等[13]的酚/氯仿抽提法。取5μL产物用1. 0%琼脂糖凝胶, 5V/cm, 进行电泳检测。然后存放于–20℃用作PCR反应的模板。
1.3 PCR扩增与序列测定
扩增采用嵌合式PCR方法, 第1次PCR的产物作为第2次PCR的模板。目的片段扩增用的引物是挑选一种海鳝科鱼类线粒体基因组序列作为模板, 利用Primer5.0软件在其16S rDNA基因序列两端的保守区设计2对引物Gym16SrRNAsen2、Gym16SrRNA- ant2、Gym16SrRNAsen3和Gym16SrRNAant3, 由上海生物技术有限公司合成, 引物序列 Gym16SrRNAsen2为5'-CGTGCAAATCGAGTCGTCC-3', Gym16SrRNAant2为5'-TCTGCCACC TTAGC ATGCC-3', Gym16SrRNAsen3为5'-AGCCTGAACACAACCGTAACC-3', Gym16SrRNA-ant3为5'-CAGGTG GCTGCTTTTAGGC-3'。反应总体积25 μL, 其中 Buffer和 dNTP 各 2.5μL, 去离子水 16.2μL,正向和反向引物各1.3 μL, mtDNA模板1.0 μL, ExTaq酶0.2 μL。扩增条件为94℃预变性4 min, 然后循环40次(94℃变性0.5 min, 50℃退火0.5 min, 72℃延伸2 min),最后 72℃延伸 10 min。PCR反应试剂盒(包括Buffer、dNTP和ExTaq酶)购自大连宝生物公司。利用未加模板 DNA的反应液作为空白对照以检查是否有污染存在。PCR产物用1.0%琼脂糖凝胶电泳检测,于–20℃保存。
1. 4 PCR产物纯化和测序
扩增得到的 PCR产物经琼脂糖凝胶电泳, 所需带切下后利用宝生物工程(大连)有限公司生产的DNA凝胶回收试剂盒(TaKaRa Anarose Gel DNA Purification Kit Ver. 2. 0)纯化回收。并将其构建到PMD18-T载体并送上海生工测序, 测序引物为通用引物M13[14]。
1. 5 DNA序列数据的处理
利用在线的blast软件及Clustal X[15]排定DNA序列并经人工核查。采用DNASP4.0软件分析10种裸胸鳝的多态位点和多态简约信息位点数; 用分子进化遗传分析软件MEGA5.0[16]软件计算不同序列间的碱基替代数、变异位点、颠换百分比、转换/颠换比率; 系统分析采用邻接法 Neighbore-Joining(简称NJ)里面的 Kimura 2-parameter法, 最小进化法Minimum Evolution(简称 ME)及最大简约法Maximum Parsimony(简称MP)构建分子系统进化树,置信度用Bootstrap工具1000次循环检验分子系统树,以花鳗鲡(GenBank登录号为: AB021760)的同源序列作为外群。
2 结果
2.1 16S rDNA基因片段序列长度及碱基组成
利用在线的blast程序和Clustal X程序对测定及下载的序列进行对位排列, 辅以手工校正, 除去多余序列, 本实验中扩增得到裸胸鳝属鱼类16S rDNA基因片段长度为516 bp。采用MEGA5.0中的统计软件计算它们的碱基组成(表2)。从表2可知, 它们平均碱基组成T为18.0%, C为22.1%, A为37.8%, G为22.2%。
2.2 16S rDNA基因片段序列
10种裸胸鳝属鱼的16S rDNA核苷酸扩增片段进行测序, 得到16S rDNA序列及从基因库检索获得的蠕纹裸胸鳝的同源序列(图1)。
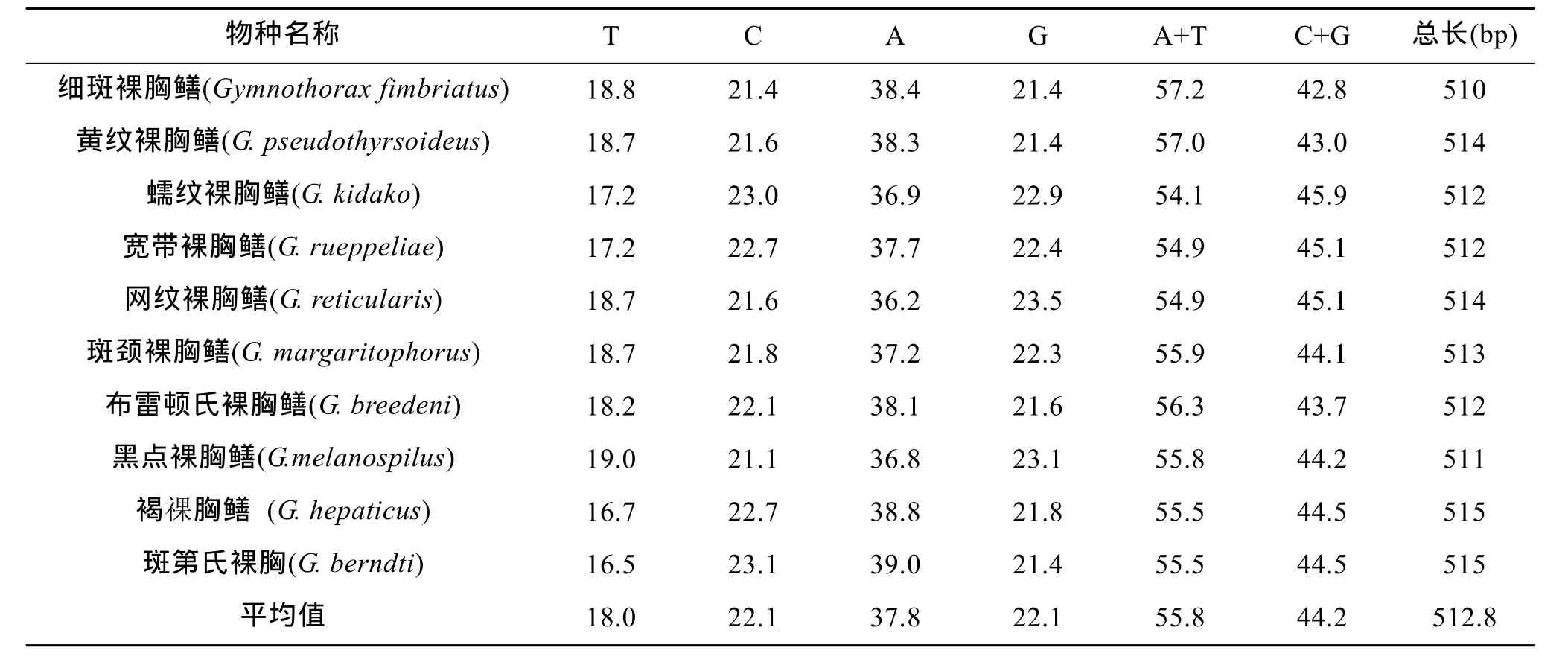
表2 10种裸胸鳝16S rDNA片段长度和碱基组成Tab.2 Base composition of 16S rDNA gene fragments of ten species of Gymnothorax
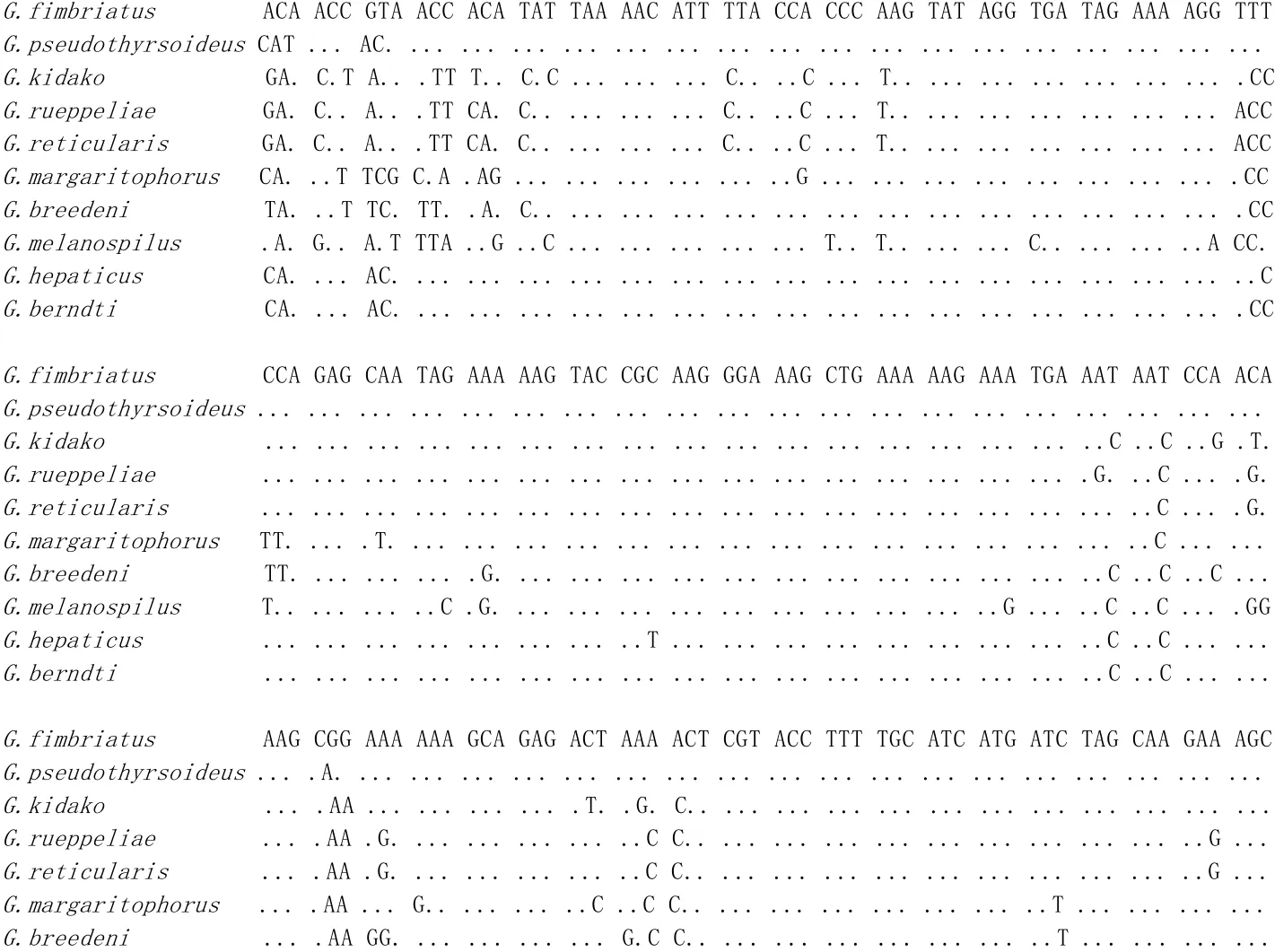
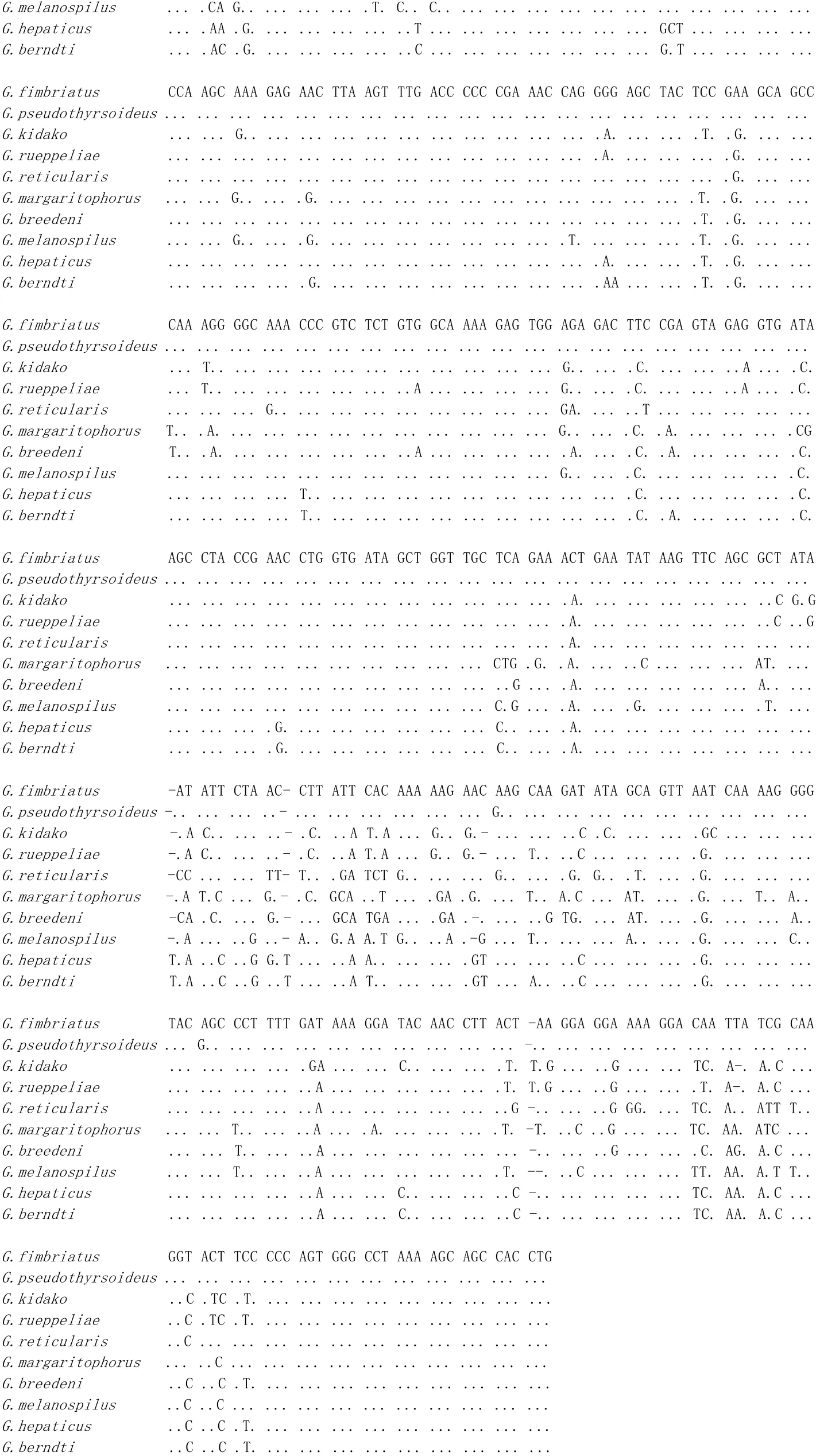
图1 裸胸鳝属鱼类16S rDNA 516bp的碱基序列Fig.1 DNA sequences of mitochondrial 16S ribosomal DNA 516 bp of ten species of Gymnothorax.
从图1可知, 对本实验所研究的10个裸胸鳝鱼类的线粒体16S rDNA基因扩增片段序列经排序, 共获得516 bp的碱基序列, 部分位点存在缺失现象。
2.3 10种裸胸鳝之间的相对遗传距离
利用MEGA5.0软件中的双参数法, 以转换加颠换、转换比颠换分别计算10种裸胸鳝之间的相对遗传距离(表3)。
从表3可知, 10种裸胸鳝之间的序列差异(转换加颠换)为0.022~0.177, 其中褐裸胸鳝和布雷顿氏裸胸鳝序列差异最小为 0.022, 网纹裸胸鳝与斑颈裸胸鳝的序列差异最大为0.177, 平均为0.123; 种间序列差异不大。此外, 10种裸胸鳝鱼类之间序列碱基转换与颠换比值为1.732~5.830, 平均为3.441。一般认为转换颠换比值小于 2.0时基因序列突变已经达到饱和状态[17-18], 这说明10种裸胸鳝大部分种类的 16S rDNA基因片段序列变异并不显著。
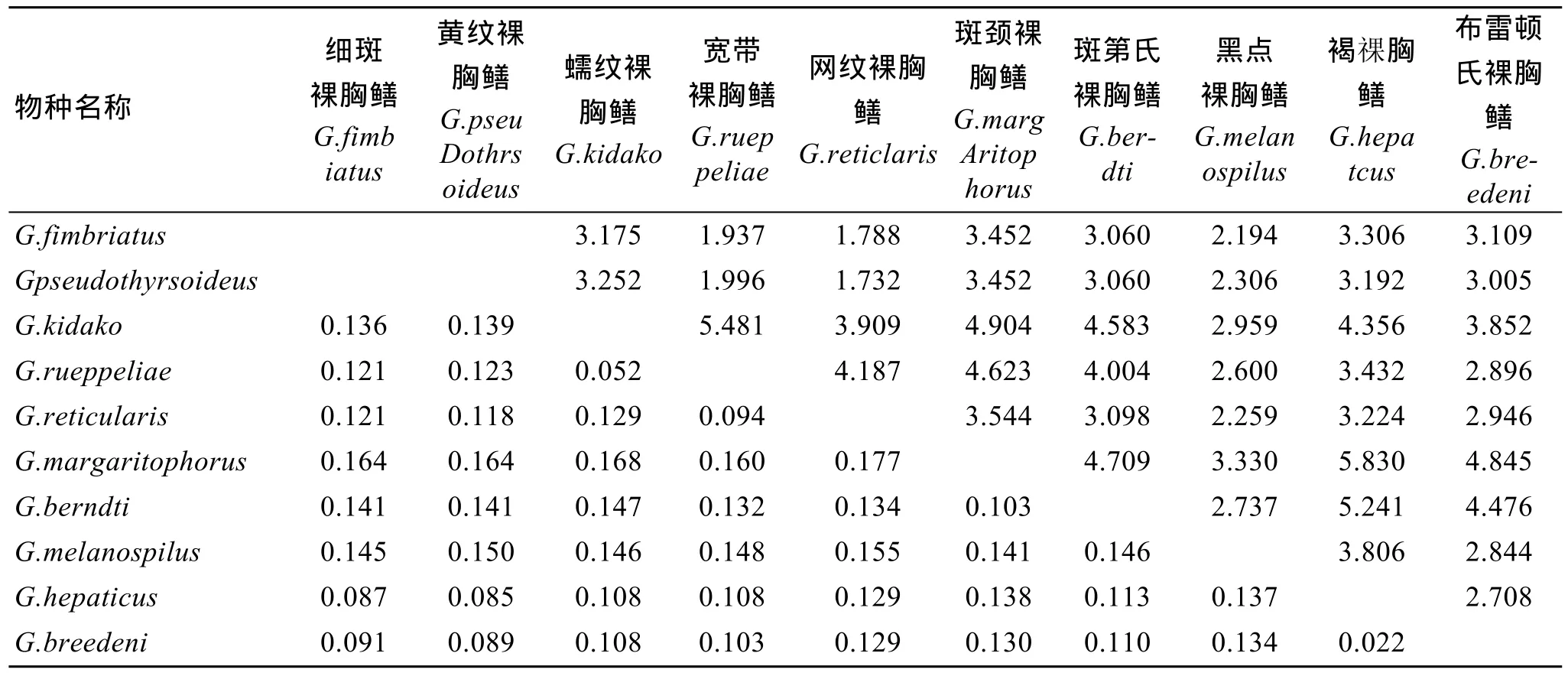
表3 相对遗传距离Tab. 3 Pairwise distance matrix
2.4 10种裸胸鳝鱼类系统进化树的建立
以花鳗鲡作为外群构建NJ树(图2A)、MP树(图2B)和 ME树(图 2C)所有系统树支上数值为 1000次自举(Bootstrap)检测得到的对该分支的支持百分数。从图2中可以看出这3种不同的分子系统进化树具有基本相同的拓扑结构。分为明显的两大分支, 花鳗鲡分为独立的一支, 本实验的10种裸胸鳝鱼类分为另外一支。
3 讨论
1795年, Bloch将裸胸鳝属定义为Gymnothorax。目前, 对裸胸鳝属鱼类的分类仅依据体色花纹等外形特征[1,12], 而且在不同的生长阶段体色花纹不尽相同, 属、种间也出现重叠, 依据形态的分类方式十分困难, 而且也容易出错。裸胸鳝属鱼类与鳗鲡属的花鳗鲡同属于鳗鲡科。本实验研究结果也支持这一观点: 利用 ClustalX软件计算 10种裸胸鳝的 16S rDNA部分序列之间的序列相似性(在本文的10个裸胸鳝属鱼类中, 云纹裸胸鳝和斑点裸胸鳝序列相似性最小值为 81%, 本研究中其余的裸胸鳝鱼类之间相似性都高于 81%)高于它们与花鳗鲡的 16S rDNA部分序列相似性(本研究的 10个裸胸鳝属鱼类与花鳗鲡相似性比较中, 斑点裸胸鳝和花鳗鲡的序列相似性属间最大为 80%, 其余裸胸鳝属鱼类与花鳗鲡相似性都低于 80%)。NJ树和 MP树的聚类图表明:花鳗鲡与裸胸鳝属其中鱼类分离, 成为单独一支,与传统分类结果一致, 证明16S rDNA是解决分类及系统进化问题最可信的分子标记之一[19]。
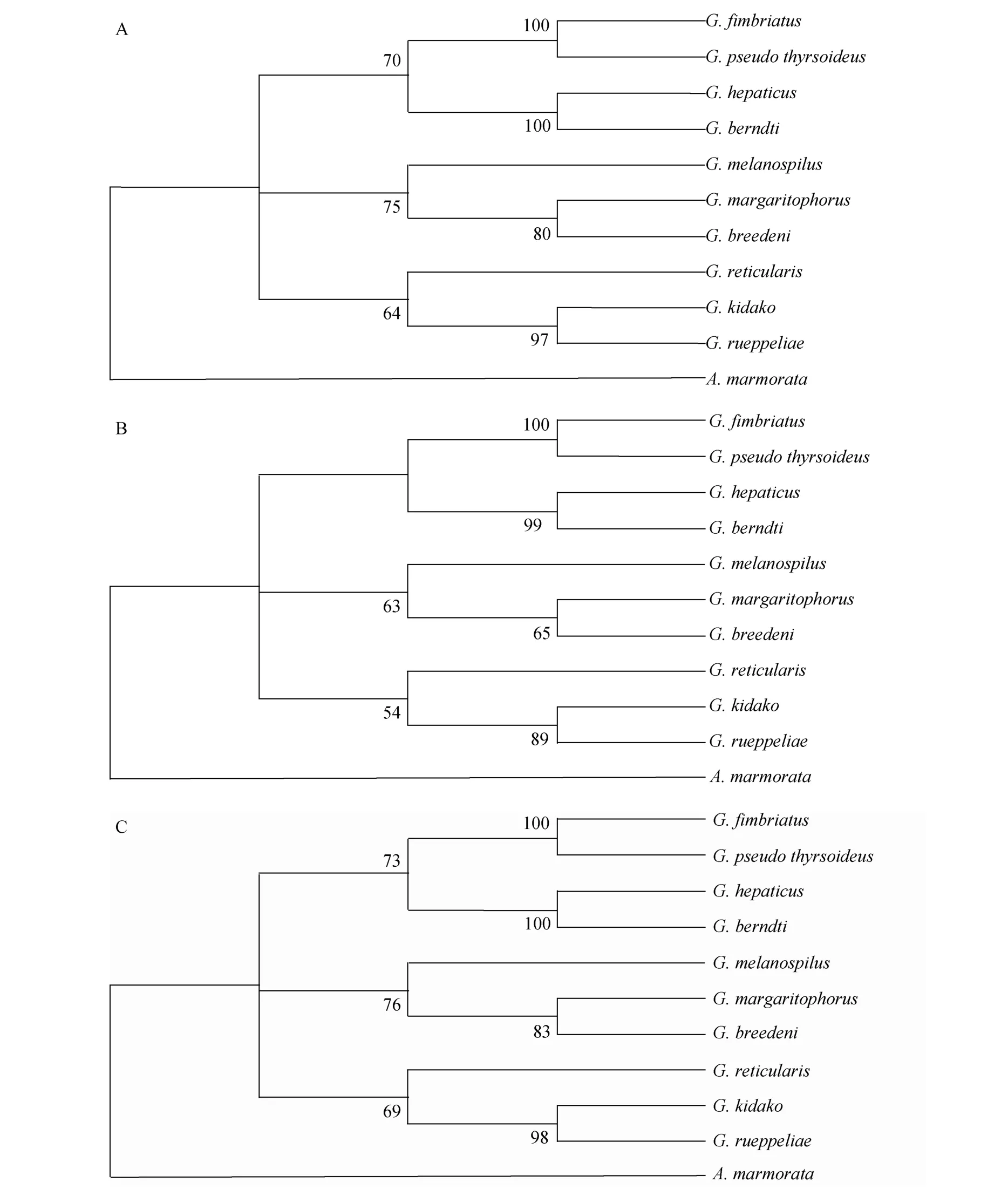
图2 基于16S rDNA基因部分序列构建的分子系统树Fig. 2 Molecular phylogenetic trees based on 16S ribosomal DNA sequences data with bootstrap test
采用DNASP4.0软件分析10种裸胸鳝的多态简约信息位点(Parsimony informative sites)和单突变位点(Singleton variable sites), 发现在所测的516个碱基序列中, 存在插入缺失现象, 共检测出 10个缺失位点, 143个核苷酸变异位点。占整段序列的27.7%。简约信息位点 (two variants)为 80个, 简约信息位点(three variants)为21个; 多态简约信息位点数(Parsimony informative sites)共计为101个, 单突变位点数(Singleton variable sites)为42个, 单突变位点 (two variants)为35个, 单突变位点(three variants)为7个。总体上看, 序列中的转换明显比颠换多, 平均为 3.441(表 3), 说明序列突变已经达到饱和。其中A-C和A-T颠换多于C-G和T-G颠换, T-C转换多于A-G转换。T、C、A、G的含量平均值分别为 18.0%、22.1%、37.8%、22.1%, 其中A+T含量(55.8%)明显高于C+G含量(44.2%), 序列表现出较明显的A偏倚, 而G偏倚并不明显[20]。
NJ树、MP树和ME树的聚类图表明: 拓扑结构分为明显的两个大分支, 其中花鳗鲡单独聚为一支,说明在所分析的11种鱼中, 它是最早分化出来的。本研究的10种裸胸鳝中的其他种类, NJ树和ME树的聚类图表明又分为 3个平行进化的小分支, 即蠕纹裸胸鳝、波纹裸胸鳝和宽带裸胸鳝先聚为一支(置信度分别为 64%、69%), 布雷氏裸胸鳝、斑颈裸胸鳝和黑点裸胸鳝聚为一支(置信度75%、76%), 细斑裸胸鳝、黄纹裸胸鳝、褐裸胸鳝和斑第氏裸胸鳝聚为一支(置信度70%、73%); 而MP树的结果表明分为四个平行进化的小分支, 即蠕纹裸胸鳝、波纹裸胸鳝和宽带裸胸鳝先聚为一支(置信度54%), 布雷氏裸胸鳝、斑颈裸胸鳝和黑点裸胸鳝聚为一支(置信度63%), 细斑裸胸鳝和黄纹裸胸鳝聚为一支(置信度100%), 褐裸胸鳝和斑第氏裸胸鳝聚为一支(置信度99%)。说明在本研究的10种裸胸鳝属鱼中, 可能存在着几种平行进化的种类。GenBank上面蠕纹裸胸鳝的样本来源于日本海, 而其余的 9种裸胸鳝来源于中国南海, 从系统树上来看, 它们之间并没有明显的地区差异; 这个研究结果与其他的研究结果相一致: 杜民等[11]利用6种裸胸鳝的细胞色素b基因的全长序列构建的系统树显示来自日本海的蠕纹裸胸鳝与来自中国海的另外 5种裸胸鳝没有地域上的差别, 齐兴柱等[9]利用6种裸胸鳝的细胞色素氧化酶亚基Ⅰ(mtDNA-COⅠ)构建的系统进化树及利用6种裸胸鳝的细胞色素氧化酶亚基ⅡmtDNA-COⅡ[10]的聚类结果也展示了被研究的裸胸鳝的基因序列并没有地域上的差异。
本研究通过用实验者自行设计的引物对其16SrDNA进行PCR扩增并对扩增的序列进行比较研究的结果表明, 设计的这对引物在裸胸鳝鱼类中具有普遍的适应性, 所扩增的序列除了宽带裸胸鳝与黄纹裸胸鳝、网纹裸胸鳝与黄纹裸胸鳝转换比颠换值小于 2外(如果某个物种的基因序列比较时, 转换与颠换的比值小于 2时, 则此基因序列的突变已经达到饱和状态, 受进化噪音的影响可能性较大, 构建系统进化树时可能会受影响)[21], 本研究裸胸鳝的其余种类之间的转换比颠换值都在 2以上且序列差异也不大, 说明本实验中裸胸鳝鱼类的16S rDNA基因片段序列突变还没有达到饱和, 构建的系统进化树具有的可信度较高。本研究所得的16S rDNA序列适合裸胸鳝鱼类的分子进化和遗传多样性分析。本研究通过对10种裸胸鳝鱼类的16S rDNA基因部分序列的比较研究, 以期为整个裸胸鳝属鱼类分子水平的系统发生和进化研究以及遗传多样性研究提供依据。裸胸鳝属鱼类分类较为复杂, 系统发生关系有待对更多的种类从分子水平来揭示。
[1]成庆泰, 郑葆珊. 中国鱼类系统检索[M]. 北京: 科学出版社, 1987: 99-108, 747-757.
[2]孟庆闻, 苏锦祥, 缪学祖. 鱼类分类学[M]. 北京: 中国农业出版社, 1995: 18, 139-141.
[3]Kochet T D, Thomas W K, Meger A, et al. Dynamics of mitochondrial DNA evolution in animals: Amplification and sequencing with conserved primers [J]. Proc Natl Acad Sci USA, 1989, 86: 6190-6200.
[4]Meyer A, Wilsom A C. Origin of tetrapods inferred from their mitochondrial DNA affilitation to lungfish[J].J Mol Evol , 1990, 31: 359-364.
[5]Irwin D M, KocherT D, Wilson A C. Evolution of the cytochrome b gene of mammals [J]. J Mol Evol, 1991,32: 128-144.
[6]Wilson A C, Cann R L, Carr S M, et al. Mitochondrial DNA and two perspectives on evolutionary genetics [J].Biol J Linn Soc, 1985, 26: 375-400.
[7]Martin A P, Bermingham E. Systematics and evolution of lower central American cichlid inferred from analysis of cytochrome b gene sequences [J]. Mol Phylogenet Evol, 1998, 9: 192-203.
[8]Briolay J, Galtie rN, Brito R M, et al. Molecular phylogeny of Cyprinidae inferred from cytochrome b DNA sequences[J].Mol Phylogenet Evol, 1998, 9: 100-108.
[9]齐兴柱, 骆剑, 刘志亮, 等. 基于 CO I序列的 DNA条形码在中国南海裸胸鳝属鱼类中的应用[J]. 热带生物学报, 2011, 1(4): 321-326.
[10]齐兴柱, 尹绍武, 张本, 等. 基于 mtDNA-COⅡ基因序列对中国南海裸胸鳝属鱼类分子系统进化关系的研究[J]. 水产科学, 2010, 29(10): 605-609.
[11]杜民, 齐兴柱, 尹绍武, 等. 基于 Cyt b 基因序列研究 6 种裸胸鳝属鱼类的进化关系[J]. 中国水产科学,2009, 16 (1): 24-30.
[12]沈世杰. 台湾鱼类检索[M]. 台北: 南天书局有限公司, 1984: 101-107.
[13]Sambroo J, Fitch E, Maniatis T. Molecular Cloning: A Laboratory Manual.2nd edn[M].New York: Cold Spring Harbor Laboratory Press, 1989.
[14]Sambroo J, Fitch E, Maniatis T. Molecular Cloning; A LaboratoryManual. 2nd ed[M]. New York: Cold Spring Harbor Laboratory Press, 1989.
[15]Thompson J S, Gibson T J, Plewniak F, et al. The Clustal_X windows interface: Flexible strategies for multiple sequences alignment aided by quality analysis tools [J].Nucleic Acids Research, 1997, 25(24): 4876-4882.
[16]Tamura K, Peterson D, Peterson N, et al. MEGA5:Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J].Mol Biol Evol, 2011, 28(10): 2731-2739.
[17]周继亮, 张亚平, 黄美华, 等. 蝮亚科蛇线粒体Cytb基因序列分析及其系统发育[J]. 动物学报, 2001, 47(4): 361-366.
[18]周发林, 江世贵, 苏天凤, 等. 6种笛鲷属鱼类Cytb基因片段序列的比较[J]. 热带海洋学报, 2004, 23(4): 87-92.
[19]Zardoya R, Meyer A. Phylogenetic performance mitochondrial protein coding genes in resolving relationship among Vertebrates[J].Mol Biol Evol, 1996, 13: 933-942.
[20]丁少雄, 王颖汇, 王军, 等. 基于 16S rDNA 部分序列探讨中国近海30种石斑鱼类的分子系统进化关系[J]. 动物学报, 2006, 52(3): 504-513.
[21]Vidal N, Lecointre G. Weighting and congruence: a case study based on three mitochondrial genes in pitvipers[J]. Mol Phyl Evol, 1998, 9(3): 366-374.

