适于鱿鱼肌肉组织中菌群特征分析的PCR-DGGE方法的建立
邓弘毅,王 月,丁林贤,王建玲,金 杨
(1.台州出入境检验检疫局,浙江 台州 318000;2.浙江师范大学,浙江 金华 321004)
鱿鱼,属软体动物的门头足纲,其肉质鲜美,风味与鲍鱼类似,国外有将鱿鱼加工成类似鲍鱼的罐头出售,因此鱿鱼又被冠以“穷人的鲍鱼”的美称,是广受欢迎的水产品之一[1]。但由于其水分含量高,蛋白质丰富,微生物含量较多,特别容易腐败变质,因此会引发一系列食品安全问题。目前关于食品中菌群特征方面的研究,国内外已有不少报道,但许多研究仍停留在传统的微生物鉴定法。由于自然界中有85%~99%的微生物至今还不可培养,因此传统的微生物鉴定法具有一定的局限性[2]。近年来,随着分子生物学的发展,越来越多的分子生物学手段被引入到食品微生物学的研究中。其中,基于传统PCR技术发展起来的PCR-变性梯度凝胶电泳 (DGGE)指纹分析技术就是一种可以有效避开微生物的分离培养阶段,直接从食品中提取总DNA,揭示样品中的微生物组成和动态变化的方法。
该方法首先由 Fischer等[3]于 1979年提出,1985年 Myers等[4]首次在 DGGE中使用 “GC夹板”和异源双链技术,使该技术日臻完善。目前,PCR-DGGE技术已广泛应用于环境微生物的微生物生态和种群群落的研究,并在食品领域尤其在水产品保鲜及加工中得到了一定的应用。如Hovda等[5]利用该技术对气调包装大马哈鱼中的优势菌群进行分析,发现传统以及改良的气调包装大马哈鱼中的优势菌群为磷发光杆菌、热杀索氏菌和假单胞菌属。Yoshikawa等[6]对大马哈鱼子酱发酵过程中的菌群情况研究时发现耐盐性酵母菌、假丝酵母等4种真菌是大马哈鱼子酱发酵过程中的优势菌群。
进行DGGE分析的首要问题就是样品中总细菌DNA的提取。作者以冻融法、匀浆法和摇床法3种方法处理鱿鱼样本,采用SDS法结合蛋白酶K法直接对鱿鱼组织中的细菌基因组DNA进行提取,建立了一套适合鱿鱼组织中菌落结构分析的方法,为进一步研究鱿鱼腐败机理提供技术支持。
1 材料与方法
1.1 试验材料
新鲜鱿鱼为购自金华市果蔬水产批发市场的冰鲜鱿鱼,无菌水洗涤,室温放置至出现轻度腐败味时进行取样分析。
1.2 鱿鱼肌肉组织中细菌总DNA的提取
在无菌操作条件下将鱿鱼肌肉组织剪碎,称取6份10 g样品,采用冻融法、摇床法和匀浆法进行前处理,各方法均重复2次[7]。
1.2.1 冻融法
取10 g样品置于无菌离心管中,液氮处理2min后放于65℃水浴中保温至完全融化,反复冻融3次。将样品转移至无菌锥形瓶中,加入90mL无菌TE缓冲液,充分摇匀,静置5min,取上清液50mL,10000 r·min-1离心10min,待用。
1.2.2 匀浆法
取10 g样品置于无菌玻璃匀浆器中充分匀浆后转移至无菌锥形瓶中,加入90mL无菌TE缓冲液,充分摇匀,静置 5min,取上清液 50mL,10000 r·min-1离心 10min,待用。
1.2.3 摇床法
取10 g样品置于无菌锥形瓶中,加入10 g无菌玻璃珠和90mL无菌TE缓冲液,200 r·min-1、4℃振荡处理30min,静置5min,取上清液50mL,10000 r·min-1离心 10min,待用。
1.2.4 细菌总DNA的提取
3种处理后所得沉淀用10mL TE悬浮,加入0.5mL 10%SDS,50μL蛋白酶 K,混匀,37℃孵育 1 h。加入 1.5mL 5 mol·L-1NaCl,1.5mL CTAB/NaCl溶液,混匀,65℃孵育20min。用等体积酚∶氯仿∶异戊醇(25∶24∶1)抽提,10000 r·min-1离心10min取上清,重复上述操作直至两相交界面无白色物质出现为止。取上清,加入等体积氯仿∶异戊醇(24∶1),10000 r·min-1离心 10min。取上清,加入等体积异丙醇,混匀,-20℃静置1 h,10000 r·min-1离心15min得到DNA沉淀。加入 700μL 75% 乙醇,70μL 3mmol NaAc漂洗 DNA沉淀后,溶解于1mL TE,-20℃保存。
1.3 16 S rDNA的PCR-DGGE图谱的构建
1.3.1 1500 bp 16 S rDNA片段的PCR扩增
DNA用无菌水分别连续作3倍梯度稀释,进行PCR扩增。
PCR引物:8 F(5’-AGAGTTTGATCCTGGCT CAG-3’)和 1510 R(5’-GGCTACCTTGTTACGA-3’)。
PCR反应体系:Primer 1μL,10×Ex Taq Buffer 5.0μL,dNTP Mixture 5.0μL, DNA 1.0μL,TaKaRa Ex Taq 0.3μL 和 ddH2O 37.7μL。
PCR扩增程序:94℃预变性2min;94℃变性1min,55℃退火1min,72℃延伸1.5min,30个循环;最后72℃延伸2min。
1.3.2 16 S rDNA V3区片段的PCR扩增
PCR 引 物:GC357F(5’-CGCCCGCCGCG CGCGGCGGGCGGGGCGGGGCACGGGCCACCTACG GGAGGCAGCAG-3’) 和 517 R(5’-ATTACCGC GGCTGCTGG-3’)。
PCR反应体系:Primer 1μL,2×GC Buffer II 25.0μL,dNTP Mixture 8.0μL, DNA 1.0μL,TaKaRa La Taq 0.5μL 和 ddH2O 14.5μL。
PCR扩增程序:94℃预变性5min;94℃变性1min,55℃退火 1min,72℃延伸 1.5min,305个循环;最后72℃延伸5min。
1.3.3 DGGE图谱的建立与分析
DGGE电泳采用 Muyzer等[8]的方法,电泳条件为恒温60℃,电压85 V,聚丙烯酰胺凝胶的浓度为8%,电泳时间14 h。电泳结束后,EB染液浸染15min,再用ddH2O漂洗5min,UVP凝胶成像系统照相。
2 结果与分析
2.1 鱿鱼肌肉组织细菌总DNA的提取
鱿鱼样品采用3种不同前处理后所提取的细菌总DNA电泳图如图1所示,结果表明经3种前处理方法后均能成功提取鱿鱼肌肉组织细菌的总DNA。其中,匀浆处理较其他2种处理所得DNA电泳条带要亮很多,说明匀浆处理提取DNA的产率最高。摇床处理次之,冻融处理提取DNA的产率最低。
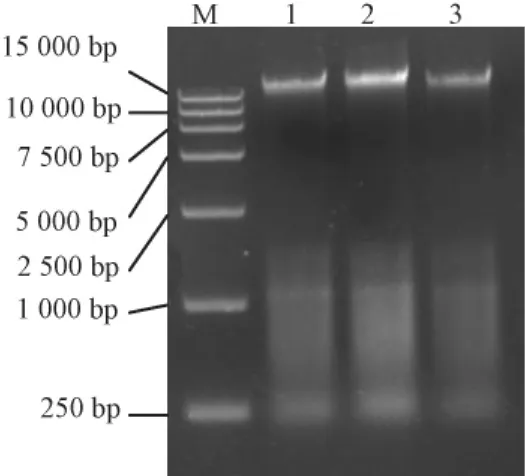
图1 鱿鱼肌肉组织细菌总DNA提取电泳图
2.2 1500 bp 16 S rDNA片段的PCR扩增
以提取的鱿鱼肌肉组织细菌总DNA及其相应的稀释梯度为模板,以8 F和1510 R为引物进行第1套PCR扩增,结果如图2所示,所得产物是大小约1500 bp的16 S rDNA片段,且随着稀释梯度的加大,条带亮度先增强后减弱。
2.3 细菌16 S rDNA V3区片段的PCR扩增

图2 1500 bp 16 S rDNA片段的PCR扩增结果
以第1套PCR扩增产物为模板,以GC357 F和517 R为引物进行第2套PCR扩增,结果如图3所示,所得产物是大小约160 bp的片段。
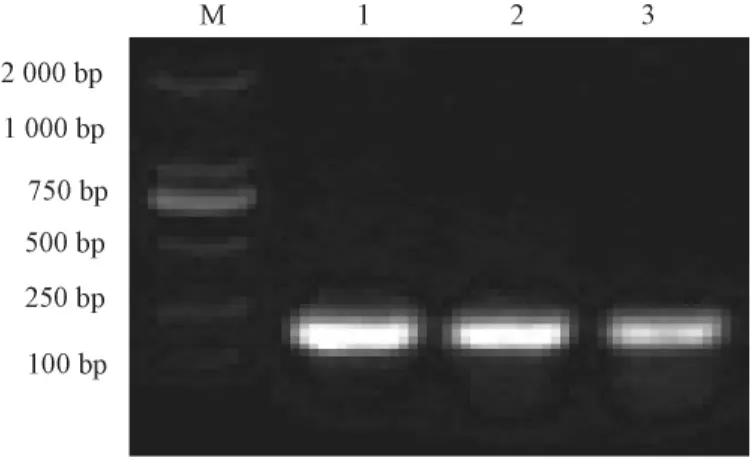
图3 细菌16 S rDNA V3区片段PCR扩增结果
2.4 DGGE图谱的建立
用第2轮PCR扩增产物在变性梯度30%~60%进行DGGE电泳,结果如图4。由图4可知,DGGE共分离产生5条较明显的条带,3种方法所得图谱均相似,且都存在明显的主次带之分。
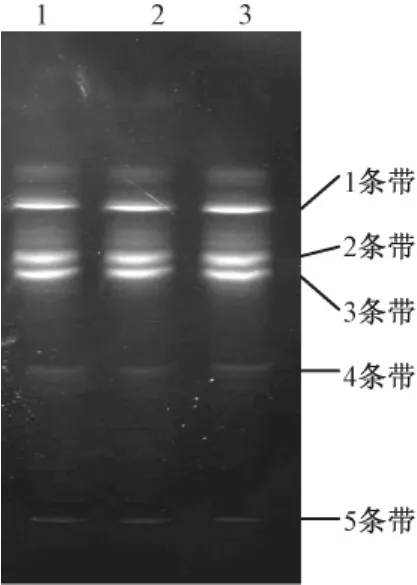
图4 细菌16 S rDNA V3区DGGE图谱
3 小结与讨论
PCR-DGGE方法避开了传统微生物培养分离的环节,直接提取微生物总DNA,获得种群的DNA信息,较为真实地揭示样品中微生物的实际组成,理论上只要实验条件适当,DGGE技术便可区分1个碱基差别的DNA片段[9]。但是,如何从样品中直接提取微生物总DNA,这是DGGE分析首先要解决的问题,可以说高质量和高产量的基因组DNA的提取是PCR-DGGE分析的前提和基础。一般来说,直接从鱿鱼组织中提取细菌DNA需要对样品进行前处理。前处理方法的不同可能会影响DNA提取的效果及后续的PCR扩增和DGGE分析。冻融法、匀浆法和摇床法3种前处理对鱿鱼组织中细菌DNA的提取效果及对DGGE分析影响实验的结果表明,匀浆处理提取DNA的产率最高,摇床处理次之,冻融处理最低。PCR-DGGE分析表明3种前处理形成的条带分布相似,且都存在明显的主次带之分,说明这3种前处理对后续PCR-DGGE分析的影响无明显差异。但是,就具体试验来说,摇床法和冻融法分别采用无菌锥形瓶和无菌离心管,所以能同时处理多个样本,而匀浆法在处理每一个样品时需对匀浆器进行灭菌,故该方法不适于处理大量样本。
鱿鱼是一类蛋白质含量丰富的水产品,100 g鱿鱼鲜品中的蛋白质含量就高达16~18 g[10],杂蛋白一方面会影响DNA的溶解,另一方面也会干扰后续的PCR反应,因此尽可能的去除杂蛋白也是建立适于鱿鱼组织的DGGE分析方法的关键问题。一般来说,为获取高纯度的DNA样本都需要对DNA样本进行纯化,但是常用的DNA纯化方法容易导致DNA样本的严重损失,最终导致微生物多样性的下降。本试验通过简单的提高抽提次数并结合DNA稀释法,降低杂质在DNA模板中的浓度,获得了高质量的DNA样本,直接用于PCR扩增,有效避开了DNA纯化步骤,在很大程度上避免了DNA样本的流失,更为真实地反映待测样品中微生物的菌群特征。
[1]吴少杰,张俊杰,姚兴存,等.我国鱿鱼的综合加工利用现状与展望 [J].食品研究与开发,2011,32(1):154-156.
[2]辜运富,张小平,涂仕华.变性梯度凝胶电泳 (DGGE)技术在土壤微生物多样性研究中的应用 [J].土壤,2008,40(3):344-350.
[3]Fischer S G,Lerman L S.DNA fragments differing by single base-pair substitutions are separated in denaturing gradient gels:correspondence with melting theory [J].Proceedings of the National Academy of Sciences of the United States of America,1983,80(6):1579-1583.
[4]Myers R M,Fischer S G,Lerman L S,et al.Nearly all single base substitutions in DNA fragments joined to a GC-clamp can be detected by denaturing gradient gel electrophoresis[J].Nucleic Acids Research,1985,13(9):3131-3145.
[5]Hovda M B, Sivertsvik M, Lunestad B T, et al.Characterisation of the dominant bacterial population in modified atmosphere packaged farmed halibut (Hippoglossus hippoglossus)basedon 16SrDNA-DGGE [J].Food Microbiology,2007,24(4):362-371.
[6]Yoshikawa S,Yasokawa D,Nagashima K,et al.Microbiota during fermentation of chum salmon(Oncorhynchus keta)sauce mash inoculated with halotolerant microbial starters:analyses using the plate count method and PCR-denaturing gradient gel electrophoresis(DGGE) [J].Food Microbiology,2010,27(4):509-514.
[7]江芸,高峰,徐幸莲,等.冷却猪肉不同前处理对细菌DNA提取及PCR-DGGE的影响 [J].食品科学,2010,31(6):258-262.
[8]Muyzer G,de Waal E C,Uitterlinden A G.Profiling of complex microbialpopulations by denaturing gradientgel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA [J].Applied and Environmental Microbiology,1993,59(3):695-700.
[9]Amann R I, Ludwig W, SchleiferK H. Phylogenetic identification and in situ detection of individual microbial cells without cultivation [J].Microbiological Reviews,1995,59(1):143-169.
[10]陈意.鱿鱼的营养及食用价值 [J].食品与药品,2006,8(6):75.

