致病疟原虫分子功能注释二级数据库的构建
许三岗 庄永龙 郝志勇 单广良 杨啸林 王 恒
(中国医学科学院基础医学研究所 北 京协和医学院基础学院,北京 1 00005)
XU San-Gang ZHUANG Yong-Long HAO Zhi-Yong SHAN Guang-Liang YANG Xiao-Lin* WANG Heng*
(Institute of Basic Medical Sciences,Chinese Academy of Medical Sciences,School of Basic Medicine,
Peking Union Medical College,Beijing 100005,China)
引言
疟疾是一种由按蚊叮咬传播疟原虫所致的寄生虫病,多发生于热带与亚热带地区,涵盖非洲、东南亚、拉丁美洲、中东等地区,全世界每年有近百万人死于疟疾。疟原虫寄生于人与动物的红细胞,通过在红细胞中发育、增殖而完成其生活周期,同时引发临床症状,造成机体损伤。目前已知的疟原虫种类繁多,达130多种[1-2],但均有严格的宿主特异性。以人类为宿主的疟原虫共5种,即恶性疟原虫、间日疟原虫、三日疟原虫、卵形疟原虫和诺氏疟原虫。
近年来,随着分子生物学、遗传学、生物信息学等的发展,为疟原虫致病的分子机制和防治研究提供了大量的数据。因此,如何快速高效地对实验数据进行功能注释成为深入研究疟原虫生物学特性及其致病分子机制过程中亟待解决的问题。目前疟原虫研究领域中整合数据资源较多的数据库是PlasmoDB(http://PlasmoDB.org),该数据库是关于疟疾寄生虫基因组计划的官方数据库,整合了实验数据和计算数据,包含已完成测序和注释的恶性疟原虫基因组,以及完成了测序和部分注释的其他7种疟原虫基因组[3]。
PlasmoDB作为疟原虫研究领域的专业数据库,其优点是数据资源较为全面。但是,在高通量分子功能注释方面,PlasmoDB数据库有一些内容未考虑周全,如:(1)对高通量的疟原虫芯片实验数据进行功能注释时,其注释分子的类型中无ProbeID(探针ID)入口,无法对探针数据进行完美注释;(2)该数据库中的ID标识与NCBI等数据库中的标识不一致,因此,若要对一个分子进行全面的注释,需要到不同的数据库中查询;(3)在对实验数据进行Gene Ontology、蛋白质相互作用及Pathway等功能注释时,其注释结果仅以列表的形式进行展示,无图形化结果显示,注释结果不直观,对注释信息尚未进行任何统计学分析筛选,既不利于研究人员的理解,也不利于从海量的注释结果中快速挖掘具有重要意义的信息。为解决以上问题,提高数据注释的效率,本研究开发了一个可对疟原虫高通量生物实验数据提供快速有效功能注释的二次数据库(Plasmodium Molecular Functional Annotation Secondary Database,简称 PlasmoFADB)。
1 材料和方法
1.1 材料
1.1.1 聚合物种信息
[1]Liu Chang,Zhou Jiliu,Lang Fangnian,et al.Generalized discriminant orthogonal non-negative matrix factorization and its applications[J].Systems Engineering and Electronics,2011,33(10):2327-2330.
[2]Zhu Yanli,Chen Jun,Qu Peixin.A novel discriminant nonnegative matrix factorization and its application to facial expression recognition [J]. Advanced MaterialsResearch,2011,143-144:129-133.
PlasmoFADB聚合了疟原虫属的多个物种的信息,包括伯氏疟原虫(plasmodium berghei str.ANKA,pbe)、夏氏疟原虫(plasmodium chabaudi chabaudi,pcb)、3D7 恶 性 疟 原 虫(plasmodium falciparum 3D7,pfa)、诺 氏 疟 原 虫(plasmodium knowlesi strain H,pkn)、间日疟原虫(plasmodium vivax SaI-1,pvx)及约氏疟原虫(plasmodium yoelii str.17XNL,pyo)。
1.1.2 聚合数据来源
PlasmoFADB聚合了多种生物学通用数据库,包括GenBank、KEGG、Gene Ontology(简称GO)、MINT、Uniprot等数据库以及Affymetrix芯片中关于疟原虫的探针注释信息(见表1)。

表1 数据源及下载地址Tab.1 Data sources and download websites
1.2 方法
图1为 PlasmoFADB平台的整体构架,分别建立了数据层(数据库表定义及数据存储)、算法插件层(结果展现功能及R等相关脚本算法集成)、结果展示层(结果展示形式及展示数据的界面排布插件配置)。通过三层架构将工作划分为数据整理、算法插件实现、结果展示配置等3个部分,在确定数据来源基础上,将后期开发工作主要集中在算法插件开发及结果展示方面,进而使数据整理与功能实现相对独立,提高了数据库的可扩展性及灵活性。
1.2.1 数据整理
1.2.1.1 数据表结构设计
PlasmoFADB以MySQL作为后台数据库,主要包括以下数据表:GeneInfo(基因的基本信息)、Gene2GO(基因与GO的映射关系)、Gene2Pathway(基因与Pathway的映射关系)、Probe(探针信息)、Probe2Gene(探针与基因的映射关系)、Probe2GO(探针与GO的映射关系)、GOTerm(GO术语)、Term2Term(GO术语关系)、Pathway(通路信息)、Genecoords(信号分子在通路中的定位信息)及Interaction(蛋白质相互作用信息)。

图1 PlasmoFADB平台整体构架Fig.1 Overall structure of the PlasmoFADB

图2 PlasmoFADB拓扑结构图Fig.2 Topological structure of PlasmoFADB
图2为PlasmoFADB的拓扑结构,各个表之间的关系以GeneID(基因ID)作为衔接纽带,与KEGG、GO、MINT等数据库通过关系表Gene2Pathway、Gene2GO、Interaction进行关系构建,使数据表之间相对独立。其他注释分子入口,如ProbeID(探针ID)及Gene Symbol(基因标识)先查询其对应GeneID再与其他表之间建立关系。
1.2.1.2 数据解析与存储
数据源的数据类型可以分为两类:一类为TAB制表符分割的文本格式文件,另一类为XML或其他格式的文件。通过对原始数据的解析、整理,最终实现关系型数据库的存储。主要流程包括3个步骤。
步骤1:根据原始数据文件格式,编写XML文件解析器和TAB文件解析器,分别用于解析XML格式的文件和TAB制表符分割的文本格式文件;
步骤2:利用数据库应用程序编程接口,将上述数据存储到MySQL数据库中,用于构建PlasmoFADB;
步骤3:随机抽取一些已导入数据库中的数据,然后进入其来源数据库中进行人工搜索核对,完成数据核查。
1.2.2 算法插件层实现
PlasmoFADB基于J2EE技术和MVC三层体系架构,采用JDBC/Hibernate实现数据库的访问,利用JAVA等语言开发分子功能注释插件,对分子注释主界面进行定义,并利用XML确定各个数据表之间的关系,以及数据查询路径等[10]。
通过调用R脚本实现在Gene Ontology、Pathway功能注释结果中集成富集分析,核心函数利用R中q值包中提供的q值方法,即调用Fisher's精确检验程序包,p-value越小说明差异越显著[11]。
利用Graphviz(Graph Visualization Software)工具对蛋白质相互作用结果(protein-protein interaction,PPI)进行图形化描述,确定各个蛋白质分子的图形布局情况,将图形中的节点在画布上比较均匀的分布,缩短节点之间的边长,将其图形化描述内容转换为SVG格式,再利用Adobe SVG Viewer软件对PPI结果进行图形化显示[12]。
1.2.3 结果展示配置
PlasmoFADB门户发布采用Portal技术,页面使用HTML和JavaScript语言开发,通过运用Ajax及Jquery等技术提高交互性和用户友好性,以XML插件配置实现PlasmoFADB分子注释结果的展示界面排布。
2 结果
PlasmoFADB整合了疟原虫分子机制研究中的常用的多种生物学通用数据库,提供多种直观的图形方式来反映疟原虫的生物学信息,支持GO树状分类图、Pathway基因定位图、蛋白质相互作用图等多种图形显示。目前,数据库中数据条目达7万余条,其构成和分布见表2。此外,PlasmoFADB还可以通过调用富集效应分析的R算法,对Gene Ontology及Pathway注释结果进行统计分析,用户可从这些注释结果中挖掘出更有意义的信息。
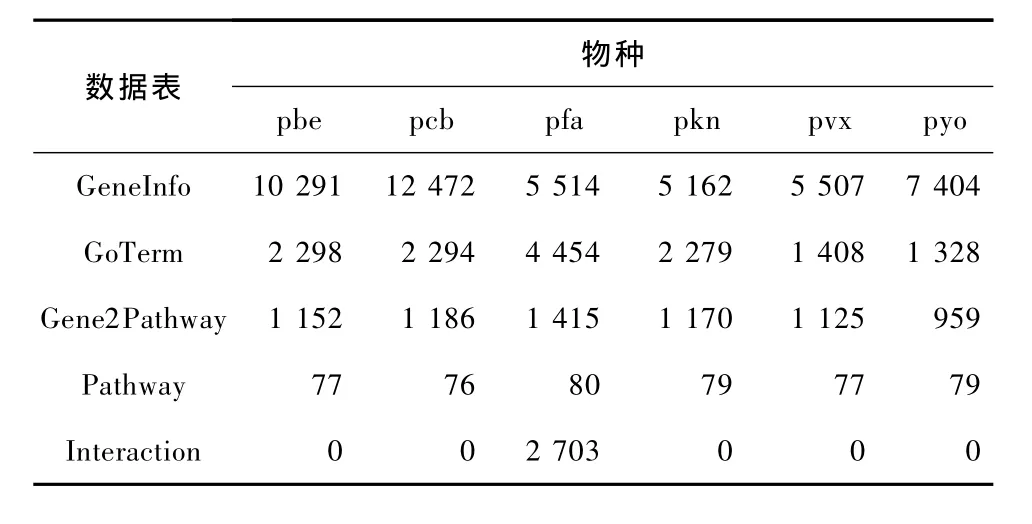
表2 PlasmoFADB数据表中数据条目数Tab.2 Data item number of PlasmoFADB data tables
图3为PlasmoFADB分子功能注释界面,通过输入注释文件、设定分子类型、选定注释文件类型等步骤后,即可查看注释结果。
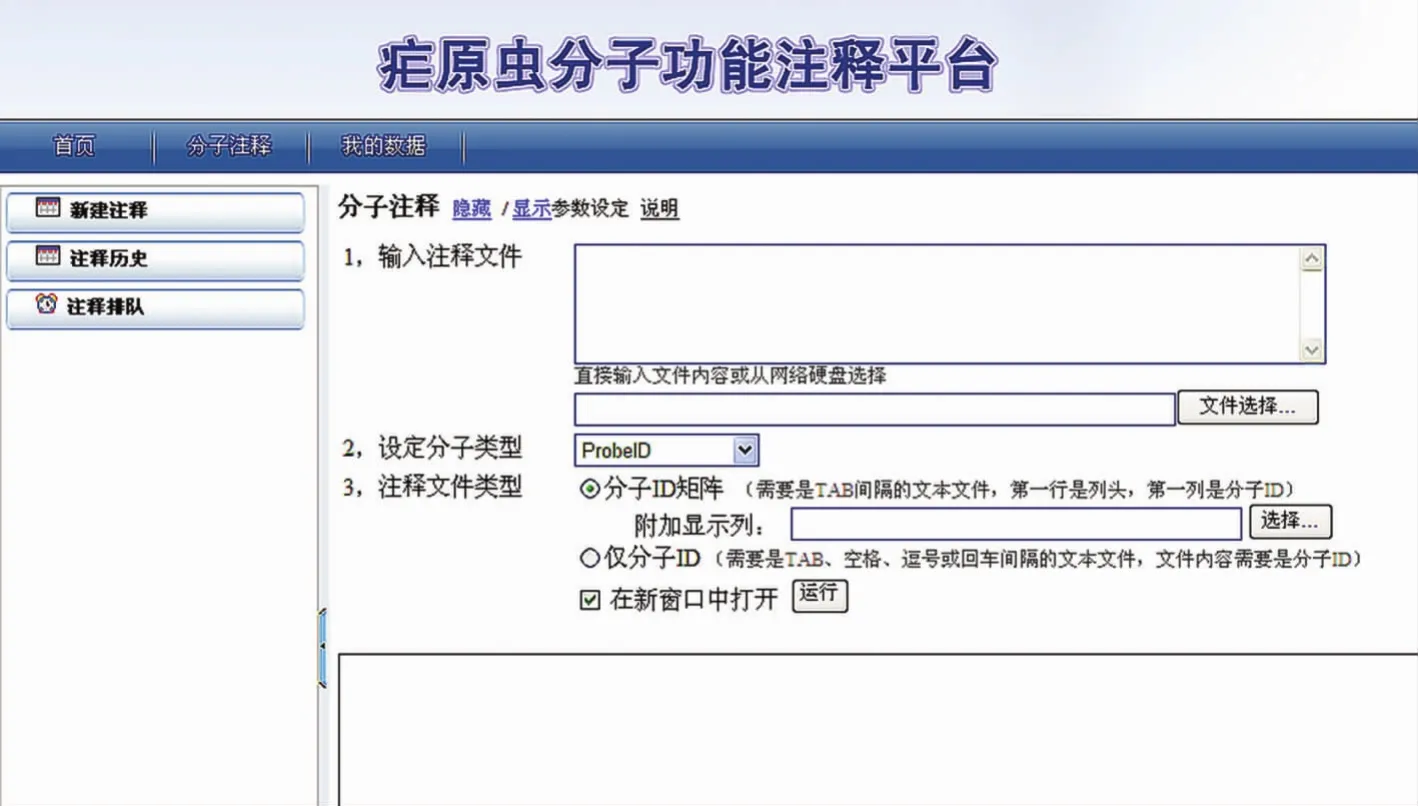
图3 PlasmoFADB分子功能注释界面Fig.3 Interface of PlasmoFADB molecular functional annotation
另外,本平台支持多用户多线程,针对每个不同的用户可点击“注释历史”以查看自己的已经注释过的信息,并可点击“注释排队”以查看整个平台有多少线程在同时运行。
2.1 GO功能注释
GO功能注释实现Gene2GO与GO2Gene的双向数据注释。利用该功能可以查看基因在GO中的映射信息,点击基因在GO中的映射数目,即可查看相应的GO信息,点击映射上的GO的名字,以树状图的形式展示GO的分类信息,底层节点以加粗形式显示(见图4)。
GO2Gene富集分析展示不同层次上GO节点注释基因情况并提供富集分析值,根据富集分析值可选择富集效果较好的基因作为下一步的研究对象,默认展示三层,点击“show level”可显示不同的层级,并可以设定相应的p值和q值对结果进行筛选,点击关注节点的“Hits”查看该GO节点下命中的基因信息(见图5)。
2.2 Pathway通路注释
Pathway通路注释实现了Gene2Pathway与Pathway2Gene的双向注释。Gene2Pathway主要展示和注释基因有关通路的基本信息列表。Pathway2Gene主要提供通路中关联基因的富集分析结果,可对相应的参数进行设定,如PathwayDB(通路来源数据库)、Hits Threshold(命中数目)、p值、q值设定,对结果进行筛选,点击列表中表头可对该列结果数据进行排序浏览,点击列表结果中Name列中的名称可浏览此通路图(见图6)。
图7为Pathway2Gene动态显示图。左侧显示该通路图包含的所有基因,左侧阴影部分即为注释基因在该通路中映射上的基因。右侧阴影部分为注释基因在通路中所处的位置,点击阴影部分可链接到NCBI网站,用户可以浏览关于该基因的详细信息。

图4 Gene2GO树状图Fig.4 Tree diagram of Gene2GO

图5 GO2Gene富集分析Fig.5 The result of GO2Gene enrichment analysis
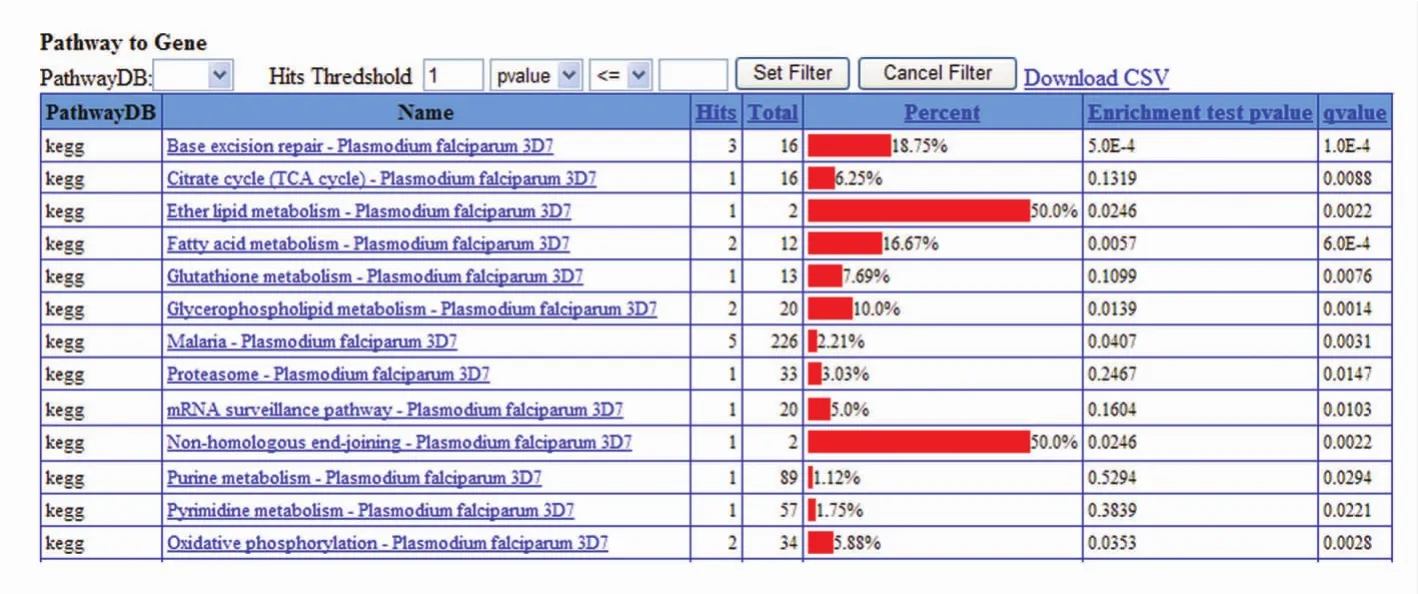
图6 Pathway2Gene富集分析Fig.6 The result of Pathway2Gene enrichment analysis
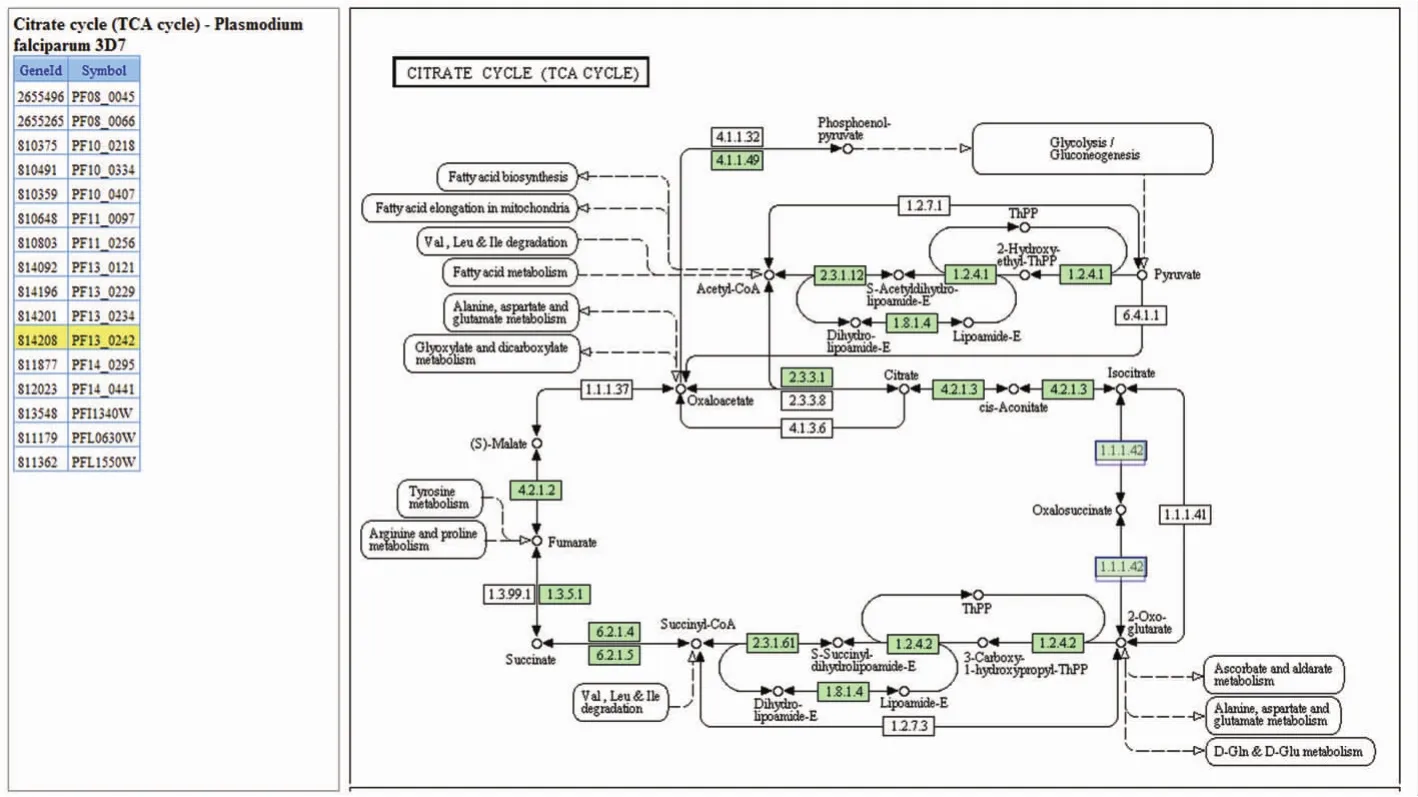
图7 Pathway2Gene图形化动态显示Fig.7 The result of Pathway2Gene graphical dynamic display
2.3 蛋白质相互作用网络
蛋白质相互作用网络以图形化的形式显示蛋白质相互作信息(见图8),用户可在图形中输入某个基因的ID,设定相应参数,如DB(来源数据库)、Hits Threshold(命中数目)、Direct(排列方向)、layout Engine(布局引擎)等,以查看该基因所对应的蛋白质分子与其他蛋白质分子的相互作用。对于注释到的蛋白质相互作用分子,点击后可动态链接到NCBI相关页面供用户查询浏览更加详尽的信息。
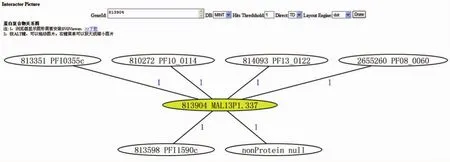
图8 蛋白相互作用网络图Fig.8 The result of protein interaction network diagram
3 讨论和结论
本研究建立了一个针对高通量疟原虫分子功能注释的二级数据库,数据库保存了疟原虫部分物种的基因信息、探针信息、Gene Ontology、Pathway及PPI等信息。除了为用户提供较好的服务外,其本身还具有较好的扩展性、灵活性、独立性,可以根据需要随时对数据库结构进行更改。
PlasmoFADB中的原始数据均来源于互联网在线数据库,为确保PlasmoFADB中数据与在线数据库数据在一定时期内的数据一致性,在表格设计时尽量与原数据库中的数据表格结构一致,各个数据表之间不建立强引用关系,保证了各个数据表之间的数据结构的独立性及整合数据的完整性。
PlasmoFADB在高通量分子注释及注释结果可视化方面对PlasmoDB进行了补充。提供多种分子注释类型入口,各个数据库中ID标识与NCBI数据库建立关联。若要对一个分子进行全面快速的注释,只需要输入唯一的ID标识即可。
PlasmoFADB利用分子注释插件实现Gene2GO树状图和Pathway2Gene图形化动态显示,可充分展示Gene Ontology的层次结构及Gene在Pathway中的位置信息,并调用R脚本对GO、Pathway功能注释结果进行富集分析,用户可利用该富集分析结果选取富集效果较好的分子作为下一步的研究目标。
PlasmoFADB利用SVG格式实现对PPI的可视化,用户可以直接利用安装了Adobe SVG Viewer的浏览器呈现PPI的信息,对PPI图形化结果放大或缩小而不改变清晰度,以便读取PPI可视化注释结果。
在疟原虫的致病分子机制研究过程中,虽然虫源性的miRNAs尚未被发现,但研究人员正试图解读该类分子疟原虫中的表达情况,并阐述其在疟原虫致病过程中的调控作用机理。随着与疟原虫相关的诸如miRNAs、Promoter、Transfactor等分子的高通量注释信息的出现和不断增加,对整合数据库的及时更新成为一个重要的任务,所构建的数据可具备了这方面的扩展能力。
本研究开发了一个可对疟原虫高通量生物实验数据提供快速有效功能注释的二次数据库,为深入挖掘和理解疟原虫致病分子机制研究中高通量数据生物学意义提供了有力的分析工具,可有效促进疟原虫基因功能、表达调控、信号通路及疟疾疫苗抗原筛选等方面的研究。
[1]Snow RW,Uerra CA,Noor AM,et al.The global distribution of clinical episodes of Plasmodium falciparum malaria[J].Nature,2005,434(7030):214-217.
[2]彭桂英,王恒.恶性疟候选疫苗研究进展[J].国际医学寄生虫病学杂志,2007,34(1):37-42.
[3]Aurrecoechea C,Brestelli J,Brunk BP,et al.PlasmoDB:a functional genomic database for malaria parasites[J].Nucleic Acids Res,2009,37:D539-D543.
[4]Benson DA,Karsch-Mizrachi I,Lipman DJ,et al.GenBank[J].Nucleic Acids Res,2011,39:D32-D37.
[5]Kanehisa M,Goto S,Sato Y,et al.KEGG for integration and interpretation of large-scale molecular datasets[J].Nucleic Acids Res,2012,40:D109-D114.
[6]The Gene Ontology Consortium.Gene ontology:tool for the unification of biology[J].Nat Genet,2000,25(1):25-29.
[7]Licata L,Briganti L,Peluso D,et al.MINT,the molecular interaction database:2012 update[J].Nucleic Acids Res,2012,40:D857-D861.
[8]The UniProt Consortium.Reorganizing the protein space at the Universal Protein Resource(UniProt)[J].Nucleic Acids Res,2012,40:D71-D75.
[9]Affymetrix Community.Plasmodium_Anopheles Annotations,CSV format,Release 32[EB/OL].http://www.affymetrix.com/estore/support/technical/annotationfilesmain.affx,2011-06-22/2012-11-02.
[10]张智,张正国.基于Web Service的生物学实体映射数据库[J].中国生物医学工程学报,2010,29(5):704-709.
[11]曹文君,李运明,陈长生.基因表达谱富集分析方法研究进展[J].生物技术通讯,2008,19(6):931-934.
[12]张智,张正国.基于元数据的异构蛋白质-蛋白质相互作用数据库整合[J].中国生物医学工程学报,2010,29(2):201-206.

