灵长类动物中TRIMCyp融合基因模式及对逆转录病毒复制的限制作用
曹 光, 刘丰亮, 张高红, 郑永唐,*
(1.中国科学院和云南省动物模型与人类疾病机理重点实验室, 中国科学院昆明动物研究所, 云南 昆明 650223; 2. 中国科学院研究生院, 北京 100049)
灵长类动物中TRIMCyp融合基因模式及对逆转录病毒复制的限制作用
曹 光1,2, 刘丰亮1, 张高红1, 郑永唐1,*
(1.中国科学院和云南省动物模型与人类疾病机理重点实验室,中国科学院昆明动物研究所,云南 昆明650223; 2.中国科学院研究生院,北京100049)
TRIM5-CypA融合基因(TRIMCyp)是一种独特的TRIM5基因形式。迄今已发现新大陆猴中包括鹰猴在内的夜猴属所有代表种,以及在北平顶猴、巽他平顶猴、食蟹猴、印度恒河猴和熊猴等旧大陆猴中均存在这种基因融合现象, 但在新大陆猴与旧大陆猴中的TRIMCyp融合基因的基因融合模式和表达剪接方式不同。新大陆猴TRIMCyp融合基因是由CypA假基因的cDNA序列通过LINE-1逆转座子介导的逆转座方式插入至TRIM5α基因的第7和第8外显子之间的内含子中形成, 而旧大陆猴TRIMCyp融合基因则是由CypA假基因的cDNA序列以相似的逆转座方式插入至TRIM5基因的3'非翻译区(untranslated regions, UTR)形成。TRIMCyp融合基因在不同灵长类动物中的存在比例、基因型、TRIMCyp融合蛋白的表达以及对逆转录病毒的限制活性均有所差异。鹰猴和平顶猴的TRIMCyp融合基因研究较多, 鹰猴TRIMCyp融合蛋白可能以与TRIM5α相似机制限制HIV-1的感染, 而平顶猴TRIMCyp融合蛋白则丧失了限制HIV-1的作用。这两个功能截然不同的融合基因为TRIM5α作用机制研究提供了难得的实验材料, 也为建立HIV-1感染的新型灵长类动物艾滋病模型奠定了科学依据。该文综述了TRIMCyp融合基因在灵长类动物中的分布、存在形式及其限制逆转录病毒复制的作用机制等方面的研究情况。
TRIM5; TRIMCyp; 限制因子; HIV; 艾滋病; 动物模型
人免疫缺陷病毒(human immunodeficiency virus, HIV)是人获得性免疫缺陷综合症(acquired immunodeficiency syndrome, AIDS), 俗称艾滋病的主要病原体。HIV主要分为2大类:HIV-1和HIV-2。HIV-1是最早被人们发现的HIV病毒, 它相比HIV-2具有更大的毒性和感染力(Gilbert et al, 2003),是导致艾滋病在全球蔓延的主要感染病毒。与HIV-1相比, HIV-2的感染力较弱, 很少会有人在一次暴露后感染HIV-2, 并且由于HIV-2的低传播能力, 使它的传播范围受到了很大局限, 目前的研究发现HIV-2 主要在西非部分地区流行(Reeves & Doms, 2002)。由于HIV-1的强致病性和全球流行趋势日益严峻, 多年以来, 科学家们一直致力于HIV-1的相关基础研究工作, 希望通过不断加深对HIV-1的了解, 来为艾滋病的预防和治疗寻找有效的方法和途径。目前研究认为, HIV-1是由猴免疫缺陷病毒(simian immunodefiency virus, SIV)跨种传播进化而来的。尽管HIV-1可以感染人和黑猩猩, 但在大多数旧大陆猴中都不能正常复制。这种跨种传播屏障存在于大多数的灵长类动物中, 也正因为如此, 使得种类繁多的SIV只能在其相应的宿主范围内才能正常复制(Sastri & Campbell, 2011)。这种针对逆转录病毒的免疫反应是由宿主细胞组成型表达的限制因子抑制了逆转录病毒在细胞内的复制所导致 (Bieniasz, 2004; Goff, 2004)。
宿主细胞表达限制逆转录病毒复制的限制因子的最早证据来源于20世纪60年代末。当时, 研究人员在小鼠中发现, 弗里德氏病毒易感基因1(friend virus susceptibility gene 1, Fv1) 决定了小鼠对2种不同鼠白血病病毒 (murine leukemia virus, MLV)易感性的差异(Lilly, 1967; Steeves & Lilly, 1977)。随着对细胞内抗逆转录病毒免疫的不断了解与深入研究, 发现其他蛋白也参与了这种抗病毒反应(Sastri & Campbell, 2011)。2004年, 通过遗传筛选方法, 在恒河猴(Macaca mulatta)中发现了能够抑制HIV-1感染的TRIM5α蛋白(rhTRIM5α) (Stremlau et al, 2004)。
TRIM5α以一种物种依赖的方式限制包括HIV-1在内的逆转录病毒的感染 (Tang et al, 2009)。TRIM5α及其他TRIM5分子(TRIM5β、γ、δ、ε)同属于三重模体蛋白(tripartite motif protein, TRIM)家族。TRIM5α除具有TRIM家族蛋白的共性结构, 即包括RING、B-box(es)和Coiled-coil 三个结构域外,在C末端还有1个B30.2/SPRY (SPla and RYanodine)结构域, 该结构域可以与病毒衣壳蛋白结合并介导逆转录病毒的特异性识别(Stremlau et al, 2004; Diaz-Griffero et al, 2009)。TRIM5同源物不仅广泛存在于包括人类、猿、旧大陆猴(Old World monkey)及新大陆猴(New World monkey)等的多种灵长类谱系中(Sawyer et al, 2005; Song et al, 2005), 而且在其他哺乳动物, 如牛(Si et al, 2006; Ylinen et al, 2006)和兔(Schaller et al, 2007)等中也存在。由于TRIM5α分子的存在和作用, 除平顶猴外的其他旧大陆猴都不能感染HIV-1。
在新大陆猴中, 只有鹰猴(owl monkey,Aotus trivirgatus)表现出对HIV-1病毒进入细胞后的复制抑制效应(Hofmann et al, 1999)。Sayah et al(2004)和Nisole et al (2004)通过克隆鹰猴的TRIM5α基因, 发现在鹰猴TRIM5α基因的第7和第8外显子之间的内含子中插入了1个cyclophilin A假基因, 形成了1个嵌合基因TRIMCyp(owl monkeyTRIM5-CypA, omTRIMCyp)。omTRIMCyp抑制 HIV-1病毒进入鹰猴细胞内后的复制。随后, 本实验室及国际上多个实验室分别在北平顶猴(M. leonina,Northern pig-tailed macaque)、熊猴(M. assamensis,Assam macaque)、巽他平顶猴(M. nemestrina,Sunda pig-tailed macaque)、食蟹猴(M. fascicularis,Crab-eating macaque)及印度恒河猴(M. mulatta,Indian rhesus macaque)等旧大陆猴中也发现存在这种TRIMCyp融合基因 (Brennan et al, 2008; Cao et al, 2011; Kuang et al, 2009; Liao et al, 2007; Newman et al, 2008; Virgen et al, 2008; Wilson et al, 2008)。不同灵长类动物来源的TRIMCyp融合蛋白对不同逆转录病毒表现出不同的限制活性。由于TRIMCyp融合基因在天然宿主因子限制HIV-1复制机制研究以及建立新型艾滋病灵长类动物模型方面可能发挥着重要作用,将会越来越多地受到人们的关注。鉴于此,本文综述和介绍近几年来TRIMCyp融合基因在灵长类动物中的分布、存在形式及其限制逆转录病毒复制的作用机制等方面的研究进展,以期理清思路,提出今后的科学研究问题。
1 TRIMCyp融合基因在灵长类动物中的分布情况
1.1 新大陆猴与旧大陆猴的区分
灵长目包括原猴亚目和猿猴亚目, 按区域分布及鼻孔构造, 猿猴亚目又分为阔鼻猴小目, 又称新大陆猴; 狭鼻猴小目, 又称旧大陆猴。新大陆猴原产于中南美洲, 包括狨科(Callitrichidae)、卷尾猴科(Cebidae)、青猴科(Aotidae)、僧面猴科(Pitheciidae)和蜘蛛猴科(Atelidae)共5个科。旧大陆猴原产于非洲和亚洲, 栖息于热带雨林、草原、灌木林及山地之中。化石记录旧大陆猴也曾在欧洲存在。旧大陆猴包括非人灵长类中的大部分相似种属, 如狒狒属(Papio)和猕猴属(Macaca)。
1.2 灵长类动物中TRIMCyp融合基因的分布情况
继首次报道在鹰猴中发现存在TRIMCyp融合基因后(Nisole et al, 2004; Sayah et al, 2004),研究人员对新大陆猴的全部代表种属进行了TRIMCyp融合基因的检测, 结果发现, 包括鹰猴在内的夜猴属(Aotus)的4个代表种均存在TRIMCyp融合基因, 而新大陆猴其它15个属中均没有发现存在TRIMCyp基因融合现象(Ribeiro et al, 2005)。提示类似鹰猴的TRIMCyp融合基因可能只在新大陆猴的夜猴属中存在。
在旧大陆猴中, 平顶猴(Pig-tailed macaque)是唯一报道可能感染HIV-1的灵长类动物。现行灵长类动物分类学, 将原来的平顶猴群(M. nemestrinagroup)分为猕猴属的3个种:巽他平顶猴(M. nemestrina,Sunda pig-tailed macaque), 北平顶猴(M. leonina,Northern pig-tailed macaque)和明打威猴(M. pagensis,Mentawai macaques)(Gippoliti, 2001; Groves, 2001; Li & Zhang, 2005; Rosenblum et al, 1997)。北平顶猴分布于我国云南南部和西藏东南部以及缅甸、泰国和印度北部地区。本实验室在国际上首次发现北平顶猴存在着与鹰猴相似的TRIMCyp融合基因现象, 并将该融合基因命名为npmTRIMCyp(Northern pig-tailed macaqueTRIMCyp)。但npmTRIMCyp基因的融合模式和表达剪接方式与鹰猴不同(Liao et al, 2007)。随后, 国际上的多个实验室证实了我们的实验结果, 并相继在同属于旧大陆猴的巽他平顶猴、食蟹猴及印度恒河猴中也发现存在TRIMCyp融合基因, 但在中国恒河猴(M. mulatta,Chinese rhesus macaque)和来源于非洲的乌白眉猴(Cercocebus atys, Sooty mangabeys)中却没有发现其存在(Brennan et al, 2008; Cao et al, 2011; Newman et al, 2008; Virgen et al, 2008; Wilson et al, 2008)。此后, 有研究人员对狒狒属(genusPapio)的2个种:草原狒狒(Papio cynocephalus anubis)和阿拉伯狒狒(Papio hamadrya)、原产于亚洲的2种猕猴:黑冠猕猴(M. nigra)和藏酋猴(M. thibetana)以及唯一一种在亚洲以外发现的猕猴: 地中海猕猴(M. sylvanus)也进行了TRIMCyp融合基因检测, 结果也没有发现有类似于北平顶猴的TRIMCyp融合基因存在(Dietrich et al, 2010)。最近, 本实验室在对藏酋猴(M. thibetana)、熊猴、红面猴(M. arctoides)及中国恒河猴(M. mulatta,Chinese rhesus macaque )进行TRIMCyp融合基因检测时发现, 来自越南西贡和中国云南的熊猴也存在类似于北平顶猴npmTRIMCyp的TRIMCyp融合基因 (Cao et al, 2011)。由于发现存在TRIMCyp融合基因的北平顶猴、巽他平顶猴、食蟹猴、印度恒河猴及熊猴同属于旧大陆猴的猕猴属, 因此, 可能类似北平顶猴的TRIMCyp融合基因只在旧大陆猴猕猴属的一些种中存在。
2 TRIMCyp融合基因在灵长类动物中的存在形式
2.1 TRIMCyp融合基因的形成方式
亲环蛋白(cyclophilin A, CypA)是1个肽基-脯氨酸顺反异构酶(peptidyl-prolylcis-transisomerase, PPIase), 它能催化靶蛋白中X(任意氨基酸)-脯氨酸肽键的顺反异构。CypA能通过与靶细胞中HIV-1 Gag蛋白结合, 掺入到新生HIV-1病毒颗粒中, 促进HIV-1复制(Franke et al, 1994; Thali et al, 1994)。对HIV-1复制来说, 靶细胞中的CypA比病毒颗粒产生细胞中的CypA更为重要(Hatziioannou et al, 2005)。CypA能催化HIV-1病毒衣壳蛋白G89-P90肽键的顺反异构, 这可能导致HIV-1感染性的改变(Towers, 2007)。
鹰猴TRIMCyp融合基因是由1个LINE-1(long interspersed nuclear element 1)逆转座子(retrotransposon)催化1个CypA假基因的cDNA序列插入到TRIM5α基因第7和第8外显子之间的内含子中形成的(图1)。这也是第1个在脊椎动物中被发现的通过外显子穿梭机制产生嵌合基因的例子。北平顶猴、巽他平顶猴、食蟹猴、印度恒河猴和熊猴TRIMCyp融合基因则均由CypA假基因通过LINE-1介导的逆转座方式插入到TRIM5基因的3'-UTR (untranslated region) 区域形成。它们均能经转录翻译产生由TRIM5α第2~6外显子和完整CypA结构域组成的TRIMCyp融合蛋白(图1) (Cao et al, 2011; Brennan et al, 2008; Kuang et al, 2009; Liao et al, 2007; Newman et al, 2008; Virgen et al, 2008; Wilson et al, 2008)。
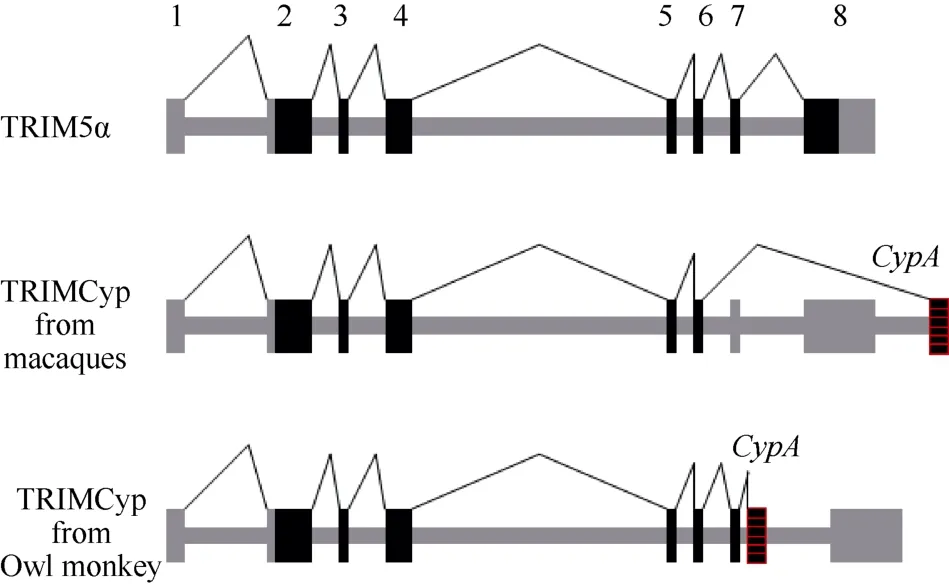
图1 TRIMCyp融合基因的形成方式及其转录剪接模式(修改自Ylinen et al, 2010)Fig. 1 The fusion and splicing modes of TRIMCyp gene (Modified from Ylinen et al, 2010)
2.2 TRIMCyp融合基因的基因型
在新大陆猴的夜猴属中,TRIMCyp融合基因均以TRIMCyp纯合子基因型存在, 不存在TRIM5/ TRIMCyp杂合子基因型(Nisole et al, 2004; Ribeiro et al, 2005; Sayah et al, 2004)。在旧大陆猴中, 北平顶猴和巽他平顶猴的TRIMCyp融合基因也为TRIMCyp纯合子基因型(Kuang et al, 2009; Liao et al, 2007; Newman et al, 2008)。在食蟹猴和印度恒河猴中,TRIMCyp融合基因以一定比例存在, 并且既存在TRIMCyp纯合子基因型, 也存在TRIM5/TRIMCyp杂合子基因型。在熊猴中,TRIMCyp融合基因以一定比例存在, 并且均为TRIM5/TRIMCyp杂合子基因型(Brennan et al, 2008; Cao et al, 2011; Dietrich et al, 2010; Newman et al, 2008; Wilson et al, 2008)。TRIMCyp融合基因在不同灵长类动物中的存在情况及其基因型的多样性提示其可能在与逆转录病毒的共同进化过程中, 受到来自不同灵长类动物种群生存的地理环境因素及古代逆转录病毒选择压力的影响, 从而产生了其在灵长类动物总群体中的过渡性存在方式及其基因型在单个种群中的过渡性存在形式, 暗示TRIMCyp融合基因的产生可能是由于特定环境因素及特定逆转录病毒选择压力对不同灵长类动物种群选择的结果。但由于相关研究极少, 证据缺乏,还有待于进一步的深入研究。
2.3 TRIM5内含子6的3'-剪接位点G-to-T突变与旧大陆猴TRIMCyp融合基因的转录剪接和形成相关
本实验室及国际上多个实验室先后发现在北平顶猴、印度恒河猴和巽他平顶猴的TRIM5内含子6的3'-剪接位点存在G-to-T的单核苷酸突变现象, 并且认为该突变可能与其转录剪接体中缺失外显子7有关(Brennan et al, 2007; Kuang et al, 2009; Wilson et al, 2008)。Newman et al (2008) 在对印度恒河猴及巽他平顶猴TRIM5基因座进行测序时也发现其内含子6的3'-剪接位点存在G-to-T突变, 并认为该突变可能会干扰mRNA的剪接过程。同时,还发现在该突变位点上游16个核苷酸处偶联了另1个能够产生NsiI限制性核酸内切酶识别位点的单核苷酸突变, 这2个突变在染色体遗传上是紧密连锁的。由于该3'-剪接位点的突变可以阻止或降低TRIM5转录形成α异构体即TRIM5α, 从而致使其可能与CypA插入TRIM53'-UTR 相关。在对北平顶猴、巽他平顶猴、印度恒河猴、食蟹猴和熊猴的TRIM5内含子6的3'-剪接位点G-to-T突变进行检测后发现, 3'-剪接位点的G-to-T突变存在与否及其存在形式与TRIM53'-UTR是否存在CypA插入现象及其形成的TRIMCyp融合基因的基因型密切相关, 即(G/G)对应TRIM5α纯合子, (G/T)对应形成TRIM5/TRIMCyp杂合子, (T/T)对应形成TRIMCyp纯合子(图2)(Cao et al, 2011; Dietrich et al, 2010; Kuang et al, 2009; Newman et al, 2008)。
3 TRIMCyp融合基因限制逆转录病毒复制的作用机制
3.1 TRIMCyp融合基因限制逆转录病毒活性的种属特异性
不同灵长类动物来源的TRIMCyp融合蛋白对不同逆转录病毒表现不同的限制活性 (表1)。如鹰猴TRIMCyp融合蛋白可限制HIV-1、HIV-2、SIVagm和猫免疫缺陷病毒(Feline immunodeficiency virus, FIV)的感染, 但不能限制SIVmac和马传染性贫血病毒(equine infectious anemia virus, EIAV)的感染(Diaz-Griffero et al, 2006; Lin & Emerman, 2006; Nisole et al, 2004; Sayah et al, 2004; Zhang et al, 2006)。北平顶猴npmTRIMCyp融合蛋白可限制HIV-2的感染, 但不能限制HIV-1和SIVmac的感染(Kuang et al, 2009; Liao et al, 2007)。
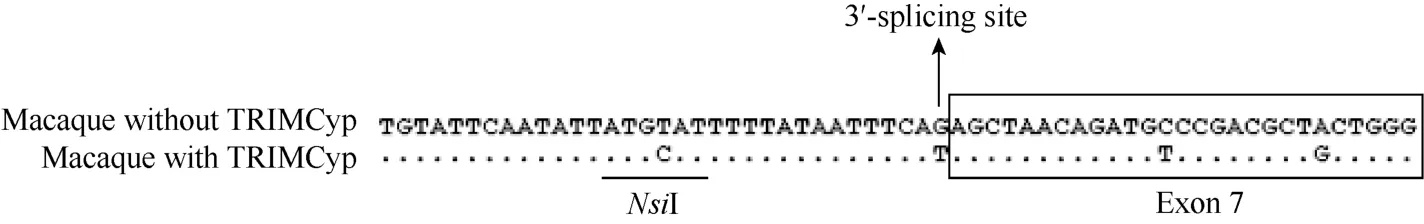
图2 TRIM5 内含子6 的3'-剪接位点突变分析(修改自Newman et al, 2008)Fig.2 Analysis of the 3'-splicing site in intron 6 at the TRIM5 locus (Modified from Newman et al, 2008)
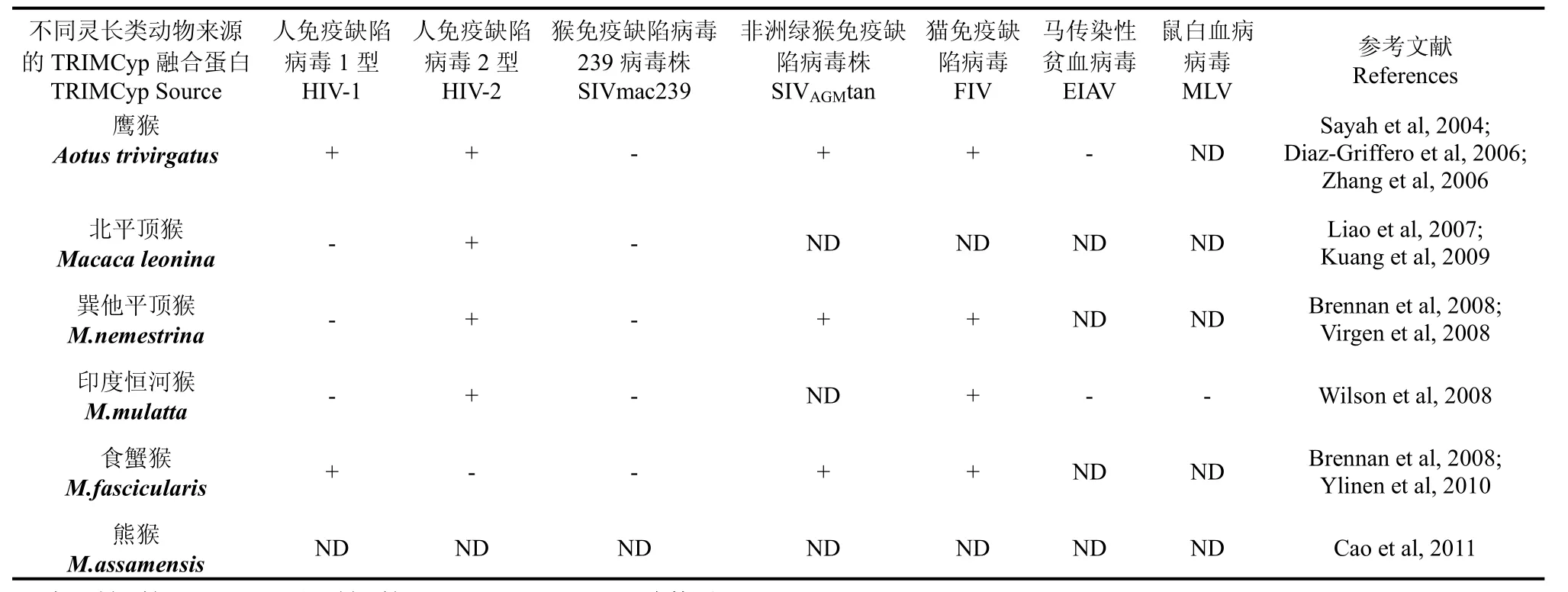
表1 灵长类动物TRIMCyp融合蛋白限制逆转录病毒的种属特异性Tab. 1 Species-specific restriction of different TRIMCyp fusion proteins to retroviruses
3.2 影响TRIMCyp限制逆转录病毒活性的因素
目前认为TRIMCyp融合蛋白限制逆转录病毒的感染是通过其CypA结构域与病毒衣壳蛋白的相互作用而发挥限制作用(Chatterji et al, 2005; Nisole et al, 2004; Sayah et al, 2004; Towers et al, 2003; Wilson et al, 2008)。研究表明, TRIMCyp融合蛋白必须与其所限制的逆转录病毒的特定组分结合才能抑制其感染(Diaz-Griffero et al, 2006)。环孢霉素A (cyclosporine A, CsA)可阻断CypA-CA间相互作用(Luban et al, 1993)并抑制TRIMCyp融合蛋白的限制作用(Chatterji et al, 2005; Nisole et al, 2004; Sayah et al, 2004; Towers et al, 2003; Wilson et al, 2008)。
TRIMCyp融合蛋白CypA结构域中的第66位和69位氨基酸在其限制不同逆转录病毒活性方面具有重要作用。鹰猴TRIMCyp 融合蛋白中这2个位点未发生突变(DR), 它可以限制HIV-1、HIV-2、 FIV和SIVAGMTan的感染, 而巽他平顶猴TRIMCyp融合蛋白在这2个位点存在双突变现象(NH), 它可以限制HIV-2、FIV和SIVAGMTan的感染, 但不能限制HIV-1的感染(Virgen et al, 2008)。有研究人员在恒河猴中对这2个位点进行突变分析后发现, 未突变(DR)的TRIMCyp蛋白和第69位单氨基酸突变R69H (DH)的TRIMCyp蛋白可以限制HIV-1, 双突变(NH)的TRIMCyp蛋白和第66位单氨基酸突变D66N(NR)的TRIMCyp蛋白不能限制HIV-1, 但可以限制HIV-2, 并且双突变(NH)的TRIMCyp蛋白限制HIV-2的能力比未突变(DR)的TRIMCyp蛋白显著增强(Price et al, 2009)。我们在北平顶猴中也发现其npmTRIMCyp的CypA结构域中存在第66位和69位氨基酸的双突变(NH)现象, 这可能是该蛋白不能够限制HIV-1的原因之一(Kuang et al, 2009)。
另有研究表明, 食蟹猴TRIMCyp 融合蛋白不能限制HIV-1和SIVmac239的感染(Brennan et al, 2008)。但是, Ylinen et al (2010)在印度尼西亚食蟹猴(Indonesian Cynomolgus macaque)中发现, 其TRIMCyp 融合蛋白可以限制HIV-1、SIVtan和FIV的感染, 但不能限制HIV-2的感染, 在对产生这种现象的原因进行分析与研究后发现, CypA结构域中第54位氨基酸的单点突变(H54R)决定了食蟹猴TRIMCyp 融合蛋白限制HIV-1和FIV的能力; 而另一个重要的单氨基酸突变E143K, 使得能够限制HIV-1和HIV-2的食蟹猴TRIMCyp蛋白丧失了限制HIV-2的能力而只能限制HIV-1, 并且使其抑制SIVAGMtan的能力大大降低(Ylinen et al, 2010)。
最近, Dietrich et al (2011)对来源于印度尼西亚、印度支那(中南半岛)、菲律宾及毛里求斯的食蟹猴(分别为18、14、44和4只)中TRIMCyp融合基因的存在情况进行了检测, 发现除来源于毛里求斯的食蟹猴不存在TRIMCyp融合基因外, 其他3种不同地区来源的食蟹猴均存在TRIMCyp融合基因;通过对5只印度尼西亚食蟹猴TRIMCyp融合基因等位基因限制逆转录病毒活性的研究, 发现TRIMCyp融合蛋白CypA结构域中的第66位和143位氨基酸与印度尼西亚食蟹猴TRIMCyp融合蛋白的限制逆转录病毒活性密切相关, 证实了Ylinen et al的研究结果, 并且他们还发现TRIMCyp融合蛋白的Coiled-coil结构域中碱基突变H178Y, 会影响TRIMCyp融合蛋白的表达和稳定性, 但并不会影响TRIMCyp融合蛋白的逆转录病毒限制能力(Dietrich et al, 2011)。由于不同灵长类动物来源的TRIMCyp融合蛋白在其CypA结构域中许多位点氨基酸的差异(图3), 从而导致其TRIMCyp融合蛋白与不同逆转录病毒的衣壳蛋白Gag的识别和结合能力出现差异, 继而导致了抑制活性上的差异。
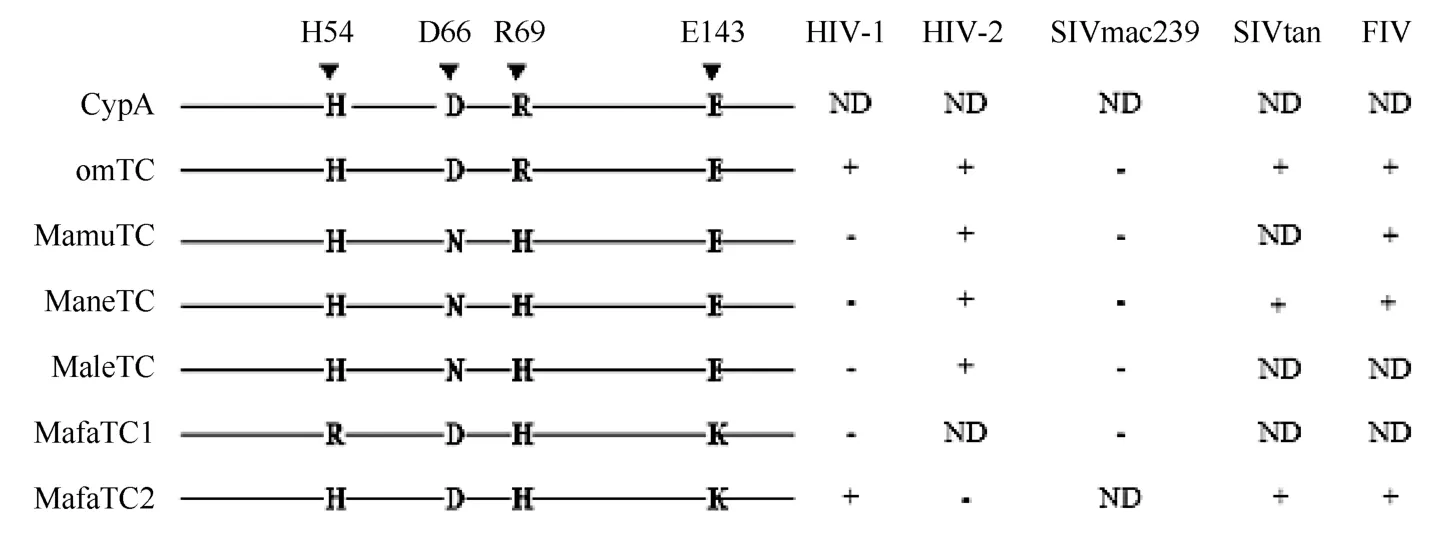
图3 灵长类动物TRIMCyp融合蛋白CypA结构域氨基酸位点差异与限制逆转录病毒活性关系Fig.3 The amino acids difference related to the restriction of different retroviruses in the CypA domain of species variants of TRIMCyps
3.3 TRIMCyp融合基因限制HIV-1感染和复制的可能机制
TRIMCyp与TRIM5α的区别只是C末端存在差异, 因此认为TRIMCyp即可能以与TRIM5α类似的机制来限制逆转录病毒的复制。虽然RING和B-box2结构域影响TRIMCyp的半衰期和抗HIV-1活性, 但它们对TRIMCyp的抗病毒活性并不是绝对必要的(Diaz-Griffero et al, 2007), 这可能是由于CypA本身就具有促进脱壳和限制HIV-1作用, 使得TRIMCyp的抗病毒活性不太依赖于由RBCC所募集而来的辅助因子(Luban, 2007)。早期研究表明, TRIMCyp在哺乳动物细胞内主要以三聚体的形式存在(Diaz-Griffero et al, 2006; Javanbakht et al, 2007)。但近年来也发现, 除三聚体外, TRIMCyp还存在二聚体、六聚体及非常复杂的高聚体等形式,并且六聚体似乎是TRIMCyp在哺乳动物细胞中的主要多聚体存在形式。六聚体TRIMCyp蛋白结构有利于识别成熟逆转录病毒衣壳的组成单元六聚体衣壳粒(capsomer), 但是TRIMCyp的多聚化与特异性病毒衣壳蛋白的识别无关(Nepveu-Traversy et al, 2009)。
目前认为鹰猴TRIMCyp融合蛋白可能以以下机制限制HIV-1的感染(图4): 首先, 在HIV-1进入细胞后, TRIMCyp的CypA结构域很快地与HIV-1衣壳蛋白结合(Diaz-Griffero et al, 2006; Nisole et al, 2004; Perez-Caballero et al, 2005; Sayah et al, 2004; Yap et al, 2006), 加速HIV-1的脱壳或衣壳蛋白的降解, 阻止HIV-1进行逆转录(Diaz-Griffero et al, 2006; Yap et al, 2006)。在这个过程中, Coiled-coil结构域和CypA结构域对TRIMCyp与HIV-1衣壳-核衣壳蛋白复合体(capsid-nucleocapsid complexes, CA-NC complexes)的相互作用至关重要, 并且含有这2个结构域的三聚体TRIMCyp融合蛋白能够更有效地与衣壳蛋白结合(Diaz-Griffero et al, 2006)。其次, 虽然蛋白酶体抑制剂能使HIV-1恢复逆转录产物的产生, 形成有整合能力的整合前复合物(pre-integration complex, PIC), 但是这种PIC被TRIMCyp以未知的方式阻止入核, 从而限制了病毒进一步复制(Anderson et al, 2006)。另外, 最近有研究发现TRIM5α蛋白除了能够通过直接抑制逆转录病毒的复制过程来抑制其感染外的另一个功能为作为模式识别受体(pattern-recognition receptors, PRR)引起细胞天然免疫反应(innate immune responses)。Pertel et al发现TRIM5α蛋白与病毒衣壳蛋白结合激活了它的泛素(ubiquitin, Ub)连接酶活性, 它与泛素交联酶复合物UBC13-UEV1A等泛素化酶一起催化合成了由泛素分子彼此间通过第63位赖氨酸(lysine 63, K63)残基连接起来的游离泛素链(K63-linked ubiquitin chains),该泛素链促使蛋白激酶复合体中的TAK1磷酸化并被激活, 激活状态下的TAK1可以激活转录因子AP-1和NF-κB, 进而通过上调相关免疫分子的表达,引起细胞的抗病毒天然免疫反应。鹰猴TRIMCyp融合蛋白在体外也可以催化游离K63-linked ubiquitin chains的合成, 并且在加入HIV-1衣壳蛋白后, 产生游离泛素链的量会大量增加, 产生的该游离泛素链也可以促使TAK1发生磷酸化(Aiken & Joyce, 2011; Pertel et al, 2011)。提示鹰猴TRIMCyp也可能具有作为模式识别受体与逆转录病毒衣壳蛋白相互作用, 从而引发细胞抗病毒天然免疫反应的功能。
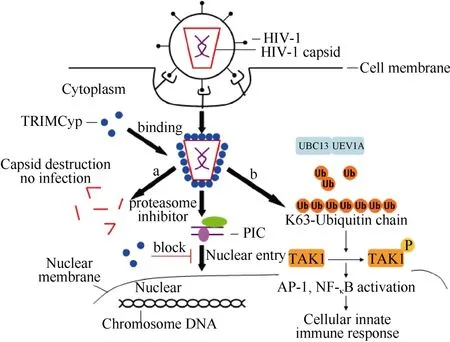
图4 鹰猴TRIMCyp融合蛋白限制HIV-1感染和复制的可能机制Fig.4 TRIMCyp fusion protein from owl monkey interfering with the HIV-1 infection and replication
4 前景和展望
迄今为止, 人们已经在新大陆猴的夜猴属和旧大陆猴猕猴属中的北平顶猴、巽他平顶猴、食蟹猴、印度恒河猴和熊猴中发现存在TRIMCyp基因融合现象。该融合基因在新大陆猴和旧大陆猴中的基因融合模式和表达剪接方式截然不同。灵长类动物种属繁多, 资源丰富, 其他灵长类动物是否也存在类似的TRIMCyp基因融合现象? 若存在, 其融合基因的形成方式和表达剪接方式怎样? 是否存在新的TRIMCyp基因融合模式?
灵长类动物TRIMCyp融合基因发现至今, 人们对鹰猴TRIMCyp融合蛋白限制HIV-1复制的作用机制有一定了解, 而旧大陆猴TRIMCyp融合蛋白限制HIV-1复制的作用机制及灵长类动物TRIMCyp融合蛋白对HIV-1以外的其他逆转录病毒的限制作用机制大都还不清楚, 许多问题还没有解决, 部分研究成果相互矛盾, 有些结论还停留在推测的水平。比如, 灵长类动物TRIMCyp融合蛋白限制逆转录病毒复制的作用机制是否具有一个普遍相似的规律?灵长类动物TRIMCyp融合蛋白对不同逆转录病毒特异性限制的原因是什么?有研究表明, TRIM5α分子是通过先形成二聚体, 然后与逆转录病毒的衣壳蛋白识别并结合, 之后在TRIM5α分子的B-box2和L2结构域的介导下发生多聚化并将单独的TRIM5α二聚体组装起来, 从而形成围绕在病毒衣壳周围的六聚体网格状结构(hexameric lattice), 继而发挥其限制功能(Sastri & Campbell, 2011)。那么灵长类动物TRIMCyp融合蛋白是否会以相似的方式来与逆转录病毒衣壳蛋白结合并发挥其抗病毒功能?大部分旧大陆猴TRIMCyp融合蛋白都不能限制HIV-1感染的具体机制是什么?鹰猴TRIMCyp与病毒衣壳蛋白结合后, 是否会招募其他TRIM家族蛋白协助其发挥抗病毒能力?存在TRIM5/TRIMCyp杂合子基因型的灵长类动物, 其TRIM5α蛋白和TRIMCyp融合蛋白在彼此限制逆转录病毒作用中所扮演的角色是什么?这些问题都是亟待研究解决的科学问题。TRIM5-CypA融合基因的发现和研究不仅将对HIV/AIDS发病机制研究、药物和疫苗研发、生物治疗策略具有重要意义, 而且将为基于TRIMCyp等固有免疫分子建立更为理想的HIV/AIDS感染的灵长类动物模型提供重要的科学思路和依据。
Aiken C, Joyce S. 2011. Immunology: TRIM5 does double duty [J]. Nature,472(7343): 305-306.
Anderson JL, Campbell EM, Wu XL, Vandegraaff N, Engelman A, Hope TJ. 2006. Proteasome inhibition reveals that a functional preintegration complex intermediate can be generated during restriction by diverse TRIM5 proteins [J]. J Virol,80(19): 9754-9760.
Bieniasz PD. 2004. Intrinsic immunity: a front-line defense against viral attack [J]. Nat Immunol,5(11): 1109-1115.
Brennan G, Kozyrev Y, Kodama T, Hu SL. 2007. Novel TRIM5 isoforms expressed by Macaca nemestrina [J]. J Virol,81(22): 12210-12217.
Brennan G, Kozyrev Y, Hu SL. 2008. TRIMCyp expression in Old World primates Macaca nemestrina and Macaca fascicularis [J]. Proc Natl Acad Sci USA,105(9): 3569-3574.
Cao G, Nie WH, Liu FL, Kuang YQ, Wang JH, Su WT, Zheng YT. 2011. Identification of the TRIM5/TRIMCyp heterozygous genotype in Macaca assamensis [J]. Zool Res,32(1): 40-49. [曹 光,佴文惠,刘丰亮,况轶群,王金焕,苏伟婷,郑永唐. 2011.熊猴存在TRIM5/ TRIMCyp杂合子基因型.动物学研究,32(1): 40-49.]
Chatterji U, Bobardt MD, Stanfield R, Ptak R G, Pallansch LA, Ward PA, Jones MJ, Stoddart CA, Scalfaro P, Dumont JM, Besseghir K, Rosenwirth B, Gallay PA. 2005. Naturally occurring capsid substitutions render HIV-1 cyclophilin A independent in human cells and TRIM-cyclophilin-resistant in Owl monkey cells [J]. J Biol Chem,280(48): 40293-40300.
Diaz-Griffero F, Vandegraaff N, Li Y, McGee-Estrada K, Stremlau M, Welikala S, Si ZH, Engelman A, Sodroski J. 2006. Requirements forcapsid-binding and an effector function in TRIMCyp-mediated restriction of HIV-1 [J]. Virology,351(2): 404-419.
Diaz-Griffero F, Kar A, Perron M, Xiang SH, Javanbakht H, Li X, Sodroski J. 2007. Modulation of retroviral restriction and proteasome inhibitorresistant turnover by changes in the TRIM5αB-box 2 domain [J]. J Virol,81(19): 10362-10378.
Diaz-Griffero F, Qin XR, Hayashi F, Kigawa T, Finzi A, Sarnak Z, Lienlaf M, Yokoyama S, Sodroski J. 2009. A B-box 2 surface patch important for TRIM5αself-association, capsid binding avidity, and retrovirus restriction [J]. J Virol,83(20): 10737-10751.
Dietrich EA, Brennan G, Ferguson B, Wiseman RW, O'Connor D, Hu SL. 2011. Variable prevalence and functional diversity of the antiretroviral restriction factor TRIMCyp in Macaca fascicularis [J]. J Virol,85(19): 9956-9963.
Dietrich EA, Jones-Engel L, Hu SL. 2010. Evolution of the antiretroviral restriction factor TRIMCyp in Old World primates [J]. PLoS One,5(11): e14019.
Franke EK, Yuan HE, Luban J. 1994. Specific incorporation of cyclophilin A into HIV-1 virions [J]. Nature,372(6504): 359-362.
Gilbert PB, McKeague IW, Eisen G, Mullins C, Guéye-NDiaye A, Mboup S, Kanki PJ. 2003. Comparison of HIV-1 and HIV-2 infectivity from a prospective cohort study in Senegal [J]. Stat Med,22(4): 573-593.
Gippoliti S. 2001.Notes on the taxonomy of Macaca nemestrina leonina blyth, 1863 (Primates: Cercopithecidae) [J]. Hystrix It J Mamm,12(1): 51-54.
Goff SP. 2004. Retrovirus restriction factors [J]. Mol Cell,16(6): 849-859.
Groves CP. 2001. Primate Taxonomy [M]. Washington, DC, USA: Smithsonian Institution Press, 222-224.
Hatziioannou T, Perez-Caballero D, Cowan S, Bieniasz PD. 2005. Cyclophilin interactions with incoming human immunodeficiency virus type 1 capsids with opposing effects on infectivity in human cells [J]. J Virol,79(1): 176-183.
Hofmann W, Schubert D, LaBonte J, Munson L, Gibson S, Scammell J, Ferrigno P, Sodroski J. 1999. Species-specific, postentry barriers to primate immunodeficiency virus infection [J]. J Virol,73(12): 10020-10028.
Javanbakht H, Diaz-Griffero F, Yuan W, Yeung DF, Li X, Song B, Sodroski J. 2007. The ability of multimerized cyclophilin A to restrict retrovirus infection [J]. Virology,367(1): 19-29.
Kuang YQ, Tang X, Liu FL, Jiang XL, Zhang YP, Gao GX, Zheng YT. 2009. Genotyping of TRIM5 locus in northern pig-tailed macaques (Macaca leonina), a primate species susceptible to human immunodeficiency virus type 1 infection [J]. Retrovirology,6: 58.
Li QQ, Zhang YP. 2005. Phylogenetic relationships of the macaques (Cercopithecidae: Macaca), inferred from mitochondrial DNA sequences [J]. Biochem Genet,43(7-8): 375-386.
Liao CH, Kuang YQ, Liu HL, Zheng YT, Su B. 2007. A novel fusion gene, TRIM5-Cyclophilin A in the pig-tailed macaque determines its susceptibility to HIV-1 infection [J]. AIDS,21(Suppl 8): S19-S26.
Lilly F. 1967. Susceptibility to two strains of Friend leukemia virus in mice [J]. Science,155(3761): 461-462.
Lin TY, Emerman M. 2006. Cyclophilin A interacts with diverse lentiviral capsids [J]. Retrovirology,3: 70.
Luban J. 2007. Cyclophilin A, TRIM5, and resistance to human immunodeficiency virus type 1 infection [J]. J Virol,81(3): 1054-1061.
Luban J, Bossolt KL, Franke EK, Kalpana GV, Goff SP. 1993. Human immunodeficiency virus type 1 Gag protein binds to cyclophilins A and B [J]. Cell,73(6): 1067-1078.
Nepveu-Traversy ME, Bérubé J, Berthoux L. 2009. TRIM5alpha and TRIMCyp form apparent hexamers and their multimeric state is not affected by exposure to restriction-sensitive viruses or by treatment with pharmacological inhibitors [J]. Retrovirology,6: 100.
Newman RM, Hall L, Kirmaier A, Pozzi LA, Pery E, Farzan M, O'Neil SP, Johnson W. 2008. Evolution of a TRIM5-CypA splice isoform in old world monkeys [J]. PLoS Pathog,4(2): e1000003.
Nisole S, Lynch C, Stoye JP, Yap MW. 2004. A Trim5-cyclophilin A fusion protein found in owl monkey kidney cells can restrict HIV-1 [J]. Proc Natl Acad Sci USA,101(36): 13324-13328.
Perez-Caballero D, Hatziioannou T, Zhang FW, Cowan S, Bieniasz PD. 2005. Restriction of human immunodeficiency virus type 1 by TRIM-CypA occurs with rapid kinetics and independently of cytoplasmic bodies, ubiquitin, and proteasome activity [J]. J Virol,79(24): 15567-15572.
Pertel T, Hausmann S, Morger D, Zuger S, Guerra J, Lascano J, Reinhard C, Santoni FA, Uchil P D, Chatel L, Bisiaux A, Albert ML, Strambio-De-Castillia C, Mothes W, Pizzato M, Grutter MG, Luban J. 2011. TRIM5 is an innate immune sensor for the retrovirus capsid lattice [J]. Nature,472(7343): 361-365.
Price AJ, Marzetta F, Lammers M, Ylinen LM, Schaller T, Wilson S J, Towers GJ, James LC. 2009. Active site remodeling switches HIV specificity of antiretroviral TRIMCyp [J]. Nat Struct Mol Biol,16(10): 1036-1042.
Reeves JD, Doms RW. 2002. Human immunodeficiency virus type 2 [J]. J Gen Virol, 83(6): 1253-1265.
Ribeiro IP, Menezes AN, Moreira MA, Bonvicino CR, Seuanez HN, Soares MA. 2005. Evolution of cyclophilin A and TRIMCyp retrotransposition in New World primates [J]. J Virol,79(23): 14998-15003.
Rosenblum LL, Supriatna J, Melnick DJ. 1997. Phylogeographic analysis of pig-tail macaque populations (Macaca nemestrina) inferred from mitochondrial DNA [J]. Am J Phys Anthropol,104(1): 35-45.
Sastri J, Campbell EM. 2011. Recent insights into the mechanism and consequences of TRIM5αretroviral restriction [J]. AIDS Res Hum Retroviruses,27(3): 231-238.
Sawyer SL, Wu LI, Emerman M, Malik HS. 2005. Positive selection of primate TRIM5αidentifies a critical species-specific retroviral restriction domain [J]. Proc Natl Acad Sci USA,102(8): 2832-2837.
Sayah DM, Sokolskaja E, Berthoux L, Luban J. 2004. Cyclophilin A retrotransposition into TRIM5 explains owl monkey resistance to HIV-1 [J]. Nature,430(6999): 569-573.
Schaller T, Hué S, Towers GJ. 2007. An active TRIM5 protein in rabbits indicates a common antiviral ancestor for mammalian TRIM5 proteins [J]. J Virol,81(21): 11713-11721.
Si ZH, Vandegraaff N, O'Huigin C, Song B, Yuan W, Xu C, Perron M, Li X, Marasco WA, Engelman A, Dean M, Sodroski J. 2006. Evolution of a cytoplasmic tripartite motif (TRIM) protein in cows that restricts retroviral infection [J]. Proc Natl Acad Sci USA,103(19): 7454-7459.
Song B, Gold B, O'Huigin C, Javanbakht H, Li X, Stremlau M, Winkler C, Dean M, Sodroski J. 2005. The B30.2 (SPRY) domain of the retroviral restriction factor TRIM5αexhibits lineage-specific length and sequence variation in primates [J]. J Virol,79(10): 6111-6121.
Steeves R, Lilly F. 1977. Interactions between host and viral genomes in mouse leukemia [J]. Annu Rev Genet,11(1): 277-296.
Stremlau M, Owens CM, Perron MJ, Kiessling M, Autissier P, Sodroski J. 2004. The cytoplasmic body component TRIM5αrestricts HIV-1 infection in Old World monkeys [J]. Nature,427(6977): 848-853.
Tang X, Kuang YQ, Zheng YT. 2009. Research advance of TRIM5αon structure and restriction mechanism to HIV-1 replication [J]. Chin J Virol,25(2): 148-153. [汤霞,况轶群,郑永唐. 2009. TRIM5α分子结构和限制HIV-1复制机制的研究进展.病毒学报,25(2): 148-153.]
Thali M, Bukovsky A, Kondo E, Rosenwirth B, Walsh CT, Sodroski J, Gottlinger HG. 1994. Functional association of cyclophilin A with HIV-1 virions [J]. Nature,372(6504): 363-365.
Towers GJ. 2007. The control of viral infection by tripartite motif proteins and cyclophilin A [J]. Retrovirology,4: 40.
Towers GJ, Hatziioannou T, Cowan S, Goff SP, Luban J, Bieniasz PD. 2003. Cyclophilin A modulates the sensitivity of HIV-1 to host restriction factors [J]. Nat Med,9(9): 1138-1143.
Virgen CA, Kratovac Z, Bieniasz PD, Hatziioannou T. 2008. Independent genesis of chimeric TRIM5-cyclophilin proteins in two primate species [J]. Proc Natl Acad Sci USA,105(9): 3563-3568.
Wilson SJ, Webb BL, Ylinen LM, Verschoor E, Heeney JL, Towers GJ. 2008. Independent evolution of an antiviral TRIMCyp in rhesus macaques [J]. Proc Natl Acad Sci USA,105(9): 3557-3562.
Yap MW, Dodding MP, Stoye JP. 2006. Trim-cyclophilin A fusion proteins can restrict human immunodeficiency virus type 1 infection at two distinct phases in the viral life cycle [J]. J Virol,80(8): 4061-4067.
Ylinen LMJ, Keckesova Z, Webb BL, Gifford RJ, Smith TP, Towers GJ. 2006. Isolation of an active Lv1 gene from cattle indicates that tripartite motif protein-mediated innate immunity to retroviral infection is widespread among mammals [J]. J Virol,80(15): 7332-7338.
Ylinen LMJ, Price AJ, Rasaiyaah J, Hue S, Rose NJ, Marzetta F, James LC, Towers GJ. 2010. Conformational adaptation of Asian macaque TRIMCyp directs lineage specific antiviral activity [J]. PLoS Pathog,6(8): e1001062.
Zhang FW, Hatziioannou T, Perez-Caballero D, Derse D, Bieniasz PD. 2006. Antiretroviral potential of human tripartite motif-5 and related proteins [J]. Virology,353(2): 396-409.
The primateTRIMCypfusion genes and mechanism of restricting retroviruses replication
CAO Guang1,2, LIU Feng-Liang1, ZHANG Gao-Hong1, ZHENG Yong-Tang1,*
(1. Key Laboratory of Animal Models and Human Disease Mechanisms of the Chinese Academy of Sciences &Yunnan Province, Kunming Institute of Zoology, the Chinese Academy of Sciences, Kunming 650223, China; 2. Graduate University of the Chinese Academy of Science, Beijing 100049, China)
TRIM5-cyclophilin A(TRIMCyp)fusion gene is an unusualTRIM5locus. At present, this fusion phenomenon has been found in the representative species which contain owl monkey (Aotus trivirgatus) ofAotusgenus that belongs to New World monkeys and Old World monkeys such as Northern pig-tailed macaque (M. leonina), Sunda pig-tailed macaque(M. nemestrina), Crab-eating macaque (M. fascicularis), Indian rhesus macaque (M. mulatta) and Assam macaque (M. assamensis), etc. But the fusion mode and transcription splicing pattern ofTRIMCypfusion gene are different between New World and Old World monkeys. TheTRIMCypfusion gene of New World monkeys is formed by inserting a CypA pseudogene cDNA sequence into the region between exon 7 and exon 8 of theTRIM5locus through retrotransposition. However theTRIMCypfusion gene of Old World monkeys results from the retrotransposition of aCypApseudogene cDNA into 3' terminal or 3'-UTR ofTRIM5gene. The distributions, genotypes, expression and restricting activities against different retroviruses of TRIMCyp were different across species of primates. Moreover, most of the researches focused on theTRIMCypfusion gene of owl monkey and pig-tailed macaque and found that they may play very important roles in restricting HIV-1 replication and determine the susceptibility to HIV-1 infection. It was reported that the TRIMCyp protein of owl monkey could inhibit HIV-1 infection in a similar way as TRIM5α, butTRIMCyp protein of pig-tailed monkey loss the restricting activity to HIV-1 infection. Here we reviewed the distributions, genotypes and restriction mechanism for inhibiting retroviruses replication ofTRIMCypfusion gene in primates.
TRIM5; TRIMCyp; Restriction factor; HIV; AIDS; Animal model
Q291; R-332; Q959.848; R512.91
A
0254-5853-(2012)01-0099-09
10.3724/SP.J.1141.2012.01099
2011-10-28;接受日期:2011-12-29
国家自然科学基金(U0832601; 81172876; 30872317; 30800113);国家科技重大专项“十一五”计划(2009ZX09501-029);中国科学院知识创新工程重要方向(KSCX1-YW-10; KSCX2-EW-R-13)
∗通信作者(Corresponding author),Tex/Fax:0871-5195684, E-mail: zhengyt@mail.kiz.ac.cn
曹光,男,硕士研究生,主要从事TRIM5α限制HIV-1复制机制研究

