树鼩干扰素家族的基本构成及分子特征分析
李明利, 田巍威, 高跃东, 郭 彦, 黄京飞, 张华堂,*
(1. 中国科学院昆明动物研究所 动物模型与人类疾病机理重点实验室, 云南 昆明 650223; 2. 中国科学院研究生院, 北京 100049; 3. 西南大学 生物技术学院, 重庆 北碚 400715; 4. 中国科学院昆明生物多样性大型仪器区域中心, 云南 昆明 650223; 5. 中国科学院昆明动物研究所 遗传资源与进化国家重点实验室, 云南 昆明 650223)
树鼩干扰素家族的基本构成及分子特征分析
李明利1,2, 田巍威1,3, 高跃东4,5, 郭 彦1, 黄京飞4,5, 张华堂1,*
(1.中国科学院昆明动物研究所 动物模型与人类疾病机理重点实验室,云南 昆明650223; 2.中国科学院研究生院,北京100049; 3.西南大学 生物技术学院,重庆 北碚400715; 4.中国科学院昆明生物多样性大型仪器区域中心,云南 昆明650223; 5.中国科学院昆明动物研究所 遗传资源与进化国家重点实验室,云南 昆明650223)
干扰素(IFN)是在“危险信号”刺激下, 由细胞分泌的具有抗病毒、抗肿瘤、抑制细胞增殖和免疫调节等多重作用的糖蛋白家族, 在机体免疫系统中具有重要地位。树鼩作为多种人类疾病研究模型的前景已受到广泛关注, 但对其IFN家族的研究尚属空白。该研究在现有的树鼩全基因组数据基础上, 应用大片段核酸序列比对、基因预测等方法, 对树鼩IFN家族的基本构成和分子特征进行预测和分析。结果显示, 树鼩具有I型IFN:α (5个亚型)、β、ω、κ、ε、δ;II型IFN-γ;III型IFN:IFN-λ1、λ2/3, 所编码的氨基酸序列及蛋白空间结构与其它哺乳动物具有较高的相似性, 但在半胱氨酸位置和N糖基化个数上具有部分差异。该研究以全基因组数据对树鼩IFN家族信息进行系统挖掘和分析, 为树鼩IFN的基因克隆以及其在感染免疫学中的作用和机理研究奠定了基础。
树鼩; 干扰素; 大片段核酸序列比对; 基因预测
干扰素(interferon, IFN)最初是由Isaacs & Lindenmann(1957)在研究病毒干扰现象时发现的一类性质待定的“可溶性物质”。直至1980年, Taniguchi et al(1980a; 1980b)首次克隆了人IFN-α和IFN-β全长cDNA, 由此,IFN成为研究最早的一类细胞因子。现已证明, IFN为多成员家族, 它们普遍存在于脊椎动物中, 是多种类细胞在受到“危险信号”刺激后分泌的糖蛋白。IFN作为机体抵抗病毒感染的第一道防线, 具有抗病毒、抗肿瘤、抑制细胞增殖及免疫调节等多重作用(Ank et al, 2006; Brand et al, 2005; Kotenko et al, 2003; Österlund et al, 2005)。依据基因序列、染色体定位和受体特异性等特点, 可将IFN分为3种类型(Kotenko et al, 2003; Samarajiwa et al, 2009)。I型IFN包括9个亚型, 即α、β、ε、κ、τ、ω、ζ、ν、δ; II型IFN仅有IFN-γ 1种; III型IFN由 IFN-λ1(IL-29)、IFN-λ2(IL-28A)和IFN-λ3(IL-28B)构成。
树鼩(tree shrew,Tupaia belangeri), 其外形酷似松鼠(Cao et al, 2003), 在进化关系上与灵长类动物较为接近(Novacek, 1992; Waddell et al, 2001)。Walter et al(1996)证实了我国学者早在80年代的发现(Su et al, 1987), 即树鼩肝细胞在体内外均能感染人乙型肝炎病毒(HBV), 表明树鼩可作为模型动物用以研究乙型肝炎病毒致病机理,但是, 目前对树鼩IFN家族的研究尚属空白状态。阐明树鼩IFN家族的基本成员及其分子结构-功能关系, 是系统研究IFN在乙型肝炎等病毒性疾病感染过程中免疫功能的前提。因此, 本研究以目前仅有的树鼩全基因组数据为基础, 通过基因预测和蛋白质模建等生物信息学方法, 对树鼩IFN家族的基本构成和分子结构特征进行了系统分析, 为进一步全面深入树鼩IFN的功能和免疫调节机理研究奠定基础。
1 材料方法
1.1 树鼩基因组及参比IFN基因
树鼩全基因组序列(BioProject Accession: PRJNA13971)取自NCBI数据库。这一序列由美国哈佛-麻省理工的博德研究所(Broad Institute of MIT and Harvard)于2006年公布, 覆盖率为2倍(http://www.ncbi.nlm.nih.gov/nuccore/108744176)。用以参比的已克隆各物种IFN基因取自GenBank数据库, 其录入号分别为:
I型IFN:人α:NM_024013, NM_000605, NM_ 021068, NM_002169-73, NM_021002, NM_021057, NM_027866-67, NM_004681, NM_006900, NM_ 021268, NM_002175, NM_036676; β:NM_002176;ε: NM_176891;κ: NM_020124;ω: NM_002177。小鼠α:NM_010502-05, NM_206871, NM_0083343, NM_010507,NM_008333,NM_177361,M_177347;β:NM_010510;ε: NM_177348;ζ: NM_197889;κ: NM_199157。大鼠α:NM_001014786, NM_233152;β:NM_019127; 猪α:NM_214393, NM_001130219, NM_001166319, NM_001164860, NM_001164848-49, NM_001195377, NM_001166310, NM_0011130246, NM_001164843, NM_001166318, NM_001164855, NM_001195375;β:NM_001003923;δ:NM_ 001002832;ε: NM_001105310; κ: NM_001164857;ω: NM_ 001130238。牛α:NM_001017411, NM_ 174085, NM_001172040-41;β:NM_174350;τ:NM_001245936;ω: NM_174351。马α:NM_ 001114537- 38, NM_001099441;β:NM_001099440;ω: NM_001083595。猫NM_001006654。鸡NM_205427。
II型IFN:人NM_000619;黑猩猩NM_ 001193665;小鼠NM_213948;大鼠NM_008337;猪NM_1388802;牛NM_174086;马NM_ 001081949;羊NM_001009803;猫NM_001009873;犬NM_ 001003174。
III型IFN:人NM_172138-40; 小鼠NM_ 001024673, NM_177396; 蝙蝠HQ201955-56; 鸡EF587763; 爪蟾FJ581033-36。
1.2 基因预测
以上述树鼩全基因组和各物种IFN编码序列为基础, 采用全基因组在线BLAST(Altschul et al, 1990)及用于大片段序列比对的工具BLASTZ (Schwartz et al, 2000, 2003) 进行序列比对。采用GENSCAN(Burge & Karlin, 1998)预测软件对树鼩IFN家族的可能成员进行预测。该基因预测方法的基础为五阶马尔可夫模型, 同时结合了六聚体频率以及编码信号如起始密码子、TATA框、帽子位点、poly-A等信息预测基因。
1.3 分子特征分析
分别采用DNAMAN(http://www.lynnon.com/)、BLASTP(Altschul et al, 1990)和t_coffee (Notredame et al, 2000)对核酸和氨基酸序列进行分析。内含子-外显子基因结构模式图用在线软件Fancy GENE v1.4(http://host13.bioinfo3.ifom-ieo-campus.it/fancyg ene/)构建。蛋白质信号肽序列采用在线SignalP 3.0软件(http://www.cbs.dtu.dk/services/SignalP/)预测。蛋白质三维结构的模建和分析则通过Discovery studio(Accelrys Software:Discovery Studio 3.1)和PyMOL(http://www.pymol.org/)完成。
2 结果与讨论
2.1 基因组数据来源与质量
尽管本研究所采用的基因组数据仅有2倍覆盖率, 但依据此基因组序列, Ensembl数据库已预测得到了树鼩IFN-λ编码基因(ENSTBET0000005887) (http://asia.ensembl.org/Tupaia_belangeri/Gene/Sum mary?g=ENSTBEG00000005902;r=scaffold_115883: 37536-39087;t=ENSTBET00000005887)。同时,Fox et al(2009)预测得到两条树鼩IFN III型基因(具体序列未公布)。2011年10月, Lindblad-Toh et al (2011)使用该2倍覆盖率树鼩基因组作为29个哺乳动物之一进行多物种全基因组分析, 得出“所有哺乳动物编码基因均受共同的DNA(包含启动子中的保守元件、蛋白编码序列中的功能元件、转录因子靶序列等)调节, 从而增强或减弱编码基因表达”的结论。此外, 我们利用此数据已成功克隆得到树鼩IFN-λ2/3的全长cDNA序列(待发表)。这些结果表明,现有的树鼩基因组数据为进一步挖掘、分析相关基因奠定了基础。进一步, 我们将使用华盛顿大学医学院的基因组测序中心(Genome Sequencing Center (GSC) at Washington University (WashU) of Medicine)测定的6倍(BioProject Accession: PRJNA20273)(待发表)和中科院昆明动物研究所测定的70倍覆盖率树鼩基因组(待发表)进行IFN基因挖掘和验证。
2.2 树鼩I型IFN的基本构成和结构特征
我们首先对树鼩I型IFN家族的基本构成进行了预测和分析。结果表明, 在现有的9种I型IFN中, 树鼩具有较为明确的编码α、β、ε、κ、ω、δ的6种基因序列(表1)。其中, IFN-α包含5个(α1、α2、α4、α9、α22)亚型, 而δ、ε及α22有2~5条可能的编码序列(有待进一步证实), 但并未发现τ、ζ、ν及α3、α5-8、α10-17、α20-21的相应序列。从表1中亦可看出, 已克隆测序的IFN基因, 在各物种中差异较大, 也进一步表明,对包括树鼩在内的各物种进行IFN研究的空间较大。
为进一步研究树鼩IFN-α1与其它物种IFN-α1之间的差异, 我们对IFN-α1的氨基酸序列进行了多序列比对(图1)。与人(Homo sapiens)、小鼠(Mus muculus)、大鼠(Rattus norvegicus)、牛(Bos taurus)、马(Equus caballus)和猪(Sus scrofa)IFN-α1相同, 树鼩IFN-α1基因可编码24个氨基酸的信号肽, 表明其同样为分泌蛋白。根据Zou et al (2007)的报道, I型IFN可以分为含2个半胱氨酸和含4个半胱氨酸两个亚类。多序列比对分析显示,树鼩与人、小鼠、大鼠、猪的IFN-α1均属于后者。 此外, 所有参与比对的IFN-α1 C末端均具有一个保守的CAWE基序, 表明树鼩与其它哺乳动物I型IFN的基本特征一致。但是, 树鼩IFN-α1的第4个半胱氨酸在位置上与人、小鼠、大鼠和猪IFN-α1的有所不同; 树鼩、牛、马在人、小鼠、大鼠的第2个半胱氨酸位置上不包含半胱氨酸。 这些都有待进一步的基因克隆和测序加以验证。牛和马IFN-α1仅含3个半胱氨酸(图1), 与斑点叉尾鮰(channel catfish)相同(Robertsen, 2006), 提示IFN-α1在多物种进化速率上存在差异。同时, 这些半胱氨酸是否会配对形成二硫键以影响IFN-α1结构和功能;是否有除半胱氨酸之外的其他氨基酸或基序对IFN的结构−功能具有重要作用。这些均值得进一步研究。
我们进一步用Dsicovery Studio对树鼩的IFN-α1进行了结构模建, 所使用的模板为实验测定的人IFN-α1三维结构(PDB ID: 1RH2), 模板与靶序列间的一致性为87%。图2表明,树鼩和人IFN-α1空间结构相似, 可形成5个α螺旋(ABCDE), 具有典型的“螺旋形细胞因子”的特征(Conklin, 2004; Karpusas et al, 1997)。Uzé et al (1994, 1995)研究表明,螺旋结构在I型IFN与受体结合中具有决定性作用。其中, 第1和第3个螺旋(Aα、Cα螺旋)是与I型IFN受体1 (interferon alpha receptor 1, IFNAR1)的结合位点; 第4个螺旋(Dα螺旋)、AB螺旋和DE螺旋之间的连接环则是与I型IFN受体2 (interferon α receptor 2, IFNAR2)的结合位点。同时, 根据现有的序列信息(未列出), 我们亦可推知, 树鼩I型IFN其他亚型与受体结合的区域与人类相应区域结构均高度近似。此外, 与人IFN-α1相比, 树鼩IFN-α1中具有两个N糖基化位点, 结合糖基化在蛋白质中的重要作用(Lis & Sharon, 1993), 推测其增强了树鼩IFN-α1与其受体的结合能力。
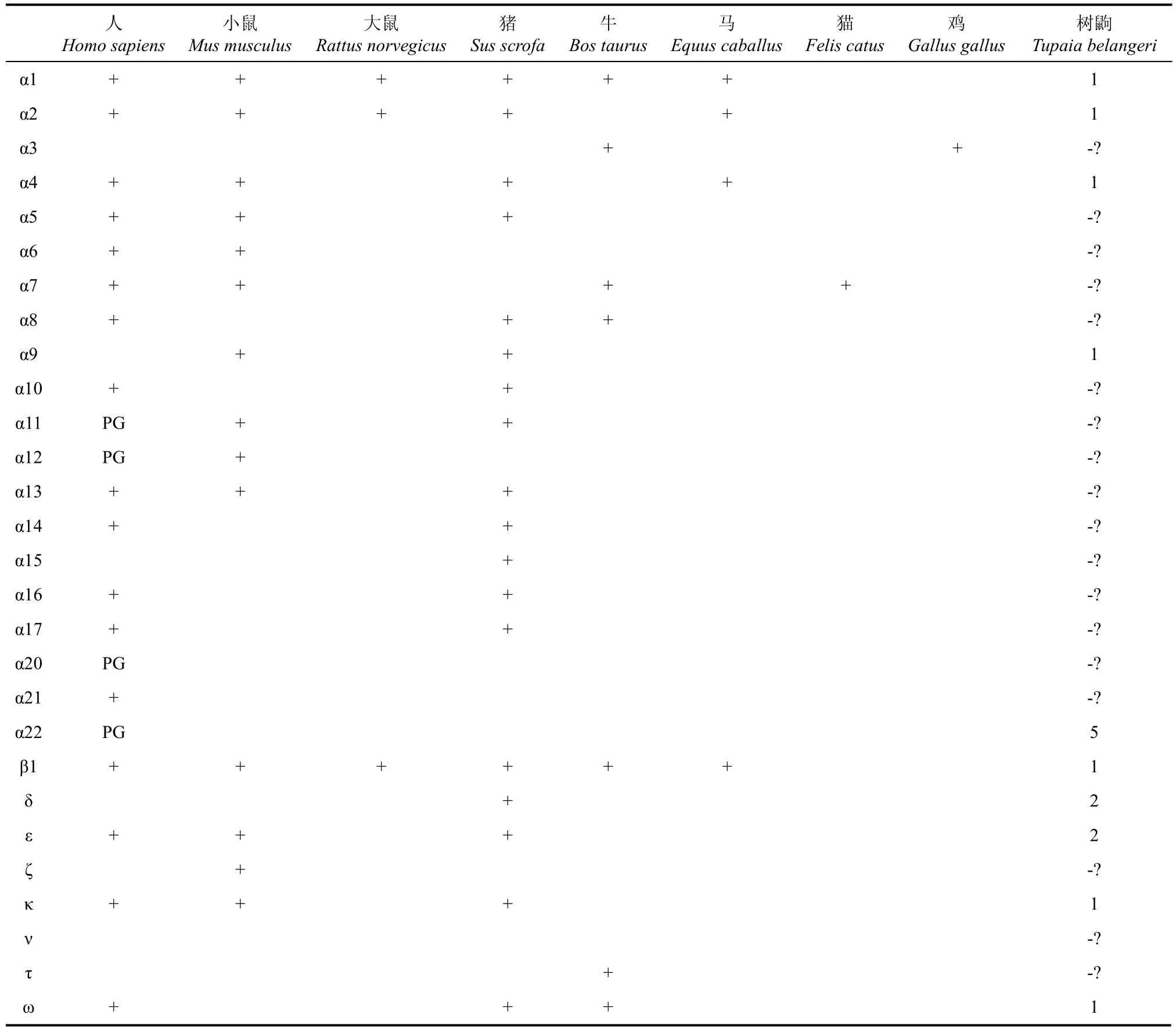
表1 NCBI中几个物种I型IFN及预测得到的树鼩I型IFNTab.1 Type I IFNs of several species in NCBI and predicted type I IFNs of Tupaia belangeri
2.3 树鼩II型IFN的基本构成和特征分析
运用上述分析方法, 我们预测得出两段与人II型IFN相近似的树鼩IFN-γ序列, 相似度分别为68%和63%。参照各物种IFN-γ的基因结构, 亦可推知, 这两段编码序列分别来自树鼩IFN-γ的第1和第4个外显子。
根据现有信息, 我们所得到的仅为2个较小的片段, 共82个氨基酸, 未能预测到中间约84个氨基酸的编码序列。这也说明现有基因组序列的局限性和不完整性,但氨基酸序列同源性和物种亲缘分析显示(表2), 除黑猩猩外, 人与树鼩的序列相似度最高, 达到了68%。这一结果显示了树鼩IFN-γ的存在, 同时也为我们克隆IFN-γ全长基因及进一步的功能分析提供了必要的线索。
2.4 树鼩III型IFN的基本构成和特征分析

图1 树鼩、人、小鼠、大鼠、牛、马、猪IFN-α1氨基酸多序列比对Fig.1 Alignment of amino acid sequences of tree shrew IFN-α1 with those from other mammals
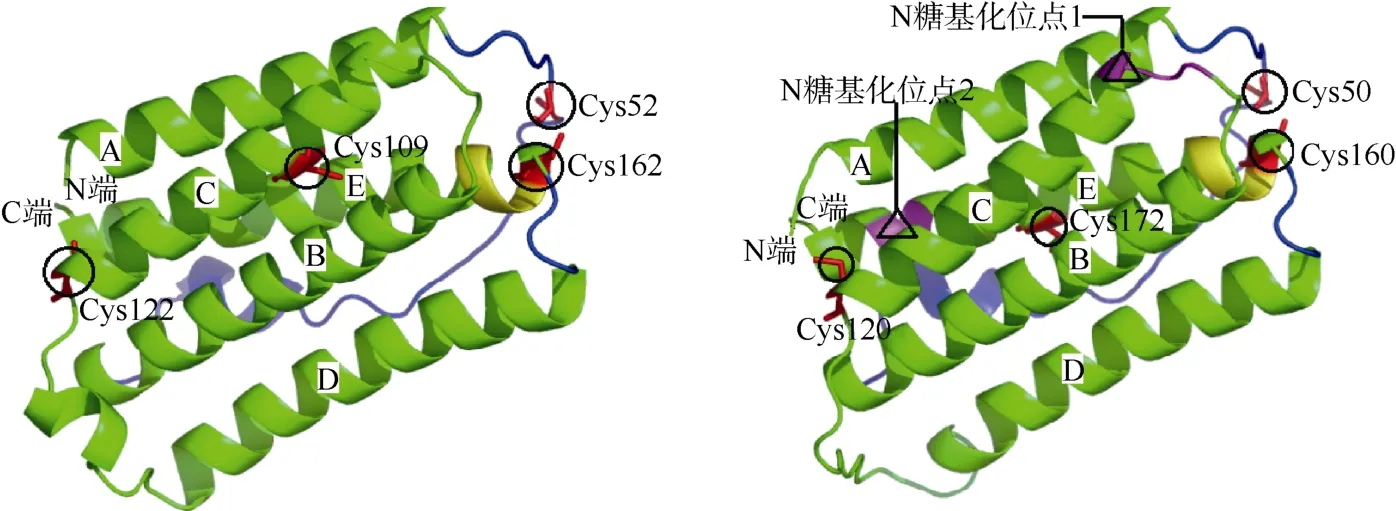
图2 人(左)、树鼩(右)IFN-α1分子蛋白质三维结构比较Fig.2 Comparison between the IFN-α1 protein three-dimensional structures of human (left) and tree shrew (right)

图 3 树鼩、人IFN-γ序列比对Fig.3 Amino acid sequence alignment of tree shrew and human IFN-γ
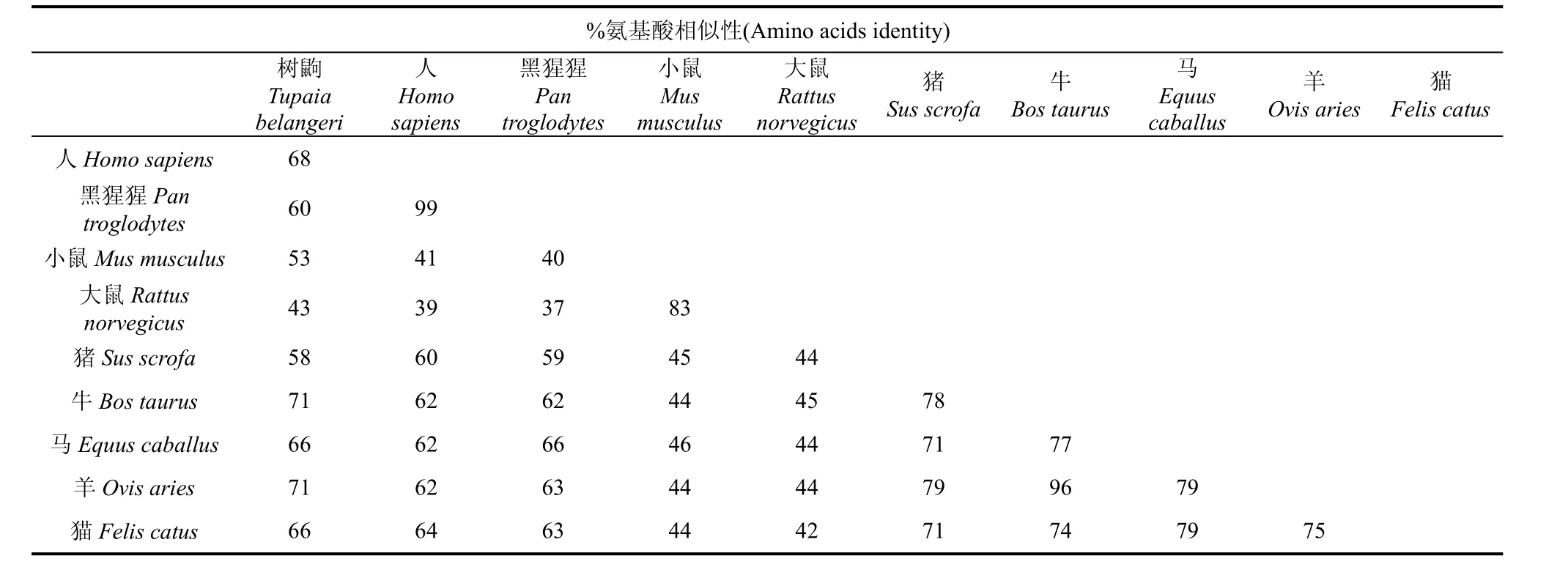
表2 物种间IFN-γ在氨基酸水平上相似性百分比Tab.2 Percentage of amino acid sequence similarity of IFN-γ between Homo sapiens, Chimpanzee, Sus scrofa, Mus musculus, Rattus norvegicus, Bos taurus, Equus caballus, Ovis aries, Felis catus, Canis lupus and Tupaia belangeri
自2003年发现III型IFN以来(Kotenko et al, 2003; Sheppard et al, 2003),在人、小鼠、蝙蝠、鱼、爪蟾、鸡等物种中已有研究报道(表3), 但各物种相应的基因结构较为复杂。人含有3个III型IFN基因, 分别为IFN-λ1(IL-29)、IFN-λ2(IL-28A)和IFN-λ3(IL-28B), 定位于19号染色体。小鼠第7号染色体上具有2个III型IFN编码基因(IFN-λ2、IFN-λ3)和1个假基因(IFN-λ1)(Lasfar et al, 2006)。蝙蝠可有效编码IFN-λ1和IFN-λ2, 而与人同源的IFN-λ3为假基因。此外, 蝙蝠IFN-λ1基因的第2内含子长达4 kb, 与人和其他哺乳动物对应区域1.1 kb的长度差异较大(Zhou et al, 2011)。值得注意的是, 鸡仅有1个III型IFN有效编码基因, 位于7号染色体(Karpala et al, 2008), 而爪蟾则有4个(IFN-λ1、IFN-λ2、IFN-λ3和IFN-λ5)有效编码基因和1个是一个假基因(IFN-λ4)(Qi et al, 2010)。因此,参照多物种数据系统研究树鼩IFN-λs基因结构和蛋白功能对充分理解树鼩III型IFN具有重要意义。
我们的预测发现, 在树鼩全基因组中有2条序列与人IFN-λs具有较高同源性。其中, 1条序列具有从起始密码子到终止密码子的全长编码框, 与人IFN-λ2和IFN-λ3均具有82%的序列相似性, 因此,暂命名为IFN-λ2/3。据此预测结果我们已将此585 bp的基因克隆并双向测序。

表3 目前已克隆的几个物种III型IFN及预测所得树鼩III型IFNTab.3 Type III IFNs of several species which have been cloned and those predicted type III IFNs in Tupai belangeri
就基因结构而言, III型IFN与I型IFN仅有1个外显子的特点明显不同, 它们均由5个(IFN-λ1、IFN-λ3)或6个外显子(IFN-λ2)构成(Uzé & Monneron, 2007)。利用Fancy GENE v1.4(Rambaldi & Ciccarelli, 2009), 我们构建了树鼩IFN-λ2/3与人IFN-λs内含子-外显子模式图(图4), 显示树鼩IFN-λ含有5个外显子和4个内含子结构, 具III型IFN的典型特征, 且其内含子−外显子的大小与人IFN-λ2基本一致。
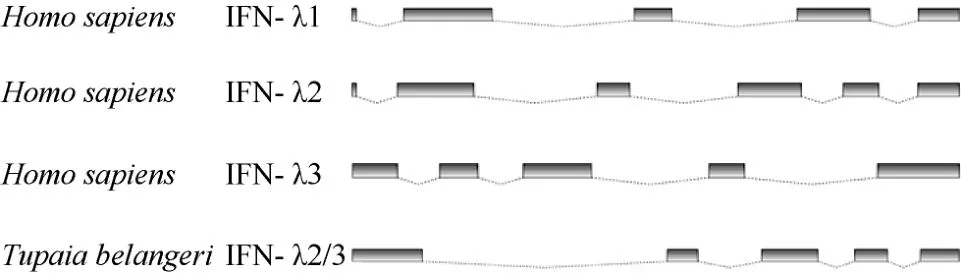
图4 人IFN-λ与树鼩IFN-λ2/λ3内含子−外显子结构比较Fig.4 Comparison of the intron-exon structure of tree shrew IFN-λ2/3 with those of human IFN-λs genes
以人IFN-λ2/3结构(PDB ID: 3HHC)为模板(模板与靶序列间的序列一致性为82%), 用Discovery Studio对树鼩IFN-λ2/3的蛋白三维结构进行了分子模建。结果显示(图5), 树鼩IFN-λ2/3结构(图5C)在整体上与人IFN-λ2/3结构(图5B)相似, 而与人IFN-λ1(图5A)差别很大。此外, 树鼩与人IFN-λs都由多个α螺旋构成, 具有典型的II型细胞因子家族蛋白结构特点(Zdanov, 2010)。人IFN-λ1具有N糖基化位点, 而IFN-λ2/3不具有相类似的糖基化位点(Dellgren et al, 2009), 结合糖基化在维持蛋白质与蛋白质之间结合中的作用(Lis & Sharon, 1993),推测IFN-λ1比IFN-λ2/3与受体结合能力更强, 从而起抗病毒中决定性作用。

图5 人IFN-λ1(A)、人IFN-λ2/3(B)和树鼩IFN-λ2/3(C)蛋白质结构模型Fig.5 Protein structural models of human IFN-λ1(A), IFN-λ2/3(B) and tree shrew IFN-λ2/3(C)
综上所述, 借助于现有的树鼩全基因组序列信息, 我们首次对树鼩IFN家族成员进行了系统挖掘, 并对其结构−功能进行了初步探讨,为进一步发现、验证树鼩IFN家族的其他成员以及相关功能研究奠定了基础。
Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ. 1990. Basic local alignment search tool[J]. J Mol Biol,215(3): 403-410.
Ank N, West H, Bartholdy C, Eriksson K, Thomsen AR, Paludan SR. 2006. Lambda interferon (IFN-λ), a type III IFN, is induced by viruses and IFNs and displays potent antiviral activity against select virus infections in vivo[J]. J Virol,80(9): 4501-4509.
Brand S, Beigel F, Olszak T, Zitzmann K, Eichhorst ST, Otte JM, Diebold J, Diepolder H, Adler B, Auernhammer CJ, Goke B, Dambacher J. 2005. IL-28A and IL-29 mediate antiproliferative and antiviral signals in intestinal epithelial cells and murine CMV infection increases colonic IL-28A expression[J]. Am J Physiol Gastrointest Liver Physiol,289(5): G960-G968.
Burge CB, Karlin S. 1998. Finding the genes in genomic DNA[J]. Curr Opin Struc Biol,8(3): 346-354.
Cao J, Yang EB, Su JJ, Li Y, Chow P. 2003. The tree shrews: adjuncts and alternatives to primates as models for biomedical research[J]. J Med Primatology,32(3): 123-130.
Conklin D. 2004. Recognition of the helical cytokine fold[J]. J Comput Biol, 11(6): 1189-1200.
Dellgren C, Gad HH, Hamming OJ, Melchjorsen J, Hartmann R. 2009. Human interferon-λ3 is a potent member of the type III interferon family[J]. Genes Immun,10(2): 125-131.
Fox BA, Sheppard PO, O'Hara PJ. 2009. The role of genomic data in thediscovery, annotation and evolutionary interpretation of the interferon-lambda family[J]. PLoS One,4(3): e4933.
Isaacs A, Lindenmann J. 1987. Virus interference. I. The interferon[J]. J Interferon Res,7(5): 429-438.
Karpala AJ, Morris KR, Broadway MM, McWaters PGD, O'Neil TE, Goossens KE, Lowenthal JW, Bean AGD. 2008. Molecular cloning, expression, and characterization of chicken IFN -λ[J]. J Interferon Cytokine Res,28(6): 341-350.
Karpusas M, Nolte M, Benton CB, Meier W, Lipscomb WN, Goelz S. 1997. The crystal structure of human interferonβat 2.2-Å resolution[J]. Proc Natl Acad Sci USA,94(22): 11813-11818.
Kotenko SV, Gallagher G, Baurin VV, Lewis-Antes A, Shen ML, Shah NK, Langer JA, Sheikh F, Dickensheets H, Donnelly RP. 2003. IFN-λs mediate antiviral protection through a distinct class II cytokine receptor complex[J]. Nat Immunol,4(1): 69-77.
Lasfar A, Lewis-Antes A, Smirnov SV, Anantha S, Abushahba W, Tian B, Reuhl K, Dickensheets H, Sheikh F, Donnelly RP, Raveche E, Kotenko SV. 2006. Characterization of the mouse IFN-λligand-receptor system: IFN-λs exhibit antitumor activity against B16 melanoma[J]. Cancer Res,66(8): 4468-4477.
Lindblad-Toh K, Garber M, Zuk O, Lin MF, Parker BJ, Washietl S, Kheradpour P, Ernst J, Jordan G, Mauceli E, Ward LD, Lowe CB, Holloway AK, Clamp M, Gnerre S, Alföldi J, Beal K, Chang J,Clawson H, Cuff J, Di Palma F, Fitzgerald S, Flicek P, Guttman M, Hubisz MJ, Jaffe DB, Jungreis I, Kent WJ, Kostka D, Lara M, Martins AL, Massingham T, Moltke I, Raney BJ, Rasmussen MD, Robinson J, Stark A, Vilella AJ, Wen JY, Xie XH, Zody MC, Baldwin J, Bloom T, Chin CW, Heiman D, Nicol R, Nusbaum C, Young S, Wilkinson J, Worley KC, Kovar CL, Muzny DM, Gibbs RA, Cree A, Dihn HH, Fowler G, Jhangiani S, Joshi V, Lee S, Lewis LR, Nazareth LV, Okwuonu G, Santibanez J, Warren WC, Mardis ER, Weinstock GM, Wilson RK, Delehaunty K, Dooling D, Fronik C, Fulton L, Fulton B, Graves T, Minx P, Sodergren E, Birney E, Margulies EH, Herrero J, Green ED, Haussler D, Siepel A, Goldman N, Pollard KS, Pedersen JS, Lander ES, Kellis M. 2011. A high-resolution map of human evolutionary constraint using 29 mammals[J]. Nature,478(7370): 476-482.
Lis H, Sharon N. 1993. Protein glycosylation. Structural and functional aspects[J]. Eur J Biochem,218(1): 1-27.
Notredame C, Higgins DG, Heringa J. 2000. T-Coffee: a novel method for fast and accurate multiple sequence alignment[J]. J Mol Biol,302(1): 205-217.
Novacek MJ. 1992. Mammalian phylogeny: shaking the tree[J]. Nature,356(6365): 121-125.
Österlund P, Veckman V, Sirén J, Klucher KM, Hiscott J, Matikainen S, Julkunen I. 2005. Gene expression and antiviral activity of alpha/beta interferons and interleukin-29 in virus-infected human myeloid dendritic cells[J]. J Virol,79(15): 9608-9617.
Qi ZT, Nie P, Secombes CJ, Zou J. 2010. Intron-containing type I and type III IFN coexist in amphibians: refuting the concept that a retroposition event gave rise to type I IFNs[J]. J Immunol,184(9): 5038-5046.
Rambaldi D, Ciccarelli FD. 2009. FancyGene: dynamic visualization of gene structures and protein domain architectures on genomic loci[J]. Bioinformatics,25(17): 2281-2282
Robertsen B. 2006. The interferon system of teleost fish[J]. Fish Shellfish Immunol, 20(2): 172-191.
Samarajiwa SA, Forster S, Auchettl K, Hertzog PJ. 2009. INTERFEROME: the database of interferon regulated genes[J]. Nucleic Acids Res,37(Database issue): D852-D857.
Schwartz S, Kent WJ, Smit A, Zhang Z, Baertsch R, Hardison RC, Haussler D, Miller W. 2003. Human-mouse alignments with BLASTZ[J]. Genome Res,13(1): 103-107.
Schwartz S, Zhang Z, Frazer KA, Smit A, Riemer C, Bouck J, Gibbs R,Hardison R, Miller W. 2000. PipMaker-a web server for aligning two genomic DNA sequences[J]. Genome Res,10(4): 577-586.
Sheppard P, Kindsvogel W, Xu W, Henderson K, Schlutsmeyer S, Whitmore TE, Kuestner R, Garrigues U, Birks C, Roraback J, Ostrander C, Dong D, Shin J, Presnell S, Fox B, Haldeman B, Cooper E, Taft D, Gilbert T, Grant FJ, Tackett M, Krivan W, McKnight G, Clegg C, Foster D, Klucher KM. 2003. IL-28, IL-29 and their class II cytokine receptor IL-28R[J]. Nat Immunol,4(1): 63-68.
Su JJ, Yan QR, Gan YQ, Zhou DN, Huang DR, Huang GH 1987. Study on the HBV infection of adult Tupaia[J]. Zhonghua Binglixue Zazhi, (16): 103-105.
Taniguchi T, Fujii-Kuriyama Y, Muramatsu M. 1980a. Molecular cloning of human interferon cDNA[J]. Proc Natl Acad Sci USA,77(7): 4003-4006.
Taniguchi T, Mantei N, Schwarzstein M, Nagata S, Muramatsu M, Weissmann C. 1980b. Human leukocyte and fibroblast interferons are structurally related[J]. Nature,285(5766): 547-549.
Uzé G, Di Marco S, Mouchel-Vielh E, Monneron D, Bandu MT, Horisberger MA, Dorques A, Lutfalla G, Mogensen KE. 1994. domains of interaction betweenα-interferon and its receptor components[J]. J Mol Biol,243(2): 245-257.
Uzé G, Lutfalla G, Mogensen KE. 1995.α-interferon andβinterferon and their receptor and their friends and relations[J]. J Interferon Cytokine Res,15(1): 3-26.
Uzé G, Monneron D. 2007. IL-28 and IL-29: newcomers to the interferon family[J]. Biochimie,89(6-7): 729-734.
Waddell PJ, Kishino H, Ota R. 2001. A phylogenetic foundation for comparative mammalian genomics[J]. Genome Inform,12: 141-154.
Walter E, Keist R, Niederost B, Pult I, Blum HE. 1996. Hepatitis B virus infection of tupaia hepatocytes in vitro and in vivo[J]. Hepatology,24(1): 1-5.
Zdanov A. 2010. Structural analysis of cytokines comprising the IL-10 family[J]. Cytokine Growth Factor Rev,21(5): 325-330.
Zhou P, Cowled C, Todd S, Crameri G, Virtue ER, Marsh GA, Klein R, Shi Z, Wang LF, Baker ML. 2011. Type III IFNs in pteropid bats: differential expression patterns provide evidence for distinct roles in antiviral immunity[J]. J Immunol,186(5): 3138-3147.
Zou J, Tafalla C, Truckle J, Secombes CJ. 2007. Identification of a second group of type I IFNs in fish sheds light on IFN evolution in vertebrates[J]. J Immunol,179(6): 3859-3871.
Genome-wide prediction of interferon family members of tree shrew and their molecular characteristics analysis
LI Ming-Li1,2, TIAN Wei-Wei1,3, GAO Yue-Dong4,5, GUO Yan1, HUANG Jing-Fei4,5, ZHANG Hua-Tang1,*
(1. Key Laboratory of Animal Models and Human Disease Mechanisms, Kunming Institute of Zoology, the Chinese Academy of Sciences, Kunming 650223, China; 2. Graduate School of the Chinese Academy of Sciences, Beijing 100049, China; 3. College of Biotechnology, Southwest University, Beibei 400715, China; 4. Kuming Biological Diversity Regional Center of Large Apparatus and Equipment, Chinese Academy of Sciences, Kunming 650223, China; 5. State Key laboratory of Genetic Resources and Evolution, Kunming Institute of Zoology, Chinese Academy of Sciences, Kunming 650223, China)
Interferons (IFNs) represent proteins with antiviral activities that are secreted from cells in response to a variety of stimuli. In addition to antiviral, antibacterial and anti-parasitic host-defense functions they are now also recognized as crucial regulators of cell proliferation, differentiation, survival and death as well as activators of specialized cell functions particularly in the immune system and play important roles in infectious and inflammatory diseases, autoimmunity and cancer. Tree shrews (Tupaia belangeri) were found to be susceptible to several human viruses and therefore are widely regarded as good models for analyzing mechanism of human diseases. In this report, we have forecasted the interferon family members of tree shrew from its genome mainly using the methods like Blast (whole genome shotgun sequence) and gene prediction. Our data show that tree shrew interferon system includes: type I IFN: α (five subtypes), β, ω, κ, ε, δ; type II IFN: γ; type III IFN: λ1, λ2/3. Furthermore, the predicted structures of α and λ have similar character with those of other mammals. However, there are some differences in cysteine position and N-glycosylation numbers between human and Tree shrew IFNs. These results provide fundamental basis for further molecular cloning and function analysis of tree shrew IFNs in future.
Tree shrews; Interferon; Whole genome sequence alignment; Gene prediction
硕士研究生, E-mail: limingli@mail.kiz.ac.cn
Q959.832; Q951.3; Q347
A
0254-5853-(2012)01-0067-08
10.3724/SP.J.1141.2012.01067
2011-10-24;接受日期:2012-01-10
国家重点基础研究发展计划(“973”计划)分课题(2007CB512807);中国科学院知识创新工程重要方向项目(KSCX2-EW-R-12);中国科学院基础前沿研究专项项目(KSCX2-EW-J-23);中科院昆明动物研究所“动物模型与人类疾病机理重点实验室”开放课题
∗通信作者(Corresponding author),E-mail: zhanght@mail.kiz.ac.cn

