甜菜亚硝酸还原酶(NiR)基因的克隆与分析
曾彦达,石晓艳,马凤鸣
(东北农业大学农学院,哈尔滨 150030)
亚硝酸还原酶(Nitrite reductase,NiR)是植物硝态氮同化过程中一个十分重要的酶,其与硝酸还原酶(Nitrate reductase,NR)偶联完成NO3-的无机同化[1],该酶催化NO2-还原为NH4+,以NADPH为电子供体在白色体中完成,在硝酸盐降解生成铵这一过程中起到承前启后的作用。NiR是植物体中NO2-同化过程的控制酶,其基因与NR都受NO3-的调节表达有关[2];Faure等研究发现,在白化烟叶片中的NR和NiR转录同时积累,当低水平的NR活性恢复后,NiR的活性水平也恢复正常,并把这种表达过程中的NR与NiR的相互作用称为NR和NiR的偶联调节[3]。目前,与NR相比较,有关NiR研究尚少。
NiR基因结构的研究始于上个世纪80年代末期,Lahners等提出了玉米NiR分子结构[4]。继后,在菠菜、水稻、烟草、拟南芥等高等植物中已克隆了NiR的基因[5-7],在甜菜上鲜有报道而且研究并不详细。本研究克隆了甜菜亚硝酸还原酶基因,对其核苷酸和氨基酸序列以及蛋白结构进行了分析,有利于研究清楚甜菜这一特定作物编码NiR的基因及其表达和调控问题,可为植物营养与分子生理学填补基础性资料;为改造基因和基因操作奠定基础,尤其是研究由环境条件引起的mRNA和特异蛋白质差异变化,将对甜菜NiR基因操作表达及表达时环境条件影响的机制提供分子生物学的解释;为全面分析NR和NiR的偶联作用提供分子生物学理论基础,有助于研究制定更为合理的施肥措施,培育甜菜氮高效利用品种,降低施肥费用,提高种植甜菜的经济效益,改善和防止因过量施肥所导致硝酸盐和亚硝酸盐积累造成的生态污染。
1 材料与方法
1.1 材料
试验所用甜菜品种为甜研7号,种子在室温下浸种12 h,然后置于培养皿中25℃恒温培养24~48 h,移栽于培养钵中,待幼苗长至4~6片叶后,剪取完全伸展的幼苗叶片,提取总RNA和基因组DNA。
1.2 方法
1.2.1 总RNA的提取
参照Trizol的说明书方法提取总RNA,电泳后通过凝胶成像系统进行观察和拍照。选择完整性好,质量高的RNA进行下一步试验。
1.2.2 甜菜NiR基因cDNA全长的克隆
使用宝生物公司的RACE试剂盒克隆基因的末端序列。参照NCBI上甜菜NiR基因cDNA的部分序列(登陆号:AF173663),以及菠菜、大豆NiR序列保守区分别设计3′/5′特异引物。设计两条正向嵌套引物3′-RACE Outer SP和3′-RACE inner SP,3′-RACE Outer SP与试剂盒引物3′Outer primer进行第一轮PCR反应,产物稀释5倍作为模板,3′-RACE Inner SP 与3′Inner primer进行第二轮巢式PCR扩增。设计5′端特异反转录引物5′-RACE Outer AP 和5′-RACE Inner AP,5′-RACE Outer AP与5′Outer primer进行第一轮PCR反应,产物直接作为模板,5′-RACE Inner AP和5′Inner primer进行第二轮巢式扩增。将已知的中间序列与3′端和5′端序列拼接后得到基因全长。克隆甜菜NiR基因引物见表1,所有引物由上海生工生物工程有限公司合成。
PCR扩增的特异片段按照宝生物胶回收试剂盒的使用说明进行回收。回收的目的片段连接到pMD19-T载体(TaKaRa)。连接产物转化感受态大肠杆菌JM109(TaKaRa),进行蓝白斑筛选,挑取白色菌落,使用菌落PCR法确认载体中插入片段的长度大小;将白斑接种于LB液体培养基中,37℃振荡培养过夜。取阳性克隆菌液由上海生物工程公司测序。根据所得甜菜NiR基因的末端序列设计引物(见表1)进行PCR,克隆NiR基因cDNA序列全长。

表1 甜菜NiR基因特异引物Table 1 Primers for cloning of sugar beet NiR gene
1.2.3 甜菜NiR基因组DNA(gDNA)的克隆
依据NiRcDNA全长序列,设计引物,进行PCR扩增NiRgDNA的全长序列。PCR扩增条件:94℃ 1 min,94℃ 30 s,57℃ 1 min,72℃ 3 min,30个循环,72℃10 min,4℃保存。PCR扩增的特异片段按照宝生物胶回收试剂盒的使用说明进行回收。回收的目的片段连接到pMD19-T载体(TaKaRa)。连接产物转化感受态大肠杆菌JM109(TaKaRa),进行蓝白斑筛选,挑取白色菌落,使用菌落PCR法确认载体中插入片段的长度大小;将白斑接种于LB液体培养基中,37℃振荡培养过夜。取阳性克隆菌液由上海生工生物工程有限公司测序。
1.2.4 序列分析
测序得到的NiRcDNA序列利用NCBI网站提供的ORF Finder找出基因的读码框,然后利用瑞士生物信息学研究所提供的ProtParam等网站进行氨基酸残基数目、组成、蛋白质相对分子质量、理论等电点、平均疏水性及三维结构等参数在线分析[8-9]。
2 结果与分析
2.1 甜菜NiR cDNA序列的克隆
以甜菜总RNA为模板,根据甜菜NiRcDNA已知的中间片段设计了3′/5′RACE的引物,分别经过两轮巢式PCR扩增,得到了一条约1 640 bp和一条约437 bp左右的条带,与预期片段大小基本相符。将两片段克隆,测序后发现,克隆所得片段与中间片段都具有重叠区,且含有poly(A)结构。同时将中间片段和RACE产物的拼接序列设计特异引物cDNA全长SP和cDNA全长AP,进行PCR扩增得到序列对比拼接序列,发现二者相同。再将该序列进入NCBI比对,结果显示全序列与同为藜科植物的菠菜NiR序列相似度达到了83%,表明所克隆的片段正是甜菜NiR的3′末端和5′末端序列。结果表明,该cDNA全长为2 014 bp,含有1 830 bp的完整开放阅读框,编码599个氨基酸的蛋白质,登录号为HQ224499(见图1)。

图1 甜菜NiR cDNA扩增Fig.1 Amplification of sugar beet NiR cDNA
2.2 甜菜NiR gDNA序列的克隆
以甜菜总DNA为模板,根据已克隆甜菜NiRcDNA序列设计特异引物gDNA全长SP和gDNA全长AP,通过PCR扩增得到甜菜NiRgDNA编码序列,登录号为HQ419065,总长度为2 815 bp,含有4个外显子和有3个内含子,内含子长度分别为532、136、270 bp(见图2)。
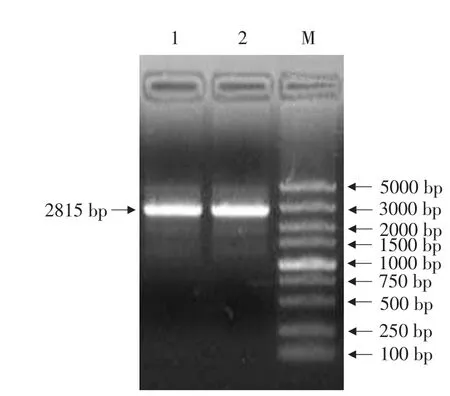
图2 甜菜NiR gDNA扩增Fig.2 Amplification of sugar beet NiR gDNA
2.3 序列分析
2.3.1 不同植物NiR蛋白分子进化树分析
在BLASTp分析的基础上[10-11],选择与其同源性较高的几条序列做N-J聚类分析,以期对Ty7 NiR蛋白和其他NiR蛋白的同源性做进一步分析。用Clustalx软件将NiR的氨基酸序列进行多重序列比对,然后将生成的系统发育树做N-J聚类分析(见图3)。
NiR进化树分析表明,甜菜与藜科植物NiR亲缘关系较近,与其他植物存在一定差别。在该进化树中同科植物表现比较近的亲缘关系,如豆科的大豆(Glycine max)、菜豆(Phaseolus vulgaris)、百脉根(Lotus japonicus)、茄科的烟草(Nicotiana tabacum)、甜椒(Capsicum annuum)、乔木科的小麦(Triticum aestivum)、水稻(Oryza sativa)、玉米(Zea mays)。推测NiR在进化过程中的变异不大,有较强的保守性。使用在线软件Pfam分析NiR蛋白的保守序列,结果表明,NiR序列在133~598位氨基酸之间保守性很强。

图3 甜菜NiR蛋白的分子进化树分析Fig.3 Phylogenetic analysis of sugar beet NiR proteins
2.3.2 甜菜NiR基因编码蛋白一级结构分析
甜菜NiR cDNA序列通过ORF finder分析,可得到完整的ORF位于核酸序列的32~1 831区域,编码蛋白包含599个氨基酸残基,分子质量为66.88 ku(Expasy protparam),理论等电点为7.21。酸性氨基酸残基总数(Asp+Glu)78个,碱性氨基酸残基总数(Arg+Lys)78个。分子式为C2938H4716N848O873S31,总原子数为9 406。根据所编码蛋白质的一级序列进行疏水性作图(见图4),疏水性较高的肽段处于蛋白质内部,反之,亲水性较高的肽段处于蛋白质分子的表面。
甜菜NiR的平均疏水性分析显示约有10个峰值,表明该肽段约有10个疏水区域。使用在线分析软件SignalP对甜菜NiR进行信号肽的分析,得知甜菜NiR无典型的信号肽分值,无信号肽,为非分泌型蛋白。

图4 甜菜NiR编码蛋白的疏水性分析Fig.4 Hydrophobicity profile of sugar beet NiR
2.3.3 甜菜NiR基因编码蛋白二级结构预测
利用ExPASy的GOR进行二级结构预测(见图5),h代表α螺旋,e代表延伸链,c代表无规则卷曲。α螺旋、延伸链、无规则卷曲分别占29.05%、21.04%和49.92%。
2.3.4 甜菜NiR基因编码蛋白立体结构预测
将所推测的甜菜NiR蛋白序列提交至ExPASy Geno3d,选取程序搜索到的第一个模板,以这一蛋白结构作为模板进行Geno3d建模。Geno3d建模如图6所示,其中包括24个α螺旋和57个转角。

图5 GOR法预测蛋白质的二级结构Fig.5 Secondary structures of protein predicted by GOR method

图6 用Geno3d预测的甜菜NiR三维结构Fig.6 Tertiary structure of NiR protein predicted by Geno3d
3 讨论
硝态氮(NO3-)是旱田作物可利用氮素的主要形式,NR是NO3-无机同化的限速酶,NiR是NO3-无机同化的控制酶。二者偶联完成NO3-的无机同化。与NR相比较,有关NiR研究尚少。本研究首次克隆了甜菜亚硝酸还原酶基因,有助于后续研究NiR与NR的偶联调节,及偶联作用下与甜菜蔗糖代谢积累的相互关系,可为种质资源鉴定、基因操作培育氮高效利用品种提供分子生物学理论依据;可从分子水平上阐明外界因子的调节作用,提高氮的转化效率,为制定施肥措施服务;同时对防止与改善农业施肥中硝酸盐及其转化过程中给地下水带来的严重污染具有重要科学意义。
4 结论
本研究以甜菜甜研7号为材料,通过简并PCR和RACE方法克隆了甜菜亚硝酸还原酶cDNA和gDNA基因;利用生物信息学手段进行NiR基因的结构及其理化特化分析。得到结论如下:
a.从甜菜中获得了NiR基因mRNA和gDNA序列,其中mRNA序列长度为2 014 bp(登陆号:HQ224499),可编码一个含有599个氨基酸,gDNA序列长度为2 815 bp(登陆号:HQ419065),包含4个外显子和3个内含子。
b.分析了NiR的理化特征,得到NiR的理论等电点为7.21,分子式为C2938H4716N848O873S31,蛋白质相对分子质量为66.88 ku,对NiR进行疏水性分析,显示该肽段有约10个疏水区域。
c.二级结构预测结果显示,NiR的α螺旋、延伸链、无规则卷曲分别占33.89%、12.85%和53.26%。三维结构预测结果显示,NiR蛋白包括24个α螺旋和57个转角。
d.构建了NiR的进化树,结果表明甜菜NiR与菠菜亲缘关系最近,在进化中相对保守。运用分析软件Pfam分析甜菜NiR氨基酸序列,显示甜菜NiR包含一个保守区域,存在于133~598位氨基酸之间。
e.运用在线分析软件SignalP对甜菜NiR进行信号肽的分析,得知甜菜NiR无典型的信号肽分值,无信号肽,为非分泌型蛋白。
[1] 王玉琴.内外生理条件下对小麦黄化幼苗叶片亚硝酸还原酶活力的影响[J].植物生理学通讯,1988(4):18-20.
[2] Takahashi M,Sasaki Y,Shoji I,et al.Nitrite reductase gene enrichment improves assimilation of NO2-inArabidopsis[J].Plant Physiology,2001,126:731-741.
[3] Faure J D,Vincentz M,Kronenberger J,et al.Co-regulated expression of nitrate and nitrite reductase[J].Plant J,1991(1):107-113.
[4] Lahners K,Kramer V,Back E,et al.Molecular cloning of complementary DNA encoding maize nitrite reductase[J].Plant Physiol,1988,88:741-746.
[5] Back E.Isolation of the spinach nitrite reductase gene promoter which confers nitrate inducibillity onGUSgene ecpression in transgenic tobacco[J].Plant Mol Biol,1991,17:9-18.
[6] Polcyn W,Lucinski R.Effect of N oxyanions on anaerobic induction of nitrite reductase in subcellular fraction ofBradyrhizobiumsp.(Lupinus)[J].Antonie van Leeuwenhoek,2009,95:159-164.
[7] Dose M M,Hirasawa M,Kleis-SanFrancisco S,et al.The ferredoxin-binding site of ferredoxin nitrite oxidoreductase differential chemical modification of the free enzyme and its comlex with ferredoxin[J].Plant Physiol,1997,114(3):1047-1053.
[8] 孙啸,陆祖宏.生物信息学基础[M].北京:清华大学出版社,2005.
[9] 钟扬,王莉,张亮.生物信息学[M].北京:高等教育出版社,2003.
[10] 张成岗,贺福初.生物信息学方法与实践[M].北京:科学出版社,2002.
[11] Baldi P,Chauvin Y,Hunkapiller T,et al.Hidden Markov models of biological primary sequence information[J].Proc Nat Acad Sci,1991(3):1059-1063,1994.

