阴离子交换晶胶层析分离质粒DNA
郭延涛,沈绍传,贠军贤,姚克俭
浙江工业大学化学工程与材料学院 绿色合成技术国家重点实验室培育基地,浙江 杭州 310032
质粒DNA (pDNA) 可作为DNA载体用于基因治疗。相比于病毒载体来说,作为非病毒的pDNA安全方便[1]。近几年,基因治疗和 DNA疫苗技术的快速发展推动了对 pDNA载体的大量需求[2],至2011年,全球开展的1 714项基因治疗临床医学试验中,有 18.7% (320项) 是以超螺旋质粒 DNA (scpDNA) 为外源基因运载载体[3]。获得pDNA一般要经过质粒转化、菌体培养扩增质粒、菌体裂解和分离纯化等几个步骤。从工程菌裂解液中分离 pDNA的实验室方法有些要用到动物源性酶,如煮沸法要用的溶菌酶和RNA酶,有些要用到有毒试剂,如碱裂解法要用苯酚和氯仿,溴化乙锭-氯化铯密度梯度离心法要用溴化乙锭和氯化铯,不适于药用pDNA的大规模制备。柱层析是生物分离中应用最广泛的方法之一,层析介质是其关键。针对蛋白质或酶等生物分子吸附分离而设计的常规商业化介质,孔径在纳米量级范围,适于分子量较小的目标物的扩散吸附。但是,对于pDNA等生物大分子物质,难以在这些介质内部扩散吸附,吸附容量和分离效率低。因此,研究适于pDNA等生物大分子的层析新技术和新介质,具有重要意义。近些年来,依据pDNA的各种特性,研究人员提出了不同的层析分离纯化方法,如离子交换色谱法[4-5]、疏水作用色谱法[6-7]、尺寸排阻色谱法[8]等。
超大孔连续床晶胶是一种新型的分离介质,内部有几微米至上百微米的相互连通的超大孔隙,可实现生物大分子的快速分离纯化[9-16]。离子交换型晶胶介质因其具有良好的选择性、传质阻力小和吸附分离迅速等优点,在分离纯化生物大分子的应用中受到广泛关注[5,9,12,14-16]。Wang等制得经二乙氨基乙基葡聚糖 (DEAE-Dextran)改性的弱阴离子交换聚甲基丙烯酸酯基连续床,并应用于蛋白混合物的分离[17]。本研究中,通过溶剂结晶致孔法制备聚丙烯酰胺基晶胶基质,然后接枝DEAE-Dextran得到阴离子交换型晶胶介质。用该晶胶介质从大肠杆菌Escherichia coli裂解液中分离pDNA。分离过程中未加入动物源性酶,也没有加入苯酚、氯仿和溴化乙锭等有毒试剂,使操作过程以及获得的pDNA产物更加安全。
1 材料与方法
1.1 材料
1.1.1 试剂
DEAE-Dextran (相对分子质量500 000)、烯丙基缩水甘油醚 (AGE) 和 N,N'-亚甲基双丙烯酰胺 (MBAAm) 购自 Sigma-Aldrich公司 (美国);丙烯酰胺 (AAm)、考马斯亮蓝G-250和琼脂糖购自 Biobasic公司 (加拿大);质粒 pUC19购自Fermentas公司 (加拿大);E. coli DH5α菌株、过硫酸铵 (APS)、LB培养基、N,N,N',N'-四甲基乙二胺 (TEMED)、十二烷基硫酸钠 (SDS)和乙二胺四乙酸 (EDTA) 购自生工生物工程(上海) 有限公司;其余试剂均为分析纯。
1.1.2 主要仪器
BT1-100E恒流泵、HD-21-88紫外检测仪 (上海琪特分析仪器有限公司),Sub-Cell GT system核酸电泳仪 (Bio-Rad,美国),Tanon-3500全自动数码凝胶图像分析系统 (上海天能科技有限公司),THCD-01低温恒温槽 (宁波天恒仪器厂),Ultrospec 3300 pro紫外/可见光分光光度计 (GE Healthcare)。
1.2 方法
1.2.1 晶胶基质的制备
将单体AAm、配基AGE和交联剂MBAAm配成反应液,在直径为10 mm的玻璃层析柱内,变温冷冻条件下聚合获得晶胶基质,具体方法见文献[18]。AAm、AGE和MBAAm的总质量浓度为7%,其中AAm与AGE质量比为10/1,(AAm+AGE) 和MBAAm质量比为10/1,引发剂APS占AAm、AGE和MBAAm总质量的1.2%,加速剂TEMED占AAm、AGE和MBAAm总质量的0.5%。具体变温过程见文献[19]中的冷冻曲线B。
1.2.2 阴离子交换基团的接枝
将DEAE-Dextran溶于0.025 mol/L Na2HPO4-NaOH缓冲液 (pH 12) 中,配成质量分数为1%的溶液。先用恒流泵将 DEAE-Dextran溶液以1.6 mL/min的速度泵过晶胶基质至pH值恒定,然后在室温下,以0.4 mL/min的速度密封循环反应48 h。反应机理见文献[17]。
1.2.3 阴离子交换晶胶介质的性能测试
阴离子交换晶胶介质的性能测试方法见文献[18-19]。通过脉冲示踪法以3% (V/V) 丙酮为示踪剂获得停留时间分布 (RTD) 曲线,根据相应公式计算得到理论塔板数和理论塔板高度(HETP)。通过测定单位时间内流经晶胶介质的水流量和对应的水力压降,利用达西方程计算得到渗透率。以牛血清白蛋白为模型蛋白考察层析性能。通过扫描电镜观察孔隙结构。
1.2.4 pDNA的扩增及初步处理
E. coli DH5α用LB液体培养基在37 ℃、150 r/min下进行培养。菌体培养至OD600达0.5左右,进行质粒 pUC19转化,经含氨苄青霉素(Amp) 平板筛选后,在含有0.1 mg/mL Amp的LB液体培养基中再培养,在OD600达到2.0时收集菌体。采用比较常用的碱法进行裂解,具体步骤如下:1) 150 mL经pDNA转化的E. coli DH5α培养液,室温下12 000 r/min离心5 min,收集菌体;2) 将菌体重悬于 10 mL预冷的溶液Ⅰ(50 mmol/L葡萄糖,10 mmol/L EDTA,25 mmol/L Tris-HCl,pH 8) 中,剧烈振荡,室温放置3 min;3) 加入 20 mL新鲜配制的溶液Ⅱ (0.2 mol/L NaOH,1% SDS),颠倒数次混匀,冰上静置3 min;4) 加入15 mL预冷的溶液Ⅲ (3 mol/L乙酸钾,2 mol/L乙酸,pH 4.8),将离心管温和颠倒数次混匀,冰上静置20 min后,4 ℃、10 000 r/min离心10 min,所得的上清液用于层析。
1.2.5 阴离子交换晶胶介质层析分离pDNA
阴离子交换晶胶层析分离 pDNA的过程如下:1) 用20 mmol/L磷酸盐缓冲液 (PB) 平衡晶胶床柱;2) 将1.2.4中所得的上清液用PB稀释至5倍作为层析上样液,以1.6 mL/min上样至吸附饱和,然后用PB进行冲洗以除去游离在晶胶介质中的杂质;3) 分别用不同浓度的NaCl溶液(用PB配制) 依次洗脱;4) 用0.5 mol/L NaOH和大量的去离子水对晶胶介质进行再生。整个层析过程在波长为254 nm的紫外检测仪下检测并记录,在上样和冲洗阶段每60 s收集1管,在洗脱阶段每30 s收集1管。
1.2.6 分析方法
选取层析过程代表性样品用1%的琼脂糖凝胶进行电泳分析,电压80 V。0.05 mg/L溴化乙锭染色20 min后用Tanon-3500全自动数码凝胶图像分析系统在紫外灯下进行观察和图像拍摄。用Quantity One (v 4.6.2) 软件对pDNA的纯度和含量进行分析。洗脱液中的蛋白含量用考马斯亮蓝染色法测定[20]。
2 结果与分析
2.1 阴离子交换晶胶介质的基本性能
阴离子交换晶胶介质直径为10 mm,长度为7.5 cm。用脉冲示踪法所测得的该晶胶介质的RTD曲线如图1所示。由图可知,表观流速 (UL)从1 cm/min到12 cm/min范围内,RTD峰型对称性较好。根据 RTD数据计算得到晶胶介质的HETP,如图 2所示。晶胶介质的 HETP在0.069 cm到0.076 cm之间,说明此晶胶介质的传质分离性能较好。晶胶介质的压降与流速的关系如图 3所示,根据达西公式计算得到渗透率为2.03×10−12m2,流体流动阻力较小。晶胶介质对牛血清白蛋白的吸附容量达到1.1 g/L晶胶介质。通过扫描电镜观察到的晶胶孔隙结构如图 4所示,由图可知晶胶介质的孔隙结构较均匀,且连通性较好。因此,该阴离子交换型晶胶介质性能较好,适用于pDNA的层析分离。
2.2 pDNA的层析分离

图1 阴离子交换晶胶介质的RTDsFig. 1 Residence time distributions at various liquid velocities in the anion-exchange cryogel.
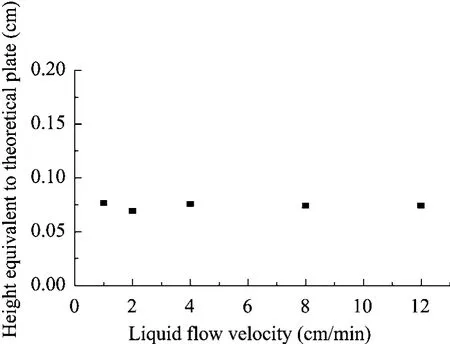
图2 阴离子交换晶胶介质的HETPFig. 2 Variation of height equivalent to theoretical plate with liquid velocity in the anion-exchange cryogel.
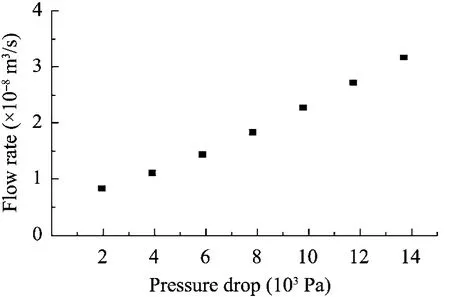
图3 阴离子交换晶胶介质压降与流速的关系Fig. 3 Flow rate vs. pressure drop in the anionexchange cryogel.

图4 阴离子交换晶胶介质孔隙结构的扫描电镜图片Fig. 4 Scanning electron microscope photograph of the anion-exchange cryogel.
通过在碱性条件下使晶胶基质上的环氧基团开环后接枝阴离子交换基团 DEAE-Dextran。由于DEAE-Dextran是长链聚合物,吸附位点多,如触手一样长在晶胶孔隙表面,可灵活地延伸到孔内料液流体中,有利于较大尺寸的pDNA的吸附,使晶胶介质有较大的吸附容量和较高的选择性。pDNA有scpDNA、开环pDNA (ocpDNA) 和线性pDNA (lnpDNA) 三种构型,其中scpDNA是有效的DNA载体[21],电泳迁移速率scpDNA大于lnpDNA和ocpDNA。E. coli经过溶液Ⅰ、Ⅱ和Ⅲ处理后,细胞被裂解,大部分蛋白被沉淀,经离心后,上清液中除了目标物scpDNA外,还含有基因组DNA (gDNA)、lnpDNA、ocpDNA、RNA、少量蛋白和细胞碎片等杂质,细胞碎片可直接通过晶胶介质流出,其他杂质均可能被吸附,需要优化层析条件以得到高纯度的scpDNA。
在pH 7.2下进行层析,分别用0.3、0.35、 0.4或0.5 mol/L NaCl进行第一步洗脱,用1 mol/L NaCl进行第二步洗脱。洗脱的scpDNA中均含有较多杂质,梯度洗脱没有将scpDNA与杂质分开。在pH 6.0、6.6或7.8条件下分别进行层析,发现在pH 6.0和7.8条件下层析得到的scpDNA均含有较多杂质,只有在pH 6.6条件下层析得到的scpDNA纯度较高。
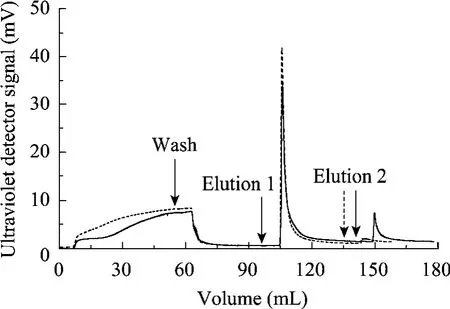
图5 不同洗脱条件下的层析过程Fig. 5 Chromatographic processes under different elution conditions. The cryogel column was equilibrated and washed with 20 mmol/L sodium phosphate buffer of pH 6.6. The liquid velocity was 1.6 mL/min. (solid line) Eluted with 0.4 mol/L NaCl (elution 1) followed by 1 mol/L NaCl (elution 2). (dotted line) Eluted with 0.5 mol/L NaCl (elution 1) followed by 1 mol/L NaCl (elution 2).
在pH 6.6时进行层析,用0.4或0.5 mol/L NaCl进行第一步洗脱,用1 mol/L NaCl进行第二步洗脱,用0.5 mol/L NaOH再生床柱,层析曲线见图5,电泳结果见图6。从图5中可知,第一步洗脱峰较高,第二步洗脱峰较低,特别是用0.5 mol/L NaCl进行第一步洗脱时,1 mol/L NaCl洗脱已几乎看不到洗脱峰,说明用 0.5 mol/L NaCl一步洗脱足以将目标物洗脱下来。从图 6中可知,洗脱物中杂质较少,晶胶柱对scpDNA取得了较理想的分离效果。上样液中scpDNA浓度较低,电泳图中未见明显条带 (图6A,泳道2),在洗脱峰中有明显的scpDNA条带,说明晶胶介质对scpDNA起到了较好的浓缩作用。在用0.4、1 mol/L NaCl洗脱时,0.4 mol/L NaCl洗脱得到的scpDNA杂质较少,纯度达到83.8% (图6A,泳道9~11),回收率为29.4%,1 mol/L NaCl洗脱只隐约见scpDNA一条条带,浓度较低 (图6A,泳道6~8);用0.5、1 mol/L NaCl洗脱时,0.5 mol/L NaCl洗脱得到的 scpDNA纯度达到 82.8% (图6B,泳道3~5),回收率为33.3%,而1 mol/L NaCl洗脱几乎看不到条带 (图6B,泳道6~8)。0.5 mol/L NaCl洗脱较0.4 mol/L NaCl洗脱得到的scpDNA纯度相差不多,但是scpDNA浓度有所提高,所以用0.5 mol/L NaCl一步洗脱更为合适,具体结果见表1。用0.5 mol/L NaOH可将残留在晶胶介质内的scpDNA和其他杂质一并洗脱下来使床柱再生 (图6A,泳道3~5,图6B,泳道9~10)。用考马斯亮蓝法对洗脱液进行检测,未检出蛋白。

图6 层析收集液的琼脂糖凝胶电泳图Fig. 6 Agarose gel electrophoresis images of chromatographic eluents. M: DNA marker (10 000, 8 000, 6 000, 5 000, 4 000, 3 500, 3 000, 2 500, 2 000, 1 500, 1 000, 750, 500, 250 bp). (A) 1: pUC19 DNA; 2: loading sample; 3−5: eluted with 0.5 mol/L NaOH; 6−8: eluted with 1 mol/L NaCl; 9−11: eluted with 0.4 mol/L NaCl. (B) 1: pUC19 DNA; 2: breakthrough; 3−5: eluted with 0.5 mol/L NaCl; 6−8: eluted with 1 mol/L NaCl; 9−10: eluted with 0.5 mol/L NaOH.
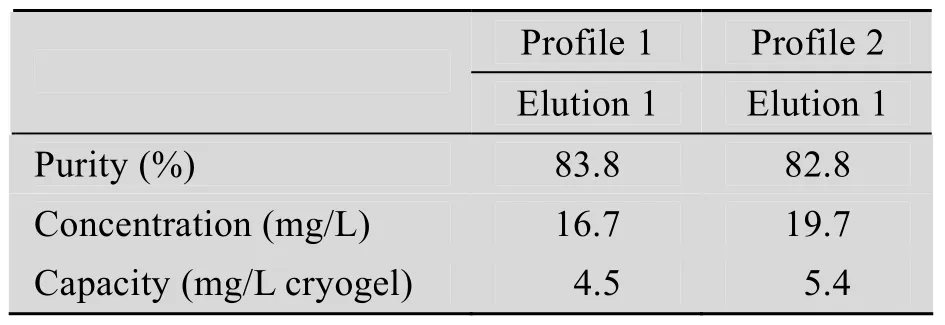
表1 pH 6.6下层析获得的scpDNA的纯度和浓度,以及晶胶的分离能力Table 1 The purity and concentration of scpDNA obtained by chromatography under pH 6.6, and the corresponding separation capacity of the cryogel
3 结论
利用自制的超大孔晶胶基质,成功接枝DEAE-Dextran得到阴离子交换晶胶介质。用该介质在pH 6.0、6.6、7.2或7.8条件下进行层析,从E. coli裂解液中分离pDNA,分别以0.3、0.35、0.4或0.5 mol/L NaCl进行第一步洗脱,1 mol/L NaCl进行第二步洗脱,最终找到了获得较高纯度scpDNA的合适层析条件。在pH 6.6条件下进行层析,用 0.5 mol/L NaCl一步洗脱得到的scpDNA纯度达到 82.8%,晶胶吸附量达到5.4 mg/L晶胶介质。整个分离过程迅速高效,没有使用动物源性酶,也没使用苯酚、氯仿和溴化乙锭等毒性试剂,使操作过程和所获得的pDNA更加安全。
[1] Williams JA, Carnes AE, Hodgson CP. Plasmid DNA vaccine vector design: Impact on efficacy, safety and upstream production. Biotechnol Adv, 2009, 27(4): 353−370.
[2] Ferreira GNM, Monteiro GA, Prazeres DMF, et al. Downstream processing of plasmid DNA for gene therapy and DNA vaccine applications. Trends Biotechnol, 2000, 18(9): 380−388.
[3] Gene Therapy Clinical Trials Worldwide [EB/OL]. [2011-06]. http://www.wiley.com/legacy/wileychi/ genmed /clinical/.
[4] Hanora A, Savina I, Plieva FM, et al. Direct capture of plasmid DNA from non-clarified bacterial lysate using polycation-grafted monoliths. J Biotechnol, 2006, 123(3): 343−355.
[5] Zhong L, Scharer J, Moo-Young M, et al. Potential application of hydrogel-based strong anionexchange membrane for plasmid DNA purification. J Chromatogr B, 2011, 879(9/10): 564−572.
[6] Li Y, Dong XY, Sun Y. High-speed chromatographic purification of plasmid DNA with a customized biporous hydrophobic adsorbent. Biochem Eng J, 2005, 27(1): 33−39.
[7] Freitas SS, Santos JAL, Prazeres DMF. Plasmid purification by hydrophobic interaction chromatography using sodium citrate in the mobile phase. Sep Purif Technol, 2009, 65(1): 95−104.
[8] Latulippe DR, Zydney AL. Size exclusion chromatography of plasmid DNA isoforms. J Chromatogr A, 2009, 1216(35): 6295−6302.
[9] Arvidsson P, Plieva FM, Savina IN, et al. Chromatography of microbial cells using continuous supermacroporous affinity and ion-exchange columns. J Chromatogr A, 2002, 977(1): 27−38.
[10] Plieva FM, Savina IN, Deraz S, et al. Characterization of supermacroporous monolithic polyacrylamide based matrices designed for chromatography of bioparticles. J Chromatogr B, 2004, 807(1): 129−137.
[11] Yao KJ, Yun JX, Shen SC, et al. Characterization of a novel continuous supermacroporous monolithic cryogel embedded with nanoparticles for protein chromatography. J Chromatogr A, 2006, 1109(1): 103−110.
[12] Yao KJ, Yun JX, Shen SC, et al. In-situ graft-polymerization preparation of cationexchange supermacroporous cryogel with sulfo groups in glass columns. J Chromatogr A, 2007, 1157(1/2): 246−251.
[13] Yao KJ, Shen SC, Yun JX, et al. Protein adsorption in supermacroporous cryogels with embedded nanoparticles. Biochem Eng J, 2007, 36(2): 139−146.
[14] Yun JX, Shen SC, Chen F, et al. One-step isolation of adenosine triphosphate from crude fermentation broth of Saccharomyces cerevisiae by anionexchange chromatography using supermacroporous cryogel. J Chromatogr B, 2007, 860(1): 57−62.
[15] Shen ZM, Shen SC, Yun JX, et al. Purification of adenosine triphosphate from crude fermentation broth by one-step isolation using a large-size weak anion-exchange supermacroporous cryogel. J Chem Eng Chinese Univ, 2009, 23(5): 768−773.沈哲明, 沈绍传, 贠军贤, 等. 大尺寸弱阴离子交换晶胶层析一步法分离 ATP的实验研究. 高校化学工程学报, 2009, 23(5): 768−773.
[16] Yan LD, Shen SC, Yun JX, et al. Isolation of lysozyme from chicken egg white using polyacrylamide-based cation-exchange cryogel. Chinese J Chem Eng, 2011, 19(5): 876−880.
[17] Wang MY, Xu J, Zhou X, et al. Modification with DEAE-dextran, an alternative way to prepare anion-exchange monolithic column with lower pressure drop. Biochem Eng J, 2007, 34(1): 76−81. [18] He XJ, Yao KJ, Shen SC, et al. Freezing characteristics of acrylamide-based aqueous solution used for the preparation of supermacroporous cryogels via cryo-copolymerization. Chem Eng Sci, 2007, 62(5): 1334−1342.
[19] Yao KJ, Shen SC, Yun JX, et al. Preparation of polyacrylamide-based supermacroporous monolithic cryogel beds under freezing-temperature variation conditions. Chem Eng Sci, 2006, 61(20): 6701−6708. [20] Bradford MM. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Anal Biochem, 1976, 72(1/2): 248−254.
[21] Diogo MM, Queiroz JA, Prazeres DMF. Chromatography of plasmid DNA. J Chromatogr A, 2005, 1069(1): 3−22.