酿酒酵母细胞表面展示技术在燃料乙醇生产中的应用及研究进展
杨非,曹萌,金怡,杨秀山,田沈
首都师范大学生命科学学院,北京 100048
随着化石能源的日益枯竭,可再生清洁能源生物乙醇的开发和利用受到了人们的广泛关注。燃料乙醇可以从蔗糖、淀粉和纤维素生物质中获得,给料的类型决定了发酵过程的复杂程度,方法技术也从简单的转化糖发酵发展到利用多种策略来转化木质纤维素生产乙醇[1]。在本领域的诸多最新研究趋势中,开发具有同步糖化发酵能力的优良酵母菌株是减少乙醇生产成本的关键因素。近年来,随着生物乙醇的生产由第一代向第二代成功过渡,大量的研究集中在利用酵母细胞表面展示技术来开发能够转化多种生物质的生物乙醇生产菌株[2-3]。
酵母细胞表面展示技术是一种真核蛋白表达系统。其基本原理是将外源靶蛋白基因与特定的载体基因序列融合后导入酵母细胞,融合蛋白诱导表达后,信号肽引导融合蛋白向细胞外分泌,外源蛋白的展示主要是借助于 GPI锚的作用,GPI锚定蛋白的C端包含疏水多肽,当细胞蛋白被合成,前体蛋白通过羧基末端的疏水序列锚定在内质网膜上,其余蛋白则位于内质网腔中。在极短的时间内,疏水序列在转酰氨基酶的作用下ω位点裂开同时被GPI锚置换,并被运输到高尔基体再通过分泌途径分泌至细胞膜外。在蛋白水解酶作用下,分泌信号序列被切除,细胞蛋白被磷脂酰肌醇特异性磷脂酶 C从细胞膜上释放并以 GPI锚定形式与葡聚糖共价相连被转运至细胞壁外。图1展示的是通过α-凝集素系统转运目的蛋白到细胞表面的分泌途径。酵母表面展示技术具有表达偏差小,展示的融合蛋白相对分子质量范围大,分子数量多,筛选、纯化、活性测定方便等优点。

图1 基于α-agglutinin的蛋白运输示意图[4]Fig. 1 Illustration of the general transportation of α-agglutinin to the cell surface[4].
目前,最常见的酵母表面展示系统包括:凝集素展示表达和絮凝素展示表达[5]。如图2所示,α-凝集素展示表达系统 (图 2A) 是将外源基因与酵母编码 α-凝集素 C端编码序列连接后插入质粒载体的分泌信号肽 (Secretion signal,SS) 下游,融合蛋白诱导表达后,信号肽引导嵌合蛋白向细胞外分泌,由于融合蛋白C-末端存在含GPI锚定信号 (GPI anchor attachment signal) 的凝集素多肽序列,可将蛋白锚定在酵母细胞壁上,从而将蛋白分子展示表达在酵母细胞表面;a-凝集素展示系统 (图 2B) 与 α-凝集素展示系统不同的是其具有由二硫键连接的Aga1和Aga2两个亚基,目的蛋白通过与Aga2形成融合蛋白,再通过Aga1末端的GPI结构使目的蛋白表达于细胞表面;絮凝素展示表达系统利用絮凝素基因(flo1p) 与外源基因融合,并将融合蛋白展示表达在细胞表面,此系统又包括两个子系统:GPI锚定系统 (图2C) 和絮凝结构域 (Flo1p flocculation functional domain) 锚定系统 (图2D)。GPI锚定系统是利用絮凝素 Flo1p的 C末端含有的 GPI结构锚定外源蛋白,此系统与 α-凝集素展示相似;絮凝结构域锚定系统是利用Flo1p的中间絮凝功能结构域与外源蛋白融合,通过絮凝功能结构域识别酵母细胞壁中的甘露聚糖链并通过非共价作用诱导细胞粘附、聚集成可逆性絮状物。进而使外源蛋白表达于酵母细胞表面。
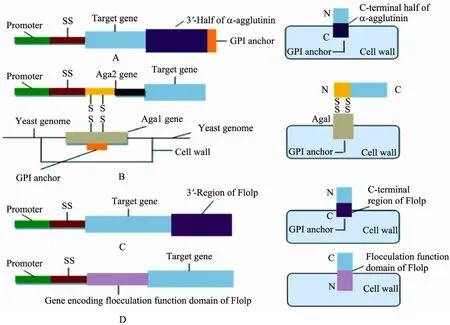
图2 酵母表面展示系统:(A) α-凝集素 (B) a-凝集素 (C) Flo1p C末端 (D) Flo1p N末端絮凝功能区[2,6]Fig. 2 Yeast surface display systems using α-agglutinin (A), a-agglutinin (B), C-terminus region of Flo1p (C), and N-terminus flocculation function domain of Flo1p (D)[2,6].
近年来,S. cerevisiae细胞表面展示技术迅速发展并在多个领域得到应用,展现出广阔的发展前景。可通过使外源蛋白固定化于 S. cerevisiae细胞表面,从而生产微生物细胞表面蛋白,包括淀粉酶、纤维素酶等在生物乙醇生产中起关键作用的酶蛋白,从而使酵母表面展示技术在生物乙醇生产应用中不断发展与完善,本文着重介绍了近年来酵母细胞表面工程在生物乙醇菌种开发中的应用与研究进展。
1 酵母细胞表面技术展示淀粉分解酶
以淀粉类生物质为主要成分的蔗糖和糖蜜可以实现大规模的乙醇生产,然而,以淀粉为原料生产生物乙醇要经历多步反应而且生产成本偏高。导致成本偏高的原因主要有两个:一是由于S. cerevisiae不能直接利用淀粉类物质,因此在发酵过程中需要额外添加大量的葡糖淀粉酶(Glucoamylase) 和 α-淀粉酶 (α-Amylase);二是为获得较高的乙醇产量,首先要将淀粉在140 ℃~180 ℃的高温下进行糊化,而这也就增加了能耗。为此,大量的研究集中尝试在 S. cerevisiae细胞表面展示淀粉分解酶,从而实现以淀粉类物质为底物的大规模乙醇生产,并取得了一定的成果。
利用细胞表面展示技术构建能利用淀粉的全细胞催化剂,可将编码淀粉酶的外源基因导入酵母细胞内使其能够分泌淀粉酶[7]。已经构建好的质粒pGA11带有编码米根霉Rhizopus oryzae的葡糖淀粉酶基因,转化子细胞能够在以1%可溶性淀粉为唯一碳源的培养基中迅速生长并产生乙醇,葡糖淀粉酶活力稳定至少100 h,而且所表达的酶,其酶学性质与天然酶相当[8]。
Murai等[9]是研究细胞表面表达淀粉酶工作的先驱,他们创造性地通过在S. cerevisiae细胞表面展示淀粉酶来开发重组酿酒酵母的研究策略,构建了带有S. cerevisiae MT8-1的GAPDH启动子及含有信号肽和glucoamylase/α-凝集素融合基因的载体,glucoamylase来自于R. oryzae,能有效切割淀粉 α-1,4和 α-1,6糖苷键。随后,Kondo[10]等在具絮凝功能特性的酵母细胞表面展示了葡糖淀粉酶,他们的工作表明,展示在细胞表面的葡糖淀粉酶对酵母的正常生长及乙醇产生无任何影响;厌氧发酵150 h后乙醇浓度达到20~30 g/L。但Kondo等构建的展示葡糖淀粉酶的重组絮凝酵母在发酵300 h的情况下还能保持较高的乙醇产率 (0.6~0.7 g/(L·h))[10],同时,发酵培养基中低浓度的葡萄糖对减少污染也极为有利。展现了在S. cerevisiae细胞表面展示淀粉酶的优越性。
在批式补料发酵过程中,由于缺少溶解酶——α-淀粉酶,单一展示葡糖淀粉酶的酵母菌株在发酵过程中会有不溶性淀粉的积累。为克服这一问题,Shigechi等[11-14]构建了共展示来自R. oryzae的glucoamylase和来自嗜热脂肪芽胞杆菌Bacillus stearothermophilus的α-amylase基因,并以可溶性淀粉为底物,进行补料批式发酵。结果表明,将淀粉颗粒处理到与酵母细胞大小相近时可极大程度地提高乙醇产率,发酵100 h后乙醇浓度达到60 g/L,而且共展示葡糖淀粉酶和α-淀粉酶菌株的絮凝能力与只展示葡糖淀粉酶的菌株基本相同,发酵过程中酵母絮凝能力并不发生改变。在后续的研究中,Shigechi等[14]构建了含有葡糖淀粉酶结合结构域 (SBD) 突变子的酵母展示株系,结果发现,带有突变子 SBD葡萄淀粉酶的菌株较野生型菌株的淀粉降解能力提高1.4倍。
Shigechi等[13]又利用低温糊化的淀粉 (80 ℃)取代可溶性淀粉作为唯一碳源进行乙醇发酵,结果表明:共展示葡糖淀粉酶和α-淀粉酶的菌株与单展示葡糖淀粉酶的菌株相比,乙醇产率明显增加,尤其是共展示菌能更快速地产生乙醇且无延滞期,说明随机水解淀粉α-1,4糖苷键的α-淀粉酶在淀粉水解过程中起关键作用。比较共展示两种淀粉酶的菌株在低温和高温糊化情况下的发酵效率,无论是最大乙醇浓度、乙醇产率还是底物消耗率,两种发酵系统都基本相同,乙醇产量为0.5 g/g,相当于理论值的97.2%。更好地说明了共展示两种淀粉酶菌株的优势。
从牛链球菌Streptococcus bovis中分离的淀粉酶为生料淀粉的高效水解提供了新的研究思路[15]。Shigechi等[12]分别以α-凝集素和絮凝素为基础,共展示来自R. oryzae的葡糖淀粉酶和来自S. bovis148的α-淀粉酶,并以生料淀粉为底物进行发酵试验,结果表明α-淀粉酶活性依赖于锚定蛋白,而葡糖淀粉酶活性则独立于锚定蛋白;对于α-淀粉酶活性,以絮凝素系统为基础构建的菌株要比以凝集素为基础构建的菌株酶活性高 40倍。目前已经报道了一些α-淀粉酶具有结合生料淀粉的能力,且降解淀粉的区域位于C-末端[16]。以 α-凝集素为基础的共展示菌能以生料淀粉为原料生产乙醇[17-19]。另一方面,基于α-凝集素和絮凝素Flo1p共展示的葡糖淀粉酶和α-淀粉酶在生料发酵过程中不需要额外添加淀粉酶,在发酵过程中,淀粉浓度迅速下降,72 h乙醇浓度达到61.8 g/L,乙醇产量为0.44 g/L,相当于理论值的86.5%。
利用酵母细胞表面展示技术从最初的单展示葡糖淀粉酶到共展示葡糖淀粉酶和α-淀粉酶,均取得了较好的成果,为利用淀粉类物质实现大规模乙醇生产提供了新的思路。不过随着生物乙醇生产技术的不断发展,人们开始着眼于利用木质纤维素生产燃料乙醇,从而避免了第一代生物乙醇存在的与人争粮的缺陷。
2 酵母细胞表面技术与第二代生物乙醇
由于第一代生物乙醇的生产存在着与人争粮、与粮争地的缺陷,发展第二代生物燃料是人们面对能源、环境与粮食问题所采取的积极措施。利用丰富廉价的可再生木质纤维素生产乙醇,是规模化发展车用燃料乙醇的基础。木质纤维素是地球上最丰富的可再生资源,占地球总生物量的40%左右,高效、廉价地利用木质纤维素转化为如燃料乙醇等化工产品,是由石油基向生物基经济社会过渡的基础。
为此大量的研究开始转向展示纤维素分解酶、木糖代谢酶等,从而实现新的突破与尝试,为实现第二代生物燃料的开发与利用提供了一定的理论依据。
2.1 展示纤维素分解酶
在纤维素乙醇生产经历的糖化步骤形成葡萄糖的过程中,S. cerevisiae不能直接利用纤维素类物质,因此在S. cerevisiae中稳定表达纤维素酶是降低生产成本的有效方法。基于α-凝集素C端构建的pCMC11含有来自棘孢曲霉Aspergillus aculeatus的内切羧甲基纤维素酶 (CMCase) 编码基因,Murai 等[20]通过一系列方法证明了cmcase基因成功地锚定在细胞表面,内切羧甲基纤维素酶活性稳定至少120 h。当pCMC11和含有β-bgl1的载体 (pBG211) 共表达时,可以快速地将纤维低聚糖转化为葡萄糖,从而显著地提高了利用纤维素的效率。目前已报道了很多在S. cerevisiae中表达多种纤维素酶基因和能够糖化可溶性纤维寡糖的重组酿酒酵母[21-22]。
Fujita等同样基于 α-凝集素系统在S. cerevisiae细胞表面展示来自丝状真菌里氏木霉 Trichoderma reesei的内切葡聚糖酶Ⅱ的融合蛋白,酵母菌株能明显提高对大麦β-葡聚糖的水解能力;随后,构建了共表达内切葡聚糖酶Ⅱ和来自A. aculeatus的β-葡糖苷酶Ⅰ的酵母株系,这种酵母能在以大麦 β-葡聚糖为唯一碳源的培养基上生长,在50 h内将β-葡聚糖发酵生成乙醇,乙醇产量为0.48 g/g,相当于理论值的93.3%,且发酵培养基中的 β-葡聚糖能在酵母细胞表面连续水解为葡萄糖,在胞内酶的作用下,立即被利用,转变为乙醇。同时,酵母细胞的酶活测定结果和流式细胞计数分析结果表明:共展示菌中内切葡聚糖酶Ⅱ和 β-葡糖苷酶Ⅰ分子的数量大于单展示菌中内切葡聚糖酶Ⅱ或 β-葡糖苷酶Ⅰ分子数的总和[23]。其后,Fujita研究小组[24]将来自T. reesei的纤维二糖水解酶Ⅱ也增加到展示系统中,从而使酵母细胞真正具有水解无定形纤维素能力,同步糖化发酵生成乙醇,结果显示共展示内切葡聚糖酶Ⅱ和纤维二糖水解酶Ⅱ的菌株比只展示内切葡聚糖酶Ⅱ的菌株对无定形纤维素的水解能力高,且主要产物是纤维二糖,单展示纤维二糖水解酶Ⅱ的菌株主要产物是少量的纤维二糖,单展示内切葡聚糖酶Ⅱ的菌株主要产物是纤维二糖、纤维三糖,而共展示 β-葡糖苷酶Ⅰ和内切葡聚糖酶Ⅱ的菌株无乙醇产生。在以葡萄糖为碳源的培养基里,虽然在40 h内所有无定形纤维素不能被完全利用,但其发酵能力也达到10 g/L纤维素可产生3 g/L的酒精。即每克碳水化合物可产 0.45 g/g酒精,相当于理论值的88.5%,可他们并没有报道在纤维素为唯一碳源的条件下该酵母的发酵能力。Shuhei等[25]以共展示三种纤维素酶的重组酵母 (内切葡聚糖酶Ⅱ、纤维二糖水解酶Ⅱ、β-葡糖苷酶Ⅰ) 和共展示两种纤维素酶的菌株 (内切葡聚糖酶Ⅱ和纤维二糖水解酶Ⅱ) 的发酵效果进行比较,结果表明前者的乙醇产量明显优于后者。
姚淑敏等[26]也成功地将来自裂殖酵母的β-葡糖苷酶Ⅰ以其活性形式展示在S. cerevisiae细胞表面,诱导表达后的 β-葡糖苷酶Ⅰ酶活测定显示,重组酵母菌株诱导培养 12 h后有酶活,到48 h酶活达到最高,最适作用的pH为7.0,最适作用温度为45 ℃。
目前,利用酵母细胞表面技术展示纤维素酶,无论单展示还是共展示,均取得了一定的进展,虽然现阶段局限于实验室阶段,但相比较而言,在底物利用上已经取得了一定的突破。
2.2 展示木糖代谢途径关键酶
S. cerevisiae是传统的乙醇发酵生产菌株,由于没有专一性转化木糖为木酮糖的酶系,不能很好地利用木糖。其中木糖的跨膜运输被认为是工程菌利用木糖的障碍之一。S. cerevisiae没有专一性木糖转运蛋白,木糖运输是由非专一性的已糖转运蛋白完成,而此类蛋白对于木糖的亲和力远低于葡萄糖[27]。目前也有部分研究集中于展示木糖代谢途径的关键酶,以填补S. cerevisiae不能利用木糖的缺陷,更好地利用底物生产燃料乙醇,提高底物利用率。Satoshi Katahira等[28]将来自 T. reesei的木聚糖酶和米曲霉 Aspergillus oryzae的β-木糖苷酶成功地展示在S. cerevisiae细胞表面,改变了S. cerevisiae的代谢途径,实现了酵母直接发酵来源于白桦木的木聚糖生产乙醇的工艺。结果表明这两个酶缺一不可,只有在两者同时存在的情况下才能将木聚糖转化为木糖。因为S. cerevisiae不能直接利用木糖生产酒精,所以要向S. cerevisiae导入来自毕赤酵母的木糖还原酶和木糖醇脱氢酶以及 S. cerevisiae本身的木酮糖激酶,它们共同作用再将木糖转化成木酮糖-5磷酸,从而使S. cerevisiae能利用木糖;该重组酵母经 62 h发酵后产生酒精浓度为7.1 g/L,表明每克碳水化合物可产0.30 g酒精,是理论值的58%。虽然该重组酵母产酒精能力明显不足,但该实验为今后开发利用半纤维素指出了一条出路。侯进等[29]基于α-凝集素系统,将来源于嗜热细菌Thermus thermophilus的木糖异构酶基因xylA,插入到S. cerevisiae蔗糖酶信号肽序列与α-凝集素的C端编码序列之间,形成融合表达框,构建重组质粒 pSY-xy222,转化S. cerevisiae H158。含重组质粒的菌株H158-SXI木糖异构酶活性测定表明,细胞壁上酶活测定值为1.53 U,木糖异构酶在S. cerevisiae细胞壁上得到活性表达。木糖葡萄糖共发酵结果显示,重组菌株木糖利用率较出发菌株提高了17.8%。陈璐菲等[30]利用S. cerevisiae表面展示系统,将来源于C. tropicalis的木糖还原酶基因xyll嵌入带有 His-Tag的 S. cerevisiaea-凝集素展示载体pICAS-His,构建重组质粒pICASHis-Ctxyll,并转化宿主菌S. cerevisiae MT8-l,通过流式细胞仪快速检测和筛选,得到重组菌株MT8-1/pICAS-His-Ctxyll。将重组酵母用于葡萄糖 (15 g/L) 和木糖 (5 g/L) 的混合糖发酵实验。结果表明,重组酵母MT8-1/pICAS-His-Ctxyll细胞具有良好的生长和产酶特性,同时能转化木糖产生木糖醇,将2.5 g/L木糖转化生成2.5 g/L木糖醇,转化率达98.7%。
通过展示木糖代谢途径关键酶的重组酿酒酵母在发酵木糖生产乙醇的过程中,木糖异构酶虽有一定的活性表达,但利用木糖产乙醇的木糖利用率并没有太大的改善,虽然此部分研究工作依旧面临着很多的问题与挑战,但这些工作为解决S. cerevisiae代谢工程菌株的木糖利用问题做出了新的研究与尝试。
2.3 基于展示技术的纤维素乙醇统合加工
为了降低生产成本,可以将纤维素糖化和发酵等几步反应过程合并在一步反应里完成,即统合生物加工 (CBP)。CBP要求某种“超级”微生物具有既能水解纤维素又能利用水解纤维素产生的糖类发酵产酒精,可自然界中没有哪个已知微生物能满足 CBP这样的要求。改造现有微生物也就成为唯一的选择[31]。在S. cerevisiae细胞表面分别表达T. reesei的两种编码纤维二糖水解酶:纤维二糖水解酶Ⅰ和纤维二糖水解酶Ⅱ的纤维素结合结构域 (CBD) 基因,同时也构建了两个串联排列的CBDI和CBDⅡ融合基因,在细胞表面表达两种CBD。表达CBDⅠ和CBDⅡ的两种酵母展示细胞的CBD具有相似的结合纤维素的能力,而表达CBDⅠ和CBDⅡ融合蛋白的酵母细胞比单表达CBD的酵母细胞具有更强的纤维素结合力。展示酵母细胞的结合率主要依赖于展示在细胞表面的CBD分子多少。有CBD和突变CBD酵母展示细胞可用于分析CBD与纤维素的结合机制。而构建共展示CBD和靶酶的酵母细胞,将可以实现在纤维素滤器上构建连续的或多步酶反应的生物反应器系统。
纤维小体是近年来研究的较热门的一个话题,根据细胞表面展示技术把纤维小体展示在产酒精微生物表面,在纤维素乙醇的生物转化中具有重要的价值,纤维小体实际上是一个微型而高效的纤维素降解机器。现在已经有学者提出了通过人工设计并通过工程手段改造天然纤维小体的新思路[32]。Tsai研究小组[33]利用来自厌氧菌的纤维素酶构建了迷你纤维小体并重组到酵母细胞中。他们首先选择热纤梭菌 Clostridium thermocellum 的内切葡聚糖酶和解纤维梭菌Clostridium cellulolyticum的内切葡聚糖酶和外切葡聚糖酶构建迷你纤维小体,这个纤维小体仍有水解纤维素的协调能力。而一旦将 C. cellulolyticum的内切葡聚糖酶换成β-葡糖苷酶,就可使新的纤维小体具有了纤维素酶的三大酶组分,其水解纤维素能力不但得到提高而且还能发酵产酒精。在以无定形纤维素 (PASC) 为唯一碳源的培养基里,该酵母发酵能力为每10 g/L的PASC可产生3.5 g/L的酒精。即每克碳水化合物可产0.49 g酒精,相当于理论值的95%。而同样是以PASC为唯一碳源,Den Haan研究小组[34]因没有采用纤维小体形式改造酵母,其效果大打折扣。Den Haan等尝试将来自T. reesei的egl1和扣囊复膜孢酵母 Saccharomycopsis flbuligera的bgl1整合到S. cerevisiae中表达。虽然都检测到有酶活并能利用PASC发酵生产酒精,但其发酵酒精的浓度只为 1 g/L。从这两个小组的实验结果可以明显看出纤维小体的优势。
开发应用这种集纤维素水解和发酵于一身的共展示酵母株系,对发酵工业的发展具有重要的意义。这些实验证明了利用共展示纤维素降解酶的S. cerevisiae发酵生产乙醇的高效性和同步糖化发酵法的可行性。表1中列出了部分研究中已构建的共展示纤维素酶的菌株或质粒。
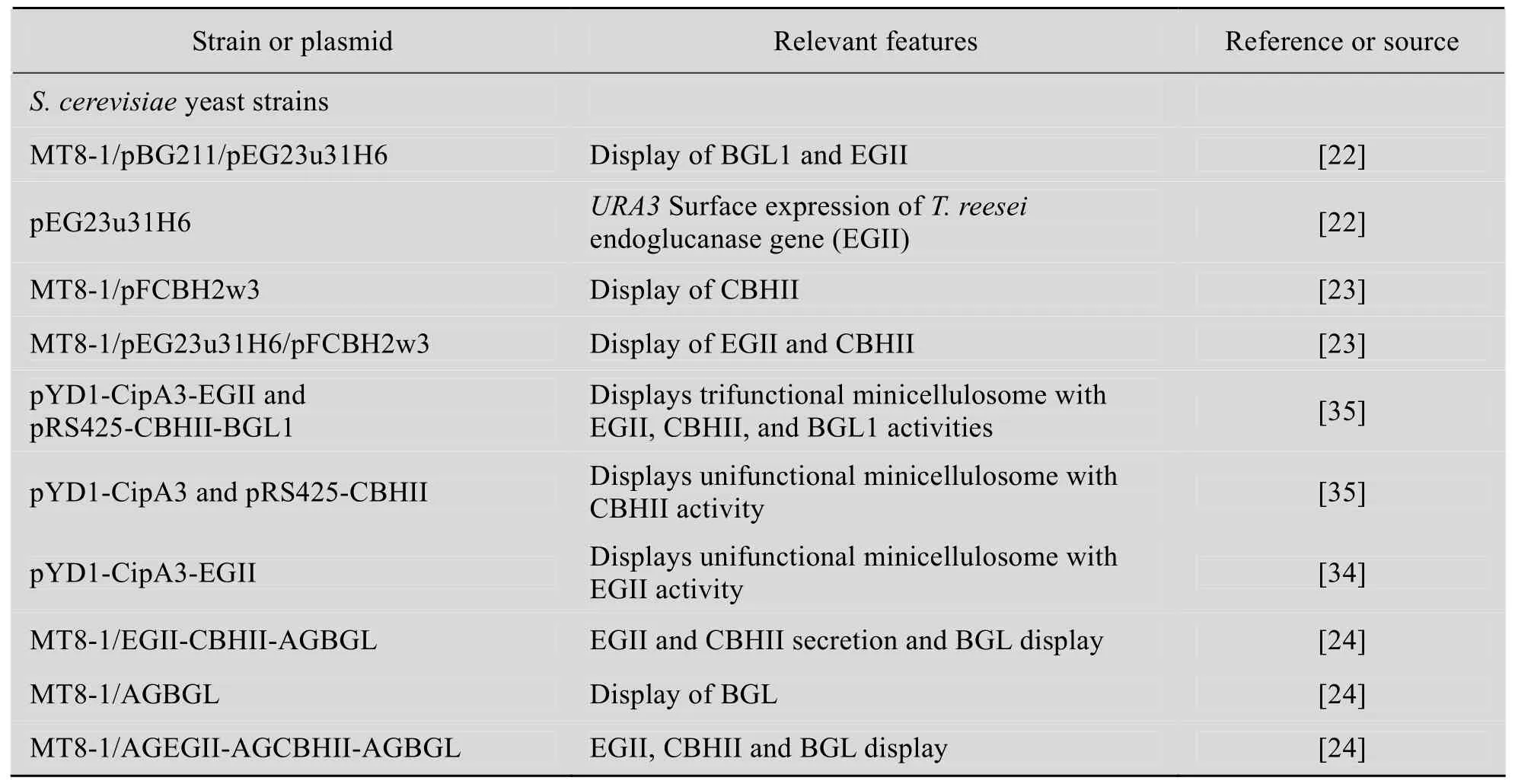
表1 共展示纤维素酶的菌株与质粒Table 1 Characteristics of strains and plasmids for codisplaying cellulolytic enzyme
本实验室一直致力于纤维素乙醇生产菌种的构建,并且已经成功地构建了基于专利菌株S. cerevisiaeY5的共代谢葡萄糖和木糖的重组酿酒酵母[36]。在以往的研究中,利用野生型工业菌株S. cerevisiae进行纤维素降解酶的展示研究还未见报道。在前期的工作中,本实验室利用了野生型二倍体工业菌株S. cerevisiaeY5,基于a-凝集素构建了一个能够有效地接受外源基因,并可以在宿主细胞表面特异性表达的展示系统,并成功展示了来自A. aculeatus的β-葡糖苷酶Ⅰ,该展示菌株能够在以纤维二糖为唯一碳源的培养基中生长,首次实现了野生型二倍体菌株为宿主的纤维素酶展示。在今后的研究工作中,我们将来自丝状真菌与纤维素降解有关的酶类展示在酵母表面,并尝试在野生型二倍体工业菌株中实现多种纤维素酶的展示,以期进一步阐述纤维素的作用机理。实现细胞的自我固定化,并研究以纤维素为唯一碳源的底物利用情况,对比展示前后酶的活性变化,通过消除或减少添加纤维素酶才能完成的糖化步骤,努力实现全细胞催化纤维素乙醇的生物转化过程,以期大大地降低生物乙醇的生产成本。
3 展望
利用S. cerevisiae细胞表面技术展示纤维素降解酶的研究已取得一定的研究成果,该系统已经得到成功开发,但是在这一领域里仍有许多亟待解决的问题,例如:展示在细胞表面的纤维素酶的活性与游离酶相比有所下降;成功展示的纤维素酶稳定性有待提高;对于该技术的研究目前仅停留在实验室阶段,还未实现工业化等。今后,此领域的研究还需更多的发展和完善,包括开发更多的具固定化展示功能的细胞壁甘露糖蛋白;进一步提高现有展示的纤维素酶的稳定性、表达量及活性;建立更多的展示酶筛选技术;克服生产中扩大培养、乙醇抑制等难题。相信随着分子生物学和生物技术的飞速发展,该领域必将取得更大的进步,以酵母细胞表面展示系统为基础的纤维素乙醇工业将更大程度地服务于人类社会。
[1] Sánchez ÓJ, Cardona CA. Trends in biotechnological production of fuel ethanol from different feedstocks. Biores Technol, 2008, 99(13): 5270−5295.
[2] Kondo A, Ueda M. Yeast cell-surface displayapplications of molecular display. Appl Microbiol Biotechnol, 2004, 64(1): 28−40.
[3] Ueda M, Tanaka A. Genetic immobilization of proteins on the yeast cell surface. Biotechnol Adv, 2000, 18(2): 121−140.
[4] Ueda M, Tanaka A. Cell surface engineering of yeast: construction of arming yeast with biocatalyst. J Biosci Bioeng, 2000, 90(2): 125−136.
[5] Ye B, Lin Y, Li SY. Development and applications in yeast cell surface display system. Ind Microbiol, 2007, 37(6): 53−58.叶波, 林影, 李双艳. 酵母细胞表面展示系统的研究进展及其应用. 工业微生物, 2007, 37(6): 53−58.
[6] Guo Q, Zhang W, Ruan H, et al. Cell surface display expression system of Saccharomyces cerevisiae and its applications. China Biotechnol, 2008, 28(12): 116–122.郭钦, 张伟, 阮晖, 等. 酿酒酵母表面展示表达系统及应用. 中国生物工程杂志, 2008, 28(12): 116–122.
[7] Murai T, Ueda M, Yamamura M, et al. Construction of a starch-utilizing yeast by cell surface engineering. Appl Environ Microbiol, 1997, 63(4): 1362−1366.
[8] Tubb RS. Amylolytic yeasts for commercial applications. Trends Biotechnol, 1986, 4(4): 98−104.
[9] Ueda M, Murai T, Shibasaki Y, et al. Molecular breeding of polysaccharide-utilizing yeast cells by surface engineering. Ann N Y Acad Sci, 1998, 864: 528−537.
[10] Kondo A, Shigechi H, Abe M, et al. High-level ethanol production from starch by a flocculent Saccharomyces cerevisiae strain displaying cell-surface glucoamylase. Appl Microbiol Biotechnol, 2002, 58(3): 291−296.
[11] Shigechi H, Koh J, Fujita Y, et al. Direct production of ethanol from raw corn starch via fermentation by use of a novel surface-engineered yeast strain codisplaying glucoamylase and α-amylase. Appl Environ Microbiol, 2004, 70(8): 5037−5040.
[12] Shigechi H, Uyama K, Fujita Y, et al. Efficient ethanol production from starch through development of novel flocculent yeast strains displaying glucoamylase and co-displaying or secreting α-amylase. J Mol Catal B, 2002, 17(3/5): 179−187.
[13] Shigechi H, Fujita Y, Koh J, et al. Energy-saving direct ethanol production from low-temperaturecooked corn starch using a cell-surface engineered yeast strain co-displaying glucoamylase and α-amylase. Biochem Eng J, 2004, 18(2): 149−153. [14] Shiraga S, Kawakami M, Ueda M. Construction of combinatorial library of starch-binding domain of Rhizopus oryzae glucoamylase and screening of clones with enhanced activity by yeast display method. J Mol Catal B: Enzym, 2004, 28(4/6): 229−234.
[15] Satoh E, Niimura Y, Uchimura T, et al. Molecular cloning and expression of two-amylase genes from Streptococcus bovis 148 in Escherichia coli. Appl Environ Microbiol, 1993, 59(11): 3669−3673.
[16] Lo HF, Lin LL, Chiang WY, et al. Deletion analysis of the C-terminal region of the α-amylase of Bacillus sp. strain TS-23. Arch Microbiol, 2002, 178(2): 115−123.
[17] Yamada R, Tanaka T, Ogino C, et al. Novel strategy for yeast construction using δ-integration and cell fusion to efficiently produce ethanol from raw starch. Appl Microbiol Biotechnol, 2010, 85(5): 1491−1498.
[18] Yamakawa SI, Yamada R, Tanaka T, et al. Repeated batch fermentation from raw starch using a maltose transporter and amylase expressing diploid yeast strain. Appl Microbiol Biotechnol, 2010, 87(1): 109−115.
[19] Yamada R, Bito Y, Adachi T, et al. Efficient production of ethanol from raw starch by a mated diploid Saccharomyces cerevisiae with integrated α-amylase and glucoamylase genes. Enzyme Microb Technol, 2009, 44(5): 344−349.
[20] Murai T, Ueda M, Atomi H, et al. Genetic immobilization of cellulase on the cell surface of Saccharomyces cerevisiae. Appl Microbiol Biotechnol, 1997, 48(4): 499−503.
[21] Van Rensburg P, Van Zyl WH, Pretorius IS. Engineering yeast for efficient cellulose degradation. Yeast, 1998, 14(1): 67−76.
[22] Cho KM, Yoo YJ, Kang HS. δ-integration of endo/exo-glucanase and β-glucosidase genes into the yeast chromosomes for direct conversion of cellulose to ethanol. Enzyme Microb Technol, 1999, 25(1/2): 23−30.
[23] Fujita Y, Takahashi S, Ueda M, et al. Direct and efficient production of ethanol from cellulosic material with a yeast strain displaying cellulolytic enzymes. Appl Environ Microbiol, 2002, 68(10): 5136−5141.
[24] Fujita Y, Ito J, Ueda M, et al. Synergistic saccharification, and direct fermentation to ethanol, of amorphous cellulose by use of an engineered yeast strain codisplaying three types of cellulolytic enzyme. Appl Environ Microbiol, 2004, 70(2): 1207−1212.
[25] Shuhei Y, Ryosuke Y, Shohei K, et al. Ethanol production from cellulosic materials using cellulase-expressing yeast. Biotechnol J, 2010, 5(5): 449−455.
[26] Yao SM, Ni N, Xu ZY, et al. Displaying of β-glucosidase on yeast cell surface and studies on propeertics of enzyme. J Qufu Normal Univ: Nat Sci, 2011, 37(3): 81−86.姚淑敏, 倪娜, 徐朝阳, 等. β-葡萄糖苷酶在酿酒酵母表面上的展示及酶学性质的研究. 曲阜师范大学学报: 自然科学版, 2011, 7(3): 81−86.
[27] Shen Y, Wang Y, Bao XM, et al. Progress in the Pathway Engineering of Ethanol Fermentation from Xylose Utilising Recombinant Saccharomyces cerevisiae. Chin J Biotech, 2003, 19(5): 636−640.沈煜, 王颖, 鲍晓明, 等. 酿酒酵母木糖发酵酒精途径工程的研究进展. 生物工程学报, 2003, 19(5): 636−640.
[28] Katahira S, Fujita Y, Mizuike A, et al. Construction of a xylan-fermenting yeast strain through codisplay of xylanolytic enzymes on the surface of xylose-utilizing Saccharomyces cerevisiae cells. Appl Environ Microbiol, 2004, 70(9): 5407−5414. [29] Hou J, Shen Y, Bao XM. Expression xylose isomerase on Saccharomyces cerevisiae cell surface and its influence on xylose metabolism. Chin J Biopro Eng, 2006, 4(1): 30−34.侯进, 沈煜, 鲍晓明. 木糖异构酶在酿酒酵母表面表达及对木糖代谢影响的初步研究. 生物加工过程, 2006, 4(1): 30−34.
[30] Chen LF, Du HL, Lin Y, et al. Display of Candida tropicalis xylose reductase on cells surface of saccharomyces cerevisiae. Food Fermen Ind, 2008, 34(5): 29−34.陈璐菲, 杜红丽, 林影, 等. 热带假丝酵母木糖还原酶在酿酒酵母细胞表面展示. 食品与发酵工业, 2008, 34(5): 29−34.
[31] Huang J, Chen D, Huang RB. Reasearch progress in cellulosome application in bio-ethnol. China Biotechnol, 2011, 31(1): 103−108.黄俊, 陈东, 黄日波. 纤维小体在燃料乙醇中的应用. 中国生物工程杂志, 2011, 31(1): 103−108.
[32] Fierobe HP, Mingardon F, Mechaly A, et al. Action of designer cellulosomes on homogeneous versus complex substrates: controlled incorporation of three distinct enzymes into a defined trifunctional scaffoldin. J Biol Chem, 2005, 280(16): 16325−16334.
[33] Tsai SL, Oh J, Singh S, et al. Functional assembly of minicellulosomes on the saccharomyces cerevisiae cell surface for cellulose hydrolysis and ethanol production. Appl Environ Microbiol, 2009, 75(19): 6087−6093.
[34] Den Haan R, Rose SH, Lynd LR, et al. Hydrolysis and fermentation of amorphous cellulose by recombinant Saccharomyces cerevisiae. Metab Eng, 2007, 9(1): 87−94.
[35] Wen F, Sun J, Zhao HM. Yeast surface display of trifunctional minicellulosomes for simultaneous saccharification and fermentation of cellulose to ethanol. Appl Environ Microbiol, 2010, 76(4): 1251−1260.
[36] Yang M, Zhang JX, Tian S. Construction of engineered saccharomyces cerevisiae for steadily metabolizing glucose and xylose to ethanol. Chin J Appl Environ Biol, 2009, 15(2): 258−261.杨态, 张金鑫, 田沈. 稳定代谢葡萄糖和木糖产乙醇的重组酿酒酵母工程菌初步构建. 应用与环境生物学报, 2009, 15(2): 258−261.