裙带菜配子体总RNA提取的比较研究
(辽宁师范大学 生命科学学院,辽宁省植物生物技术重点实验室,辽宁 大连 116029)
裙带菜(Undaria pinnatifida)是我国重要的养殖经济海藻之一,年产量仅次于海带和紫菜居第三位。裙带菜分类地位隶属于褐藻门(Phaeophyta)、海带目(Laminariales)、翅藻科(Alariaceae)、裙带菜属(Undaria),为温带性海藻,野生种类主要分布于中国、韩国和日本等亚洲北部地区[1]。裙带菜具有二倍的孢子体和单倍的配子体所组成的异型世代交替的生活史,具有独立的生殖器官和简单的根茎叶分化,因此被认为是较高等的大型海藻。由于裙带菜配子体个体微小,细胞壁较薄,黏液腺等分泌结构不发达,种质保存技术成熟等特殊的优势,近些年来被作为首选的大型海藻研究材料应用于遗传育种、细胞工程化育苗和分子生物学等相关研究领域[2]。
裙带菜及其配子体富含多糖、多酚和次生代谢物质,它们易与核酸结合形成不溶于水的复合物,同时在提取过程中这些物质也是多种酶类的抑制剂,因此裙带菜RNA的提取是广泛开展裙带菜分子生物学研究的主要难点之一。目前一些商业化的试剂盒(如Trizol等)被广泛应用于植物总RNA的提取纯化,但专门用于大型海藻总RNA提取的试剂盒还没有被开发应用。本文以裙带菜配子体为材料,对几种常见的总RNA提取方法进行了比较研究,优化了提取过程,为大型海洋藻类分子生物学研究的开展提供了基本技术。
1 材料与方法
1.1 实验材料
裙带菜(U.pinnatifida)配子体由本实验室自行保存,扩大培养方法:温度20℃,光照强度20 μmol/(m2·s),光周期 12 h :12 h,培养液 PES 每7天更换一次。
条斑紫菜(Porphyra yezoensis)丝状体由辽宁省植物生物技术重点实验室海洋藻类研究室提供,培养条件:温度 20℃,光照强度 30 μmol/(m2·s),光周期14 h :10 h,培养液PES每7天更换一次。
海带(Laminaria japonica)配子体为实验室自行保存,扩大培养方法:温度 18℃,光照强度 20 μmol/(m2·s),光周期 12 h :12 h,培养液 PESI每 7 天更换一次。
1.2 实验方法
1.2.1 裙带菜配子体总RNA提取
1.2.1.1 改进SDS法
取材料0.1g用蒸馏水冲洗2次,灭菌滤纸吸干水分,液氮研磨,转移至离心管中,加入 80℃预热的SDS提取液1mL(100 mmol/L LiCl,100 mmol/L Tris,50 mmol/L EDTA,2% SDS,使用前加16% PVP和1% β-巯基乙醇),充分振荡混匀,置冰上5 min。4 ℃、12 000g离心 10 min。取上清,加 200 μL 3mol/L KAc(pH5.5),匀冰上放置5 min。4℃、12 000g离心10 min。取上清,加200 μL氯仿,充分振荡抽提,冰上放置5 min。4 ℃、12 000g离心15 min。小心吸取上层水相于新离心管中,加600 μL预冷的异丙醇,混匀后-20℃沉淀20 min。4℃、12 000g离心10 min。1mL 75%乙醇(DEPC处理)洗涤 2次,室温干燥,20 μLDEPC水溶解,-80℃保存。
1.2.1.2 CTAB法
取液氮研磨好的材料 0.1 g于离心管中,加入CTAB提取液1mL(100 mmol/L Tris-HCl(pH8.0),50 mmol/L EDTA,2 mol/L NaCl,2% CTAB,使用前加1% DTT),充分混匀,置冰上5 min。4℃、12 000g离心10 min。取上清,加等体积氯仿:异戊醇(24:1),充分振荡抽提,冰上放置5 min。4℃、12 000g离心15 min。小心吸取上层水相于新离心管中,加1/4体积10 mol/L LiCl,混匀后-20℃沉淀过夜。4℃、12 000g离心10 min。弃上清,1 mL 75%乙醇(DEPC处理)洗涤2次,室温干燥,20 μL DEPC水溶解,-80℃保存。
1.2.1.3 Trizol试剂盒法
根据Invitrogen公司 Trizol试剂盒提供的操作流程提取。每 0.1g液氮研磨的材料加 1mL Trizol试剂,充分混匀室温放置5 min,加200 μL氯仿,用力震荡混匀,室温放置5 min。4℃、12 000g离心15 min。小心吸取上层水相于新离心管中,加 500 μL预冷的异丙醇,混匀后室温沉淀 5 min。4℃、12 000g离心10 min。弃上清,1 mL 75%乙醇(DEPC处理)洗涤两次,室温干燥,20 μLDEPC 水溶解,-80℃保存。
1.2.1.4 RNAiso Reagent 植物总RNA提取试剂盒
实验方法根据宝生物公司(TAKARA)RNA提取试剂盒(RNAiso Reagent)操作流程。每0.1g液氮研磨的材料加 1mL RNAiso Reagent,混匀室温静置5 min。12 000g4℃离心5 min。取上清,加入氯仿(RNAiso Reagent的1/5体积量),用力震荡,室温静置5 min。12 000g4℃离心15 min。取上清液转移至另一离心管中,加入等体积的异丙醇,充分混匀后在室温静置10 min。 12 000g4℃离心10 min。弃上清,75%乙醇(DEPC处理)洗涤2次,室温干燥,20 μL DEPC水溶解,-80℃保存。
1.2.2 RNA样品中DNA的去除
按照下列反应体系在离心管中依次加入 RNA 20 μL,10×buffer 10 μL,DNaseI 4μL,RNase Inhibitor 1μL,DEPC-H2O 65μL。37℃反应 30 min,然后加入等体积氯仿抽提,4℃、12 000g离心15 min。取上层水相,加 1/5体积 3 mol/LNaAc和 2倍体积无水乙醇,-80℃沉淀2 h。75%乙醇(DEPC处理)洗涤两次,室温干燥,20 μL DEPC水溶解,-80℃保存。
1.2.3 RNA纯度的检测
1μLRNA样品于1%琼脂糖凝胶电泳,1×TAE缓冲液中120 V电泳20 min。EB浸泡胶后凝胶成像系统成像。取1μL RNA样品稀释100倍后于紫外分光光度计测定A260和A280的值,计算A260/A280确定RNA纯度。
1.2.4 RT-PCR检测
将消化处理后的 RNA样品用 AMV反转录酶(TAKARA)按常规的方法进行反转录反应,以GST(谷胱甘肽硫转移酶)基因设计引物进行 PCR反应。20 μL PCR 反应体系组成如下:10×buffer 2μL;Taq 酶 0.2μL;dNTP 3.2 μL;模板 5 μL;上下游引物各 0.2 μL;DEPC-H2O 9.2 μL。反应条件:94℃ 5 min,94℃1 min,55℃1 min,72℃ 30 s,35 个循环,72℃10 min。PCR产物经1%琼脂糖凝胶电泳检测。
2 结果
2.1 RNA电泳检测
各种方法提取获得的总RNA电泳结果见图1。其中图1-A为改进SDS法提取总RNA的电泳图谱,可见28s、18s rRNA条带分离明显清晰,28s、18s条带亮度比 5s大,表明其完整性良好;点样孔中无DNA残留痕迹,28s条带上方无弥散拖尾现象,没有多糖、蛋白等杂质的污染;A260/A280比值为1.98,表明这种方法获得的RNA纯度较高;但电泳图谱中可见有少许的基因组 DNA残留,用 DNaseI消化处理后再电泳结果见图2-A,基因组DNA消失,rRNA条带保持完整,A260/A280为 1.85。从图1-B泳道可见CTAB法提取的总RNA明显有降解,18s带亮度很大,向下呈现弥散状态;总 RNA中有蛋白和多糖污染;基因组DNA残留非常明显。用两种试剂盒提取则没有能够得到完整的RNA(见图1-C和1-D),只有少量的5s条带存在。
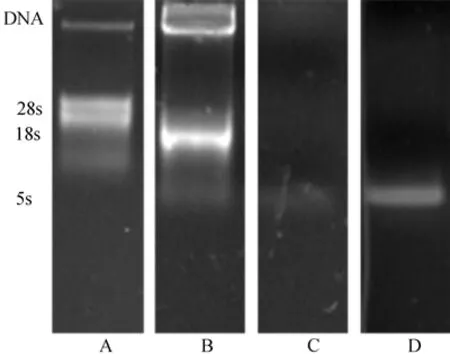
图1 裙带菜配子体总RNA琼脂糖凝胶电泳图谱Fig.1 Agarose gel electrophoresis of Total RNAs from U.pinnatifida gametophyte
2.2 RT-PCR检测
选择改进SDS法提取的总RNA用DNaseI消化处理后,进行反转录和 PCR扩增反应,结果见图2-B。扩增结果特异性强,无非特异性条带出现,说明提取的总RNA质量较好,可用于基因克隆等后续实验研究。

图2 DNaseI消化处理后的总RNA及RT-PCR琼脂糖凝胶电泳图谱Fig.2 Agarose gel electrophoresis of DNaseI digested total RNA and RT-PCR products
3 分析与讨论
近几年来已经有了一些关于大型海藻 RNA提取的报道,但裙带菜等褐藻类的研究报道数量有限(表1)。很多研究表明,大型海藻总RNA提取所采用的方法主要是 CTAB法,多用于基因克隆、文库构建和提取方法的比较等。CTAB和SDS是比较常用的蛋白质变性剂,能有效地裂解细胞膜并抑制核酸酶的活性,释放出细胞内容物并使核酸-蛋白复合物分解以便释放核酸。韩峰等[3]认为CTAB 去除海藻多糖效果显著,结合有效的抽提和纯化方法能得到高纯度的DNA。杨婧等[4]利用几种不同的方法提取绿色巴夫藻(Pavlova viridis)总RNA,比较分析后认为CTAB-酸酚法最为适合。但作者在实验中发现,CTAB法提取的总RNA含有基因组DNA的量普遍比SDS方法高,且实验耗时较长,一次提取实验需两天才能完成。
改进的SDS法采用16% PVP和1%β-巯基乙醇相结合的方法来抑制酚类物质的氧化作用并将其有效的去除。PVP是多酚的螯合剂,β-巯基乙醇则是高效的还原剂,为PVP和酚的结合提供了良好的环境,二者结合防止多酚氧化的效率大大提高[5]。高浓度的盐能够沉淀多糖,3 mol/L KAc(pH5.5)沉淀多糖的同时能提供偏酸性的环境,这时 RNA稳定而 DNA则容易变性沉淀下来,利用后续的抽提即可将其除去,从而分离得到较高质量的RNA。虽然仍有少量基因组DNA残留,但利用DNaseI可有效去除DNA污染。该方法耗时短,约5~6 h即可完成总RNA提取。为检验该方法有效性的适应范围,作者用这种方法提取了海带配子体、条斑紫菜丝状体的总 RNA,均获得了较好的结果(见图3-A和3-B)。
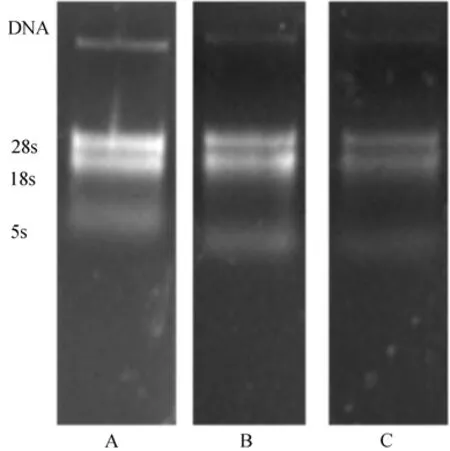
图3 海带配子体和条斑紫菜丝状体总 RNA琼脂糖凝胶电泳图谱Fig.3 Agarose gel electrophoresis for total RNA extracted from L.japonica gametophyte and P.yezoensis conchocelis
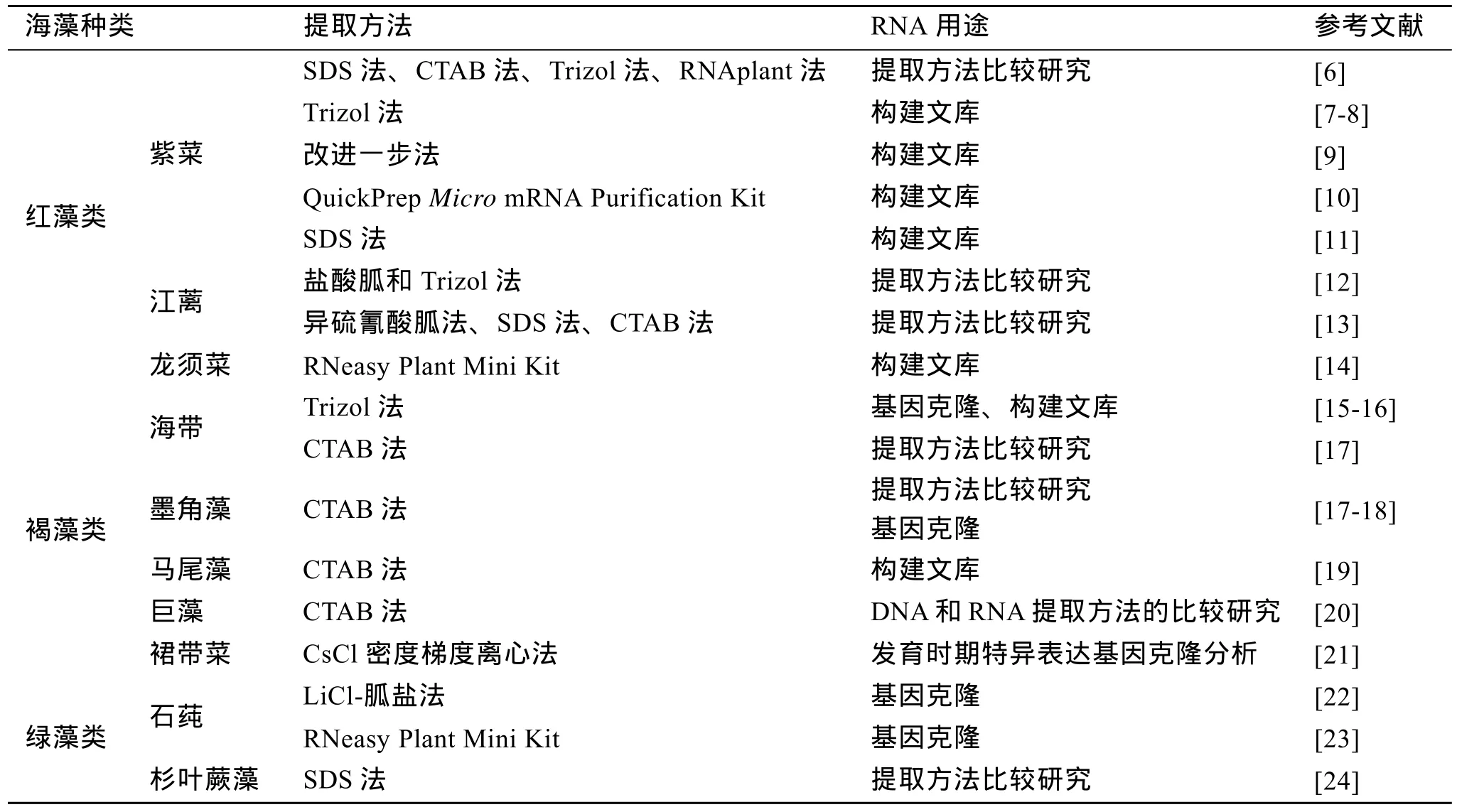
表1 大型海藻总RNA提取主要方法及其用途Tab.1 Methods to extract total RNA from seaweeds
Trizol和RNAiso Reagent是基于异硫氰酸胍法的原理而设计配制的高效RNA提取试剂盒,更适合于动物组织和细胞总RNA提取,也适用于某些植物总RNA提取。我们的实验结果表明这2种试剂盒对条斑紫菜 RNA提取效果很好(图3-C)。而对裙带菜配子体总RNA提取效果不佳。
研究结果表明,改进的 SDS法是裙带菜配子体总RNA提取行之有效的方法,并可以将该方法应用于褐藻类和其他大型海藻的RNA提取。该方法表现出提取过程耗时少,得到的RNA质量较高等明显优势,获得的RNA可以用于基因克隆、差异显示分析、分子杂交等各种分子生物学研究。
[1]钱树本,刘东艳,孙军.海藻学[M].青岛:中国海洋大学出版社,2005.
[2]胡晓燕.褐藻配子体培养的应用研究及意义[J].海洋科学,2002,26(7):30-31.
[3]韩峰,宫倩红,史晓翀,等.海藻核酸提取的难点及对策[J].海洋科学,2004,28(10):71-74.
[4]杨婧,牛艳,张可炜,等.一种适用于 RT-PCR的微藻总 RNA 提取方法[J].海洋科学,2010,01,34(1):6-10.
[5]张今今,王跃进,王西平,等.葡萄总RNA提取方法的研究[J].果树学报,2003,20(3):178-181.
[6]胡晓静,何培民.条斑紫菜丝状体总 RNA提取方法比较[J].生物技术通讯.2007,18(4):604-607.
[7]徐民俊,茅云翔,庄筠昀,等.条斑紫菜孢子体cDNA文库构建及表达序列标签的初步分析[J].大连水产学院学报,2006,21(1):7-12.
[8]杨官品,刘永健,孙雪,等.条斑紫菜丝状孢子体cDNA文库构建及抗病相关基因鉴定[J].青岛海洋大学学报,2003,33(1):47-52.
[9]庞国兴,王广策,胡松年,等.坛紫菜丝状孢子体EST的获取、生物信息学分析[J].海洋与湖沼,2005,36(5):452.
[10]Kakinuma M,Kaneko I,Coury D A,et al.Isolation and identification of gametogenesis-related genes inPorphyra yezoensis(Rhodophyta) using subtracted cDNA libraries[J].Journal of Applied Phycology,2006,18:489-496.
[11]Nikaido I,Asamizu E,Nakajima M,et al.Generation of 10154 expressed sequence tags from a leafy gametophyte of a marine red alga,Porphyra yezoensis[J].DNA Research,2000,7:223-227.
[12]Falcão V D R,Tonon A P,Oliveira M C,et al.RNA Isolation method for polysaccharide rich algae:agar producingGracilaria tenuistipitata(Rhodophyta) [J].Journal of Applied Phycology.2008,20(1):209-212.
[13]Chan C X,Ho C L,Othman R Y,et al.Total RNA Ex-traction for the Red SeaweedGracilaria changii(Gracilariales,Rhodophyta) [J].Asia-Pacific Conference in Marine Science and Technology,2002,5:12-16.
[14]孙雪,张学成.龙须菜四分孢子体cDNA文库的构建[J].青岛海洋大学学报,2003,33(5):727-732.
[15]史西志,毕艳会,周志刚.海带雄性配子体差异表达基因片段的克隆及筛选[J].水产学报,2005,29(5):666-669.
[16]胡乐琴,陆广琴,刘士成,等.利用 SMART技术构建海带配子体 cDNA文库[J].安徽农业科学,2006,34(13):2975-2976,2979.
[17]Graeth P,Asuncion L L,Marta V,et al.Simple and rapid RNA extravtion from freeze-dried tissue of brown algae and seagrass[J].European Journal of Phycology,2006,41:97-104.
[18]Morris C A,Nicolaus B,Sampson V,et al.Identification and characterization of a recombinant metallothionein protein from a marine alga,Fucus vesiculosus[J].Biochemistry Journal,1999,338:553-560.
[19]Wong T K M,Ho C L,Lee W W,et al.Analyses of expressed sequence tags fromSargassum binderi[J].Journal of Phycology,2007,43:528-534.
[20]Kirk E A,Stephanie K C,Dennis A P,et al.The gene family encoding the fucoxanthin chlorophyll proteins from the brown algaMacrocystis pyrifera[J].Molecular and General Genetics,1995,246:455-464.
[21]Hou H S,Li N,Wu C Y.Identification and sequence of a cDNA clone corresponding to a gene involved in develepment ofUndaria pinnatifida[J].Chinese Journal of Oceanology and Limnology,1998,16:25-29.
[22]Kang S E,Jin L G,Choi J S,et al.Isolation of pollutant(pine needle ash)-responding genes from tissues of the seaweedUlva pertus[J].Journal of Applied Phycology,2006,18:483-487.
[23]Kakinuma M,Coury D A,Itoh S,et al.Isolation and characterization of a single-copy actin gene from asterile mutant ofUlva pertusa(Ulvales,Chlorophyta) [J].Gene,2004,334:145-155.
[24]La Claire J W,Herrin D L.Co-isolation of high-quality DNA and RNA from coenocytic green algae[J]. Plant Molecular Biology Reporter,1997,15:263-272.

