坛紫菜磷酸烯醇式丙酮酸羧化酶基因的克隆与序列分析
张晓娟,王广策,何林文,陈昌生
(1.中国科学院 海洋研究所,山东 青岛 266071;2.中国科学院 研究生院,北京 100049;3.天津科技大学海洋学院,天津 100039;4.集美大学 渔业学院,福建 厦门361020)
磷酸烯醇式丙酮酸羧化酶(phosphoenolpyruvate carboxylase,PEPC;EC 4.1.1.31)在二价金属离子Mg2+或 Mn2+存在下,能够催化磷酸烯醇式丙酮酸(PEP)与反应生成草酰乙酸(OAA)这一不可逆反应[1]:。该酶广泛分布在高等植物、藻类、蓝细菌、细菌、原生生物中,在动物和真菌中尚未发现[2-4]。磷酸烯醇式丙酮酸羧化酶存在于所有的光合生物中,是光合作用中的关键酶,主要功能是为三羧酸循环补充四碳酸,在 C4植物和景天科植物光合代谢途径中扮演着浓缩固定 CO2[4-5]。此外该酶还参与氨基酸代谢中碳骨架的回补、参与种子形成与萌发、果实成熟、植物保卫细胞气孔开闭、豆科植物根瘤固氮过程、维持离子和pH值平衡等诸多作用[6-10]。
在种族和系统演生进化过程的研究中,分子特征已经成为一种重要工具。近年来,PEP羧化酶的全氨基酸序列和全核苷酸序列甚至全序列的一部分能够作为分子遗传标记已为诸多研究者所报道[3,6,11-14],PEP羧化酶的序列特征能够在生物体和代谢途径之间建立系统发生关系。
坛紫菜是我国南方特有的深受亚洲人民欢迎的经济红藻,也是我国出口创汇的主要农产品之一。它属于原红藻纲(Protoflorideae)、红毛菜目(Bangiales),是一种原始红藻,具有特殊的生活史和世代交替现象,包含叶状体(单倍配子体)和丝状体(二倍孢子体),叶状体在冬春季生长旺盛,主要分布于潮间带礁石上。丝状体渡夏,而且钻入贝壳等碳酸钙基质中生长,主要分布于潮下带,是一种石内生藻类(endolithic algae)。坛紫菜pH值补偿机制高,具有低的Km值,能利用作为光合作用时的碳源,其低的无机碳补偿点表明坛紫菜具有浓缩碳的作用[15-16]。本实验室的 Fan等[17]构建了坛紫菜丝状体(孢子体)的11000条EST,同时在坛紫菜代谢途径分析中,主要分析了无机碳的固定途径,结果发现坛紫菜丝状体阶段可能存在类C4的固碳途径。本实验克隆了坛紫菜 PEP羧化酶基因的部分序列,对其做了系统进化分析,对进一步深入研究坛紫菜光合作用、碳代谢等具有重要的参考价值,同时为进一步研究其功能奠定了基础。
1 材料与方法
1.1 坛紫菜丝状体的获得
试验中所用的坛紫菜丝状体为本实验室(中国科学院海洋研究所藻类分子生理与发育调控实验室)保留藻株,培养基为海水加富培养基,培养条件为18℃,光强约为 1 800 lux,光:暗周期为 12 :12,通入过滤法消毒后的空气进行培养,每周更换 1次培养基。用蒸馏水冲洗干净后进行提取RNA。
1.2 试剂、试剂盒及仪器
总RNA提取采用天根生物公司的RNAprep pure试剂盒,cDNA合成采用Superscript ΙΙ® 反转录酶试剂盒(Invitrogen公司,USA)。 PCR扩增目的基因片段、DNA纯化、连接所用试剂盒和载体为pMD-18T购自东盛生物公司和TaKaRa生物技术有限公司,菌株为E.coliTop 10。药品均为进口分装或国产分析纯。序列测定委托上海生工生物技术公司完成。
1.3 坛紫菜PCPC同源序列的克隆
EST 的钓取及组装:从 http://est.kazusa.or.jp/en/plant/porphyra/EST/上获得了与条斑紫菜PEP羧化酶相关的 EST 序列:AU193218、AU193318、AU191558、AU188467、AV430322、AU195387、AU195597、AU192608、AU196318、AU191989、AU191907、AU195415、AU191278、AU192140、AU192294、AU196648、AU191070、AU190685、AV430724、AU192096、AU192037、AV431839、AU194305、AV433323、AU188200[18-19]。利用BioEdit生物学软件对这些 EST序列进行拼接,从中设计 PEPCS1(5′-CTCCAGCATGTCGAATGTAGCG-3′)和 PEPCA1(5′-CCGTCGTGTACGTCTCAAGGGT-3′)一 对 引 物 ,以坛紫菜的cDNA为模板,进行PCR扩增,PCR仪器购自德国Eppendorf 公司。
PCR 反应条件为:94℃预变性 5min;94℃变性30s,60°C退火 30s,72℃延伸 2min,30个循环;72°C延伸10min;4°C保存。
反应体系见表1。PCR产物经1%琼脂糖凝胶电泳检测,目的条带用琼脂糖凝胶纯化试剂盒纯化(购自北京天根生物公司)后克隆入 pMD18-T载体(TaKaRa),转化到大肠杆菌 Top 10感受态细胞中,转化后,于氨苄LB培养基上挑取单克隆培养,用上述引物再次进行 PCR 扩增,以进一步验证克隆为阳性,将阳性克隆送去上海生工生物技术公司采用ABI3730自动测序仪测序。
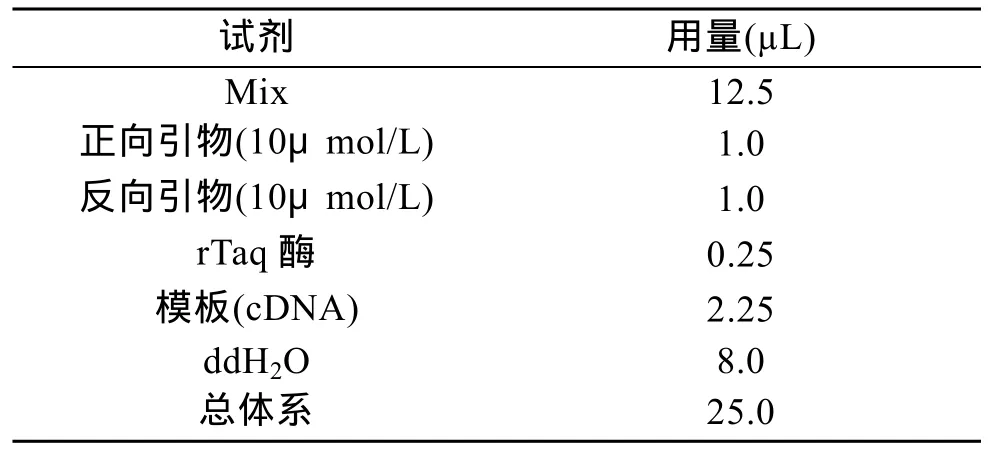
表1 PCR扩增体系Tab.1 PCR amplification system
1.4 RACE PCR克隆PEPC的3′末端
以接头引物 AdapterdT(5′-GGCCACGCGTCGACTAGTAC 1qzT17-3′)引导反转录合成。用通用引物up(5′-GGCCACGCGTCGACTAGTAC-3′) 和 基 于PEPC部分序列设计的基因特异引物 E31(5′-GCCGCCCAAGACTATCAATG-3′)和 E32(5′-TCTACCGCTTCGTACACCCACT-3′),采用半巢式 PCR 的方法(semi-nested PCR)扩增基因3′末端。应用Touch down的PCR程序:25μL体系(同表1);94℃预变性5min;94℃变性 30s,66℃退火 30s(每一循环降低 0.6℃),72℃延伸1min,10个循环;94℃变性30s,60℃退火30s,72℃延伸1min,25个循环;72°C延伸10min;4°C保存。
PCR产物经1%琼脂糖凝胶电泳检测,目的条带用琼脂糖凝胶纯化试剂盒纯化(购自北京天根生物公司)后克隆入 pMD18-T载体(TaKaRa,大连宝生物公司),转化到大肠杆菌Top 10感受态细胞中,转化后,于氨苄 LB培养基上挑取单克隆培养, 用上述引物再次进行 PCR 扩增,以进一步验证克隆为阳性,将阳性克隆送去测序。
1.5 序列分析
序列同源性对比采用 NCBI中的 BLAST软件(http://www.ncbi.nlm.nih.gov/blast)进行;氨基酸序列分析采用 ExPASy在线工具(http://www.expasy.org/);结构域预测采用Pfam HMM软件(http://pfam.sanger.ac.uk/search);信号肽预测采用 SignalP(http://www.cbs.dtu.dk/services/signalP/);TMHMM(http://www.cbs.dtu.dk/services/TMHMM-2.0/)用于寻找跨膜结构;根据PROSITE数据库对坛紫菜PEP羧化酶氨基酸序列进行活性位点的分析。重建系统进化树所用的PEP羧化酶核苷酸序列通过对GenBank的BLAST检索获得。重建PEP羧化酶的系统进化树时,选取了16个有代表性的序列(来自不同类群,如表2所示)。
2 结果与讨论
2.1 序列分析
所克隆的序列长为 2 538 bp,与原从条斑紫菜EST数据库中拼接出的 PEPC序列片段相似性高达89.5%。该片段是一个连续的读码框,在阅读框中不存在起始密码子,但还有一个 TAG终止密码子,191bp的 3′端非编码区(UTR)和 ploy A 尾巴,编码846个氨基酸。3′端非编码区(UTR)没有找到传统的AAUAAA的加尾信号,这在其他红藻中也很少有发现,如Porphyra yezoensis的β-tubulin基因和carbonic anhydrase基因[20-21],以及Gracilaria gracilis的galactose1-phosphate uridyl transferase 基因[22]。
BLASTx检索发现所有的同源序列均为磷酸烯醇式丙酮酸羧化酶(phosphoenolpyruvate carboxylase,PEPC),且同源性很高。以Blast软件对坛紫菜PEPC基因氨基酸序列的相似性比对结果表明,PEPC 与被子植物,裸子植物,藻类和细菌均有一定的一致性,如与玉米(Zea mays)为 46%,与芦荟(Aloe arborescens)为 46%,与水稻(Oryza sativa Japonica Group)为47%,与大肠杆菌的(Escherichia coli)为 35%,一致性范围在 35%~50%之间,由于 C4型的 PEPC的 C末端第774位或附近的一个氨基酸均为丝氨酸(S),而所有C3或CAM型的PEPC相应位置均为丙氨酸(A)[1,4,23-24],本研究中的PEPC在相对应位置为丙氨酸(A),所以不可能是一个C4型的PEPC(图1)。
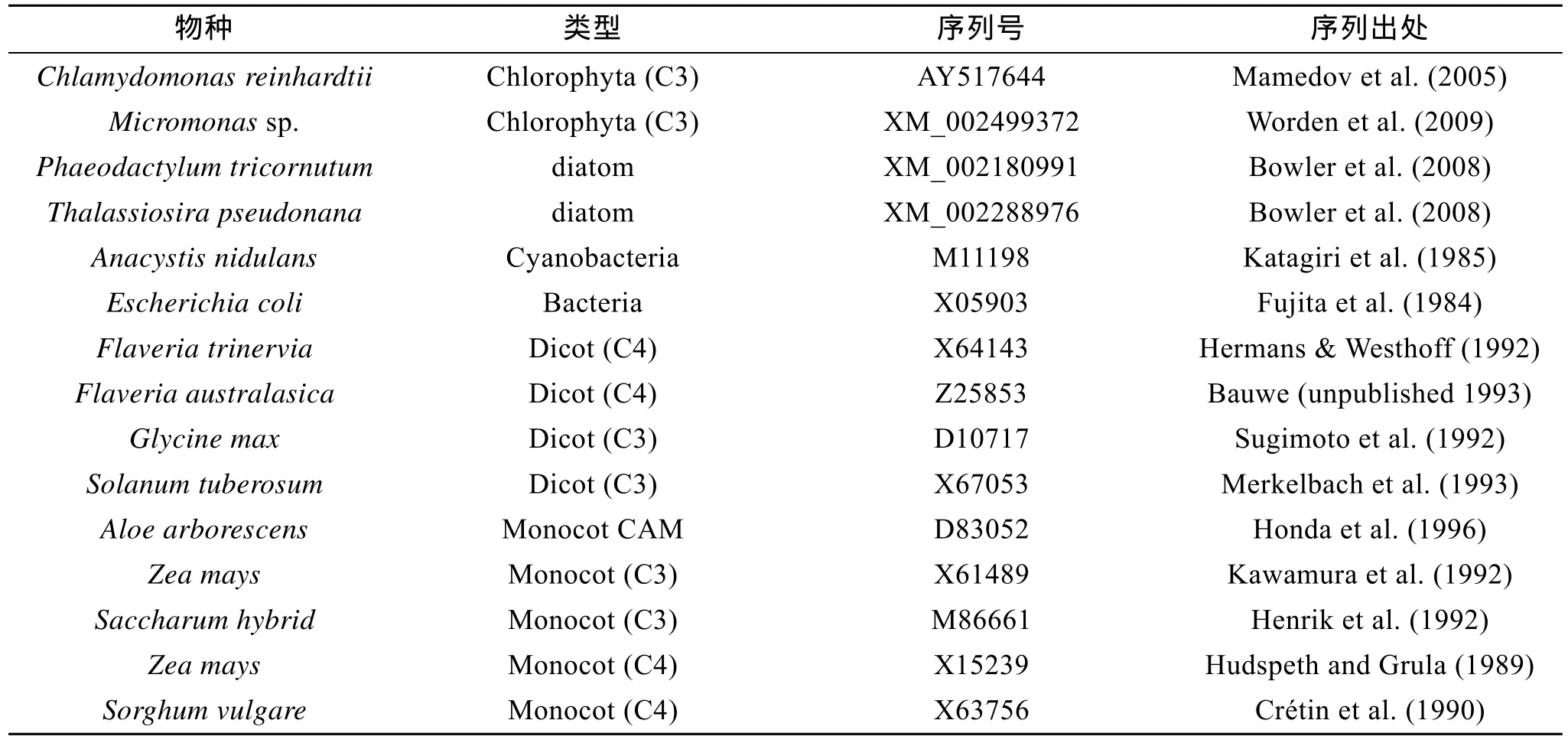
表2 构建系统进化树所用序列Tab.2 PEPC isoforms used for phylogenetic analysis

图1 部分C3,C4植物PEPC氨基酸部分序列的比较Fig.1 Partial sequence alignment of amino acid residues of PEPC from some C3 and C4 plants
Pfam HMM 软件分析显示氨基酸序列中的第130至第841个氨基酸之间为PEP羧化酶的结构域;信号肽预测SignalP和TMHMM在线软件分析显示该段序列既没有信号肽序列,也没有跨膜结构。

图2 坛紫菜PEPC核苷酸及氨基酸序列Fig.2 The nucleotide sequence and deduced amino acid sequence of PEPC from Porphyra haitanensis
根据PROSITE数据库分析,其氨基酸序列含有两个大的 PEP羧化酶活性位点,分别为 PS00781(VLTAHPTQAVRR)和PS00393(VMVGYSDSGKDGG),另外还含有4种模式的其他功能位点,分别为:依赖于 cAMP和 cGMP的蛋白激酶磷酸化位点(cAMP-and cGMP-dependent protein kinase phosphorylation site,PS00004),蛋白激酶 C磷酸化位点(Protein kinase C phosphorylation site,PS00005),酪蛋白激酶Ⅱ磷酸化位点(Casein kinase II phosphorylation site,PS00006)和 N-端十四烷酰化位点(N-myristoylation site,PS00008)。
2.2 系统进化分析
多序列比对采用 Clustal X 程序,采用 Clustal X 程序和MEGA 4.0 软件,以临位相联法(Neighbor-Joining)法来重建,计算方法重复1 000次,进化距离选择 p距离(p-distance),空缺或者缺失(gaps/missing data)的处理采用完全删除(complete deletion),以此构建的系统进化树如图3所示。
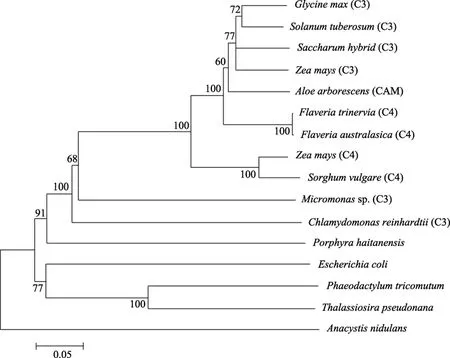
图3 采用N-J 法以Clustal X和MRGA4.0软件构建的系统进化树(计算重复1000次)Fig.3 Phylogenetic analysis of 17 PEPC isoforms.In this Neighbor-joining consensus tree,numbers on internal branches are percent bootstrap values of 1000 replicates
对 PEP羧化酶进行进化分析,同时选取了裸子植物、被子植物、藻类及微生物中的16个序列,结果如图2所示。坛紫菜PEPC羧化酶氨基酸序列以及绿藻中的莱因衣藻(C.reinhardtii)和绿藻(Micromonassp.),都不和其他的聚类。种子植物和裸子植物中的C3型的PEPC及CAM型的PEPC聚为一个大的进化支,虽然种子植物和裸子植物中的 C4型的PEPC各聚为一支,但他们的进化距离相当。大肠杆菌与硅藻(Phaeodactylum tricornutum和Thalassiosira pseudonana)聚为同一支,是与他们之间的进化关系密切相连的,硅藻中有数百个基因是来自于细菌的[25],所以他们都可以归为细菌型的 PEP羧化酶。本系统进化图与 Gehrig等[13-14]1998年和 2001年构建的PEP羧化酶部分氨基酸序列基本上是一致的。
如前人所述, PEP羧化酶可以分为四种类型:C3型、C4型,CAM型及细菌型,C3亚型最先出现,C4和CAM亚型被认为是由C3亚型进化而来,而且二者分别多次独立起源[1,6,26],本实验构建的系统进化树图很好的诠释了PEPC的代谢途径与进化关系。
在本图中,从大的分支上来说,坛紫菜 PEP羧化酶氨基酸序列是与绿色植物聚在一支的,是完全有别于细菌类型的 PEP羧化酶的一支,从进化上来看,坛紫菜PEP羧化酶氨基酸序列更接近于C3型(这在之前的序列分析中也有所体现),进化上更为古老。红藻最初依据林曼系统分类法归为植物,但后来归为最古老的真核生物,关于红藻的进化和起源一直是困扰科学家的一个问题。本进化图中坛紫菜PEP羧化酶氨基酸序列的进化可能也与红藻特殊的进化地位有关系。
[1]Svensson P,Blasing O E,Westhoff P.Evolution of C4 phosphoenolpyruvate carboxylase [J].Arch Biochem Biophys,2003,414:180-188.
[2]Lepiniec L,Vidal J,Chollet R,Gadal P,Crétin C.Phosphoenolpyruvate carboxylase:structure,regulation and evolution [J].Plant Sci,1994,99:111-124.
[3]Toh H,Kawamura T,Izui K.Molecular evolution of phosphoenolpyruvate carboxylase[J].Plant Cell Environ,1994,17:31-43.
[4]Izui K,Matsumura H,Furumoto T,et al.Phosphoenolpyruvate carboxylase:A new era of structural biology [J].Annu Rev Plant Biol,2004,55:69-84.
[5]Izui K,Ishijima S,Yamaguchi Y,et al.Cloning and sequence analysis of cDNA encoding active phosphoenolpyruvate carboxylase of the C4-pathway from maize[J].Nucleic Acids Research,1986,14:1614-1628.
[6]Lepiniec L,Keryer E,Philippe H,Gadal P,et al.The phosphoenolpyruvate carboxylase gene family of sorghum:Structure,function and molecular evolution [J].Plant Mol Biol,1993,21:487-502.
[7]Melzer E,O’Leary M.Anaplerotic fixation by phosphoenolpyruvate carboxylase in C3 plants [J].Plant Physiol,1987,84:58-60.
[8]Leegood R C,Osmond C B.The flux of metabolites in C4 and CAM plants.In Plant Physiology,Biochemistry and Molecular Biology,ed.DT Dennis,DH Turpin,Essex:Longman Sci.Tech.1990,274-298.
[9]Vance C P,Gregerson R G,Robinson D L,et al.Primary assimilation of nitrogen in alfalfa nodules:molecular features of the enzymes involved [J].Plant Sci,1994,101:51-64.
[10]Outlaw W H Jr.Kinetic properties of guard-cell phosphoenolpyruvate carboxylase [J].Biochem Physiol Pflanzen,1990,186:317-325.
[11]Cushman J C,Bohnert H J.Transcriptional activation of CAM genes during development and environmental stress [J].Ecol Stud,1996,114:135-158.
[12]Honda H,Okamoto T,Shimada H.Isolation of a cDNA for a phosphoenolpyruvate carboxylase from a monocot CAM-plant,Aloe arborescens:Structure and its gene expression [J].Plant Cell Physiol,1996,37:881-888.
[13]Gehrig H H,Heute V,Kluge M.Towards a better knowledge of the molecular evolution of phosphoenolpyruvate carboxylase by comparison of partial cDNA sequences [J].J Mol Evol,1998a,46:107-114.
[14]Gehrig H H,Heute V,Kluge M.New Partial Sequences of Phosphoenolpyruvate Carboxylase as Molecular Phylogenetic Markers [J].Molecular Phylogenetics and Evolution,2001,20:262-274.
[15]邹定辉,高冲山.坛紫菜光合作用对重碳酸盐的利用[J].科学通报,2002,47(12):926-930.
[16]骆其君,裴鲁青,潘双叶,等.坛紫菜自由丝状体对无机碳的利用[J].水产学报,2001,26(5):477-480.
[17]Fan X L,Fang Y J,Hu S N,Wang G C.Generation and analysis of 5318 Expressed Sequenced Tags from the fliamentous sporophytes ofPorphyra haitanensis(Rhodophyta) [J].J Phycol,2007,43:1287-1294.
[18]Asamizu E,Nakajima M,Kitade Y,et al.Comparison of RNA Expression Profiles between the Two Generations ofPorphyra yezoensis(Rhodophyta),Based on Expressed Sequence Tag Frequency Analysis [J].Journal of Phycology,2003,39:923-930.
[19]Nikaido I,Asamizu E,Nakajima M,et al.Generation of 10,154 Expressed Sequence Tags from a Leafy Gametophyte of a Marine Red Alga,Porphyra yezoensis[J].DNA Research,2000,7:223-227.
[20]Gong Q H,Han F,Dai J X,et al.Rapid isolation and sequence analysis of the beta-tubulin gene fromPorphyra yezoensis(Rhodophyta) [J].J Appl Phycol,2005,17:1-5.
[21]Zhang B Y,Yang F,Wang G C,et al.Cloning and quantitative analysis of carbonic anhydrase gene fromPorphyra yezoensisUEDA [J].J Phycol,2009,45:290-296.
[22]Lluisma A O,Ragan M A.Characterization of a galactose1-phosphate uridyl transferase gene from the marine red algaeGracilaria gracilis[J].Curr Genet,1998(b),34:112-119.
[23]张桂芳,赵明,丁在松,等.稗草磷酸烯醇式丙酮酸羧化酶(PEPCase) 基因的克隆与分析 [J].作物科学,2005,31(10):1365-1369.
[24]Latzko E,Kelly G J.The many-faceted function of phosphoenolpyruvate carboxylase in C3 plants [J].Physiol,1983,21:805-815.
[25]Bowler C,Allen A E,Badger J H,et al.the Phaeodactylum genome reveals the evolutionary history of diatom genomes [J].Nature,2008,456:239-244.
[26]Chollet R,Vidal J,O’Leary M H.PHOSPHOENOLPYRUVATE CARBOXYLASE:AUbiquitous,Highly Regulated Enzyme in Plants [J].Plant Mol Biol,1996,47:273-298.

