食蟹猴微卫星DNA标记遗传监测及多态性分析
李瑞生,李晓娟,高 蓉,白云峰,鲍龙涛,战大伟
(1.解放军第302医院动物实验中心,北京 100039;2.解放军总医院第一附属医院实验动物科,北京 100048)
猕猴是国家二级保护动物,在猕猴属中共有12个种46个亚种,其中食蟹猴主要分布在泰国、老挝、柬埔寨和缅甸北部等国家的热带雨林地区[1],而国内主要是通过人工饲养、驯化和繁殖,建立了人工生产繁殖种群。猕猴在生物进化上与人类的遗传物质有98.5%的同源性,其组织结构,生理代谢等功能与人类比较相似成为了最接近的实验动物,在生命科学以及其它相关学科研究中占据其它实验动物无法替代的重要位置[2,3]。目前应用于实验动物研究的猕猴主要是恒河猴,随着实验需求量的不断增加,恒河猴种群数量已远远不能满足日益增长的实验需求,而食蟹猴遗传背景特性研究又甚少,其生理、生化指标及遗传概貌特点还不十分清楚,研究掌握食蟹猴种群的遗传背景特性对大量应用食蟹猴进行各种实验研究具有十分重要的意义。因此,本实验利用所选取的100个微卫星DNA位点对食蟹猴种群进行了DNA多态性分析和遗传监测,了解食蟹猴种群的遗传背景特性,为今后建立食蟹猴种群资源库和遗传质量监测方法提供可靠的理论依据。
1 材料与方法
1.1 实验动物
食蟹猴来源于军事医学科学院实验动物中心自行繁殖的种群,随机抽取成年猴50只,雌雄各半,体重为1.5~3kg,年龄为2~4岁,具有实验动物质量合格证书(SCXK(军)-2007-004)。股静脉采血提取DNA,-20℃保存。
1.2 引物设计与合成
选取100个具有多态性高、等位基因数多和分布广的猕猴微卫星DNA位点,引物序列均来自Genbank和文献[4,5],由上海生物工程技术有限公司合成。
1.3 实验方法
利用血液提取基因组DNA作为模板,每个PCR扩增体系为25μL,35个循环,10μL扩增产物行8%非变性聚丙烯酰胺凝胶电泳,凝胶于固定液中固定20min,硝酸银染色30min,用无水碳酸钠显色,条带清晰后,用10%乙酸终止,最后用紫外凝胶成像系统进行凝胶成像,并对图带进行相关分析。
1.4 统计分析
用BioRad凝胶成像系统对8%非变性聚丙烯酰胺凝胶电泳图带结果进行分析,确定其核酸条带的分子量大小,然后再利用POPGEN32软件计算基因频率(gene frequency)、观察等位基因数(observed number of alleles,Na)、有效等位基因数(effective number of alleles,Ne)、基因多样性(gene diversity,H)和香隆信息指数(Shannon’s information index,I)[6]。
2 结果
2.1 根据等位基因数目筛选出20个多态性高的微卫星DNA位点
利用100个微卫星DNA位点对50只食蟹猴进行了DNA多态性分析,根据监测结果其等位基因数目的分布情况,从中筛选出20个等位基因数目在5条以上的微卫星DNA位点,并通过对比实验确定了各位点PCR扩增最佳退火温度、Mg2+浓度和图带片断大小的范围值,各位点的具体情况(表1)。
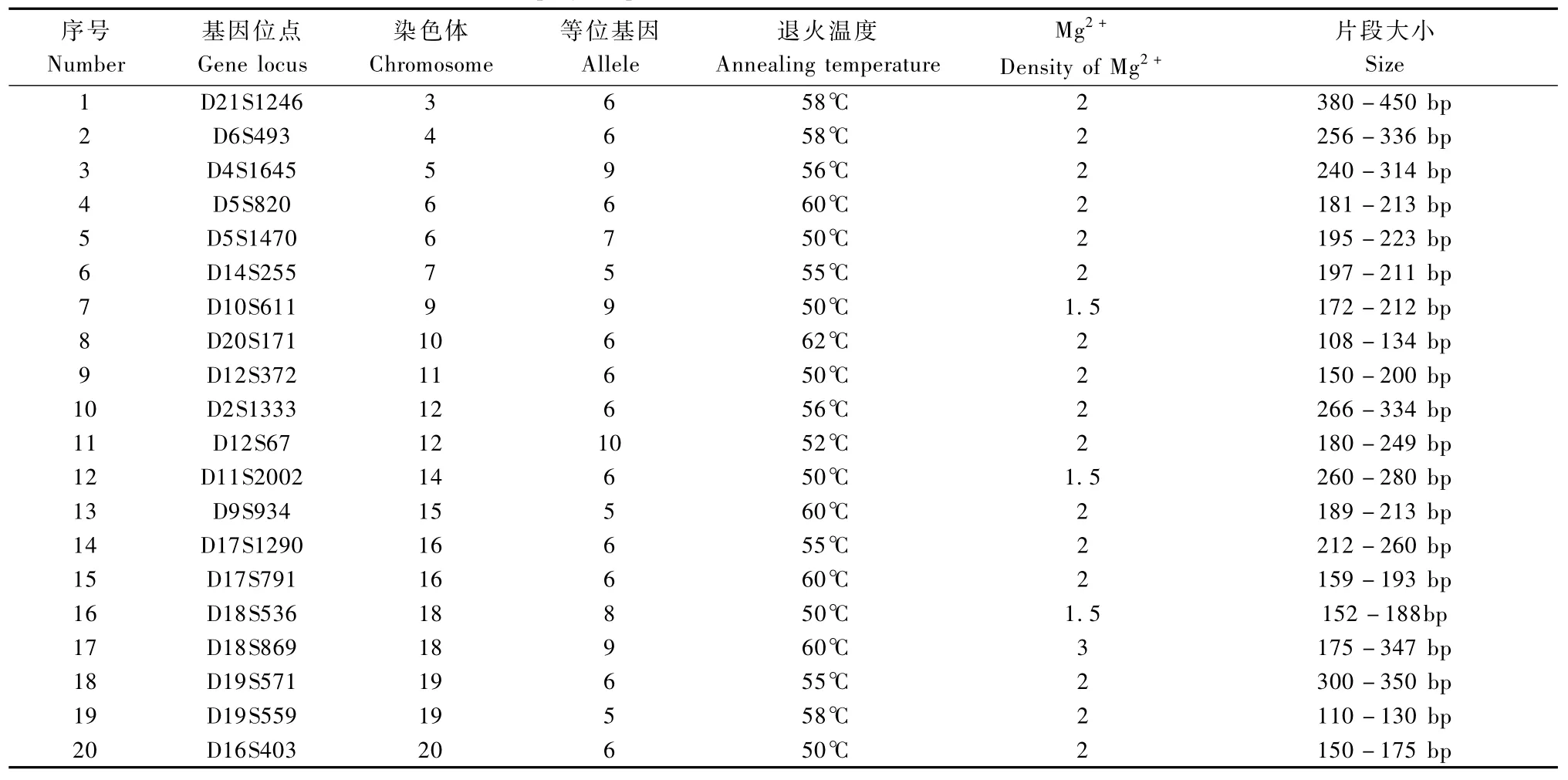
表1 20个食蟹猴多态性微卫星DNA位点Tab.1 20 polymorphic microsatellite DNA loci in Macaca mulatta
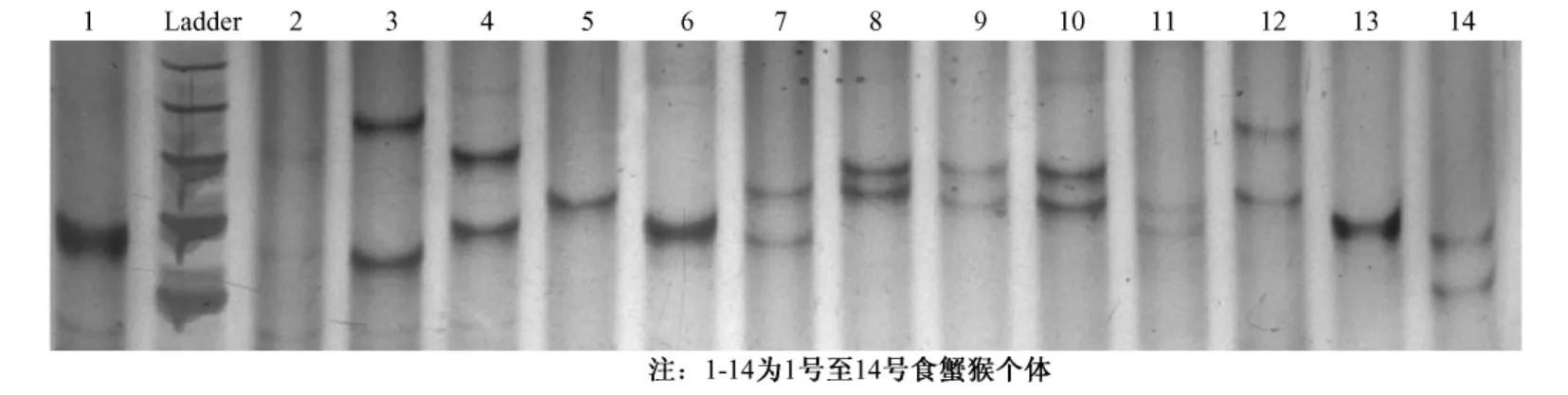
图1 D18S536位点扩增的DNA图谱注:1-14为1号至14号食蟹猴个体Fig.1 DNA mapping of amplification of D18S536 locusNote:1-14:individuals of Macaca fascicularis
2.2 食蟹猴种群不同个体间DNA多态性的分析
利用筛选出的20个多态性微卫星DNA位点对食蟹猴种群进行了DNA多态性分析,结果20个微卫星DNA位点均具有显著多态性,等位基因数最少达到5条,最多为10条,这些多态性位点能够进行个体间DNA多态性分析,并能较好地反映出群体的遗传概貌(图1)。
2.3 食蟹猴种群雌雄不同个体间多态性的统计分析
利用POPGEN32软件计算雌雄个体其观察等位基因数(observed number of alleles,Na)、有效等位基因数(effective number of alleles,Ne)、基因多样性(gene diversity,H)和香隆信息指数(Shannon’s information index,I),其分析结果(表2),其中雌雄个体间差异无显著性。
3 讨论
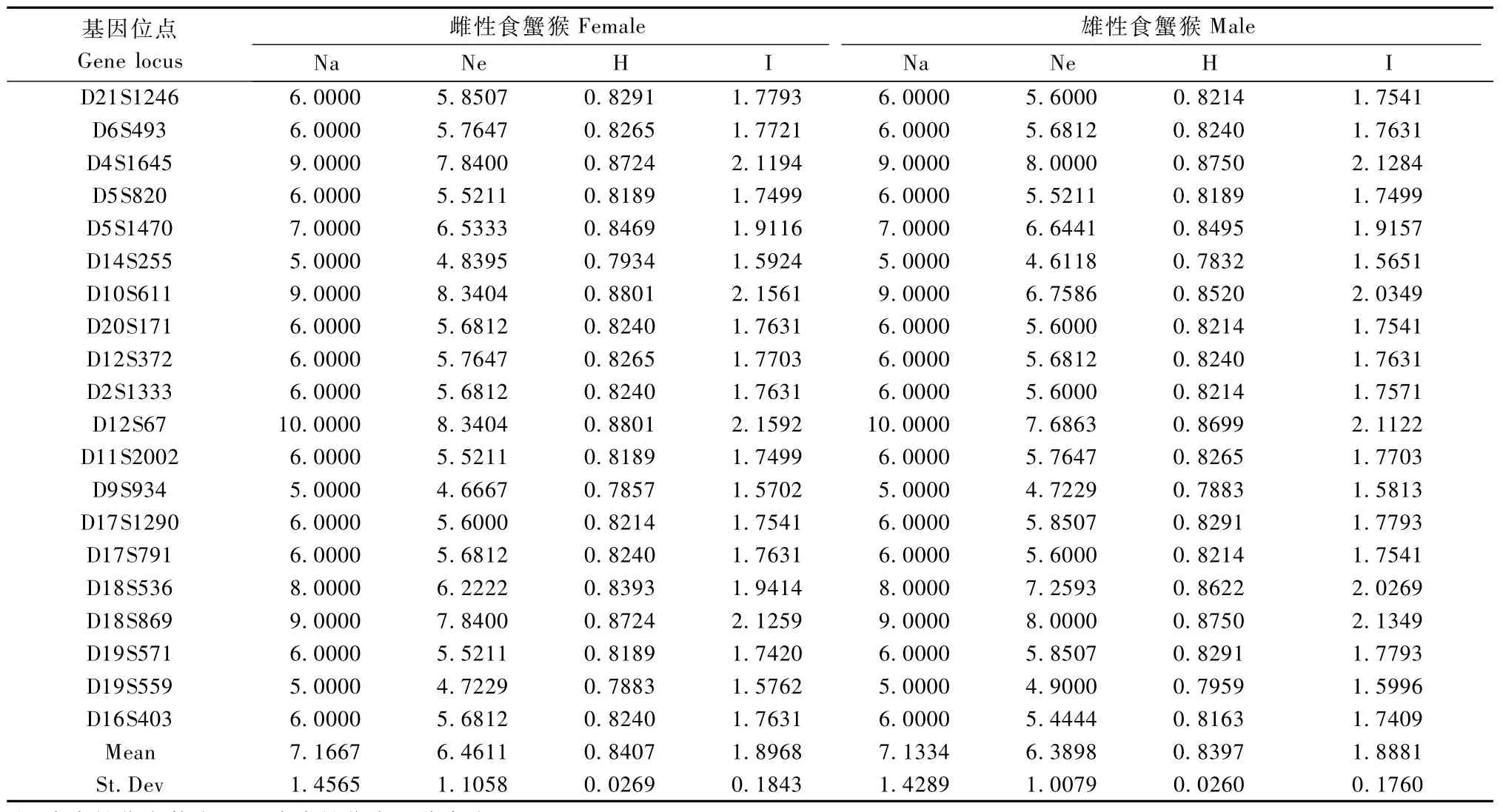
表2 雌雄个体观察等位基因数(Na)、有效等位基因数(Ne)、基因多样性(H)和香隆信息指数(I)的对比Tab.2 Observed number of alleles(Na),effective number of alleles(Ne),gene diversity(H),Shannon’s information index(I)between male and female Macaca fascicularis
猕猴在生物学、医学和药学等领域的实验研究中起到了非常关键性的作用,近年来国内外学者利用微卫星DNA标记技术对常用实验动物近交系大小鼠、家兔、犬和恒河猴进行了大量的实验研究[7-9],目前微卫星DNA标记技术已在近交系大、小鼠遗传质量监测中得到了有效的应用,在法医鉴定和人类的亲子鉴定中也同样得到了证实,可有效地运用于种群和个体间的等位基因、基因配型、亲子鉴定和遗传距离分析,使微卫星DNA标记技术得到了迅猛的发展,目前已被广泛地应用到实验动物的遗传质量监测研究中。Kanthaswamy S等[10-12]利用14个短串联重复位点(STRs)对加州国立灵长类研究中心的3种不同饲养类型的3382只具有遗传特征的纯种猕猴动物进行了遗传组成分析,同时利用13个STRs对亚洲食蟹猴、东南亚本土与岛国长尾食蟹猴(M.fascicularis)种群内以及种群间的遗传差异进行了评估。Flynn S[13]等对食蟹猴和长尾食蟹猴(Macaca fascicularis)TNF-α启动子的遗传变异性以及疟疾敏感性进行了分析。李瑞生等[14]应用微卫星DNA位点对恒河猴种群进行了DNA多态性分析,筛选出具有DNA多态性高的微卫星DNA位点,分析了恒河猴种群的遗传背景情况,证实了相同种群不同地区的恒河猴群体之间也存在差异。
本实验所选的食蟹猴群体主要是来源于军事医学科学院实验动物中心封闭繁殖5年以上的群体,没有引入外来血缘,繁殖严格按照实验动物的规定来进行生产繁殖,该群体没有出现遗传污染。利用筛选的20个多态性微卫星DNA位点对随机抽取的50只食蟹猴进行了DNA多态性和遗传监测分析,其等位基因数目都在5条以上,各位点均呈现显著的DNA多态性,均能较好地反映出食蟹猴群体中不同个体间的DNA多态性分布情况,完全符合封闭群实验动物的基本要求。因此,利用这些多态性微卫星DNA位点可有效地分析食蟹猴群体的遗传多样性,为今后建立完整的食蟹猴遗传背景资源库,分析研究基因配型、亲子鉴定及遗传距离分析,指导食蟹猴的生产繁殖,建立血缘系谱明确、遗传背景清楚的食蟹猴种群提供了理论依据。同时也为进一步筛选食蟹猴与恒河猴种群间的特异性微卫星DNA位点以及分析研究食蟹猴与恒河猴种群间遗传背景差异特性提供了理论基础。
[1]季芳,刘晓明,饶军华.分子标记在猕猴遗传多样性研究中的应用[J].中国实验动物学报,2006,14(1):75-78.
[2]Smith DG,Kanthaswamy S,Viray J,et al.Additional highly polymorphic microsatellite(STR)loci for estimating kinship in rhesus macaques(Macaca mulatta)[J].Am J Primatol.2000,50(1):1-7.
[3]李瑞生,赵爽,高荣,等.恒河猴与食蟹猴微卫星DNA多态性的比较分析[J].中国实验动物学报,2009,17(1):57-60.
[4]Kanthaswamy S,Smith DG.Effects of geographic origin on captive Macaca mulatta mitochondrial DNA variation[J].Comp Med.2004,54(2):193-201.
[5]Hadfield RM,Pullen JG,Davies KF,et al.Toward developing a genome-wide microsatellite marker set for linkage analysis in the rhesusmacaque(Macaca mulatta):identification of 76 polymorphic markers[J].Am J Primatol.2001,54(4):223-231.
[6]季芳,饶军华,刘晓明.应用微卫星标记对海南和广西恒河猴遗传多样性的研究[J].实验动物科学与管理,2006,23(2):26-30.
[7]Rogers J,Bergstrom M,Garcia R,et al.A panel of 20 highly variable microsatellite polymorphisms in rhesus macaques(Macaca mulatta)selected for pedigree or population genetic analysis[J].Am J Primatol.2005,67(3):377-383.
[8]Morin PA,Kanthaswamy S,Smith DG..Simple sequence repeat(SSR)polymorphisms for colony management and population genetics in rhesus macaques(Macaca mulatta)[J].Am J Primatol,1997,42:199-213.
[9]Kanthaswamy S,von Dollen A,Kurushima JD,etal.Microsatellite markers for standardized genetic management of captive colonies of rhesus macaques(Macaca mulatta)[J].Am J Primatol.2006,68(1):73-95.
[10]Kanthaswamy S,Kou A,Smith DG.Population genetic statistics from rhesus macaques(Macaca mulatta)in three different housing configurations at the California National Primate Research Center[J].J Am Assoc Lab Anim Sci.2010,49(5):598-609.
[11]Kanthaswamy S,Kou A,Satkoski J,et al.Genetic characterization of specific pathogen-free rhesus macaque(Macaca mulatta)populations at the California National Primate Research Center(CNPRC)[J].Am J Primatol.2010,72(7):587-599.
[12]Kanthaswamy S,Satkoski J,George D,et al.Interspecies hybridization and the stratification of nuclear genetic variation of rhesus(macaca mulatta)and long-tailed macaques(macaca fascicularis)[J].Int J Primatol.2008,29(5):1295-1311.
[13]Flynn S,Satkoski J,Lerche N,et al.Genetic variation at the TNF-alpha promoter and malaria susceptibility in rhesus(Macaca mulatta)and long-tailed(Macaca fascicularis)macaques[J].Infect Genet Evol.2009,9(5):769-777.
[14]李瑞生,孙琪云,孙岩松,等.恒河猴群微卫星DNA多态性的分析[J].中国实验动物学报,2005,13(1):51-54.

