miRNA检测技术进展
周殿明,蒋健晖
(化学生物传感与计量学国家重点实验室,湖南大学化学化工学院,湖南长沙410082)
0 引言
RNA是除了DNA外的基因分子生物学的又一庞大家族,miRNA就是这个家族中的一个非常重要的成员。1993年,Lee等在线虫(C.elegans)发现了第一个定时调控胚胎后期发育的基因lin-4[1],也就是第一个miRNA;同时发现它不编码任何蛋白质,但是可以抑制Lin-14蛋白表达,然而当时的这个发现并未引起太多的关注。直到2000年,Reinhart等又在线虫C.elegan中发现第二个miRNA-let-7[2],miRNA这一非编码基因家族的重要成员才开始在人们的眼前逐渐清晰起来。
miRNA是一类约22个碱基左右的非编码蛋白的RNA,通常在18~25个碱基之间,成熟的miRNA 5'端有一个磷酸基团,3'端是羟基。它主要通过信使RNA的直接剪切,或者间接抑制翻译来实现基因调控作用。miRNA广泛存在于各种真核生物中,它在生物体的产生过程如图1所示。
首先携带miRNA信息的基因通过RNA聚合酶Ⅱ转录生成较长的RNA,也就是通常所说的pri-miRNA,然后pri-miRNA会被一种叫做Drosha的RNA内切酶Ⅲ剪切成60~70个碱基的含有茎环结构的RNA,即pre-miRNA。随后,premiRNA被载体蛋白Exportin-5从细胞核运输到细胞质中。到了细胞质以后,pre-miRNA会从载体蛋白中脱落下来,被一种叫做Dicer的RNA内切酶Ⅲ最终剪切成成熟的miRNA。此时的miRNA是双链的结构,其中5'端热稳定性较差的一条链将被特异性地整合到RNA诱导的沉默复合物(RISC)中,再与完全匹配的目标信使RNA结合,或者通过诱导miRNA的降解,或者通过间接调节翻译,从而实现蛋白质表达的调控功能。

图1 miRNA在细胞内的产生过程及调控原理图Fig.1 Schematic of miRNA production and regulation in cell
从上面可以看出,miRNA由于其特殊的调控功能,不仅对生物的生长、发育和凋亡有着重要的影响,而且还与各种疾病比如神经疾病、血管疾病、肿瘤和免疫方面[3~5]的疾病有着密切的关系。因此miRNA的检测,对于医学、分子生物学都有着非常重要的意义。近年来已有大量的文章涉及了这方面的工作,该文就是基于此对于目前世界上各个研究小组关于miRNA检测方法的研究进展做了一个简要的综述。
1 基于表面杂交的检测技术
1.1 印迹杂交技术
印迹杂交技术(即nothern blotting)是最常用也是最早用于miRNA检测的方法[6~9],主要原理是将标记的与目标miRNA互补的DNA探针杂交到硝酸纤维素膜上,然后显影检测。该方法虽然已成为miRNA检测的金标准,但是仍然存在很多问题,比如灵敏度低,费时等。Zolta′n Havelda小组于2004年将通过引入锁核酸(LNA)对该方法进行了改进[10],由于杂交效率得到改善,所以灵敏度提高了大约10倍。虽然如此,高灵敏度,高通量,特异性好的检测技术仍然是个挑战。
1.2 阵列平台检测技术
miRNA的阵列检测技术主要是构建含有与目标miRNA互补的DNA的阵列,然后与目标杂交,再通过各种各样的信号检测方法实现检测目的。最常用的是通过聚合酶延伸的方法产生信号。2004年,Nelson等发展了一种名为基于阵列RNA引物介导的Klenow酶反应(RAKE)的检测技术[11](图2),该技术将DNA探针的5'端固定于玻璃表面,探针的3'端是与目标miRNA互补的序列,紧挨着互补序列的是三个胸腺嘧啶核苷酸残基,因此当目标存在时,将被探针捕获到基底表面,单链的探针可以通过外切酶I切除,此时加入生物素化的dATP和Klenow聚合酶可以延伸出生物素化的腺嘌呤碱基,再与染料标记的亲和素结合产生荧光信号。另外,依赖于Poly(A)聚合酶的延伸方法也被引入到miRNA的检测,Shingara等[12](图3)利用Poly(A)聚合酶以RNA为引物延伸出氨基修饰的尿嘧啶核苷酸,再利用染料与氨基修饰的尿嘧啶核苷酸交联,获得荧光检测信号。2006年,Fang等[13]同样用Poly(A)聚合酶的延伸方法实现miRNA的检测,不同的是延伸出的Poly(A)通过与Poly(T)修饰的纳米金粒子结合做为信号获得方式。
Liang等[14](图4)采用了对miRNA直接修饰的方法实现对其检测,将miRNA的3'端直接生物素修饰,然后再与亲和素化的量子点结合,从而得到荧光信号。该阵列的检测限可以达到0.4 fmol,检测的动力学范围为两个数量级。

图2 基于阵列RNA引物介导的Klenow酶反应(RAKE)的检测技术原理图(RNA-primed,array-based Klenow enzyme(RAKE)assay)Fig.2 Schematic of RNA-primed,array-based Klenow enzyme assay(RAKE)
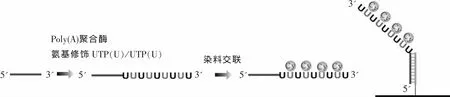
图3 基于Poly(A)聚合酶延伸修饰碱基U的miRNA阵列检测Fig.3 Schematic of miRNA microarray detection based on extension modified nucleotides U by Poly(A)

图4 生物素化的miRNA通过量子点进行阵列检测原理图Fig.4 Schematic of biotin modified miRNA profiling microarray detected with QD
DNA/RNA杂交体的特异性抗体被以一种新的标记方式引入到miRNA的检测中来,Hu等[15]构建了一个传感阵列,直接将未标记的目标miRNA与固定的DNA探针杂交,然后加入不依赖于序列信息的抗DNA/RNA杂交体的单克隆抗体,该抗体与染料标记的二抗结合,或者与生物素标记二抗和染料标记的亲和素反应,都可以得到检测信号。
除了上述的阵列检测平台以外,还有很多这方面的报道,比如利用生物发光酶标记的miRNA与待测miRNA竞争杂交到固定化的DNA探针上[16],通过检测生物发光强度实现目标检测的方法等等[17~20]。
1.3 电化学检测技术
硅纳米线[21](SiNWs)以其优越的性能被广泛应用于化学生物传感器,将肽核酸固定在其表面上可以作为检测miRNA的探针,当与检测对象miRNA进行杂交时,将会引起SiNWs界面电阻的变化,其变化值与miRNA浓度相关.该技术无需标记,检测miRNA浓度的下限可达1 fmol/L,具有良好的特异性。Lusi等[22](图5)发展了另一种无标记电化学检测技术,将用次黄嘌呤修饰的不含鸟嘌呤的DNA做为固定探针,目标miRNA存在时可以直接检测目标中鸟嘌呤的电化学信号,实现miRNA的检测。此外,Fan等[23]利用目标介导在纳米间隙电极上沉积导电聚合物纳米线的方法进行miRNA的检测。该方法首先将肽核酸固定于电极间隙中,与完全互补的目标链杂交以后,由于肽核酸不含电荷,而目标呈负电性,可以通过静电作用结合带有正电的苯胺,在酶催化的条件下,形成导电的聚苯胺纳米线,电极的导电性与目标物的含量成比例。该传感装置可以检测低至5 fmol/L的miRNA。除了无标记电化学的检测技术外,还可以通过目标miRNA直接修饰具有电化学催化活性的物质,比如氧化锇纳米粒子[24]和Ru(PD)2Cl2[25],这些电化学催化物质都可以催化肼的氧化反应,产生电化学信号,从而达到检测miRNA的目的。

图5 电化学检测次黄嘌呤修饰的捕获探针与目标miRNA杂交Fig.5 Electrochemical detection of the hybridization between the Inosine-modified capture probe and target miRNA
2 均相miRNA检测技术
2.1 基于PCR信号放大的检测技术

图6 基于茎环反转录引物的实时定量PCR技术(stem-loop RT-PCR)Fig.6 Real-time PCR based on stem-loop RT primer
PCR,即聚合酶链式反应,是一种DNA的指数扩增技术。美国科学家Mulis因发明了该技术获得了1995年诺贝尔化学奖。PCR技术通过两个短的称为引物(primer)的DNA分子,大约20个碱基左右,在一种耐热的DNA聚合酶的作用,可以在较短的时间内把极少的DNA量提高千万倍之多。PCR技术对分子生物学研究起到了巨大的推动作用,而且随着PCR技术的日趋完善,PCR在人类社会生活中的应用也越来越广泛。2005年,Chen等[26](图6)将反转录定量PCR技术引入到miRNA的检测当中,建立了名为茎环的反转录定量PCR技术(stem-loop RT-PCR)。他首先设计了一个茎环结构的反转录的探针,据报道这种结构大大提高了反转录的效率和特异性。当目标miRNA存在时,该探针与其杂交进行反转录产生cDNA,再加入正反引物和Taqman探针,进行定量PCR。该方法可以检测总RNA中低至25 pg的miRNA,实际上,高的灵敏度和特异性可以使该方法用于单个细胞的miRNA检测而不需要核酸纯化。该方法有很强的实用性,可以用于各种miRNA检测[27]。与此类似的,Raymond等[28]用一个普通的基因特异性线性探针进行杂交反转录,然后利用一个短的序列与被测物相关含有锁核酸的引物,和另一个通用的引物进行PCR反应,在一定程度上也可以提高特异性。与上面两种反转录PCR技术不同,Soroush及其研究团队[29]首先用一个含有六个碱基和目标互补的探针与miRNA杂交,该探针同时含有5'端多余的碱基,进行反转录后,产生含有5'端突出cDNA,此时加入同样5'端突出的与反转录产生部分序列互补的探针,该探针与反转录产物杂交相互为模板延伸,这样就相当于在miRNA的两测引入了通用序列,可以用通用引物扩增。该技术检测的动力学范围可达6~8个数量级,下限可达0.2 fmol/L。不过这些方法都是通过探针与目标miRNA直接杂交进行反转录的,由于miRNA序列短,所以需要用各种办法来提高杂交效率,但是有一种检测技术可以避免这个问题[30],通过Poly(A)聚合酶延伸miRNA产生反转录引物结合序列Poly(A),进行反转录,然后用同样的两个引物,一个是通用的,一个是miRNA特异的进行定量PCR扩增。2009年,Li等[31](图7)建立一种不依赖反转录的PCR检测技术,该技术设计了两个探针,分别由两部分组成,一部分可以与目标miRNA完全互补,另一部分是引物结合序列。这样,两个探针在与miRNA互补配对后,由连接酶连接成一条完整的定量PCR模板,加入上下游引物,则可以进行定量PCR反应。
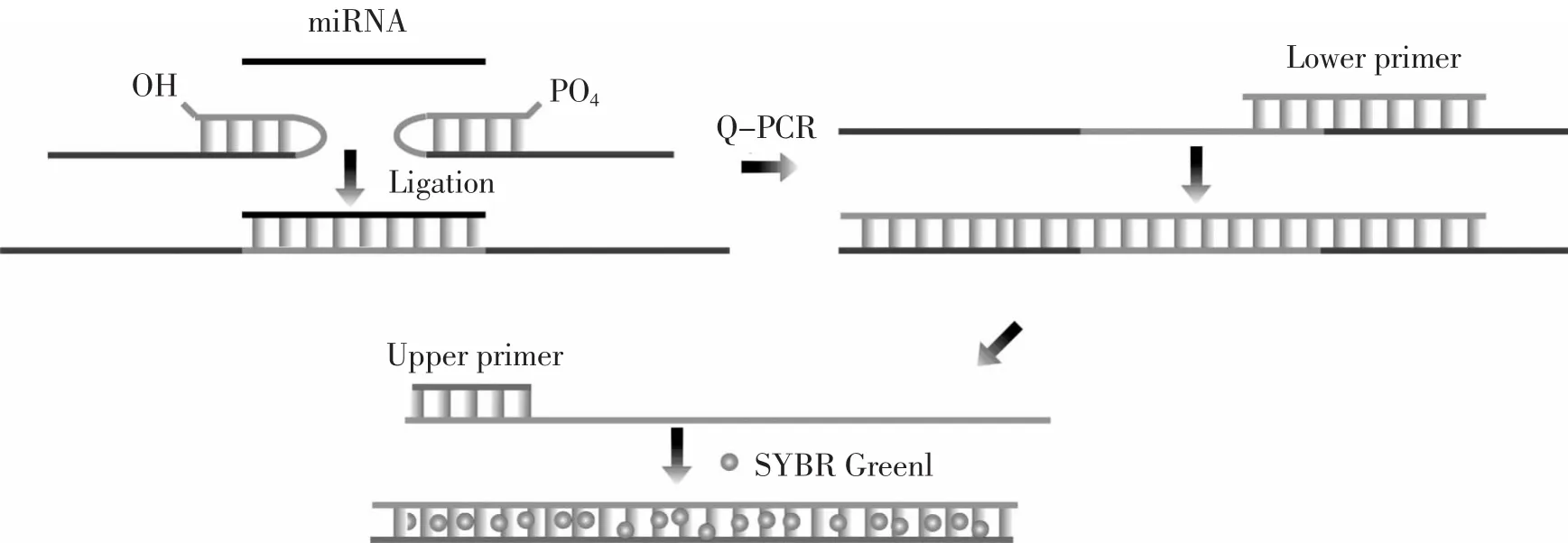
图7 基于酶连接实时PCR用于检测miRNAFig.7 Real-time PCR assay for detection of miRNA based on enzymatic ligation
2.2 基于RCA的检测技术
滚环扩增技术(Rolling Circle Amplification)也叫滚环复制技术,是一种等温扩增方法,它以环状DNA为模板,在引物存在时,聚合酶将连续复制出无数个环的互补序列拷贝。该技术已经成为生物分析领域的核心技术,得到了广泛的应用。2006年,Jonstrup等[32]将RCA技术用于miRNA的检测,miRNA首先做为模板将探针连成环,同时做为引物在聚合酶的作用下延伸聚合放大。Cheng等[33]在此基础上发展了miRNA检测的分支滚环扩增技术(branched rolling-circle amplification)。仍旧以miRNA为模板和引物,不同的是,引入了第二个引物,序列与环的一部分相同,可以与滚环产物杂交并延伸扩增,再通过聚合酶的链置换活性不仅可以扩增出环互补序列的无数个拷贝,还可以在引物2的作用下延伸出和环相同序列的无数个拷贝。最后通过染料嵌入实现信号检测。与前两种方法不同的是,Yao等[34]首先将引物与目标杂交进行反转录,反转录后的产物将以另一个桥探针为模板,连成环状分子。再加入引物1进行滚环扩增,加入引物2进行分支扩增。每次反应体系可以检测103~1010个拷贝的miRNA,动力学范围很宽,而且可以区分单碱基错配和区分成熟的miRNA及其前体(图8)。
2.3 其它均相检测技术
除了基于PCR和RCA为基础的检测技术外,还有很多均相检测技术,比如连接酶链式反应[35],阳离子聚合物[36],invader反应[37],基于核酶的信号放大检测技术[38],同位素标记连接技术[39],和将反转录与转录结合的信号检测技术等等[40]。

图8 基于RCA的miRNA检测技术(A[32],B[33],C[34])Fig.8 miRNA detection based on RCA
3 结论
综上所述,miRNA做为一种重要的基因表达调控手段,参与各种生物活动,和控制多种疾病的发生发展。其检测手段也越来越受到人们的重视,各种各样的检测技术已被运用到miRNA的检测之中,但是无论是界面检测技术还是均相检测技术,存在自身优点的同时都或多或少存在不足,获得高灵敏度,高通量,高选择性,高特异性的检测技术仍然需要生物分析专业研究者的进一步努力。
[1]Lee R C,Feinbaum R L,Ambros V.The C.elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lim-14[J].Cell,1993,75:843~854.
[2]Reinhart B J,Slack F J,Basson M,et al.The 21-nucleotide let-7 RNA regulates developmental timing in Caenorhabditis elegants[J].Nature,2000,403(6772):901~906.
[3]Lukiw W J,Zhao Y,Cui J G.An NF-kappaB-sensitive micro RNA-146a-mediated inflammatory circuit in Alzheimer disease and in stressed human brain cells[J].J Biol Chem,2008,283(46):31 315~31 322.
[4]Thum T,Gross C,Fiedler J,et al.MicroRNA-21 contributes to myocardial disease by stimulating MAP kinase signalling in fibroblasts[J].Nature,2008,456(7 224):980~984.
[5]Xiao C,Rajewsky K.MicroRNA control in the immune system:basic principles[J].Cell,2009,136(1):26~36.
[6]Reinhart B J,Slack F J,Basson M,et al.The 21-nucleotide let-7 RNAregulates developmental timing in Caenorhabditis elegans[J].Nature,2000,403(6 772):901~906.
[7]Lee R C,Ambros V.An extensive class of small RNAs in Caenorhabditis elegans[J].Science,2001,294(5 543):862~864.
[8]Lagos-Quintana M,Rauhut R,Lendeckel W,et al.Identification of novel genes coding for small expressed RNAs[J].Science,2001,294(5 543):853~858.
[9]Calin G A,Dumitru C D,Shimizu M,et al.Frequent deletions and down-regulation of micro-RNA genes miR15 and miR16 at 13q14 in chronic lymphocytic leukemia[J].Proc Natl Acad Sci U S A,2002,99(24):15 524~15 529.
[10]Válóczi A,Hornyik C,Varga N,et al.Sensitiveand specific detection of microRNAs by northern blot analysis using LNA-modified oligonucleotide probes[J].Nucleic Acids Res,2004,32(22):e175.
[11]Nelson P T,Baldwin D A,Scearce L M,et al.Microarraybased,high-throughput Gene expression profiling of microRNAs[J].Nat.Methods,2004,1:155~161.
[12]Shingara J,Keiger K,Shelton J,et al.An optimized isolation and labeling platform for accurate microRNA expression profiling[J].RNA,2005,11(9):1 461~1 470.
[13]Fang S,Lee H J,Wark A W,et al.Attomole microarray detection of microRNAs by nanoparticle-amplified SPR imaging measurements of surface polyadenylation reactions[J].J Am Chem Soc,2006,128(43):14 044~14 046.
[14]Liang R Q,Li W,Li Y,et al.An oligonucleotide microarray for microRNA expression analysis based on labeling RNA with quantum dot and nanogold probe[J].Nucleic Acids Res,2005,33(2):e17.
[15]Hu Z,Zhang A,Storz G,et al.An antibody-based microarray assay for small RNA detection[J].Nucleic Acids Res,2006,34(7):e52.
[16]Cissell K A,Rahimi Y,Shrestha S,et al.Bioluminescence-based detection of MicroRNA,miR21 in breast cancer cells[J].Analytical Chemistry,2008,80:2 319~2 325.
[17]Baskerville S,Bartel D P.Microarrayprofilingof microRNAs reveals frequent coexpression with neighboring miRNAs and host genes[J].RNA,2005,11(3):241~247.
[18]Wang H,Ach R A,Curry B.Direct and sensitive miRNA profiling from low-input total RNA[J].RNA,2007,13(1):151~159.
[19]Chen J,Lozach J,Garcia E W,et al.Highly sensitive and specific microRNA expression profiling using BeadArray technology[J].Nucleic Acids Res,2008,36(14):e87.
[20]Li J,Schachermeyer S,Wang Y,et al.Detection of microRNA by fluorescence amplification based on cationexchange in nanocrystals[J].Anal Chem.,2009,81(23):9 723~9 729.
[21]Zhang G J,Chua J H,Chee R E,et al.Label-free direct detection of MiRNAs with silicon nanowire biosensors[J].Biosens Bioelectron,2009,24(8):2 504~2 508.
[22]Lusi E A,Passamano M,Guarascio P,et al.Innovative electrochemical approach for an early detection of microRNAs[J].Anal Chem,2009,81(7):2 819~2 822.
[23]Fan Y,Chen X,Trigg A D,et al.Detection of MicroRNAs using target-guided formation of conducting polymer nanowires in nanogaps[J].J Am Chem Soc,2007,129(17):5 437~5 443.
[24]Gao Z,Yang Z.Detection of microRNAs using electrocatalytic nanoparticle tags[J].Anal Chem,2006,78(5):1 470~1 477.
[25]Gao Z,Yu Y H.Direct labeling microRNA with an electrocatalytic moiety and its application in ultrasensitive microRNA assays[J].Biosens Bioelectron,2007,22(6):933~940.
[26]Chen C,Ridzon D A,Broomer A J,et al.Real-time quantification of microRNAs by stem-loop RT-PCR[J].Nucleic Acids Res,2005,33(20):e179.
[27]Tang F,Hajkova P,Barton S C,et al.MicroRNA expression profiling of single whole embryonic stem cells[J].Nucleic Acids Res,2006,34(2):e9.
[28]Raymond C K,Roberts B S,Garrett-Engele P,et al.Simple,quantitative primer-extension PCR assay for direct monitoring of microRNAs and short-interfering RNAs[J].RNA,2005,11(11):1 737~1 744.
[29]Sharbati-Tehrani S,Kutz-Lohroff B,Bergbauer R,et al.miR-Q:a novel quantitative RT-PCR approach for the expression profiling of small RNA molecules such as miRNAs in a complex sample[J].BMC Mol Biol,2008,9:34.
[30]Shi R,Chiang V L.Facilemeansforquantifying microRNA expression by real-time PCR[J].Biotechniques.2005,39(4):519~525.
[31]Li J,Yao B,Huang H,et al.Real-time polymerase chain reactionmicroRNAdetectionbased on enzymatic stem-loop probes ligation[J].Anal Chem,2009,81(13):5 446~5 451.
[32]Jonstrup S P,Koch J,Kjems J.A microRNA detection system based on padlock probes and rolling circle amplification[J].RNA,2006,12(9):1 747~1 752.
[33]Cheng Y,Zhang X,Li Z,et al.Highly sensitive determination of microRNA using target-primed and branched rolling-circle amplification[J].Angew ChemInt Ed Engl,2009,48(18):3 268~3 272.
[34]Yao B,Li J,Huang H,et al.Quantitative analysis of zeptomole microRNAs based on isothermal ramification amplification[J].RNA,2009,15(9):1 787~1 794.
[35]Yan J,Li Z,Liu C,et al.Simple and sensitive detection of microRNAs with ligase chain reaction[J].Chem Commun(Camb),2010,46(14):2 432~2 434.
[36]Zhang Y,Li Z,Cheng Y,et al.Colorimetric detection of microRNA and RNase H activity in homogeneous solution with cationic polythiophene derivative[J].Chem Commun(Camb),2009,(22):3 172~3 174.
[37]Allawi H T,Dahlberg J E,Olson S,et al.Quantitation of microRNAs using a modified Invader assay[J].RNA,2004,10(7):1 153~1 161.
[38]Hartig J S,Grüne I,Najafi-Shoushtari S H,et al.Sequence-specific detection of MicroRNAs by signal-amplifying ribozymes[J].J Am Chem Soc,2004,126(3):722~723.
[39]Maroney P A,Chamnongpol S,Souret F,et al.A rapid,quantitative assay for direct detection of microRNAs and other small RNAs using splinted ligation[J].RNA,2007,13(6):930~936.
[40]Siva A C,Nelson L J,Fleischer C L,et al.Molecular assays for the detection of microRNAs in prostate cancer[J].Mol Cancer,2009,8:17.

