日本蟳线粒体16SrRNA和CO I基因序列比较分析*
高俊娜,刘 萍,李 健,潘鲁青,高保全,陈 萍
(1.中国水产科学院黄海水产研究所,山东青岛266071;2.中国海洋大学,山东青岛266003)
日本蟳线粒体16SrRNA和CO I基因序列比较分析*
高俊娜1,2,刘 萍1**,李 健1,潘鲁青2,高保全1,陈 萍1
(1.中国水产科学院黄海水产研究所,山东青岛266071;2.中国海洋大学,山东青岛266003)
对采自莱州湾和胶州湾的日本蟳野生群体的线粒体16S rRNA和CO I基因片段进行了PCR扩增和测序,分别得到长度为519和658 bp的碱基序列。研究结果显示这2个基因片段在种内的变异都较低,对2个基因同源序列分析表明,在线粒体16SrRNA基因片段中共检测到7个变异位点(包括6个单一变异位点,1个简约信息位点)和7种单倍型;在CO I基因中共检测到8个变异位点(包括6个单一变异位点,2个简约信息位点)和7种单倍型。通过统计变异位点、平均核苷酸差异数和核苷酸多样性指数,分析比较了两群体间的序列差异和遗传多样性水平,结果显示2个日本蟳野生群体的遗传多样性比较丰富。用M EGA 4.0软件构建了NJ和UPM GA系统树,基于16SrRNA基因片段的系统树显示梭子蟹科的4个属聚为两大支:日本蟳不同的单倍型先聚在一起,然后与青蟹属的3种蟹聚为一支;梭子蟹属的三疣梭子蟹、远海梭子蟹及塞氏梭子蟹聚在一起;再与美青蟹属的美洲蓝蟹、巴西蓝蟹等聚为一支。基于CO I基因显示日本蟳不同的单倍型先聚在一起,再与其他3种蟳聚为一支;梭子蟹属的远海梭子蟹和三疣梭子蟹相聚,然后与美青蟹属的2种蟹相聚为一支。该结果与传统分类一致。这些数据为我国日本蟳的种质资源保护和利用提供了基础的分子生物学依据。
日本蟳;16S rRNA基因;CO I基因;遗传多样性
日本蟳(Charybdis japonica)隶属甲壳纲(Crustacea),梭子蟹科(Portunidae),梭子蟹亚科(Portuninae),蟳属(Charybdis),俗称海蟳、石蟹、靠山红等,是一种中型海产食用蟹类,广泛分布于我国渤海、黄海、东海、南海沿岸岛礁区及浅海水域,是我国最普遍的常见蟹种类之一,在国外分布于日本、朝鲜、东南亚等地,属广温广盐性广布种。因其具有肉质鲜嫩,味道鲜美,营养丰富,生长快等特点,已逐渐成为我国重要的海水养殖品种之一[1-2]。
国内外对日本蟳的研究主要集中在形态、繁殖、行为、组织学、生理、生化遗传等方面[3-11],对日本蟳线粒体DNA基因方面的研究,仅有关于12S rRNA基因[12]和16S rRNA基因[13]的报道。线粒体DNA由于具有分子量小、结构简单、母系遗传、进化速度快等特点,已被广泛应用于动物群体遗传学和系统进化方面的研究。本实验拟通过对线粒体16S rRNA和CO I基因序列的扩增和测序,对采自胶州湾和莱州湾的日本蟳野生群体进行遗传多样性分析,以期为我国日本蟳种质资源的管理和可持续利用提供数据参考。
1 材料与方法
1.1 实验材料
实验用日本蟳分别于2008年采自我国胶州湾和莱州湾,均为野生群体,群体大小为60个样本,活体带回实验室,-80℃保存。
1.2 实验方法
1.2.1 基因组DNA的提取与检测 从每个群体中随机选17~20个样本,每个样本取约100 g螯足肌肉,剪碎组织,加入475μL组织匀浆缓冲液(10 mmol/L Tris-HC1,p H=8.0;50 mmol/L EDTA,p H=8.0),充分混匀,依次加入终浓度为10%的SDS和20μg/mL的蛋白酶K,55℃裂解至澄清。采用苯酚-氯仿法提取。
依次加入终浓度为10%的SDS和20μg/mL的蛋白酶K,55℃裂解至澄清。采用苯酚-氯仿法提取基因组DNA。用DNA定量仪测定样品DNA的浓度和纯度,-20℃保存备用。1%琼脂糖凝胶电泳检测,全自动凝胶成像系统拍照。
1.2.2 PCR扩增 16S rRNA基因片段扩增的引物[14]为:16S AR:5-CGCCTGTTTA TCAAAAACA T-3′,16S BR:5′-CCGGTCTGAACTCAGA TCACG-3′;CO I基因片段扩增的引物为:CO IL1490:5′-GGTCAACAAA TCA TAAAGA TA TTGG-3′,CO IH2198:5′-TAAACTTCA GGGTGACCAAAAAA TCA-3′。
PCR反应总体积为50μL,其中含模板DNA 50-100ng,10×PCR buffer 2.5μL,上、下游引物(浓度10μmol/L)各1.2μL,dN TPs(浓度2 mmol/L)4μL,M gCl2(浓度25 mmol/L)4μL,Taq酶1 U。在eppendorf-PCR仪上进行PCR反应:94℃预变性2 min;94℃变性45 s,48℃退火1 min,72℃延伸1 min,35个循环;72℃延伸5 min。
1.2.3 DNA序列测定 扩增产物用1.5%琼脂糖凝胶电泳检测,将PCR产物送往上海生物工程有限公司,纯化后直接进行双向测序。
1.2.4 序列分析 将测得的全部序列用Bioedit软件进行编辑并辅以人工核查,用Clustal X 1.83软件比对,并确定序列长度。用DnaSP 5.0软件计算群体的遗传多样性参数。M EGA 4.0软件计算序列的碱基组成、变异位点、简约信息位点以及不同群体间的Kimura2-param ter遗传距离,用邻接法(NJ)构建系统树,系统树各结点的支持率以序列数据集1 000次重复抽样检验的自引导值(Bootstrap value)表示。用于平均核苷酸差异比对的GenBank上11种蟹为:远海梭子蟹(Portunus pelagicus)、塞氏梭子蟹(Portunus sayi)、三疣梭子蟹(Portunus trituberculatus)、Scy lla tranquebarica、锯缘青蟹(Scy lla serrata)、榄绿青蟹(Scy lla olivacea)、美洲蓝蟹(Callinectes sapidus)、巴西蓝蟹(Callinectes bellicosus)、Callinectes exasperatus、Callinectes bocourti、Callinectes arcuatus和4种蟳:钝齿蟳(Charybdis hellerii)、锈斑蟳(Charybdis feriatus)、Charybdis vadorum和锐齿蟳(Charybdis acuta),其外缘序列登录号及片段长度信息见表1。
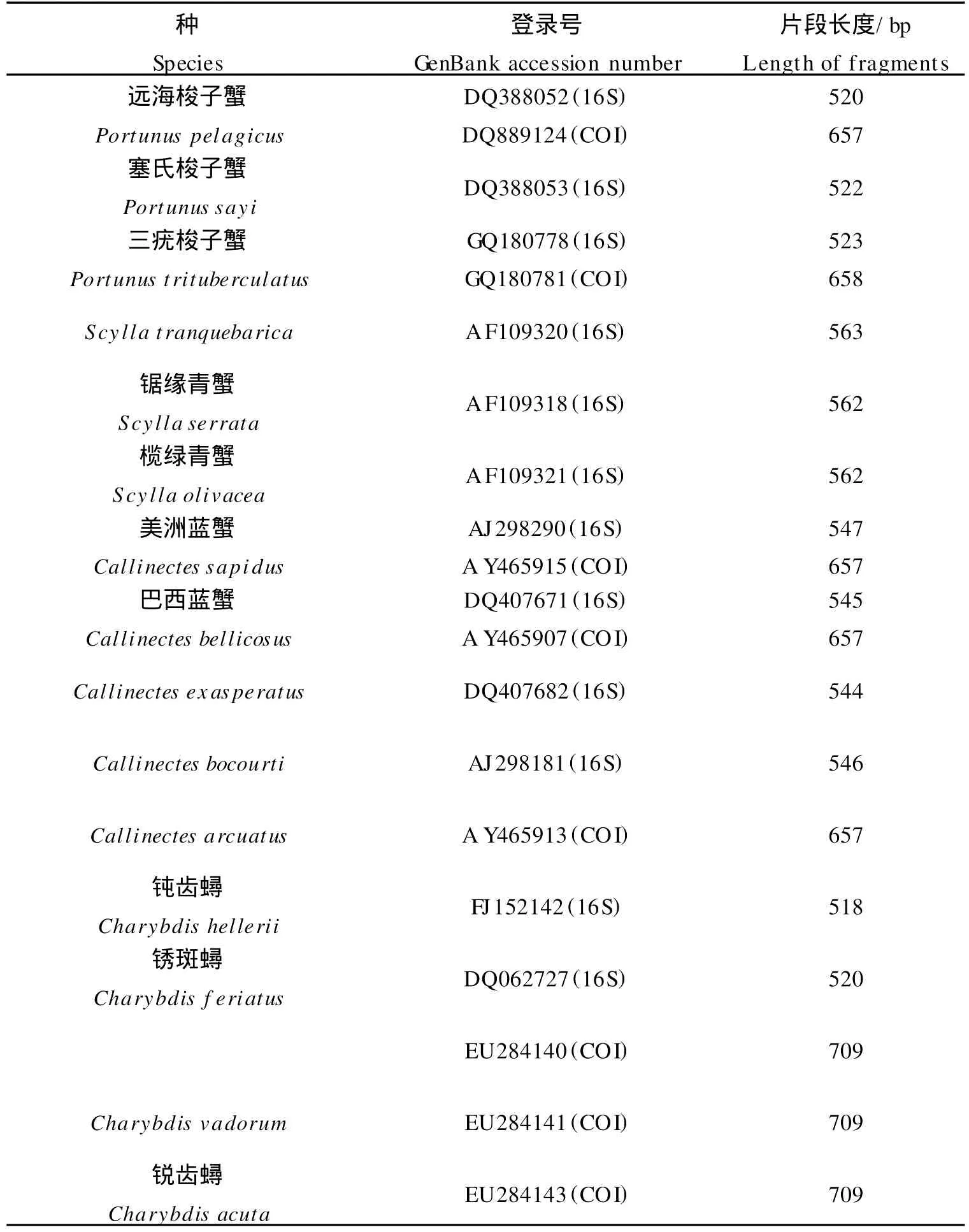
表1 外源序列物种名称,登录号和长度Table 1 Species,accession number and length of outgroup sequences
2 结果与分析
2.1 线粒体16SrRNA基因片段序列分析
对2个群体39个样品的线粒体16S rRNA基因片段进行PCR扩增和序列测定,得到长度为519 bp的基因片段,共出现7种单倍型(Hap lotype 1,2,3,4,5,6,7),已在GenBank中注册(登陆号码为HM 237590-HM 237596)。莱州湾群体检测到5个单倍型(Hap lotype 1,3,4,6,7),胶州湾检测到4个单倍型(Hap lotype 1,2,5,6),其中Hap lotype1和Hap lotype6为共享单倍型。共检测到7个变异位点,包括6个单一变异位点,1个简约信息位点。莱州湾群体检测到5个多态位点:4个单一变异位点和1个简约信息位点;胶州湾群体检测到3个多态位点:2个单一变异位点和1个简约信息位点。2个群体各碱基含量基本一致,A、T、G、C含量平均为34.6%、36%、17.7%、11.8%,A+T含量明显大于G+C含量。统计发现(见表2),莱州湾群体的各项遗传多样性参数略高于胶州湾,相差不大。

图1 日本蟳不同单倍型16S rRNA序列的变异位点Fig.1 Variable sites of 16SrRNA sequence from hap lotypesof Charybd is japonica
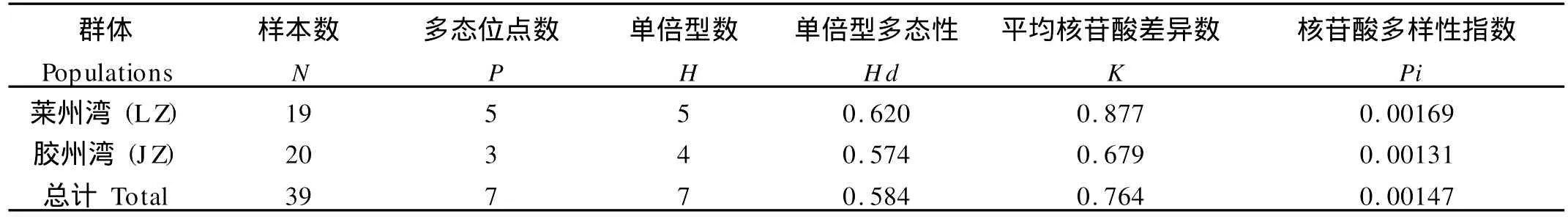
表2 日本蟳2个野生群体16SrRNA基因片段的遗传多样性参数Table 2 Genetic diversity parameter of 16S rRNA gene fragments among 2 w ild populations of Charybdis japonica
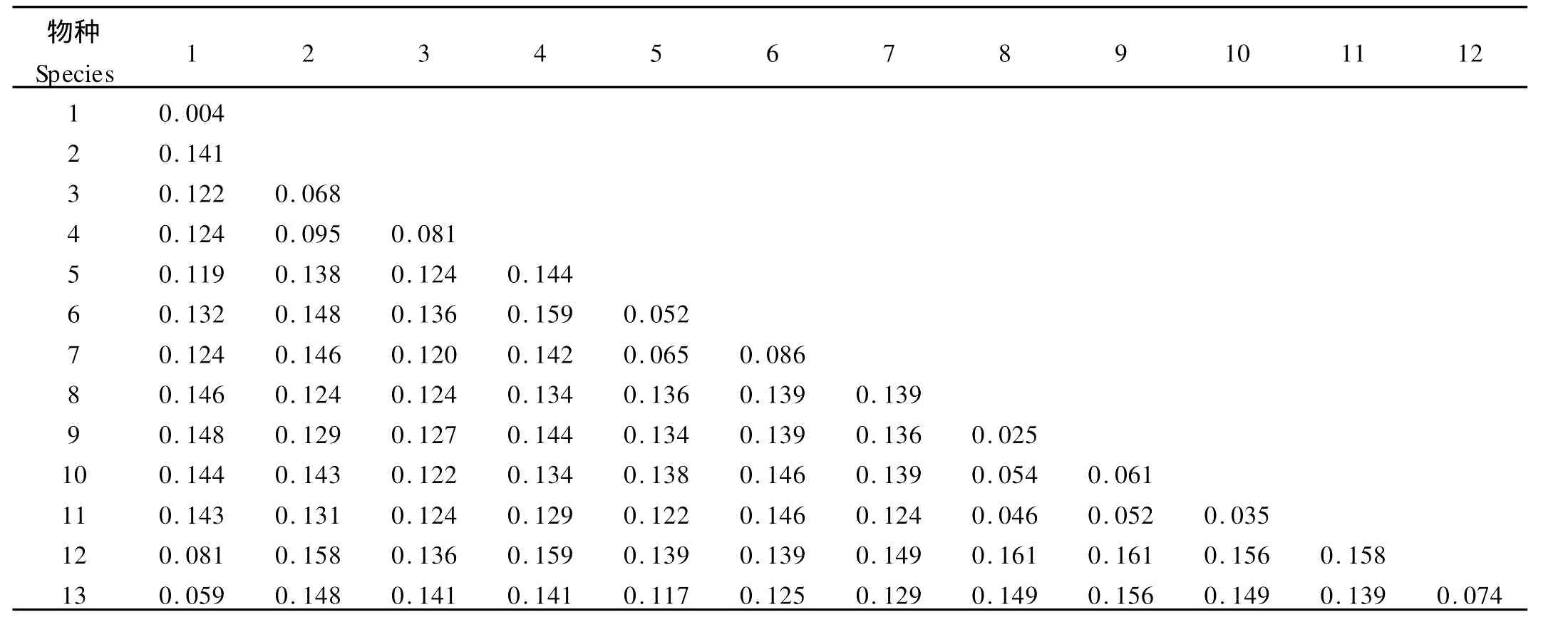
表3 基于16SrRNA基因片段计算的日本蟳各单倍型及外群间的遗传距离Table 3 Genetic distances of 16SrRNA gene fragments among Charybdis japonicas hap lo types and outgroups
用M EGA 4.0软件,将2群体的7个单倍型分别与远海梭子蟹等10种蟹和2种蟳的对应16S rRNA基因片段序列分别用NJ法和UPM GA法进行聚类分析,经1 000次bootsrap检验后获得置信度,再用Kimura双参法算出个体间遗传距离(见表3)。从表中可以看出日本蟳种内的平均遗传距离为0.004,与梭子蟹属的3种蟹之间的遗传距离0.122~0.141,与青蟹属的3种蟹之间的遗传距离0.119~0.132,与美青蟹属之间的遗传距离0.143~0.148,而与蟳属其他2种之间的遗传距离0.059和0.081,种内遗传距离远小于种间遗传距离,同属间物种的遗传距离远小于不同属间物种的遗传距离。图2为基于16SrRNA基因片段构建的NJ和UPM GA系统树,2种方法构建的系统树趋势一致,梭子蟹科的4个属聚为两大支:日本蟳不同的单倍型先聚在一起,再和青蟹属的3种蟹聚为一支;梭子蟹属的、三疣梭子蟹、远海梭子蟹及塞氏梭子蟹聚为一支;再和美青蟹属的美洲蓝蟹、巴西蓝蟹等聚为一支。
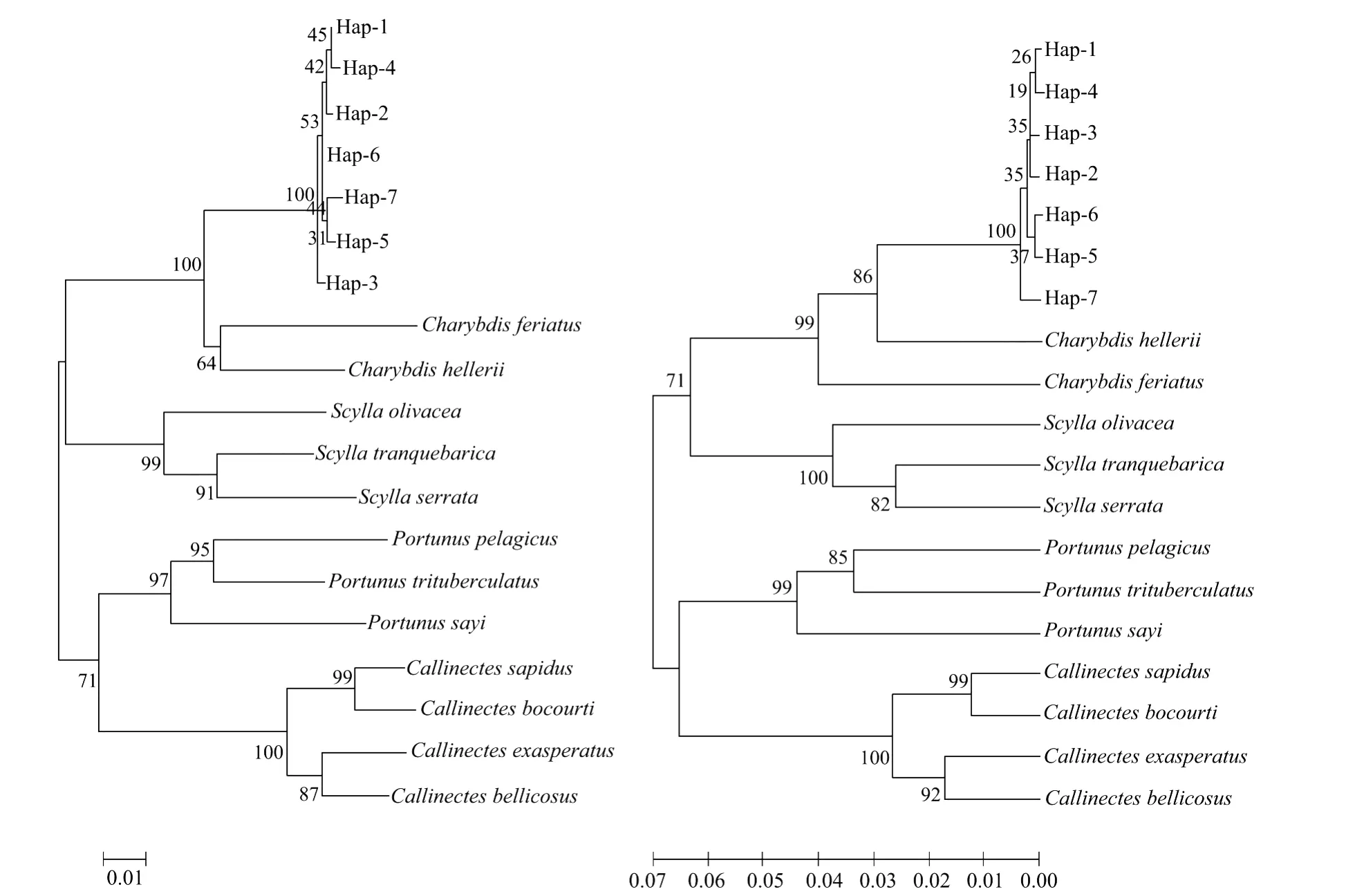
图2 基于16S rRNA基因构建的NJ系统树和UPM GA系统树Fig.2 NJ and UPGMA phylogenetic tree based on 16SrRNA gene sequences
2.2 线粒体CO I基因片段序列分析
对2个群体35个样品的线粒体CO I基因片段进行PCR扩增和序列测定,得到长度为658 bp的基因片段,共出现7种单倍型(Hap lotype 1,2,3,4,5,6,7),已在GenBank中注册(登陆号码为HM 237597-HM 237603)。莱州湾群体检测到4个单倍型(Hap lotype 1,2,5,6),胶州湾检测到6个单倍型(Hap lotype 1,2,3,4,6,7),其中Haplotype 1和Haplotype 6为共享单倍型,共检测到8个变异位点,包含6个单一变异位点,2个简约信息位点,莱州湾群体检测到3个多态位点,胶州湾群体检测到7个多态位点。2个群体各碱基含量基本一致,A、T、G、C含量平均为26.7%、36.9%、17.8%、18.6%,A+T含量明显高于G+C含量,符合无脊椎动物线粒体DNA序列特征。表4为日本蟳2个野生群体CO I基因片段的遗传多样性参数结果,可见莱州湾群体的各项遗传多样性参数值低于胶州湾。表5为基于CO I基于片段计算的日本蟳种内和种间遗传距离结果,可见日本蟳种内平均遗传距离为0.004,种间的遗传距离在0.140~0.241之间。图4为基于日本蟳CO I基因序列采用NJ法和UPM GA法构建分子进化树,2种方法构建的系统树趋势一致:日本蟳不同的单倍型先聚在一起,再和其他3种蟳聚为一支;梭子蟹属的远海梭子蟹和三疣梭子蟹相聚,然后和美青蟹属的2种蟹相聚为一支。

图3 日本蟳不同单倍型COⅠ序列的变异位点Fig.3 Variable sites of COⅠsequence from hap lotypes of Charybd is japonica
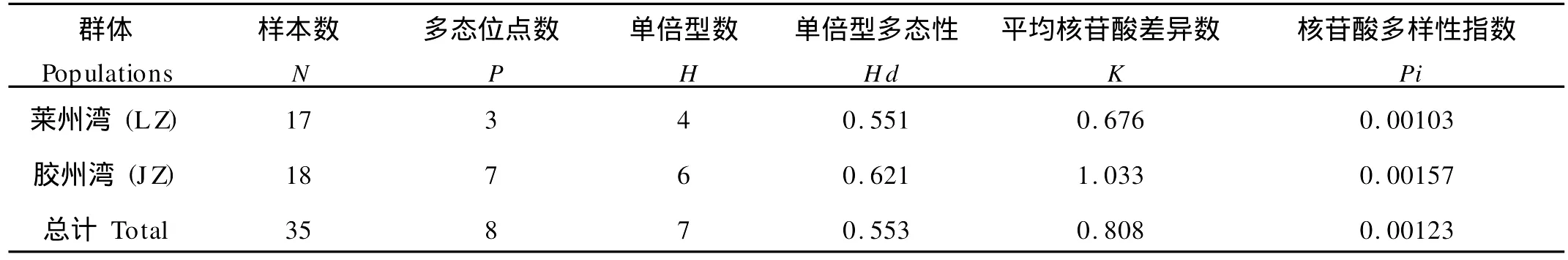
表4 日本蟳2个野生群体CO I基因片段的遗传多样性参数Table 4 Genetic diversity parameter of COIgene fragments among 2 wild populations of Charybd is japonica
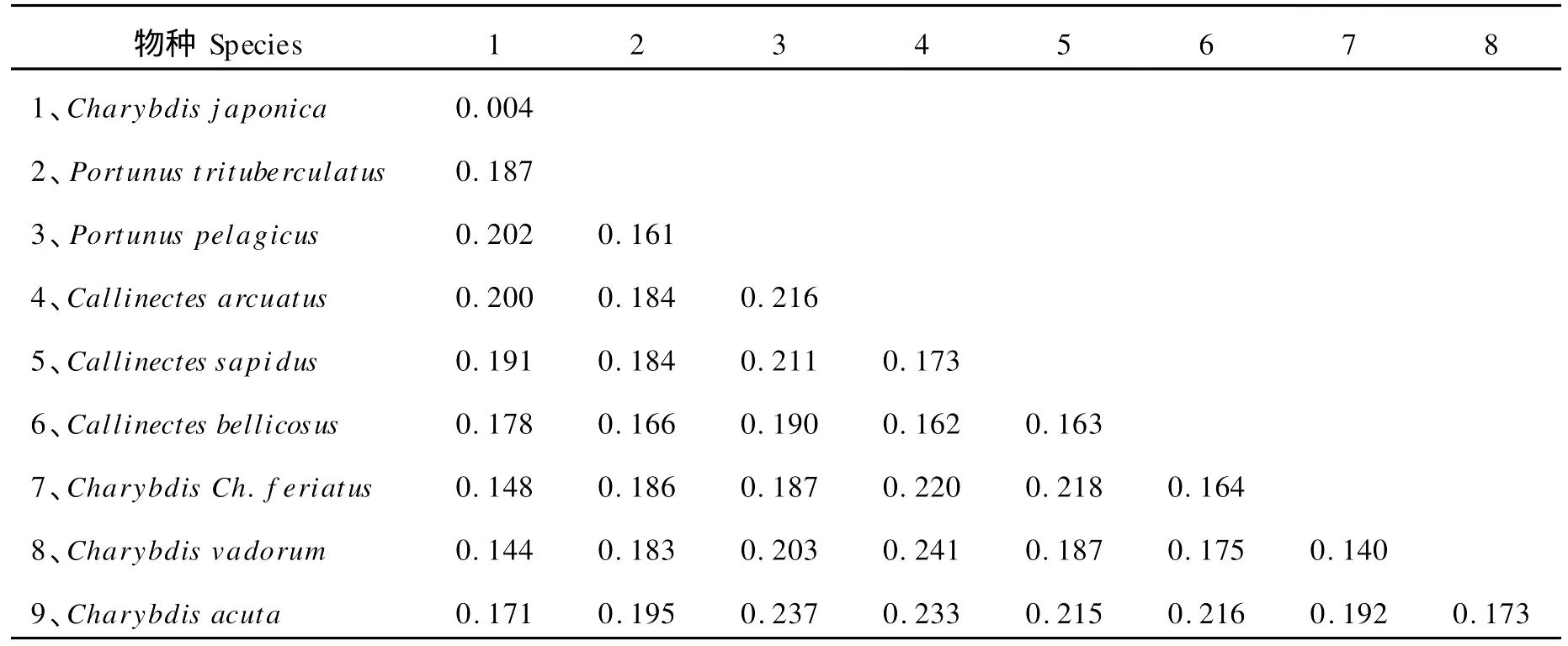
表5 基于CO I基因片段计算的日本蟳群体内和群体间遗传距离Table 5 Intraspecefic and interspecefic genetic distances based on CO Igene
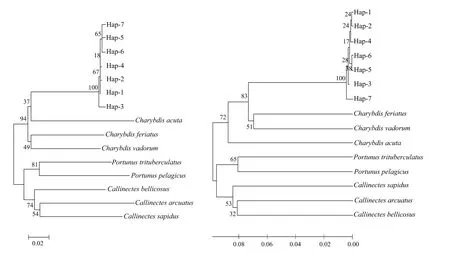
图4 基于COI基因构建的NJ和UPM GA系统树Fig.4 NJ and UPGMA phylogenetic tree based on CO Igene sequences
3 讨论
线粒体DNA由于具有进化速度快、变异性大、母系遗传、易于扩增等优点,成为动物群体进化研究中的一种重要的分子标记[15-17]。线粒体序列分析成为DNA多态性分析技术中最有效的方法之一,因为不论是个体还是群体,任何遗传变异或多态最终都是DNA序列的差异,DNA序列分析可以检测到碱基替换、插入和缺少等变异信息,从而在分子水平检测个体、群体及种间的多态及遗传差异[18-23]。因此,研究线粒体DNA的多态性,对生物不同群体间的遗传结构变异和遗传多样性的研究有着重要的意义。本文对日本蟳16S rRNA和CO I基因片段进行了PCR扩增和测序,分别得到519和658 bp的碱基序列,2个基因同源序列分析表明,A+T碱基含量明显高于G+C,这与许多研究者在虾类、蟹类等无脊椎动物的线粒体基因中观察到的结果相似[24-25]。本文对日本蟳16S rRNA和CO I 2个基因片段遗传特征的研究,显示两者在种内的变异都比较低。
本研究从线粒体16S rRNA和CO I基因部分序列获得了莱州湾和胶州湾野生日本蟳的遗传多样性参数,由于基于16S rRNA获得的遗传多样性参数较少,仅以日本囊对虾(M arsupenaeus japonicus)和三疣梭子蟹的核苷酸多样性指数(0.0022,0)[14,26]做对比,基于CO I基于的遗传多样性参数,对比中国对虾(Fenneropenaeus chinensis)、保罗美对虾(Penaeus pau lensis)、巴西美对虾(Farf antepenaeus brasiliensis),日本囊对虾和南方滨对虾(Penaeus schmitti)的COⅠ基因片段核苷酸算多样性指数(0.000 40,0.000 00,0.000 40,0.004 70)[26-28],以及野生三疣梭子蟹COⅠ基因片段核苷酸算多样性指数(0.001 09)[14],在一定程度上说明了这2个采样地的日本蟳的遗传多样性相对来说比较丰富。
通过与来自GenBank的11种蟹和4种蟳序列比较,分别采用NJ法和UPM GA法构建了分子系统发育树,结果显示,2种方法构建的系统树趋势一致:基于16SrRNA基因片段梭子蟹科的4个属聚为两大支:日本蟳不同的单倍型先聚在一起,再和青蟹属的3种蟹聚为一支;梭子蟹属的三疣梭子蟹、远海梭子蟹及塞氏梭子蟹聚在一起,再和美青蟹属的美洲蓝蟹、巴西蓝蟹等聚为一支。基于CO I基因序列显示日本蟳不同的单倍型先聚在一起,再和其他3种蟳聚为一支;而梭子蟹属的远海梭子蟹和三疣梭子蟹相聚,然后和美青蟹属的2种蟹相聚为另一支。该结果与传统分类一致。
本文测定的日本蟳线粒体16SrRNA和CO I基因片段序列,为更准确地探讨日本蟳群体遗传变异与分化及其与梭子蟹科其他种之间的系统进化关系以及为进行日本蟳种质资源保护和遗传多样性研究提供了新的材料。
[1] 戴爱云,杨思谅,宋玉枝,等.中国海洋蟹类[M].北京:海洋出版社,1986:194-196.
[2] 吴常文,王志铮,王伟洪,等.舟山近海日本蟳Charybdis japonica生物学、资源分布以及开发利用[J].浙江水产学院学报,1998,17(1):13-18.
[3] 王春琳,薛良义,刘凤燕,等.日本蟳繁殖生物学的初步研究-雌性繁殖习性[J].浙江水产学院学报,1996,15(4):261-266.
[4] 俞存根,宋海棠,姚光展.东海日本蟳的数量分布和生物学特性[J].上海水产大学学报2005,14(1):40-45.
[5] 崔龙波,刘萍,王延波,等.日本蟳消化系统的组织学研究[J].烟台师范学院学报:自然科学版,2000,16(1):51-54.
[6] 赵青松,陈惠群,王春琳,等.日本蟳血淋巴细胞的初步研究[J].水利渔业,2005,25(3):13-15.
[7] 王春琳,丁爱侠.硫酸铜蓄积对日本蟳体内保护酶系统的影响[J].大连水产学院学报,2005,20(4):278-282.
[8] 樊廷俊,于苗苗,杨玲玲,等.四种免疫促进剂对日本蟳酚氧化酶和血细胞的影响[J].中国海洋大学学报:自然科学版,2009,39(3):421-427.
[9] 张红霞,潘鲁青,刘静.重金属离子对日本蟳血淋巴抗氧化酶(SOD,CA T,GPx)活力的影响[J].中国海洋大学学报:自然科学版,2006,36增刊:49-53.
[10] 宋微微,王春琳,宋超霞,等.日本蟳同工酶组织特异性及生化遗传分析[J].水利渔业,2006,26(5):17-20.
[11] Seong Ho Ryu,Yoon Kyung Do,Ui Wook Hwang.Ribosomal DNA Intergenic Spacerof the Swimming Crab,Charybdis japonica[J].Molecular Evolution.1999,49:806–809.
[12] 高天翔,张秀梅,渡边精,等.日本蟳和底栖短桨蟹线粒体DNA 12S rRNA基因序列的初步研究[J].中国海洋大学学报:自然科学版,2004,34(1):43-47.
[13] 马春艳,马凌波,沈盎绿,等.浙江三门湾日本蟳群体线粒体16S rRNA基因序列多态性[J].海洋渔业,2009,31(2):134-138.
[14] 戴艳菊,刘萍,高保全,等.三疣梭子蟹四个野生群体线粒体16S rRNA和COI片段的比较分析[J].中国海洋大学学报:自然科学版,2010,40(3):1-7.
[15] Imai H,Cheng J H,Hamasaki K,et al.Identification of four mud crab species(genus Scy lla)using ITS-1 and 16SrDNA markers[J].Aquatic Living Resources,2004,17:31-34.
[16] Klinbunga S,Boonyapaladee A,Pratoomchat B.Genetic diversity and species-diagnostic markers of mud crabs(genus Scy lla)in eastern Thailand determined by RAPD analysis[J].Marine Biotechno1ogy,2000,2:180-187.
[17] 陈丽梅,李琪,李赟.4种海参16S rRNA和COI基因片段序列比较及系统学研究[J].中国水产科学,2008,15(6):935-942.
[18] Grant W S,Bowen B W.Shallow population histo ries in deep evolutionary lineages of marine fishes:insights from sardines and anchovies and lessons for conservation[J].Heredity,1998,(89):145-426.
[19] 孔晓瑜,喻子牛,刘亚军,等.中华绒螯蟹与日本绒螯蟹线粒体COl基因片段的序列比较研究[J].青岛海洋大学学报:自然科学版,2001,31(6):861-86.
[20] 季维智,宿兵.遗传多样性研究的原理与方法[M].杭州:浙江科学技术出版社,1999:3-7.
[21] 邱高峰,常林瑞,林巧婷,等.中国对虾16S rRNA基因序列多态性的研究[J].动物学研究,2000,21(1):35-40.
[22] 高天翔,李健,王清印,等.中国对虾线粒体16S rRNA基因序列分析[J].中国水产科学,2003,10(5):59-62.
[23] 麦维军,谢珍玉,张吕平,等.国明对虾与5种虾类线粒体16S rRNA和COI基因片段的序列比较及其系统学初步研究[J].海南大学学报:自然科学版,2009,27(1):15-23.
[24] 马凌波,张凤英,乔振国,等.中国东南沿海青蟹线粒体COI基因部分序列分析[J].水产学报,2006,30(4):463-468.
[25] 郑芳,吕秀玲,孙红英,等.基于线粒体COI基因序列探讨长江华溪蟹的遗传分化[J].南京师大学报:自然科学版,2006,29(2):103-105.
[26] 谭树华,王桂忠,李少菁.中国沿海日本囊对虾线粒体16S rRNA基因序列变异及遗传分化[J].生态学报,2009,29(12):6805-6810.
[27] Quan J X,Zhuang ZM,Deng J Y,et al.Low genetic variation of Penaeus chinensis as revealed by mitochondrial DNA COⅠand 16SrRNA gene sequences[J].Biochemical Genetics,2001,39:279-284.
[28] Gusmao J,Lazoski C,Sole-Cava A M.A new speciesof Penaeus(Crustacea:Penaeidae)revealed by allozyme and cytochrome oxidase Ianalyses[J].Marine Biology,2000,137:435-446.
Compared Analysis on the Sequences of Mitochondrial 16S rRNA Gene and CO IGene Fragments of Charybdis japonica
GAO Jun-Na1,2,L IU Ping1,L IJian1,PAN Lu-Qing2,GAO Bao-Quan1,CHEN Ping1
(1.Yellow Sea Fisheries Research Institute,Chinese Academy of Fishery Sciences,Qingdao 266071,China;2.Ocean University of China,Qingdao 266003,China)
519 base sequence of the 16S rRNA gene and 658 base sequence of the cytochrome oxidase 1 gene were amp lified and sequenced in specimens of Charybdis japonica from Laizhou Bay and Jiaozhou Bay.The analysis of the homology sequences of 16S and CO Igene indicated that little sequence variation wasobserved within species.A total of 7 or 8 nucleotide siteswere detected variable,w hich defined both 7 haplotypes in 16S and CO Igene,respectively.Variable sites,nucleotide diversity,average number of nucleotide differences and average number of nucleotide substitutions per site were calculated and compared.Rich levels of nucleotide diversities were detected in two Charybdis japonica wild populations.The molecular phylogenetic trees were constructed with NJ and UPGMA method using softw are M EGA 4.0 based on 16S rRNA and CO I gene respectively.The phylogenetic tree based on 16SrRNA gene sequences showed that Portunidae are clustered into two branches:different hap lotypes of Charybdis japonica together first,and then with the three kinds of Scylla;and three crabs of Portunus and two of Callinectes clustered into the o ther branch;While the phylogenetic tree based on CO I gene sequences show ed that different hap lotypes of Charybd is japonica together first,and then with three crabs of Charybdis;Portunus trituberculatus and Portunus pelagicus of Portunus and two of Callinectes clustered into the other branch.The results are consistent with classical taxonomy.These findings are expected to provide important information for the protection and utilization of Charybd is japonica resources of China.
Charybdis japonica;16S rRNA gene;CO Igene;genetics diversity
Q953
A
1672-5174(2010)12-057-07
国家高技术研究发展计划项目(2006AA 10A 406);国家自然科学基金项目(30871933);青岛市科技计划项目(07-2-3-5-jch)资助
2010-03-03;
2010-07-14
高俊娜(1984-),女,硕士生,主要从事海水养殖生物种质资源与遗传育种研究。E-mail:happygao123@126.com
**通讯作者:Tel:0532-85823291;E-mail:liuping@ysfri.ac.cn
责任编辑 于 卫

