草酸乙酰化降解途径酶基因密码子偏好性分析






摘要:【目的】分析100种植物中参与草酸乙酰化降解途径的草酰-CoA合成酶(AAE3)、草酰-CoA脱羧酶(OCD)和甲酸脱氢酶(FDH)基因的密码子偏好性及其影响因素,并分析其适配的异源表达受体,为揭示AAE3、OCD和FDH基因在植物中的遗传修饰和进化规律及提高其异源表达效率提供理论依据。【方法】运用CodonW 1.4.2、CUSP及Python等软件工具,通过PR2-plot分析、ENC-plot分析、相对同义密码子使用量(RSCU)分析、中性绘图分析及密码子使用频率比较分析,深入解析AAE3、OCD和FDH基因编码区(CDS)序列的碱基组成特征、密码子偏好及其主要影响因素、高频密码子、最优密码子及异源表达适配受体。【结果】AAE3、OCD和FDH基因CDS序列在不同位置上GC含量差异较大,但三者均偏好使用以G/C结尾的密码子。AAE3、OCD和FDH基因表达水平较低,且密码子偏好性均较弱,偏好性排序:FDHgt;AAE3gt;OCD。在进化过程中AAE3、OCD和FDH基因的密码子偏好性主要受自然选择的影响,突变压力对其影响则较小。RSCU分析结果显示,AAE3、OCD和FDH基因分别有31、26和27个高频密码子,分别有19、14和9个最优密码子,其中AAE3和FDH基因的最优密码子均以G/C结尾,而OCD基因的最优密码子中以G/C结尾的有8个,占最优密码子的57.14%。拟南芥和烟草可作为AAE3、OCD和FDH基因的异源表达适配受体,当选择大肠杆菌和酿酒酵母作为异源表达受体时,则需要优化较多的密码子(17~19个)。【结论】AAE3、OCD和FDH基因在进化过程中表现出较相似的密码子偏好特征,选择异源表达受体时应优先考虑拟南芥和烟草。
关键词:草酰-CoA合成酶;草酰-CoA脱羧酶;甲酸脱氢酶;密码子偏好性;最优密码子
中图分类号:S503.53文献标志码:A文章编号:2095-1191(2024)10-2990-13
Codon usage bias for genes involved in oxalate acetylation degradation pathway
LUO Yu1,LI Qi2,SHI Hui-neng2,YANG Hong-lei2,SONG Zhan-hua2,YANG Jian-li3,FAN Wei3*
(1College of Food Science and Technology,Yunnan Agricultural University,Kunming,Yunnan 650201,China;2College of Resources and Environment,Yunan Agricultural University,Kunming,Yunnan 650201,China;3College of Landscape and Horticulture,Yunnan Agricultural University/Key Laboratory of VegetableBiology of Yunnan,Kunming,Yunnan 650201,China)
Abstract:【Objective】This study aimed to explore the condon preference of oxalyl-CoA synthase(AAE3),oxalyl-CoA decarboxylase(OCD)and formate dehydrogenase(FDH)genes involved in the oxalate acetylation degradation pathway among 100 plant species,as well as the factors that influenced condon preference,and analyzed their suitable heterologous expression receptors,which could provide scientific basis for understanding the genetic modification and evolution ofAAE3,OCD and FDH genes in plants,and for enhancing their heterologous expression efficiency.【Method】Comprehensively used CodonW 1.4.2,CUSP and Python softwares,through PR2-plot analysis,ENC-plot analysis,rela-tive synonymous codon usage(RSCU)analysis,neutral plot analysis and codon frequency comparison analysis,the base composition characteristics,codon preference and its main influencing factors,high-frequency codons,optimal codons and heterologous expression adaptation receptors of AAE3,OCD,and FDH coding sequences(CDS)were deeply eluci-dated.【Result】The GC content of the CDS sequences of AAE3,OCD and FDH genes varied greatly at different posi-tions,but they all preferred codon ending with G/C.The gene expression levels of AAE3,OCD and FDH genes were low,and the codon preference was weak,with the degree ofFDHgt;AAE3gt;OCD.During evolution,the codon preference of AAE3,OCD and FDH genes was mainly influenced by natural selection,with mutation pressure having little effect.RSCU analysis showed that AAE3,OCD and FDH genes had 31,26 and 27 high-frequency codons,and 19,14 and 9 op-timal codons respectively,among which the optimal codons ofAAE3 and FDH genes allendedin G/C,and 8 of the opti-mal codons of OCD gene ended in G/C,accounting for 57.14%of the optimal codons.Arabidopsis thaliana and Nico-tiana tabacum could serve as adaptive receptors for heterologous expression ofAAE3,OCD and FDH genes.When Esche-richia coli and Saccharomyces cerevisiae were selected as heterologous expression receptors,more codons(17-19)needed to be modified.【Conclusion】AAE3,OCD and FDH genes exhibit highly similar codon preference characteristics during evolutionary process,and the selection of heterologous expression receptors should prioritize A.thaliana and N.tabacum.
Key words:oxalyl-CoA synthase;oxalyl-CoA decarboxylase;formate dehydrogenase;codon preference;optimal codon
Foundation items:National Natural Science Foundation of China(31760584);Fok Ying Tung Education Founda-tion for Young Teachers in Colleges and Universities(161100)
0引言
【研究意义】乙酰化途径是一条依赖于CoA和ATP的草酸降解途径(Giovanelli and Tobin,1961)。草酸首先在草酰-CoA合成酶(Oxalyl-CoA synthase,AAE3)乙酰化作用下生成草酰-CoA,再经过草酰-CoA脱羧酶(Oxalyl-CoA decarboxylase,OCD)生成甲酰-CoA,之后被甲酰-CoA水解酶(Formyl-CoA hydrolase)水解生成甲酸,最后在甲酸脱氢酶(For-mate dehydrogenase,FDH)作用下降解为CO2。目前,虽然已从许多植物中鉴定出编码催化该途径第1、2、4步的关键酶基因,但这些基因也同时存在于细菌和真菌等多种生物中,在基因进化、表达效率及密码子使用偏好性方面往往存在较大差异。密码子偏好性(Codon usage bias)作为物种进化的重要特征之一,广泛存在于各种生物体的基因组中,且在编码基因的非均衡使用和调控基因表达效率等方面发挥关键作用(Brule and Grayhack,2017;Zhang et al.,2018)。因此,研究植物草酸乙酰化代谢途径中关键酶基因的密码子偏好性不仅有助于解释不同物种间或同一物种内的基因进化规律和突变规律,还对优化基因表达、载体构建和未知基因的功能解析具有重要的研究意义。【前人研究进展】草酸是一种简单的低分子量二羧酸,作为碳代谢途径的副产物广泛存在于藻类、真菌、地衣和植物中。草酸在植物的许多生物学和代谢进程中扮演着重要的作用,例如参与调控细胞内pH和无机离子的稳态(Osmond,1963;Nakata,2003)、螯合解毒活性铝离子和铅离子(Yang et al.,2000;Klug and Horst,2010)及防御病虫害(Korth etal.,2006)等。虽然植物中的草酸具有多方面有益作用,但当细胞过多的积累或长期暴露于强酸环境下则会导致一系列生理问题的发生,包括干扰细胞膜完整性和线粒体代谢进程、诱发金属离子沉淀和自由基的形成等(Li etal.,2022b)。这就要求植物组织中的草酸水平必须被精细的调控才能维持合理的细胞代谢和健康。自然界中的细菌、真菌和植物已进化出一系列的草酸降解方式,主要包括氧化、脱羧和乙酰化3种途径(Li etal.,2022b),其中氧化(存在于细菌、真菌和单子叶植物)和脱羧(存在于细菌和真菌)途径介导的草酸降解并不存在于双子叶植物中。因此,乙酰化途径的发现为调节植物中的草酸降解提供了一种替代途径,从一定程度上弥补了植物调节草酸代谢功能。目前,关于植物草酸乙酰化代谢途径中的AAE3、OCD和FDH基因已在不同物种中被鉴定,并在提高植物耐铝性(Han-son and Roje,2001;Wang et al.,2016,2018a;Lou et al.,2016b;He etal.,2023)、诱导植物真菌抗性(Fos-teretal.,2012)及促进种子萌发、胚乳发育和籽粒品质提高(Yang et al.,2018;Foster et al.,2021)等方面发挥重要作用。然而,当这些基因在蛋白表达时的效率存在较大差异,会导致酶的催化活性高低不一(Km为81µmol/L~1.73 mmol/L)(Foster et al.,2016;Lou et al.,2016a;Peng et al.,2016;Xian et al.,2020;Li etal.,2022a)。例如番茄中SlAAE3-2基因(Solyc 06g035960.2.1)的表达量远远低于SlAAE3-1基因(Solyc03g025720.2.1)及其他已报道的AAE3基因表达量(Li etal.,2022a)。饭豆VuFDH(Km=121µmol/L)体外酶催化活性明显高于番茄SlFDH(Km=2.087 mmol/L)(Lou et al.,2016b;He et al.,2023)。近年来,对不同植物中某一特定基因的密码子偏好性探索已成为基因研究的热点,并且利用密码子优化的外源基因已获得了重组蛋白高表达的转基因植株(Agarwal et al.,2008;Jabeen et al.,2010)。研究发现,经过密码子优化的cry1Ah基因与Cry1Ah蛋白靶向叶绿体相结合后,赋予烟草更强的抗虫能力(Li etal.,2013);将ThIPK2基因的密码子优化后转入普通小麦中,转基因小麦的抗旱性显著提高(Zhang et al.,2020)。此外,在不改变氨基酸序列的情况下,通过改变278个核苷酸,将GC含量降低到51.05%,红景天UGT72B14蛋白表达量显著提高4.8倍,且具有同样高的活性,同时红景天苷的产量提高了3.2倍(Xue et al.,2016)。【本研究切入点】由于不同物种间基因碱基的差异限制了植物草酸乙酰化降解途径中关键酶基因表达的效率。可通过密码子优化调控植物草酸乙酰化代谢途径中关键酶基因的表达,提高植物中这些基因的表达效率,增强其酶的催化活性。但目前未见草酸乙酰化降解途径关键酶基因密码子偏好性的研究报道。【拟解决的关键问题】对100种植物中AAE3、OCD和FDH基因的碱基组成特征、密码子偏好性及其主要影响因素进行分析,并筛选最优密码子和异源表达适配系统,为揭示植物草酸降解乙酰化途径中关键酶基因的遗传修饰和进化规律及在翻译水平上提高其异源表达效率提供理论参考。
1材料与方法
1.1数据来源及处理
从NCBI和Phytozome数据库中检索下载100种植物基因组的编码区(CDS)序列,以番茄AAE3、OCD和FDH基因作为Query序列,检索100种植物的AAE3、OCD和FDH基因序列信息。从获得的CDS序列筛选出完整的CDS序列(同时具备起始密码子和终止密码子,且碱基数为3的整数倍),且CDS序列长度大于300 bp,最后去除重复的序列(Gómez et al.,2020)。最终在100种植物中分别获得AAE3(185个)、OCD(125个)和FDH(174个)基因CDS序列用于后续分析。100种植物包括81种双子叶植物、9种单子叶植物、7种木兰纲植物、1种苔纲植物、1种石松纲植物和1种真藓植物纲,具体信息见表1。
从密码子使用数据库(http://www.kazusa.or.jp/codon/)下载大肠杆菌、拟南芥、本氏烟草和酿酒酵母4种模式生物的密码子使用频率数据用于后续分析。
1.2密码子相关参数计算
通过CodonW 1.4.2(http://codonw.sourceforge.net/)、CUSP(http://www.bioinformatics.nl/emboss-explorer/)及Python分析CDS序列的密码子使用情况。密码子偏好使用参数标包括GC含量、密码子适应指数(Codon adaptation index,CAI)、密码子偏好性指数(Codon bias index,CBI)、同义密码子第3位(A、T、G和C)的含量(A3s、T3s、G3s和C3s)、同义密码子第3位的GC含量(GC3s)、密码子有效个数(Effective number of codons,ENC)和相对同义密码子使用量(Relative synonymous codon usage,RSCU)(张扬等,2022)。其中,ENC是评价基因整体密码子偏好性的重要参数,可反映密码子家族中同义密码子非均衡使用的偏好程度,当ENClt;35.00,表示密码子偏好性强;当ENCgt;50.00,表示密码子偏好性弱(Gómezetal.,2020)。此外,利用Python程序分别计算筛选出的AAE3、OCD和FDH基因平均GC含量及密码子第1、2、3位的GC含量(GC1、GC2、GC3)(早浩龙等,2023)
1.3中性绘图分析
突变压力和自然选择是影响密码子偏好性的2个重要因素。分别计算100种植物中AAE3、OCD和FDH基因CDS序列的GC1和GC2平均值(GC12)。以GC12为纵坐标,GC3为横坐标作中性分析散点图,对其进行直线拟合分析,确定影响AAE3、OCD和FDH基因密码子偏好性的主要因素。当直线回归斜率接近1,GC12与GC3相关性越高,即基因几乎沿对角线分布,则密码子偏好性主要受突变影响,反之则主要受自然选择的影响(Zu et al.,2022)。
1.4 PR2-plot分析
PR2-plot是考察突变压力和自然选择对密码子偏好性影响的方法之一。如果密码子偏好性主要受突变压力的影响,则A/T与G/C比例在基因或基因组的简并密码子中是平衡的,若A/T与C/G比例相差过大,则密码子偏好性可能受自然选择及其他因素影响(李亚麒等,2023)。为避免密码子第3位碱基A/T和G/C突变的不平衡,通过统计100种植物185个AAE3、125个OCD和174个FDH基因CDS序列的T3s、C3s、A3s和G3s,并以A3s/(A3s+T3s)为纵坐标,G3s/(G3s+C3s)为横坐标绘制散点图,从而分析CDS序列中嘌呤和嘧啶间的相对关系(尹为治等,2020)。当DNA 2条互补链之间存在碱基突变或选择偏性时,则A3/(A3+T3)和G3/(G3+C3)的坐标数值会偏离0.5的中心位置(甘颖等,2023)。
1.5 ENC-plot分析
利用ENC-plot分析除了碱基组成限制外是否还有其他因素影响密码子偏好性。分别以100种植物中各基因GC3s为横坐标、ENC为纵坐标绘制散点图,并与ENC期望值进行比较,ENC期望值=2+GC3s+29/[GC3s2+(1-GC3s)2],ENC期望值代表密码子偏好性仅受碱基组成的影响。如果每个CDS序列的真实ENC完全落在期望曲线(按ENC期望值公式绘制)上,或者落在更接近标准曲线的区域内,则GC3s可能是密码子使用的唯一决定因素;若CDS序列的ENC真实值完全分布于期望曲线的上方,其密码子偏好性仅受突变压力影响;若CDS序列的ENC真实值完全分布于期望曲线的下方,密码子偏好性仅受自然选择影响(Novembre,2002)。
1.6 RSCU分析
为考察同义密码子的偏好性,以RSCU表示特定密码子在编码对应氨基酸的同义密码子间的相对使用度(Wang et al.,2018b)。当RSCUgt;1.00时,表示该密码子使用度较高,偏好性较强;RSCUlt;1.00时,表示该密码子使用度较低,偏好性较弱;当RSCU=1.00时,表示该密码子无偏好性(Sharp and Li,1986)。
1.7高频密码子和最优密码子确定
对CDS序列的ENC进行排序,从两极各选出5个基因作为高、低表达组,分别计算对应的密码子RSCU,所筛选高、低表达组内对应密码子△RSCU之差gt;0.08时确定为高频密码子,并与整体密码子的RSCU进行结合比较。将△RSCUgt;0.08,且整体密码子的RSCUgt;1.00(除起始密码子、终止密码子及色氨酸外)的高频密码子定义为最优密码子(尹为治等,2020;赵振宁等,2023)。
1.8 AAE3、OCD和FDH基因与模式生物间密码子使用频率比较分析
2个物种密码子使用频率(某密码子个数占该物种编码基因总密码子个数的千分比)的比值是衡量不同物种间密码子使用偏好性的重要指标之一。当2种物种密码子使用频率的比值≥2.0或≤0.5时(不等于0),表示这2种物种的密码子偏好性较大,反之密码子偏好性差异较小。为充分了解100种植物中AAE3、OCD和FDH基因密码子偏好性,将其与拟南芥、烟草、大肠杆菌和酿酒酵母的密码子使用频率进行比较(早浩龙等,2023)。通过在线程序CUSP计算密码子使用频率,在Excel 2019进行各生物间密码子使用频率比较。
2结果与分析
2.1基因CDS序列碱基组成分析结果
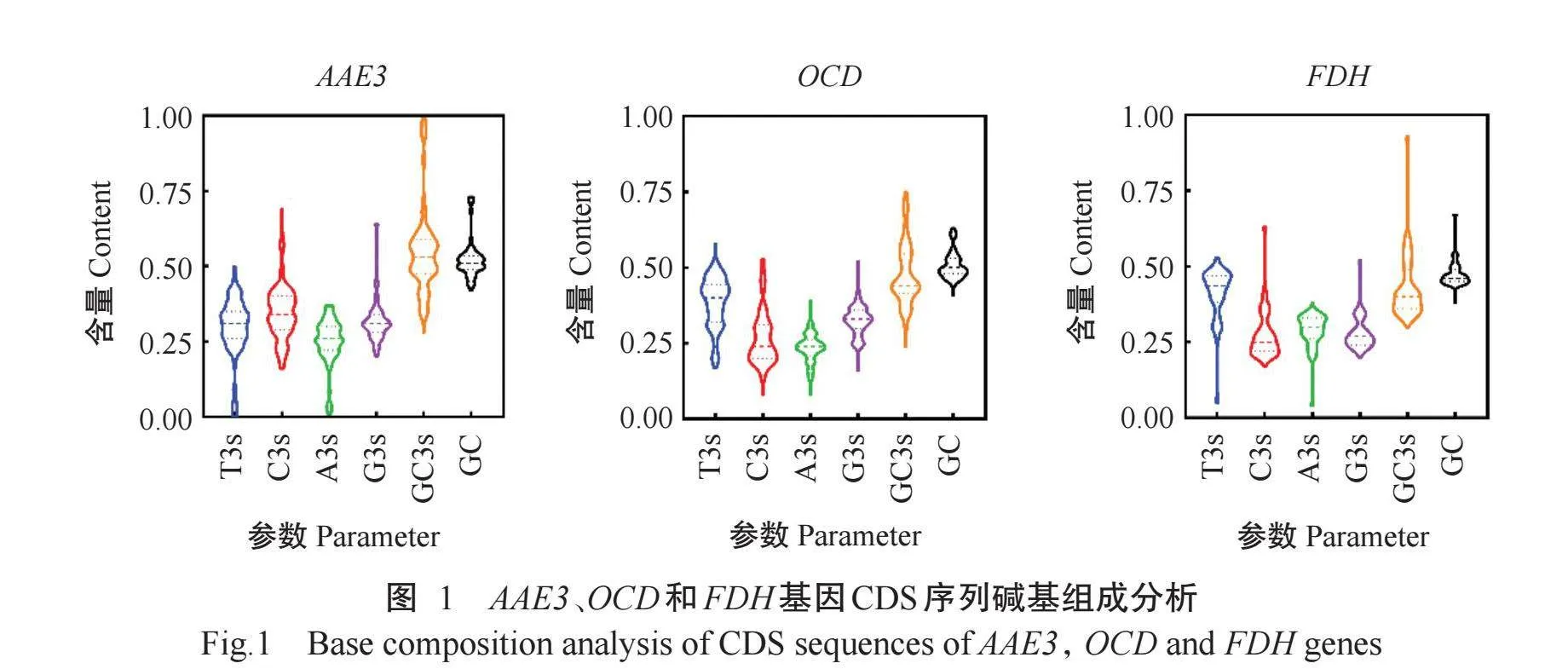
通过CodonW 1.4.2、CUSP及Python对筛选出的185个AAE3、125个OCD和174个FDH基因CDS序列进行碱基组成分析,结果如图1所示。AAE3、OCD和FDH基因CDS序列的T3s分别为0.30、0.39、0.41,A3s分别为0.25、0.24和0.28,而C3s分别为0.35、0.26和0.28,G3s分别为0.32、0.32和0.28,表明AAE3、FDH和OCD基因密码子第3位碱基均偏好使用G/C。
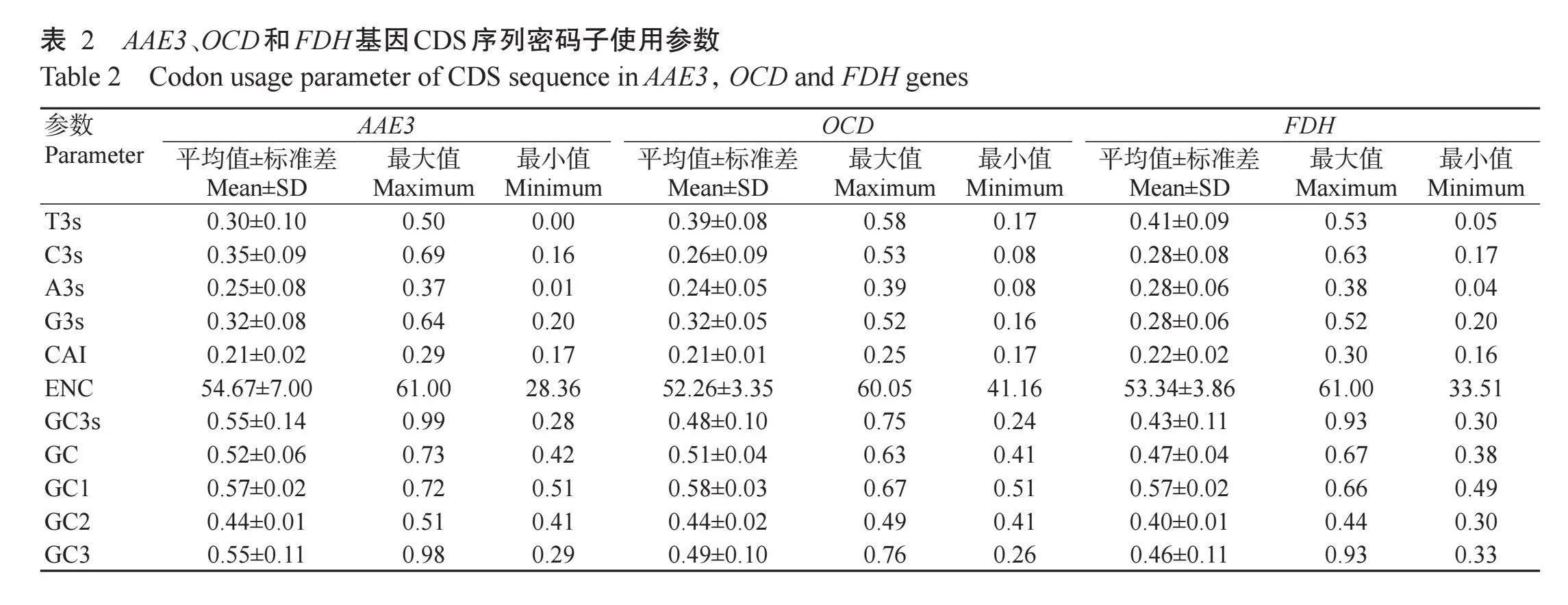
CAI反映了编码某个蛋白的所有密码子相对于这个基因均使用最优密码子的情况下的适应系数。该值越大表示适应性越强,其基因表达水平越高。AAE3、OCD和FDH基因的CAI分别为0.21、0.21和0.22,表明AAE3、OCD和FDH基因表达水平均较低。AAE3、OCD和FDH基因的ENC分别为28.36~61.00、41.16~60.05和33.51~61.00,平均值分别为54.67、52.26和53.34,其中ENCgt;50.00的CDS序列较多(表2),表明AAE3、OCD和FDH基因的密码子偏好性均较弱,其偏好性排序为FDHgt;AAE3gt;OCD。
AAE3、OCD和FDH基因的GC含量平均值分别为0.52、0.51和0.47,GC3s平均值分别为0.55、0.48和0.43。AAE3、OCD和FDH基因的CDS序列在不同位置上GC含量差异较大,GC1最高,分别为0.57、0.58和0.57,其次为GC3,分别为0.55、0.49和0.46,最低为GC2,分别为0.44、0.44和0.40,表明AAE3、OCD和FDH基因均偏好使用以G/C结尾的密码子(表2)。
2.2高频密码子和最优密码子确定
利用CodonW 1.4.2程序对100种植物AAE3、OCD和FDH基因的CDS序列进行RSCU分析,结果显示,AAE3、OCD和FDH基因的高频密码子(RSCUgt;1.00)各有31、26和27个。对AAE3、OCD和FDH基因的ENC进行排序,各选出两极的5个基因(约10%)作为高、低表达基因库,计算RSCU和△RSCU值,比较分析其最优密码子,结果如表3所示。高表达AAE3基因为PvAAE3-1、PvAAE3-2、PhAAE3、OsAAE3和BdAAE3,低表达AAE3基因为HuAAE3-1、PtAAE3-1、PeAAE3-1、PdAAE3-1和PdAAE3-2;高表达OCD基因为AqcOCD、SvOCD、HuOCD、PvOCD和EgOCD,低表达OCD基因为JrOCD、SmOCD1、SmOCD2、SmOCD3和SmOCD4;高表达FDH基因为SvFDH2、SvFDH3、SbFDH2、ZmFDH2和CpuFDH4,低表达FDH基因为ItFDH2、SmFDH1、SmFDH2、CpuFDH3和PjFDH。通过计算筛选出整体密码子的RSCUgt;1.00且高低表达库之差△RSCUgt;0.08的密码子,最终确定AAE3、OCD和FDH基因的最优密码子分别有19、14和9个。其中,AAE3和FDH基因的最优密码子均以G/C结尾,占最优密码子的100.00%;OCD基因的最优密码子中以G/C结尾的有8个,占最优密码子的57.14%。上述结果表明AAE3、OCD和FDH基因在密码子使用上对G/C有较强的偏好性,其中OCD基因的密码子偏好性最弱。
2.3密码子偏好性影响因素分析结果
利用PR2-plot分析AAE3、OCD和FDH基因的密码子偏好性影响因素,结果如图2所示。AAE3基因的大部分CDS序列位于图中左下位置,说明G/C和A/T使用频率是不平衡的,其中Cgt;G、Tgt;A;OCD和FDH基因的大部分CDS序列位于图中平面右下位置,说明G/C和A/T使用频率也是不平衡的,其中Ggt;C、Tgt;A,表明AAE3、OCD和FDH基因的密码子偏好性不仅受突变压力的影响,还受到自然选择的影响。此外,AAE3基因中G和C含量主要分布在0.40~0.58的范围,A和T含量主要分布在0.35~0.52的范围;OCD基因中G和C含量主要分布在0.45~0.65的范围,A和T含量主要分布在0.30~0.45的范围;FDH中G和C含量主要分布在0.45~0.58的范围,A和T含量主要分布在0.34~0.48的范围,表明AAE3、OCD和FDH在嘌呤碱基和嘧啶碱基的使用上分别偏好使用以G/T结尾的密码子。
ENC-plot分析结果(图3)显示,AAE3、OCD和FDH基因的大部分CDS序列位于期望曲线下方只有极少量数据点位于期望曲线上方,表明AAE3、OCD和FDH基因在进化过程中密码子偏好性的主要影响因素为自然选择,其次为突变压力及其他因素的影响,与PR2-plot分析结果一致。
通过中性绘图分析GC12含量与GC3含量相关性,如图4所示。绝大多数AAE3、OCD和FDH基因CDS序列分布在远离黑色虚线的两侧,进一步表明AAE3、OCD和FDH的密码子偏好性主要受自然选择的影响。此外,由回归斜率可知,突变压力对AAE3、OCD和FDH基因影响均小于0.5且接近于0(绝对值),表明突变压力对AAE3、OCD和FDH基因的密码子偏好性影响较小,而自然选择对其密码子偏好性的影响较大。
2.4密码子使用频率比较分析结果
利用拟南芥、烟草、大肠杆菌和酿酒酵母4种模式生物的密码子使用频率与AAE3、OCD和FDH基因进行比较分析,结果如图5所示。AAE3基因使用偏差较大的密码子分别有10、15、18和18个,与拟南芥的密码子使用频率差异最小(10个),与其余3种模式生物的密码子使用频率差异较大;OCD基因使用偏差较大的密码子分别有12、9、19和18个,其中与烟草的密码子使用频率差异最小(9个),与大肠杆菌的密码子使用频率差异最大(19个);FDH基因与这4种模式生物密码子使用偏差较大的密码子分别有4、5、17和18个,与拟南芥的密码子使用频率差异最小(4个),与酿酒酵母的密码子使用频率差异最大(18个)。以上结果表明,AAE3和FDH基因的异源表达受体优先选择拟南芥,其次为烟草;而OCD基因优先选择烟草,其次为拟南芥;选择大肠杆菌和酿酒酵母作为AAE3、OCD和FDH基因的异源表达受体时,则需对较多的密码子(17~19个)进行优化,以提高其基因高效转化表达。
3讨论
密码子偏好性代表密码子在基因编码中的非均衡使用,在物种基因进化和调控及环境适应性中起着重要作用(Foster et al.,2012;Brule and Gray-hack,2017)。大量研究表明,自然选择和突变压力(Sueoka and Kawanishi,2000)、氨基酸组成(Wan et al.,2004)、基因长度及其表达水平(Ingvarsson,2007)等众多因素均会影响密码子偏好性。通过对植物基因进行密码子偏好性分析能帮助判断其来源和进化历程,也为揭示基因家族的进化提供了更具说服力的理论依据。本研究对100种植物乙酰化草酸降解途径中3个关键酶基因AAE3、OCD和FDH进行密码子偏好性分析,结果发现,AAE3、OCD和FDH基因GC含量和GC3较高,在密码子使用上更倾向于使用以G/C结尾的密码子,说明核苷酸组成在AAE3、OCD和FDH基因密码子偏好性中起着重要作用,与Li等(2017b)、Li等(2019)、Liu等(2019)的研究结果一致。此外,不同植物CaM1基因中均偏好G/C结尾(李晨辉等,2019),可能是由于这些基因均参与调控植物新陈代谢。研究表明,参与调控新陈代谢的基因偏好G/C结尾,而结构基因或功能基因更偏好A/T结尾(Liu et al.,2005)。植物草酸乙酰化降解途径中关键酶基因的GC3s较高可能是为了适应环境。
偏好性起源假说普遍认为,突变压力和自然选择是影响密码子偏好性的2个主要因素。如果突变发生在中性的第3密码子位置,将导致随机的同义密码子选择,其中GC和AT将在基因或基因组的简并密码子群中按比例使用(Zhang et al.,2018)。PR2-plot和ENC-plot分析结果显示,自然选择可能主导AAE3、OCD和FDH基因密码子偏好性的形成因素,突变压力对密码子偏好性的形成只起到很小的作用。研究发现,自然选择是形成豆科植物IPI基因密码子偏好性的主要原因(蒋瑞平等,2022);茶树氧甲基转移酶基因密码子偏好性同时受选择压力和突变压力的影响,且自然选择压力的影响更大(谭淳月等,2023)。因此,推测来自同一代谢途径的关键酶基因密码子使用偏好性及其形成原因可能具有一定的相似性(严子成等,2020)。本研究中性绘图分析结果也表明,AAE3、OCD和FDH基因的密码子在进化过程中可能更多地受到自然选择的影响,且对FDH基因影响更大,与Li等(2019)、Liu等(2019)的研究结果一致,但与Li等(2017a)的研究结果不同,推测是由于采取的分析方法不同造成了结果的差异,本研究采用目前主流的分析依据,即按照相关性及回归曲线斜率判断,而Li等(2017a)则根据中性绘图中的CDS序列呈对角分布来判断的。此外,造成这种差异还可能是不同物种中不同基因在较大环境条件差异下受到的选择压力不同所致;DNA变异(Suzuki,2011)、基因序列长度(Ribeiro et al.,2012)、RNA结构(Belalov and Lukashev,2013)等也可能对其产生影响,在一定程度上解释了基于叶绿体基因组的分子标记能有效对其进行鉴定和遗传谱系分析。
当重组蛋白异源表达时,密码子使用偏好性对蛋白表达水平有复杂的影响,根据密码子的使用规则进行密码子优化,可有效提高蛋白表达水平(李亚麒等,2023)。本研究确定了AAE3、OCD和FDH基因高频密码子和最优密码子,不仅有利于对密码子进行优化改造,还有助于深入理解基因表达与密码子使用偏好性之间的关系。此外,本研究还发现,AAE3和FDH基因的最优密码子均以G/C结尾,OCD基因以G/C结尾的密码子占最优密码子的57.14%,进一步验证了AAE3、OCD和FDH基因对G/C结尾的密码子有更强的偏好性。密码子使用频率也和基因的表达量相关,如果外源基因使用了与转基因受体基因相似的密码子,可减少与对应分子匹配的时间,使其具有较高表达量(早浩龙等,2023)。本研究发现,AAE3、OCD和FDH基因的CAI值均较低,说明其表达量相对较低。因此,通过分析AAE3、OCD和FDH基因与拟南芥、烟草、大肠杆菌和酿酒酵母的密码子使用频率比值发现,AAE3、OCD和FDH基因与大肠杆菌和酿酒酵母的密码子偏好性差异最大,与拟南芥和烟草的密码子使用频率差异最小。因此,拟南芥和烟草可优先考虑作为AAE3、OCD和FDH基因的异源表达适配受体,以提高其基因表达水平,为表达系统中引入外源基因、优化改造密码子组成以及提高外源基因的表达效率提供理论参考。
4结论
AAE3、OCD和FDH基因的GC含量、GC3及GC3s较高,在密码子使用上更倾向于使用以G/C结尾的密码子,但在密码子偏好性上存在一定差异。AAE3、OCD和FDH基因在进化过程中主要受自然选择的影响,选择异源表达受体时应优先考虑拟南芥和烟草。
参考文献(References):
甘颖,袁滨,韩星,林俊彬,苗人云,段盺莲,严俊杰,甘炳成.2023.三种侧耳属真菌基因组密码子偏好性比较分析[J].菌物学报,42(6):1285-1297.[Gan Y,Yuan B,Han X,Lin J B,Miao R Y,Duan X L,Yan J J,Gan B C.2023.Comparative analysis of codon preference pattern in threespecies of Pleurotus based on genome data[J].Mycosys-tema,42(6):1285-1297.]doi:10.13346/j.mycosystema.220291.
蒋瑞平,赵辰晖,李文杰,安秋菊,李佳伦,周嘉裕,李遂焰,廖海.2022.豆科植物IPI基因密码子偏好性[J].浙江农业学报,34(6):1114-1123.[Jiang R P,Zhao C H,Li W J,An Q J,Li J L,Zhou J Y,Li S Y,Liao H.2022.Codon bias of IPI gene in leguminous plants[J].Acta Agriculturae Zhe-jiangensis,34(6):1114-1123.]doi:10.3969/j.issn.1004-1524.2022.06.02.
李晨辉,赵子捷,陈文烨,焦义然,杨帆,刘永伟,董福双,杜进民,周硕.2019.43种植物CaM1基因密码子使用特征及遗传差异分析[J].江苏农业科学,47(5):28-32.[Li C H,Zhao Z J,Chen W Y,Jiao Y R,Yang F,Liu Y W,Dong F S,Du J M,Zhou S.2019.Analysis of codon usage cha-racteristics and genetic difference of CaM1 gene in 43 plant species[J].Jiangsu Agricultural Sciences,47(5):28-32.]doi:10.15889/j.issn.1002-1302.2019.05.008.
李亚麒,严炜,娄予强,黄家雄,胡发广,付兴飞,李亚男,程金焕.2023.高产小粒咖啡叶绿体基因组密码子偏好性分析[J].南方农业学报,54(8):2330-2339.[Li Y Q,Yan W,Lou Y Q,Huang J X,Hu F G,Fu X F,Li YN,Cheng J H.2023.Codon usage bias in the chloroplast genome of high production Coffea arabica L.[J].Journal of Southern Agriculture,54(8):2330-2339.]doi:10.3969/j.issn.2095-1191.2023.08.014.
谭淳月,刘勇,赖章凤,周子豪,李明玺.2023.茶树氧甲基转移酶基因密码子偏好性分析[J].江西农业大学学报,45(3):652-662.[Tan C Y,Liu Y,Lai Z F,Zhou Z H,Li M X.2023.Codon bias analysis of O-methyltransferase genein Camellia sinensis[J].Acta AgriculturaeUniversitatisJiangxiensis,45(3):652-662.]doi:10.13836/j.jjau.2023 061.
严子成,蒋瑞平,梁浩伟,陈渏,周嘉裕,廖海.2020.川芎咖啡酸-O-甲基转移酶基因密码子偏好性与进化分析[J].应用与环境生物学报,26(4):894-901.[Yan Z C,Jiang R P,Liang H W,Chen Y,Zhou J Y,Liao H.2020.Codon bias and evolution analysis of the caffeic acid-O-methyltransferase gene in Ligusticum chuanxiong Hort[J].Chinese Journal of Applied and Environmental Biology,26(4):894-901.]doi:10.19675/j.cnki.1006-687x.2019.09045.
尹为治,方正,黄良鸿,龙文兴,李佳灵.2020.海南2种龙脑香科植物叶绿体基因组密码子偏好性分析[J].林业调查规划,45(6):19-26.[Yin W Z,Fang Z,Huang L H,Long W X,Li J L.2020.Codon usage bias in chloroplast genomes of two species of Dipterocarpaceae in Hainan[J].Forest Inventory and Planning,45(6):19-26.]doi:10.3969/j.issn.1671-3168.2020.06.005.
早浩龙,刘春兰,黃珍华,徐瑞,施辉能,龙光强,罗瑜,范伟.2023.基于全基因组的三七HMA基因家族鉴定与生物信息学分析[J].分子植物育种,21(17):5642-5653.[Zao H L,Liu C L,Huang Z H,Xu R,Shi H N,Long G Q,Luo Y,Fan W.2023.Identification and bioinformatics analysisof HMA gene family based on Panax notoginseng genome[J].Molecular Plant Breeding,21(17):5642-5653.]doi:10.13271/j.mpb.021.005642.
张扬,孙曙光,李晴,魏珍.2022.莴苣线粒体基因组密码子使用偏好性分析[J].河南农业科学,51(10):114-124.[Zhang Y,Sun S G,Li Q,Wei Z.2022.Codon usage bias analysis of cultivated and wild lettuce mitochondrial genomes[J].Journal of Henan Agricultural Sciences,51(10):114-124.]doi:10.15933/j.cnki.1004-3268.2022.10.013.
赵振宁,孙浩田,宋雨茹,余潇.2023.山楂属植物叶绿体基因组特征与密码子偏好性分析[J].江苏农业学报,39(2):504-517.[Zhao Z N,Sun H T,Song Y R,Yu X.2023.Chloroplast genome characteristics and codon usage bias analysis of Crataegus L.[J].Jiangsu Journal of Agricul-tural Sciences,39(2):504-517.]doi:10.3969/j.issn.1000-4440.2023.02.024
Agarwal S,Singh R,Sanyal I,Amla D V.2008.Expression of modified gene encoding functional human alpha-1-anti-trypsin protein in transgenic tomato plants[J].Transgenic Research,17(5):881-96.doi:10.1007/s 11248-008-9173-8.
Belalov I S,Lukashev A N.2013.Causes and implications of codon usage bias in RNA viruses[J].PLoS One,8(2):e56642.doi:10.1371/journal.pone.0056642.
Brule C E,Grayhack E J.2017.Synonymous codons:Choose wisely for expression[J].Trends in Genetics,33(4):283-297.doi:10.1016/j.tig.2017.02.001.
Foster J,Cheng N H,Paris V,Wang L F,Wang J,Wang X Q,Nakata PA.2021.An Arabidopsis oxalyl-CoA decarboxyla-se,AtOXC,is important for oxalate catabolism in plants[J].International Journal of Molecular Sciences,22(6):3266.doi:10.3390/ijms22063266.
Foster J,Kim H U,Nakata P A,Browse J.2012.A previously unknown oxalyl-CoA synthetase is important for oxalate catabolism in Arabidopsis[J].The Plant Cell,24(3):1217-1229.doi:10.1105/tpc.112.096032.
Foster J,Luo B,Nakata P A,2016.An oxalyl-CoA dependent pathway of oxalate catabolism plays a role in regulating calcium oxalate crystal accumulation and defending against oxalate-secreting phytopathogens in Medicago truncatula[J].PLoS One,11(2):e0149850.doi:10.1371/journal.pone.0149850.
Giovanelli J,Tobin N F.1961.Adenosine triphosphate-and coenzyme A-dependent decarboxylation of oxalate by extracts of peas[J].Nature,190:1006-1007.doi:10.1038/1901006a0.
Gómez M M,de Mello Volotão,Assandri I R,Peyrou M,Cris-tina J.2020.Analysis of codon usage bias in potato virus Y non-recombinant strains[J].Virus Research,286:198077.doi:10.1016/j.virusres.2020.198077.
Hanson A D,Roje S.2001.One-carbon metabolism in higher plants[J].Annual Review of Plant Physiology and Plant Molecular Biology,52:119-137.doi:10.1146/annurev.arplant.52.1.119.
He Q Y,Jin J F,Li P F,Zhu H H,Wang Z Q,Fan W,Yang J L.2023.Involvement of SlSTOP1 regulated SlFDHexpres-sion in aluminum tolerance by reducing NAD+to NADH in the tomato root apex[J].The Plant Journal,113(2):387-401.doi:10.1111/tpj.16054.
Ingvarsson P K.2007.Gene expression and protein lengthinfluence codon usage and rates of sequence evolution in Populus tremula[J].Molecular Biology and Evolution,24(3):836-844.doi:10.1093/molbev/msl212.
Jabeen R,Khan M S,Zafar Y,Anjum T.2010.Codon optimiza-tion of cry1Ab gene for hyper expression in plant organe-lles[J].Molecular Biology Reports,37(2):1011-1017.doi:10.1007/s 11033-009-9802-1.
Klug B,Horst W J.2010.Oxalate exudation into the root-tip water free space confers protection from aluminum toxici-ty and allows aluminum accumulation in the symplast in buckwheat(Fagopyrum esculentum)[J].The New Phy-tologist,187(2):380-391.doi:10.1111/j.1469-8137.2010.03288.x.
Korth K L,Doege S J,Park S H,Goggin F L,Wang Q,Gomez S K,Liu G J,Jia L L,Nakata P A.2006.Medicago trun-catula mutants demonstrate the role of plant calcium oxa-late crystals as an effective defense against chewing insects[J].Plant Physiology,141(1):188-195.doi:10.1104/pp.106.076737.
Li G L,Pan Z L,Gao S C,He Y Y,Xia Q Y,Jin Y,Yao H P.2019.Analysis of synonymous codon usage of chloroplast genome in Porphyra umbilicalis[J].Genes and Genomics,41(10):1173-1181.doi:10.1007/s 13258-019-00847-1.
Li G,Zhang Y J,Lu J Q,Lu X X,Han T,Ru Y.2017a.Codon usage study on chloroplast genome in medicinal plant Panax Ginseng[J].International Journal of Homeopathyamp;Natural Medicines,3(5):40-44.doi:10.11648/j.ijhnm.20170305.11.
Li J,Li H Y,Zhi J K,Shen C Z,Yang X S,Xu J C.2017b.Codon usage of expansin genes in Populus trichocarpa[J].Current Bioinformatics,12(5):452-461.doi:10.2174/157 4893611666161008195145.
Li P F,He Q Y,Jin J F,Liu Y,Wen Y X,Zhao K,Mao G Q,Fan W,Yang J L.2022a.Tomato oxalyl-CoA synthetase degrades oxalate and affects fruit quality[J].Frontiers in Plant Science,13:951386.doi:10.3389/fpls.2022.951386.
Li P F,Liu C L,Luo Y,Shi H N,Li Q,PinChu C E,Li X J,Yang J L,Fan W.2022b.Oxalate in plants:Metabolism,function,regulation,and application[J].Journal of Agricul-tural and Food Chemistry,70(51):16037-16049.doi:10.1021/acs.jafc.2c04787.
Li X Y,Li S Y,Lang Z H,Zhang J,Zhu L,Huang D F.2013.Chloroplast-targeted expression of the codon-optimized truncated cry1Ah gene in transgenic tobacco confers a high level of protection against insects[J].Plant Cell Reports,32:1299-1308.doi:10.1007/s00299-013-1444-z.
Liu Q P,Dou S J,Ji Z J,Xue Q Z.2005.Synonymous codon usage and gene function are strongly related in Oryzasativa[J].Biosystems,80(2):123-131.doi:10.1016/j.bio-systems.2004.10.008.
Liu X F,Zhu G F,Li D M,Wang X J.2019.Complete chloro-plast genome sequence and phylogenetic analysis of Spathiphyllum‘Parrish’[J].PLoS One,14(10):e0224038.doi:10.1371/journal.pone.0224038.
Lou H Q,Fan W,Xu J M,Gong Y L,Jin J F,Chen W W,Liu L Y,Hai M R,Yang J L,Zheng S J.2016a.An oxalyl-CoA synthetase is involved in oxalate degradation and alumi-num tolerance[J].Plant Physiology,172(3):1679-1690.doi:10.1104/pp.16.01106.
Lou H Q,Gong Y L,Fan W,Xu J M,Liu Y,Cao M J,Wang M H,Yang J L,Zheng S J.2016b.A formate dehydrogenase confers tolerance to aluminum and low pH[J].Plant Physiology,171(1):294-305.doi:10.1104/pp.16.01105.
Nakata P A.2003.Advances in our understanding of calcium oxalate crystal formation and function in plants[J].Plant Science,164(6):901-909.doi:10.1016/S0168-9452(03)00120-1.
Novembre J A.2002.Accounting for background nucleotide composition when measuring codon usage bias[J].Mole-cular Biology and Evolution,19(8):1390-1394.doi:10.1093/oxfordjournals.molbev.a004201.
Osmond B.1963.Oxalates and ionic equilibria in Australian saltbushes(Atriplex)[J].Nature,198:503-504.doi:10.1038/198503a0.
Peng C,Liang X,Liu E E,Zhang J J,Peng X X,Mock H P.2016.The oxalyl-CoA synthetase-regulated oxalate and its distinct effects on resisitance to bacterial blight and alu-minium toxicity in rice[J].Plant Biology,19(3):345-353.doi:10.1111/plb.12542.
Ribeiro A S,Häkkinen A,Lloyd-Price J.2012.Effects of gene length on the dynamics of gene expression[J].Computa-tional Biology and Chemistry,41:1-9.doi:10.1016/j.compbiolchem.2012.10.002.
Sharp P M,Li W H.1986.An evolutionary perspective on sy-nonymous codon usage in unicellular organisms[J].Jour-nal of Molecular Evolution,24(1-2):28-38.doi:10.1007/BF 02099948.
Sueoka N,Kawanishi Y.2000.DNA G+C content of the third codon position and codon usage biases of human genes[J].Gene,261(1):53-62.doi:10.1016/s0378-1119(00)00480-7.
Suzuki Y.2011.Statistical methods for detecting natural selec-tion from genomic data[J].Genes and Genetic Systems,85(6):359-376.doi:10.1266/ggs.85.359.
Wan X F,Xu D,Kleinhofs A,Zhou J Z.2004.Quantitative rela-tionship between synonymous codon usage bias and GC composition across unicellular genomes[J].BMC Evolu-tionary Biology,4:19.doi:10.1186/1471-2148-4-19.
Wang LY,Xing H X,Yuan Y C,Wang X L,Saeed M,Tao J C,Feng W,Zhang G H,Song X L,Sun X Z.2018a.Genome-
wide analysis of codon usage bias in four sequenced cotton species[J].PLoS One,13(3):e0194372.doi:10.1371/journal.pone.0194372.
Wang R,Zeng Z D,Guo H X,Tan H,Liu A,Zhao Y,Chen LM.2018b.Over-expression of the Arabidopsis formatedehydrogenase in chloroplasts enhances formaldehydeuptake and metabolism in transgenic tobacco leaves[J].Planta,247(2):339-354.doi:10.1007/s00425-017-2790-9.Wang R,Zeng Z D,Liu T,Liu A,Zhao Y,Li K Z,Chen L M.2016.A novel formaldehyde metabolic pathway plays an important role during formaldehyde metabolism and detoxification in tobacco leaves under liquid formaldehyde stress[J].Plant Physiology and Biochemistry,105:233-241.doi:10.1016/j.plaphy.2016.04.028.
Xian P Q,Cai Z D,Cheng Y B,Lin R B,Lian T X,Ma Q B,Nian H.2020.Wild soybean oxalyl-CoA synthetase degrades oxalate and affects the tolerance to cadmium and aluminum stresses[J].International Journal of Molecular Sciences,21(22):8869.doi:10.3390/ijms21228869.
Xue F Y,Guo H L,Hu Y Y,Liu R,Huang L N,Lv H S,Liu C M,Yang M F,Ma L Q.2016.Expression of codon-optimized plant glycosyltransferase UGT72B14 in Esche-richia coli enhances salidroside production[J].Biomed Research International,2016:9845927.doi:10.1155/2016/9845927.
Yang J,Fu M M,Ji C,Huang Y C,Wu Y R.2018.Maize oxalyl-coa decarboxylase 1 degradesoxalate and affects the seed metabolome and nutritional quality[J].The Plant Cell,30(10):2447-2462.doi:10.1105/tpc.18.00266.
Yang YY,Jung J Y,Song WY,Suh H S,Lee Y.2000.Identifi-cation of rice varieties with high tolerance or sensitivity tolead and characterization of the mechanism of tolerance[J].Plant Physiology,124(3):1019-1026.doi:10.1104/pp.124.3.1019.
Zhang R Z,Zhang L,Wang W,Zhang Z,Du H H,Qu Z,Li X Q,Xiang H.2018.Differences in codon usage bias between photosynthesis-related genes and genetic system-related genes of chloroplast genomes in cultivated and wild solanum species[J].International Journal of Molecu-lar Sciences,19(10):3142.doi:10.3390/ijms 19103142.
Zhang S J,Li Y L,Song G Q,Gao J,Zhang R Z,Li W,Chen M L,Li G Y.2020.Heterologous expression of the ThIPK2 gene enhances drought resistance of common wheat[J].Journal of Integrative Agriculture,19(4):941-952.doi:10.1016/S2095-3119(19)62714-0.
Zu Z P,Lin H J,Hu Y F,Zheng X,Chen C R,Zhao Y S,He N.2022.The genetic evolution and codon usage pattern of severe fever with thrombocytopenia syndrome virus[J].Infection Genetics and Evolution,99:105238.doi:10.1016/j.meegid.2022.105238.
(责任编辑陈燕)

