小麦遗传构成及重要性状功能基因组成解析



摘要:【目的】解析小麦品种信麦136的遗传物质构成和重要功能基因组成,以期为小麦新品种选育及种质资源创制提供理论参考。【方法】对信麦136及其母本扬麦15-1和父本信麦79进行55K SNP芯片扫描和KASP功能标记检测,明确其遗传组成及适应性、产量、籽粒品质、面粉品质、抗逆性等相关功能基因的组成。【结果】信麦136及其亲本中共获得40465个有效SNP位点,其中,有9399个位点在母本扬麦15-1和父本信麦79间呈丰富的多态性,在21条染色体和A、B、D 3个基因组上均呈现不均匀分布状态。母本扬麦15-1和父本信麦79对信麦136的平均遗传贡献率分别为49.26%和50.74%。母本扬麦15-1的遗传贡献率大于父本信麦79的染色体有9条,而其他12条染色体表现为父本信麦79的遗传贡献率大于母本扬麦15-1。适应性相关基因方面,信麦136与母本扬麦15-1和父本信麦79在开花基因(1个)、春化基因(3个)、光周期基因(3个)和芒基因(1个)上完全一致,仅在株高Rht-D1基因上有差异,信麦136及其母本扬麦15-1表现为矮秆,父本信麦79表现为高秆。产量相关基因方面,信麦136与其亲本在TaGW2-6B、TGW7A和Sus1-7A基因上均表现为高千粒重;信麦136及其父本信麦79在TaGS2-B1基因上均表现为高生物量。籽粒品质相关基因方面,信麦136与亲本均含有高分子量谷蛋白亚基含量基因(Glu-A3)、低分子量谷蛋白亚基含量基因(Glu-B3、Glu-B3e和Glu-A3b)、正常蛋白质含量基因(GCP)和增加蛋白质含量基因(NAM-6A)。面粉品质相关性状方面,信麦136与亲本在TaPds-B1、PsyB1c和PsyA1基因上均表现为低黄色素含量,而在ALPb7A基因上均表现为高面团强度。抗逆相关基因方面,信麦136与亲本在Sr36基因上均表现为感秆锈病,在Lr34基因上均表现为抗叶锈病,在TaMFT基因上均表现为感穗发芽,在Vp1B1和TaSdr-B1基因上均表现为抗穗发芽,在TaDreb-B1基因上均表现为抗旱;在1fehw3基因上均表现为低表达。【结论】母本扬麦15-1和父本信麦79的遗传物质向子代信麦136传递过程中受育种定向选择的影响发生严重偏分离,同时遗传物质传递的主要表现方式为染色体间大片段重组和整合,且信麦136的遗传物质主要来自父本信麦79,重要性状功能基因解析结果与其在实际生产中的表现(早熟、抗倒伏、稳产、品质优良、中筋偏弱、抗病、抗旱、抗穗发芽)基本一致。
关键词:小麦;信麦136;遗传构成;SNP;功能基因
中图分类号:S512.103.53文献标志码:A文章编号:2095-1191(2024)10-2980-10
Genetic composition and important trait functional genes composition of wheat
CHEN Zhen-zhen,SHEN Guan-yu,WANG Ke,CHEN Jin-ping,LI Mei,ZHOU Guo-qin*
(Xinyang Academy of Agricultural Sciences,Xinyang,Henan 464000,China)
Abstract:【Objective】To investigate the composition of genetic material and important functional genes of wheat varie-ty Xinmai 136,with a view to providing theoretical reference for the selection and breeding of new wheat varieties and the creation of germplasm resources.【Method】Xinmai 136 and its female parent Yangmai 15-1 and male parent Xinmai 79 were detected by 55K SNP chip scanning and KASP functional markers to determine the genetic composition and the composition of related functional genes such as adaptability,yield,grain quality,flour quality and stress resistance.【Re-sult】Atotal of 40465 effective SNP loci were obtained in Xinmai 136 and its parents,among which 9399 loci showed rich polymorphism between the female parent Yangmai 15-1 and male parent Xinmai 79,with heterogeneous distribution on 21 chromosomes and 3 genomes,A,B and D.The average genetic contributions of the female parent Yangmai 15-1 andmale parent Xinmai 79 to Xinmai 136 were 49.26%and 50.74%respectively.There were 9 chromosomes in which the ge-netic contribution of female parent Yangmai 15-1 was greater than that of male parent Xinmai 79,while the other 12 chro-mosomes showed that the genetic contribution of male parent Xinmai 79 was greater than that of female parent Yangmai 15-1.Regarding the genes related to adaptation,Xinmai 136 was completely identical to female parent Yangmai 15-1 and male parent Xinmai 79 in terms of the genes for flowering(1 gene),vernalisation(3 genes),photoperiodi-city(3 genes)and awn(1 gene).There was only difference in the plant height Rht-D1 gene,with Xinmai 136 and its female parent Yangmai 15-1 showing dwarf culms,male parent Xinmai 79 showing high culms.In terms of yield related genes,both Xinmai 136 and its parents showed high thousand-kernel weight in TaGW2-6B,TGW7A and Sus1-7A genes,while both Xinmai 136 and its male parent Xinmai 79 showed high biomass in TaGS2-B1 gene.For grain quality related genes,both Xinmai 136 and its parents contained high molecular weight glutenin subunit content gene(Glu-A3),low molecular weight glutenin subunit content genes(Glu-B3,Glu-B3e and Glu-A3b),normal protein content gene(GCP)and in-creased protein content gene(NAM-6A).For flour quality related traits,Xinmai 136 and its parents showed low yellow pigment content in TaPds-B1,PsyB1c and PsyA1 genes and high dough strength in ALPb7A gene.As for the resistance re-lated genes,both Xinmai 136 and its parents showed culm rust susceptibility on Sr36 gene,leaf rust resistance on Lr34 gene,spike germination susceptibility on TaMFT gene,spike germination resistance on Vp1B1 and TaSdr-B1 genes,and drought resistance on TaDreb-B1 gene;and showed low expression on 1fehw3 gene.【Conclusion】In the process of trans-mission of genetic material from the female parent Yangmai 15-1 and the male parent Xinmai 79 to the offspring Xinmai 136,severe segregation occurs under the influence of directional selection inbreeding,and the main way of transmission of genetic material is recombination and integration of large segments of chromosomes,and the genetic materials of Xinmai 136 mainly come from the male parent Xinmai 79.The results of gene analysis for important traits are basically consistent with their actual production performance(early maturity,resistance to failure,stable yield,good quality,weak gluten,disease resistance,drought resistance,and resistance to spike germination).
Key words:wheat;Xinmai 136;genetic composition;SNP;functional gene
Foundation items:China Agriculture Research System—Wheat(CARS-03-01A);“Leading the Charge with Open Competition”Project of Henan Key Science and Technology Special Project(21110110800)
0引言
【研究意义】小麦(Triticumaestivum L.)是我国重要的粮食作物,种植面积广、总产量高(赵吉平等,2019)。随着小麦品种产量潜力不断提高,新育成品种出现了品种类型相似和抗病、抗逆性较差的问题,在产量和适应性上无突破性进展(林作楫等,2005;李振声,2010),其主要原因是育种的基础为遗传物质的重组和突变,育种家定向选择会导致遗传多样性下降和遗传基础越来越窄。信麦136是2021年国审小麦品种,适宜在长江中下游冬麦区种植。因此,利用SNP和KASP分子标记对信麦136进行遗传物质构成及功能基因解析,探究小麦品种选育过程中的遗传变异,对小麦新品种选育及重要种质资源创制具有理论指导意义。【前人研究进展】近年来,随着生物技术和信息技术的迅猛发展,越来越多的SNP分子标记技术被开发和利用。中国农业科学院作物科学研究所在660K SNP的基础上结合1000多份本土小麦材料开发出55K SNP,与小麦其他SNP芯片相比,55K SNP有效性更高,且其在QTL定位(Liu et al.,2018;Ren etal.,2018)、遗传多样性分析(李聪等,2019;Zhang et al.,2019a,2019b)和农艺性状关联分析(Ye etal.,2019a,2019b;马艳明等,2020)等研究中得到广泛应用。近年来,利用KASP分子标记技术对各种作物种质资源基因分型或作物辅助育种的研究越来越多,Rasheed等(2016)对普通小麦品质、适应性、产量及抗逆相关的152对KASP标记进行开发与鉴定,结果表明这些标记在子代、杂交亲本及高代品系的鉴定效率显著高于PCR标记;Qureshi等(2018)对小麦Yr34和Yr48基因进行KASP检测,结果表明二者为同一个基因;Su等(2018)开发出与小麦赤霉病相关的TaHRC-KASP分子标记,并用这些标记对中国和日本的小麦品种进行抗赤霉病筛选,结果表明这个标记可有效筛选出Fhb1基因;王君婵等(2019)通过对扬麦系列品种(系)重要性状功能基因进行KASP检测,结果显示,扬麦系列品种(系)含有矮秆、光周期不敏感、抗病虫、抗穗发芽、抗旱等优异基因;Ur Rehman等(2021)利用16个与小麦抗旱性相关基因的等位基因差异开发出KASP分子标记,并用这些标记对47份小麦材料进行籽粒产量等相关性状的功能分析,可有效区分出47份小麦籽粒产量相关的基因;张维军等(2022)对宁夏小麦种质资源粒重基因进行KASP检测,结果显示,从209份参试材料中共筛选到14份千粒重大于50 g的高粒重材料,其中有9份材料聚合了4个粒重相关基因的优异单倍型;杜莹莹等(2023)通过对江苏淮北小麦品种(系)重要性状功能基因进行KASP检测,结果显示,江苏淮北小麦品种(系)聚合了产量、品质、抗病虫性及抗穗发芽等相关基因;隋建枢等(2023)对75份贵州小麦品种(系)抗病基因进行KASP检测,结果显示,同时含有抗叶锈病、白粉病抗病基因的小麦品种(系)共计26份材料,占34.7%。【本研究切入点】信麦136为春性品种,抗性表现为中感纹枯病和赤霉病,高感白粉病、叶锈病和慢条锈病,具有较大的推广种植潜力。但信麦136的遗传组成及重要性状功能基因分析尚未见有研究报道。SNP和KASP分子标记可从基因位点、基因多态性和基因功能等方面对信麦136的遗传结构解析提供更好的基础。【拟解决的关键问题】对信麦136及亲本(扬麦15-1和信麦79)进行55K SNP芯片扫描和KASP功能标记检测,明确其遗传组成及适应性、产量、籽粒品质、面粉品质、抗逆等相关功能基因的组成,为小麦新品种选育及种质资源创制提供理论参考。
1材料与方法
1.1试验材料
供试材料为信麦136及其母本扬麦15-1和父本信麦79,均为异源六倍体(含有A、B、D 3个基因组),均由信阳市农业科学院小麦研究所提供。DP305植物基因组DNA提取试剂盒购自生工生物工程(上海)股份有限公司;NanoDrop 1000超微量分光光度计(ThermoFisher Scientific)。
1.2基因组DNA提取
选取信麦136、扬麦15-1和信麦79的50粒小麦种子置于培养皿内培养,待小麦种子长出幼嫩的叶片,选取各材料的幼嫩叶片于-80℃超低温冰箱冷冻保存,用液氮研磨后采用DNA提取试剂盒提取信麦136及其母本扬麦15-1和父本信麦79的基因组DNA。
1.3 55K芯片分析
样品经处理、核酸提取、核酸扩增、核酸标记和芯片杂交后,获得原始下机数据,共有53007个SNP位点。对Dish QC(DQC)≥0.82且Call Rate(CR)≥95的样品进行SNP位点质控,筛选出有效SNP位点,再从中挑选出母本扬麦15-1和父本信麦79之间呈多态性的位点。
1.4亲本对信麦136遗传贡献率分析及信麦136基因型图谱绘制
统计信麦136与亲本(扬麦15-1和信麦79)存在差异的位点数。采用PLANT GMAP(http://183.223.252.63:3333/)查找信麦136及其亲本(扬麦15-1和信麦79)相同的SNP位点;参考小麦RefSeq v1.0基因组信息,采用Python(https://www.python.org)绘制信麦136的基因型图谱。
1.5信麦136及其亲本的KASP分子标记检测
信麦136与亲本(扬麦15-1和信麦79)的KASP检测委托中玉金标生物技术有限公司进行。整个过程在Douglas Scientific Array Tape平台上完成。首先用NanoDrop 1000超微量分光光度计检测DNA的含量及纯度,根据小麦基因组大小,利用TECAN液体自动化工作站进行DNA的稀释,然后配制PCR反应体系1.622μL,包括DNA工作液0.800μL(3 ng/μL)、2×KSP Master Mix 0.800μL、Assay Mix 0.022μL。在Soellex®高通量PCR水浴热循环系统中完成PCR扩增,扩增程序:95℃预变性15 min;95℃变性20 s,65℃逐渐降至55℃退火1min,进行10个循环;95℃变性20 s,55℃延伸1 min,进行30个循环;95℃预变性5 min;95℃变性20 s,55℃延伸1 min,进行5个循环。最后在Araya内联荧光检测系统上进行荧光强度扫描检测并读取数据。
2结果与分析
2.1信麦136及其亲本SNP位点在基因组上的差异分析结果
对53007个SNP位点进行质控后,共获得40465个有效SNP位点,其中有9399个位点在母本扬麦15-1与父本信麦79之间呈丰富的多态性,占比25.1%,这些位点在21条染色体和A、B、D 3个基因组上均呈不均匀分布状态。但总体上来看表现为扬麦15-1与信麦79的相同位点数多,差异位点数少;B基因组的差异位点数占比为25.50%,其次是A基因组,占比为25.24%,D基因组的差异位点数占比为24.60%,表明B基因组的差异位点数最多(图1)。
2.2亲本对信麦136的遗传贡献分析结果
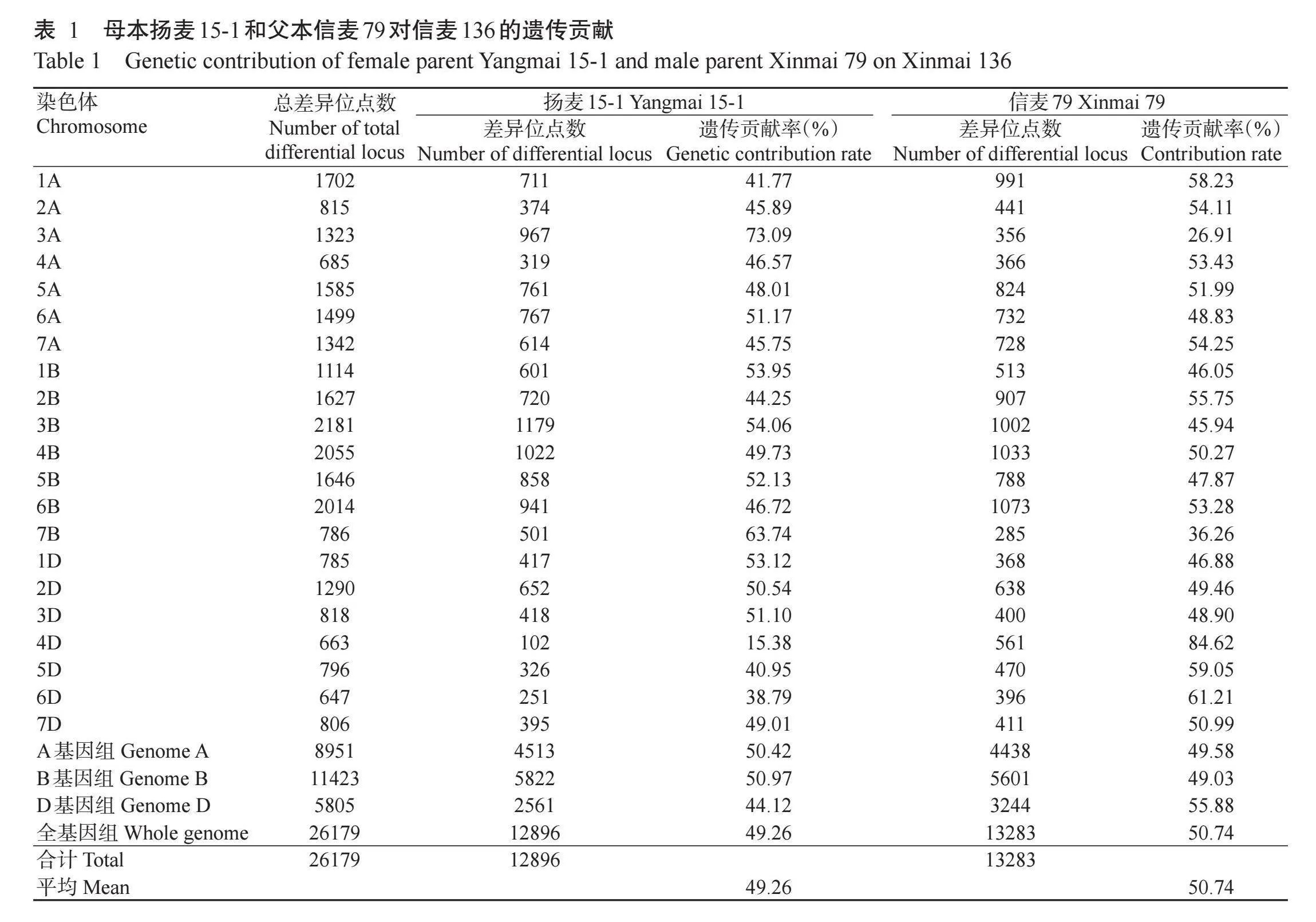
在染色体水平上,母本扬麦15-1对信麦136的遗传贡献率为15.38%~73.09%,母本3A染色体有73.09%的遗传物质传递给信麦136;父本信麦79对信麦136的遗传贡献率为26.91%~84.62%,其中4D染色体有84.62%的遗传物质传递给信麦136,6D染色体有61.21%的遗传物质传递给信麦136(表1)。母本扬麦15-1对信麦136的平均遗传贡献率为49.26%,父本信麦79对信麦136的平均遗传贡献率为50.74%,说明父本信麦79对信麦136的遗传贡献较大(表1)。母本扬麦15-1的遗传贡献率大于父本信麦79的染色体分别为3A、6A、1B、3B、5B、7B、1D、2D、3D等9条染色体(表1和图2),父本信麦79的遗传贡献率大于母本扬麦15-1的染色体有1A、2A、4A、5A、7A、2B、4B、6B、4D、5D、6D和7D等12条染色体(表1和图3)。在A、B、D基因组上,母本扬麦15-1的遗传贡献率分别为50.42%、50.97%和44.12%;父本信麦79的遗传贡献率分别为49.58%、49.03%和55.88%。
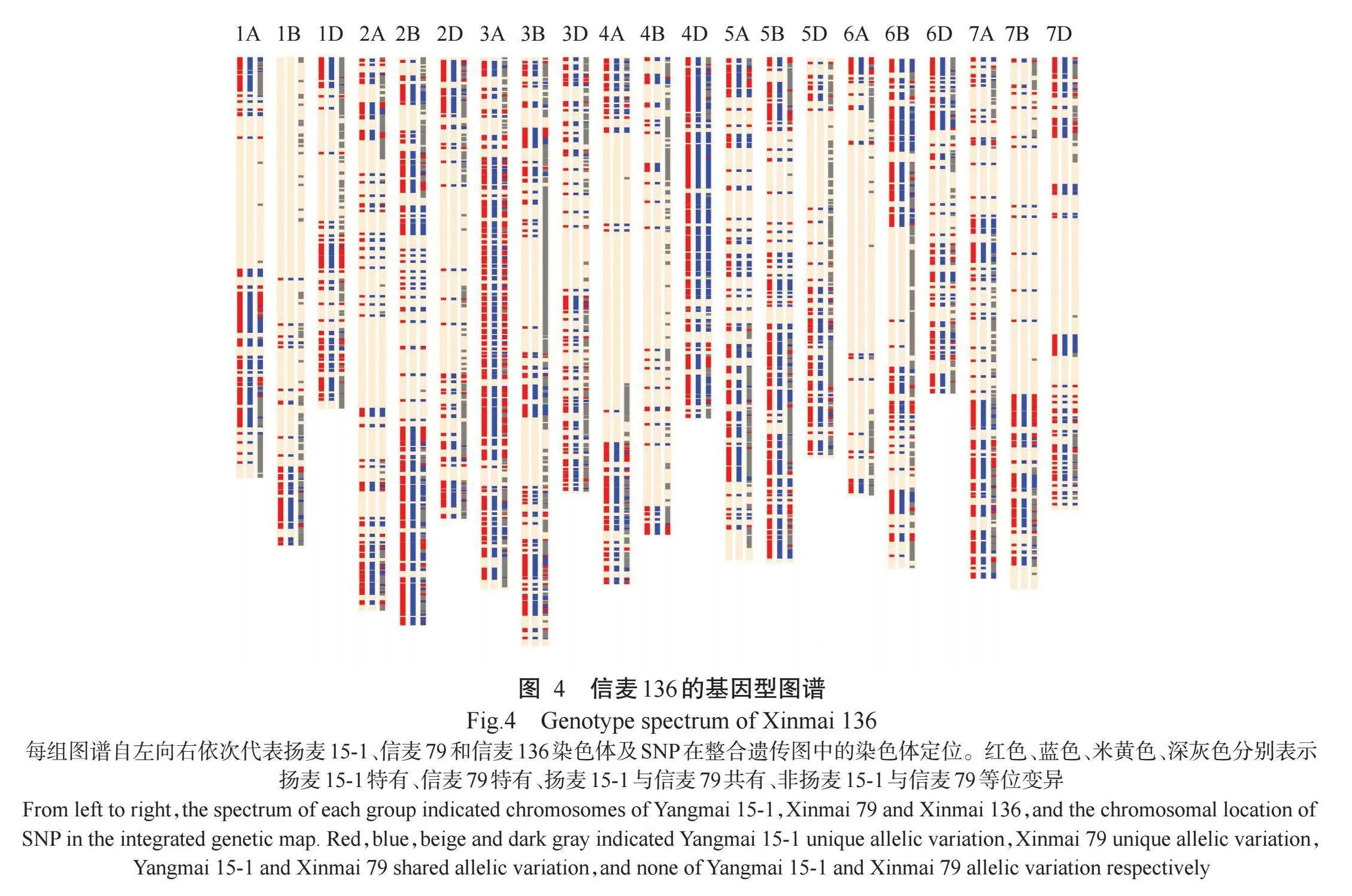
2.3亲本遗传信息在信麦136染色体上的分布情况从信麦136的基因图谱(图4)可看出,母本扬麦以染色体大片段形式来实现,如1A、1D、2B、4D、6B、7A、7D等染色体;信麦136只有在1A、1D、2D、3B、4A、5A、5B、5D、6B等染色体上有大片段非亲本等位基因的变异,大多表现为重组,其中3B、5D、6B染色体上的大片段重组较多,超过50.00%;另外的13条染色体上未出现较多的染色体大片段重组在整合遗传图谱中。信麦136在D组染色体集成亲本的区段较为集中,而且是父本信麦79遗传给信麦136的区段较多。在4D和6D染色体上分布着父本信麦79较多的遗传位点,即这2条染色体遗传信息绝大部分(超过60.00%)是由父本信麦79传递给信麦136;在3A、7B等染色体上分布着扬麦15-1较多的遗传位点,即这2条染色体遗传信息绝大部分(超过60.00%)是由母本扬麦15-1传递给信麦136。可见,信麦136 SNP基因型图谱、SNP位点分析结果、遗传贡献率分析结果三者均呈现出较好的统一性。
2.4重要性状功能基因挖掘结果
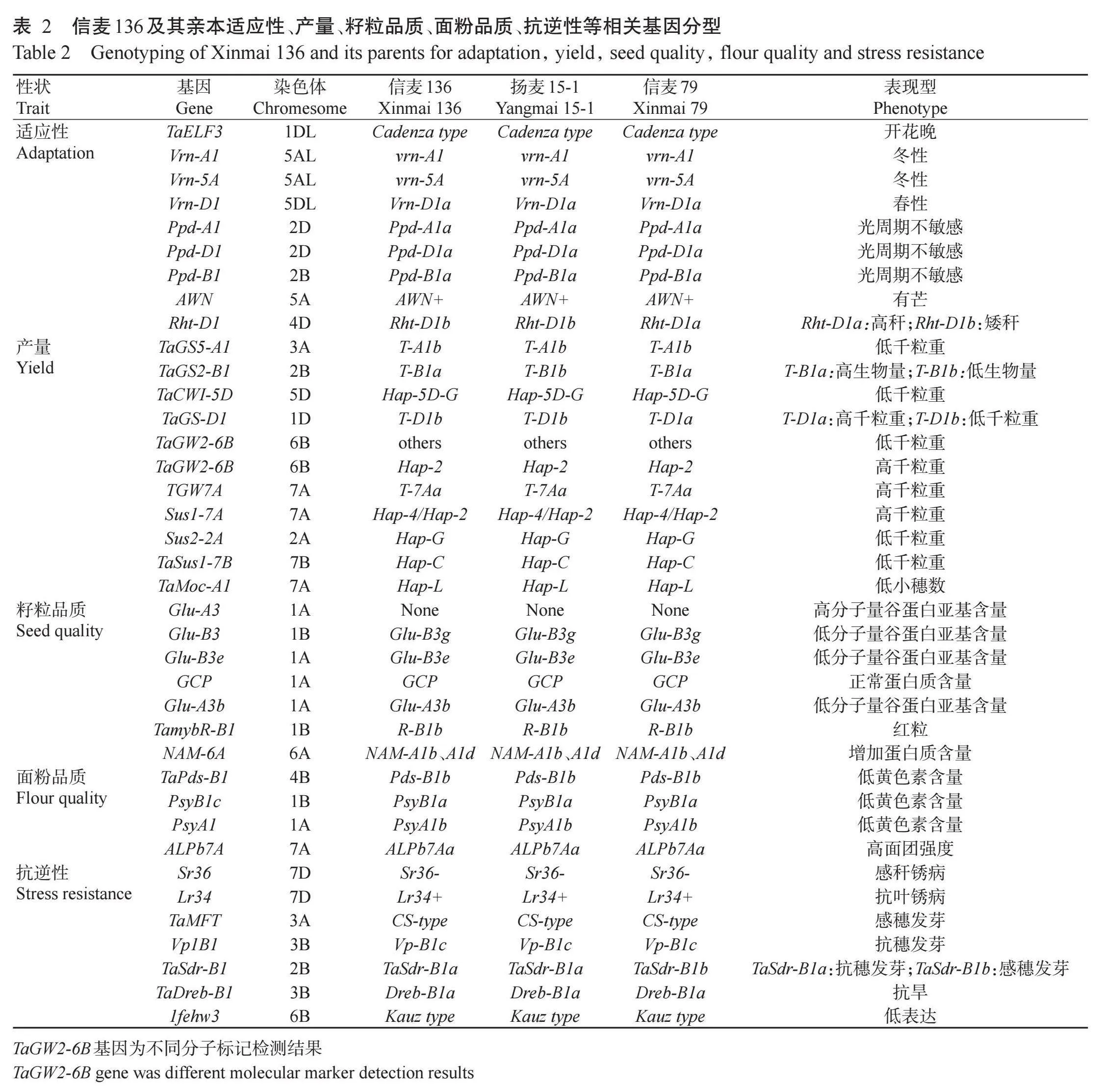
从信麦136及其母本扬麦15-1和父本信麦79中共检测到9个适应性相关基因,包括1个开花基因(TaELF3)、3个春化基因(Vrn-A1、Vrn-5A、Vrn-D1)、3个光周期不敏感基因(Ppd-A1a、Ppd-B1a和Ppd-D1a)、1个芒基因(AWN)和1个株高基因(Rht-D1),结果显示三者的开花基因、春化基因、光周期基因和芒基因完全一致,仅株高基因Rht-D1存在差异(表2),信麦136表现为矮秆,而母本扬麦15-1表现为矮秆,父本信麦79表现为高秆。
从信麦136及其母本扬麦15-1和父本信麦79中共检测到TaGS5-A1、TaGS2-B1、TaCWI-5D、TaGS-D1、TaGW2-6B、TaGW2-6B、TGW7A、Sus1-7A、Sus2-2A、TaSus1-7B、TaMoc-A1共11个产量相关基因(表2)。在TaGS5-A 1、TaC WI-5D、TaG W2-6B、Sus 2-2A和TaSus1-7B基因上,信麦136及其亲本均表现为低千粒重;在TaGW2-6B、TGW7A和Sus1-7A基因上,信麦136及其亲本均表现为高千粒重;在TaMoc-A1基因上,信麦136及其亲本均表现为低小穗数。在TaGS2-B1基因上,信麦136与父本信麦79表现为高生物量,而母本扬麦15-1表现为低生物量;在TaGS-D1基因上,信麦136与母本扬麦15-1表现为低千粒重,而父本信麦79表现为高千粒重。
从信麦136及其母本扬麦15-1和父本信麦79中共检测到Glu-A3、Glu-B3、Glu-B3e、GCP、Glu-A3b、TamybR-B1和NAM-6A等7个与籽粒品质相关基因(表2)。在Glu-A3基因上,信麦136及其亲本均表现为高分子量谷蛋白亚基含量;在Glu-B3、Glu-B3e和Glu-A3b基因上,信麦136及其亲本均表现为低分子量谷蛋白亚基含量;在GCP基因上,信麦136及其亲本均表现为正常蛋白质含量;在TamybR-B1基因上,信麦136及其亲本均表现为红粒;在NAM-6A基因上,信麦136及其亲本均表现为增加蛋白质含量。
从信麦136及母本扬麦15-1和父本信麦79中共检测到TaPds-B1、PsyB1c、PsyA1和ALPb7A等4个面粉品质相关基因(表2)。在TaPds-B1、PsyB1c和PsyA1基因上,信麦136及其亲本均表现为低黄色素含量;在ALPb7A基因上,信麦136及其亲本均表现为高面团强度。
从信麦136及母本扬麦15-1和父本信麦79中检测到Sr36、Lr34、TaMFT、Vp1B1、TaSdr-B1、TaDreb-B1和1fehw3等7个抗逆性相关基因。在Sr36基因上,信麦136及其亲本均表现为感秆锈病;在Lr34基因上,信麦136及其亲本均表现为抗叶锈病;在TaMFT基因上,信麦136及其亲本均表现为感穗发芽;在Vp1B1基因上,信麦136及其亲本均表现为抗穗发芽;在TaSdr-B1基因上,信麦136和母本扬麦15-1表现为抗穗发芽,而父本信麦79表现为感穗发芽;在TaDreb-B1基因上,信麦136及其亲本均表现为抗旱;在1fehw3基因上,信麦136及其亲本均表现为低表达(表2)。
3讨论
近年来,随着分子生物学技术的快速发展,高通量DNA芯片技术常用于小麦品种的多态性位点、遗传差异等研究。亓佳佳等(2005)利用63对SSR引物从小麦品种小偃6号及其衍生品种(系)中共检测到175个等位变异;王珊珊等(2007)利用20对SSR引物从小麦品种矮孟牛及其衍生后代中共检测到120个SSR多态性位点;李玉刚等(2018)利用350对SSR引物从小麦品种青农2号及其亲本(鲁麦14/烟农15//矮秆麦)中检测146个SSR多态性位点。而利用高通量SNP芯片能检测到更多的多态性位点,孔子明和宋晓明(2020)利用90K SNP芯片从小麦品种周麦16及其亲本(周麦9号和周8425B)中共检测到81587个SNP位点,检测到7780个差异SNP位点;陈晓杰等(2021)利用小麦50K SNP芯片从小麦品种郑品麦24号及其亲本(豫麦34-6和豫同194)中共检测到41382个纯合SNP位点,检测到8322个纯合差异SNP位点;吴胜男等(2021)利用55K SNP芯片从小麦品种陕农33及其亲本(陕农981和新麦18)中共检测到50430个SNP位点,其中有17024个多态性SNP位点;王瑞霞等(2022)采用90K SNP芯片从小麦品种泰山22及其亲本(鲁麦18和鲁麦14)中共获得20139个SNP位点,其中有7513个SNP在泰山22及其亲本(鲁麦18和鲁麦14)上表现出差异。本研究利用小麦55K SNP育种芯片从信麦136及其亲本(扬麦15-1和信麦79)中检测到9399个多态性SNP位点,与SSR、RFLP和RAPD等分子标记相比,55K SNP育种芯片呈现出高通量、速度快、广覆盖的特点,故利用55K SNP芯片检测信麦136遗传组成及其亲本(扬麦15-1和信麦79)的遗传贡献更全面更详细。在品种选育过程中,一般会选择当地主推的品种作为亲本,以更好地适应当地的生态类型,信阳地区小麦赤霉病和锈病发生频繁,是影响信阳小麦生产发展的重要因素。信麦79是信阳市农业科学院选育的高代稳定品系,具有灌浆速度快、熟相较好、高抗白粉病,中感抗纹枯病、中抗赤霉病、耐寒性和耐湿性较好等优点;扬麦15-1是从扬麦15选育出的优异变异株,扬麦15在信阳地区具有较大的推广种植面积、具有中感赤霉病、中感纹枯病和白粉病等优点。因此,信麦136在抗病选择压力下遗传了信麦79的大部分遗传物质。从本研究绘制的信麦136的基因图谱可知,母本扬麦15-1和父本信麦79杂交育成信麦136过程中在A、B、D基因组发生不均匀基因重组,如在1B、2D、3B、3D、4B和5D等染色体上,多为信麦136的特异位点,仅有少数重组;母本扬麦15-1的3A染色体几乎整条传递至信麦136,父本信麦79的4D染色体几乎整条传递至信麦136,且1D、2B、5A、6B、6D、7A和7D等染色体也有较多遗传物质进行重组和整合。Lai等(2010)研究发现,小麦品种Zheng 58的遗传组成图谱也是通过百万数量级SNP计算完成的,更加客观真实地证明染色体传递主要是通过大片段传递的现象,进一步验证了本研究发现遗传物质从亲本向信麦136的传递规律。千粒重是小麦产量三大构成要素之一,高千粒重对产量提升具有重要的作用。本研究发现,信麦136及其亲本(扬麦15-1和信麦79)均含有TaGW2-6B、TGW7A和Sus1-7A等3个高千粒重基因,表明信麦136从亲本遗传了高千粒重基因,其产量还有提升的空间;在TaPds-B1、PsyB1c和PsyA1等3个基因上,三者均表现出低黄色素含量;在ALPb7A基因上,三者均表现出高面团强度,表明TaPds-B1、PsyB1c、PsyA1和ALPb7A基因均从亲本传递给信麦136,使得信麦136的面粉白度和品质明显提高,商品性更好。
本研究通过KASP标记检测发现,信麦136含有优良的功能基因,包括3个光周期不敏感基因(Ppd-A1a、Ppd-B1a和Ppd-D1a)、1个矮秆基因(Rht-D1)、1个高生物量基因(TaGS2-B1)、3个高千粒重基因(TaGW2-6B、TGW7A和Sus1-7A)、1个高分子量谷蛋白亚基含量基因(Glu-A3)、3个低分子量谷蛋白亚基含量基因(Glu-B3、Glu-B3e和Glu-A3b)、1个正常蛋白质含量基因(GCP)和1个增加蛋白质含量基因(NAM-6A)。此外,信麦136还含有3个低黄色素含量基因(TaPds-B1、PsyB1c和PsyA1)、1个高面团强度基因(ALPb7A)、1个抗叶锈病基因(Lr34)、2个抗穗发芽基因(Vp1B1和TaSdr-B1)、1个抗旱基因(TaDreb-B1)。正是由于含有这些优良的功能基因使信麦136表现出春性、播期弹性大、综合抗性强、中早熟、分蘖力强、品质优良、中大粒、灌浆快、籽粒饱满、丰产稳产性好、广适等特性。但信麦136的赤霉病抗性表现为中感,小麦赤霉病的发病和流行导致产量和品质损失,危害人类和动物健康,因此,今后应加强对信麦136赤霉病抗性基因的研究,挖掘其抗赤霉病潜力,也是该品种改良的主攻方向。
4结论
母本扬麦15-1和父本信麦79的遗传物质向子代信麦136传递过程中受育种定向选择的影响发生严重偏分离,同时遗传物质传递的主要表现方式为染色体间大片段重组和整合,且信麦136的遗传物质主要来自父本信麦79,重要性状功能基因解析结果与其在实际生产中的表现(早熟、抗倒伏、稳产、品质优良、中筋偏弱、抗病、抗旱、抗穗发芽)基本一致。
参考文献(References):
陈晓杰,范家霖,杨保安,杨科,张福彦,程仲杰,王嘉欢,白鹤峰,张建伟.2021.郑品麦24号的遗传构成及重要性状功能基因组成解析[J].河南农业科学,50(11):15-27.[Chen X J,Fan J L,Yang BA,Yang K,Zhang F Y,Cheng Z J,Wang J H,Bai H F,Zhang J W.2021.Genetic compo-nent and important traits gene composition of new wheat variety Zhengpinmai 24[J].Journal of Henan Agricultural Sciences,50(11):15-27.]doi:10.15933/j.cnki.1004-3268.2021.11.003.
杜莹莹,顾正中,周羊梅,王安邦,杨子博,张勇.2023.江苏淮北小麦品种(系)重要性状功能基因的KASP检测[J].麦类作物学报,43(3):279-287.[Du Y Y,Gu Z Z,Zhou Y M,Wang A B,Yang Z B,Zhang Y.2023.KASP detection of functional genes related to important traits in wheat varieties(lines)in Huaibei region of Jiangsu Province[J].Journal of Triticeae Crops,43(3):279-287.]doi:10.7606/j.issn.1009-1041.2023.03.03.
孔子明,宋晓朋.2020.小麦品种周麦16的遗传构成分析[J].种子,39(9):117-119.[Kong Z M,Song X P.2020.Genetic composition analysis of wheat variety Zhoumai 16[J].Seed,39(9):117-119.]doi:10.16590/j.cnki.1001-4705.2020.09.117.
李聪,马建,刘航,丁浦洋,杨聪聪,张涵,秦娜娜,兰秀锦.2019.基于小麦55K SNP芯片检测小麦穗长和株高性状QTL[J].麦类作物学报,39(11):1284-1292.[Li C,Ma J,Liu H,Ding PY,Yang C C,Zhang H,Qin N N,Lan X J.2019.Detection of QTLS for spike length and plant height in wheat based on 55K SNP array[J].Journal of Triticeae Crops,39(11):1284-1292.]doi:10.7606/j.issn.1009-1041.2019.11.03.
李玉刚,任民,孙绿,王圣健,韩梅,李振清,翟晓灵,代小雁,侯元江,盖红梅.2018.利用SSR和SNP标记分析鲁麦14对青农2号的遗传贡献[J].作物学报,44(2):159-168.[Li Y G,Ren M,Sun L,Wang S J,Han M,Li Z Q,Zhai X L,Dai X Y,Hou Y J,Gai H M.2018.Genetic contribution of Lumai 14 to Qingnong 2 revealed by SSR and SNP markers[J].Acta Agronomica Sinica,44(2):159-168.]doi:10.3724/SP.J.1006.2018.00159.
李振声.2010.我国小麦育种的回顾与展望[J].中国农业科技导报,12(2):1-4.[Li Z S.2010.Retrospect and prospect of wheat breeding in China[J].Journal of Agricultural Science and Technology,12(2):1-4.]doi:10.3969/j.issn.1008-0864.2010.02.01.
林作楫,揭声慧.2005.近50年河南省小麦育种工作的回顾[J].河南农业科学,(9):5-8.[Lin Z J,Jie S H.2005.Re-view of wheat breeding in Henan Province in the past 50 years[J].Journal of Henan Agricultural Sciences,(9):5-8.]doi:10.15933/j.cnki.1004-3268.2005.09.001.
马艳明,娄鸿耀,陈朝燕,肖菁,徐麟,倪中福,刘杰.2020.新疆冬小麦地方品种与育成品种基于SNP芯片的遗传多样性分析[J].作物学报,46(10):1539-1556.[Ma Y M,Lou H Y,Chen Z Y,Xiao J,Xu L,Ni Z F,Liu J.2020.Genetic diversity assessment of winter wheat landraces and cultivars in Xinjiang via SNP array analysis[J].Acta Agronomica Sinica,46(10):1539-1556.]doi:10.3724/SP.J.1006.2020.91077.
亓佳佳,韩芳,马守才,张莉莉,余欣欣,陈蕴文,毕晓静,史秀秀,牛娜,张改生.2015.小麦骨干亲本小偃6号及其衍生品种(系)的遗传解析[J].西北农林科技大学学报(自然科学版),43(11):45-53.[Qi J J,Han F,Ma S C,Zhang L L,Yu X X,Chen Y W,Bi X J,Shi X X,Niu N,Zhang G S.2015.Genetic dissection of wheat mile stone parent Xiaoyan 6 and its derivatives[J].Journal Northwest Aamp;F University(Natural Science Edition),43(11):45-53.]doi:10.13207/cnki.nwafu.2015.11.008.
隋建枢,陈天青,罗永露,吴文强,程斌,王伟,何庆才.2023.75份贵州小麦品种(系)抗病基因的KASP标记检测[J].山地农业生物学报,42(3):80-85.[Sui J S,Chen T Q,Luo Y L,Wu W Q,Cheng B,Wang W,He Q C.2023.KASP marker detection of disease resistance genes in 75 Guizhou wheat varieties(lines)[J].Journal of Mountain Agriculture and Biology,42(3):80-85.]doi:10.15958/j.cnki.sdnyswxb.2023.03.014.
王君婵,吴旭江,胡文静,张晓,张勇,高德荣,别同德,张伯桥.2019.扬麦系列品种(系)重要性状功能基因的KASP检测[J].江苏农业学报,35(6):1271-1283.[Wang J C,Wu X J,Hu W J,Zhang X,Zhang Y,Gao D R,Bie T D,Zhang B Q.2019.Kompetitive allele specific PCR(KASP)assays for functional genesof important trait in Yangmai series wheat cultivars(lines)[J].Jiangsu Journal of Agricultural Sciences,35(6):1271-1283.]doi:10.3969/j.issn.1000-4440.2019.06.002.
王瑞霞,翟晓灵,李玉刚,牟秋焕,孙盈盈,孙宪印,米勇,吕广德,盖红梅,钱兆国.2022.利用小麦90K SNP芯片研究泰山22的遗传组成[J].中国农业科技导报,24(4):21-29.[Wang R X,Zhai X L,Li Y G,Mou Q H,Sun Y Y,Sun X Y,Mi Y,LüG D,Gai H M,Qian Z G.2022.Genetic composition of Taishan 22 using high-density 90K SNP array[J].Journal of Agricultural Science and Techno-logy,24(4):21-29.]doi:10.13304/j.nykjdb.2021.0721.
王珊珊,李秀全,田纪春.2007.利用SSR标记分析小麦骨干亲本“矮孟牛”及衍生品种(系)的遗传多样性[J].分子植物育种,5(4):485-490.[Wang S S,Li X Q,Tian J C.2007.Genetic diversity of mainparent of wheat‘Aimeng-niu’and its pedigree on SSR markers[J].Molecular Plant Breeding,5(4):485-490.]doi:10.3969/j.issn.1672-416X.2007.04.007.
吴胜男,李英壮,王娜,刘录祥,谢彦周,王成社.2021.小麦新品种陕农33的遗传构成分析[J].麦类作物学报,41(2):134-139.[Wu S N,Li Y Z,Wang N,Liu L X,Xie Y Z,Wang C S.2021.Dissection of genetic components in the new high-yielding wheat cultivar Shaannong 33[J].Jour-nal of Triticeae Crops,41(2):134-139.]doi:10.7606/j.issn.1009-1041.2021.02.02.
张维军,赵俊杰,何进尚,郝晨阳,王小亮,亢玲,张富国,张学勇,陈东升.2022.宁夏小麦种质资源粒重基因KASP标记检测及验证[J].植物遗传资源学报,23(2):493-504.[Zhang W J,Zhao J J,He J S,Hao C Y,Wang X L,Kang L,Zhang F G,Zhang X Y,Chen D S.2022.Detection and validation of grain weight related genes using KASP assays in Ningxia wheat germplasm[J].Journal of Plant Genetic Resources,23(2):493-504.]doi:10.13430/j.cnki.jpgr.20211225001.
赵吉平,任杰成,郭鹏燕,许瑛,任超.2019.小麦常规育种技术与改进策略研究[J].中国种业,(7):13-15.[Zhao J P,
Ren J C,Guo P Y,Xu Y,Ren C.2019.Study on wheat general breeding techniques and improvement strategies[J].China Seed Industry,(7):13-15.]doi:10.19462/j.cnki.1671-895x.20190627.011.
Lander E S.1996.The new genomics:Global views of biology[J].Science,274(5287):536-539.doi:10.1126/science.274.5287.536.
Liu J J,Luo W,Qin N N,Ding P Y,Zhang H,Yang C C,Mu Y,Tang H P,Liu Y X,Li W,Jiang Q T,Chen G Y,Wei Y M,Zheng Y L,Liu C J,Lan X J,Ma J.2018.A 55K SNP array based genetic map and its utilization in QTL map-ping for proroductive tiller numbern in common wheat[J].Theoretical and Applied Genetics,131(11):2439-2450.doi:10.1007/s00122-018-3164-9.
Lai J S,Li R Q,Xun X,Jin W W,Xu M L,Zhao H N,Xiang Z K,Song W B,Ying K,Zhang M,Jiao Y P,Ni P X,Zhang J G,Li D,Guo X S,Ye K X,Jian M,Wang B,Zheng H S,Liang H Q,Zhang X Q,Wang S C,Chen S J,Li J S,Fu Y,Springer N M,Yang H M,Wang J,Dai J R,Schnable P S,Wang J.2010.Genome-wide patterns of genetic variation among elite maize inbred lines[J].Nature Genetics,42(11):1027-1030.doi:10.1038/ng.684.
Qureshi N,Bariana H S,Zhang P,McIntosh R,Bansal U K,Wong D,Hayden M J,Dubcovsky J,Shankar M.2018.Genetic relationship of stripe rust resistance genes Yr34 and Yr48 in wheat and identification of linked KASP mar-kers[J].Plant Disease,102(2):413-420.doi:10.1094/PDIS-08-17-1144-RE.
Ren T H,Hu Y S,Tang Y Z,Li C S,Yan B J,Ren Z L,Tan F Q,Tang Z X,Fu S L,Li Z.2018.Utilizaion of a wheat 55K SNP Array for mapping of major QTL for temporal expression of the tiller number[J].Frontiers in Plant Scien-ce,9:333.doi:10.3389/fpls.2018.00333.
Rasheed A,Wen W E,Gao F M,Zhai S N,Jin H,Liu J D,GuoQ,Zhang Y J,Dreisigacker S,Xia X C,He Z H.2016.Development and validation of KASP assays for genes underpinning key economic traits in bread wheat[J].Theo-retical and applied genetics,129(10):1843-1860.doi:10.1007/s00122-016-2743-x.
Su Z Q,Jin S J,Zhang D D,Bai G H.2018.Development and validation of diagnostic markers for Fhb1 region,a major QTL for Fusarium head blight resistance in wheat[J].Theoretical and Applied Genetics,131(11):2371-2380.doi:10.1007/s00122-018-3159-6.
Ur Rehman S,Ali Sher M,Saddique MA B,Ali Z,Khan MA,Mao X G,Irshad A,Sajjad M,Ikram R M,Naeem M,Jing R.2021.Development and exploitation of KASP assays for genes underpinning drought tolerance among wheat cultivars from Pakistan[J].Frontiers in Genetics,12:684702.doi:10.3389/FGENE.2021.684702.
Ye X L,Li J,Cheng Y K,Yao F J,Long L,Wang Y Q,Wu Y,Li J,Wang J R,Jiang Q T,Kang H Y,Li W,Qi P F,Lan X J,Ma J,Liu Y X,Jiang Y F,Wei Y M,Chen X M,Liu C J,Zheng Y L,Chen G Y.2019.Genome-wide association study reveals new loci for yield-related traits in Sichuanwheat germplasm under stripe rust stress[J].BMC Geno-mics,20(1):640.doi:org/10.1186/s 12864-019-6005-6.
Ye X L,Li J,Cheng Y K,Yao F J,Li L,Yu C,Wang Y Q,Wu Y,Li J,Wang J R,Jiang Q T,Li W,Ma J,Wei Y M,Zheng Y L,Chen G Y.2019.Genome wide association study of resistance to stripe rust(Puccinia striiformisf.sp.tritici)in Sichuan wheat[J].BMC Plant Biology,19(1):147.doi:10.1186/s 12870-019-1764-4.
Zhang P P,Li X,Gebrewahid T W,Liu H X,Xia X C,He Z H,Li Z F,Liu D Q.2019a.QTL mapping of adult-plant resis-tance to leaf and seripe rust in wheat cross SW 8588/Thatcher using the wheat 55K SNP array[J].Plant Disea-se,103(12):3041-3049.doi:10.1094/PDIS-02-19-0380-RE.
Zhang P P,Lan C X,Asad M A,Gebrewahid T W,Xia X C,He Z H,Li Z F,Liu D Q.2019b.QTL mapping of adult plant resistance to leaf rust in the chinese landraces Pinyuan 50/Mingxian 169 using the wheat 55K SNP array[J].Molecular Breeding,39(7):1-14.doi:10.1007/s11032-019-1004-5.
(责任编辑陈燕)

