黄曲条跳甲短链气味结合蛋白基因PstrOBP-sc分子和功能特点分析


摘要:【目的】分析黄曲条跳甲(Phyllotretastriolata)短链气味结合蛋白(Odorant binding protein,OBP)基因PstrOBP-sc分子和功能特点,为揭示昆虫短链OBP基因的功能提供参考。【方法】利用逆转录聚合酶链反应(RT-PCR)克隆黄曲条跳甲短链Classic OBP基因,并对其进行生物信息学分析;通过逆转录聚合酶链反应技术分析短链OBP基因在黄曲条跳甲雌、雄成虫不同组织中的表达情况;构建重组表达载体异源表达短链OBP基因,以聚丙烯酰胺凝胶电泳(SDS-PAGE)和蛋白质印迹法(Western blotting)验证目的蛋白;运用荧光结合试验检测短链OBP的配体结合能力。【结果】通过逆转录聚合酶链反应克隆验证,成功获得1条具有完整开放阅读框的黄曲条跳甲短链OBP基因PstrOBP-sc。PstrOBP-sc基因含有1个357 bp的开放阅读框,编码118个氨基酸残基,N末端具有1个由20个氨基酸残基组成的信号肽,成熟蛋白PSTROBP-SC仅由98个氨基酸残基组成,序列中含有6个保守的半胱氨酸,属于典型短链OBP基因。蛋白二级结构预测结果显示,PSTROBP-SC蛋白仅存在5个α-螺旋,且在第5个α-螺旋的下游仅有2个氨基酸残基。PstrOBP-sc基因组织表达模式及异源表达的PSTROBP-SC蛋白配体结合能力研究结果表明,PstrOBP-sc基因在黄曲条跳甲雌、雄成虫的所有供试组织中均有表达,而非特异性表达于嗅觉器官。同时,异源表达PSTROBP-SC不能结合荧光探针1-NPN。【结论】PstrOBP-sc基因的分子和功能特点不同于当前广泛研究的中长链OBP基因,其在生物体内很可能行使嗅觉以外的其他生理功能。
关键词:黄曲条跳甲;短链气味结合蛋白;分子特征;组织表达模式;荧光结合试验
中图分类号:S433.5文献标志码:A文章编号:2095-1191(2024)08-2464-09
Molecular and functional characteristics of a short-chain odorant binding protein gene PstrOBP-sc from Phyllotretastriolata
TIAN Xiao-li SONG Xu-rong MAO Yong-na3,LI Chuan-ren ZHANG Guo-hui2*
(1College of Life Science,Yangtze University,Jingzhou,Hubei 434025,China;2College of Agriculture,YangtzeUniversity/Institute of Entomology,Yangtze University,Jingzhou,Hubei 434025,China;3Jingzhou Branch,Hubei Academy of Forestry,Jingzhou,Hubei 434020,China)
Abstract:【Objective】The study aimed to analyze the molecular and functional characteristics of the short-chain odo-rant binding protein(OBP)gene PstrOBP-sc from Phyllotretastriolata,providing reference for exploring the function of short-chain OBP genes in insects.【Method】The short-chain Classic OBP gene from P.striolata was cloned using reverse transcription-polymerase chain reaction(RT-PCR)and subjected to bioinformatics analysis.The expression of the short-chain OBP gene in different tissues of male and female adult P.striolata was analyzed by RT-PCR.A recombinant expres-sion vector was constructed for the heterologous expression of the short chain OBP gene,and the target protein was veri-fied using sodium dodecyl sulfate-polyacrylamide gel electrophoresis(SDS-PAGE)and Western blotting techniques.Theligand-binding ability of the short-chain OBP was detected using fluorescence binding assay.【Result】A short-chain OBP gene(PstrOBP-sc)from P.striolata with a complete open reading frame was successfully obtained through RT-PCR clo-ning and verification.ThePstrOBP-sc gene contained an open reading frame of 357 bp,encoding 118 amino acid resi-dues,with a signal peptide of 20 amino acid residues at the N-terminal end.The mature protein PSTROBP-SC consisted of only 98 amino acid residues and contained 6 conserved cysteines in its sequence,making it a typical short-chain OBP gene.Protein secondary structure prediction showed that the PSTROBP-SC protein contained only 5α-helixes,and there were only 2 amino acid residues in the downstream of the fifthα-helix.The tissue expression pattern of the PstrOBP-sc gene and the ligand-binding ability of the heterologously expressed PSTROBP-SC protein showed that the PstrOBP-sc gene was expressed in all tested tissues of male and female adult P.striolata,and was not specifically expressed in olfac-tory organs.Additionally,the heterologously expressed PSTROBP-SC could not bind to the fluorescence probe 1-NPN.【Conclusion】The molecular and functional characteristics of PstrOBP-sc gene are different from those of the currently widely studied medium-long chain OBP genes,and it is likely to perform other physiological functions besides olfactionin the organism.
Key words:Phyllotretastriolata;short-chain odorant binding protein;molecular characteristics;tissue expression pattern;fluorescence binding assay
Foundation items:National Natural Science Foundation of China(31972274);The Outstanding Youth Science and Technology Innovation Team Project of Colleges and Universities in Hubei(T2022009);Laboratory Research Project of Colleges and Universities in Hubei(HBSY2023-016)
0引言
【研究意义】气味结合蛋白(Odorant binding pro-tein,OBP)是昆虫的重要嗅觉蛋白。OBP功能研究已成为昆虫嗅觉识别机制研究的热点之一,并被视为筛选新型高效引诱剂和驱避剂的理想靶标(Tian et al.,2016;da Costa et al.,2019;Li etal.,2021)。目前,学者们对OBP功能研究主要集中在Classic OBP(即多肽链中存在6个保守的半胱氨酸残基的OBP)的中长链OBP(120~160个氨基酸残基)(Rana et al.,2024)。有关短链OBP(约100个氨基酸残基)分子和功能研究鲜有报道。因此,深入探究短链OBP分子和功能特点对全面认识昆虫OBP家族,以及利用该家族成员应用于害虫防治具有重要的理论和实践意义。【前人研究进展】嗅觉对昆虫的生存和繁殖至关重要。昆虫利用嗅觉感受环境中挥发性化合物,并根据这些化合物携带的信息寻找配偶、食物、产卵地点以及躲避天敌(Zhao et al.,2023)。触角是昆虫主要的嗅觉器官,触角的嗅觉功能通过着生于其表面的化学感受器实现(杨成都等,2024)。昆虫化学感受器内的OBP可结合进入化学感受器内的气味分子,这个过程是昆虫识别环境气味分子的第1步生化反应(Kaissling,2009;Leal,2013)。OBP可结合并运送气味分子至化学感受器内嗅觉神经元树突上的嗅觉受体,此时神经电信号产生并传入昆虫大脑,调控昆虫各种嗅觉行为反应(Leal,2013;刘伟和王桂QTbuSeUhE/GD2qC1QqH8bFrGyZ7GIeHczSjrmHse/4k=荣,2020)。自从多音天蚕蛾(Antheraea poly-phemus)雄虫触角中首次发现OBP(Vogt and Riddi-ford,1981)后,大量OBP从超过100种昆虫中被鉴定出来(Rihani et al.,2021),如果蝇(Drosophila mela-nogaster)(Hekmat-Scafe et al.,2002)、赤拟谷盗(Tri-boliumcastaneum)(Dippel etal.,2014)和家蚕(Bom-byx mori)(Gonget al.,2009)等。昆虫OBP是一类小分子量(15~20 kD)水溶性分泌蛋白。根据OBP一级结构长度可将其分为长链OBP(约160个氨基酸残基)、中链OBP(约120个氨基酸残基)和短链OBP(约100个氨基酸残基)(Rihani et al.,2021)。典型的昆虫OBP最明显的标志是其氨基酸序列中存在6个保守的半胱氨酸残基(C1~C6),其中第2和第3位(C2和C3)以及第5和第6位(C5和C6)半胱氨酸残基之间的氨基酸残基数一般固定不变,分别为3和8个氨基酸残基(Hull et al.,2014)。在此基础上,根据氨基酸序列中半胱氨酸残基的缺失或增加,昆虫OBP可分为3个亚类:(1)Classic OBP,即序列中具有上述6个保守半胱氨酸残基;(2)Plus-C OBP,这类OBP序列中除了具有Classic OBP中的6个保守半胱氨酸残基外,在第6个保守半胱氨酸下游还存在1个独特的C端结构,该结构中包含3个保守的半胱氨酸和1个保守的脯氨酸(Hekmat-Scafe et al.,2002)。2004年学者们提出了这类OBP的基序(motif)为C1-X20-41-C2-X3-C3-X41-46-C4-X19-29-C4a-X9-C5-X8-C6-P-X9-10-C6a-X9-10(Zhou et al.,2004);(3)Minus-C OBP,这类OBP序列中缺少Classic OBP的6个保守半胱氨酸残基中的2个,一般是第2和第5位的半胱氨酸残基缺失,这2个半胱氨酸被认为在昆虫OBP三维结构中会形成二硫键以维持OBP的立体结构稳定(Hekmat-Scafe et al.,2002)。【本研究切入点】目前,学者们对Classic OBP的研究最多,并主要集中于中长链Classic OBP,关于黄曲条跳甲(Phyllotretastriolata)短链Classic OBP的分子和功能特点研究鲜有报道。【拟解决的关键问题】通过逆转录聚合酶链反应(RT-PCR)克隆黄曲条跳甲短链气味结合蛋白基因PstrOBP-sc,明确PstrOBP-sc基因生物信息学及其在成虫不同组织中的表达情况;构建重组表达载体异源表达PstrOBP-sc,以聚丙烯酰胺凝胶电泳(SDS-PAGE)和蛋白质印迹法(Western blotting)验证目的蛋白,运用荧光结合试验验证细菌表达的PSTROBP-SC配体结合能力,分析PstrOBP-sc基因的分子和功能特点,为揭示昆虫短链OBP基因的功能提供参考。
1材料与方法
1.1试验材料
1.1.1供试虫源本研究所用虫源采集自湖北省枝江市郊白菜田(30°43′N,111°82′E)。
1.1.2主要试剂Trans-T1和BL21(DE3)感受态细胞(北京全式金生物技术股份有限公司);Mini-BEST Universal RNA Extraction Kit、DL2000 DNA Marker、限制性内切酶BamHⅠ和HindⅢ、Prime-ScriptTM RT reagent Kit with gDNA Eraser、Premix Taq试剂和DNA Ligation Kit[宝生物工程(大连)有限公司];质粒提取试剂盒(美国Omega公司);透析袋和Bradford蛋白浓度测定试剂盒(北京索莱宝科技有限公司);Tris-甘氨酸转模缓冲液(pH 8.3,10×)、BSA封闭液、抗体稀释液、TBST缓冲液(pH 8.0,10×)和丽春红染色液(江苏康为世纪生物科技有限公司);pGEM-T easy vector(美国Promega公司);化学发光检测试剂盒和纯化树脂Ni-NTA His·Bind Resin(上海七海复泰生物科技有限公司);重组肠激酶(上海近岸科技有限公司)。
1.1.3主要仪器Gel Doc XR+凝胶成像系统、Mini-PROTEAN Tetra cell电泳仪、T100TM PCR仪(美国Bio-Rad公司);UV-5100B紫外可见分光光度计(上海元析仪器有限公司);F-7000荧光分光光度计(日本日立公司);Scientz-IID超声波细胞粉碎机(宁波新芝生物科技股份有限公司);Tanon 5200化学发光成像系统(上海天能生命科学有限公司)。
1.2黄曲条跳甲总RNA提取及第一链cDNA合成
按照MiniBEST Universal RNA Extraction Kit说明书依次对黄曲条跳甲雌、雄成虫的头、胸、腹3个部位进行总RNA提取。RNA完整性和浓度经1%琼脂糖凝胶电泳和核酸蛋白测定仪检测验证后,参照反转录试剂盒说明书合成第一链cDNA。
1.3 PstrOBP-sc基因克隆鉴定
根据转录组数据中筛选到的1条短链OBP候选基因(PstrOBP-sc)序列,利用SnapGene设计引物扩增PSTROBP-SC蛋白的编码序列(表 OBPscF和OBPscR)。PCR反应体系20μL:cDNA模板(黄曲条跳甲雌成虫头部组织cDNA)1μL、Premix Taq 10μL、正、反向引物(10μmol/L)各1μL、ddH2O补足至20μL。扩增程序:94℃预变性3 min;94℃30 s,56℃30 s,72℃15 s,进行35个循环;72℃延伸5min。PCR产物经切胶回收目的片段、连接T载体、转化感受态细胞涂板培养。挑取单菌落置1a7784f562a600eeb597cb5bdd50fc59于LB液体培养基中振荡(37℃,200 r/min)培养12h,并进行菌液PCR验证,将验证正确的菌液送至武汉华大基因科技有限公司测序。
1.4 PstrOBP-sc基因生物信息学分析
利用DNAMAN 7.0预测PstrOBP-sc基因编码的氨基酸序列;采用在线工具SignalP 5.0(http://www.cbs.dtu.dk/services/SignalP-5.0/)预测PSTROBP-SC蛋白氨基端信号肽;采用ExPASy的ProtParam(https://web.expasy.org/protparam/)进行蛋白理化性质分析;采用TMHMM Server v2.0(https://services.healthtech.dtu.dk/service.php?TMHMM-2.0)分析蛋白跨膜情况;使用在线工具PredictProtein(http://www.predictpro-tein.org)预测蛋白二级结构;利用MEGA 5.1中的邻接法构建系统发育进化树。
1.5 PstrOBP-sc基因在黄曲条跳甲成虫不同组织中的表达分析
以1.2中各组织cDNA为模版,分别使用表1中引物(OBPscF和OBPscR),对PstrOBP-sc基因序列进行PCR扩增。反应体系20μL:cDNA模板1μL,Premix Taq 10μL,正、反向引物(10μmol/L)各0.5μL、ddH2O补足至20μL。扩增程序:94℃预变性3min;94℃30 s,56℃30 s,72℃15 s,进行35个循环;72℃延伸5min。以黄曲条跳甲Actin作为内参基因(表 PstrActinF和PstrActinR),PCR反应体系同上。扩增程序:94℃预变性3 min;94℃30 s,60℃30 s,72℃15 s,进行35个循环;72℃延伸5 min。利用1%琼脂糖凝胶电泳检测PCR产物,以此判断PstrOBP-sc基因在供试各组织中的表达情况。
1.6 PstrOBP-sc基因表达载体构建及其原核表达与纯化
将1.3测序验证无误的重组克隆载体以及表达载体pET-32a(+)进行双酶切(BamHⅠ和HindⅢ)。凝胶回收目的基因片段和表达载体的长片段。参照T4连接酶说明书连接二者的回收产物。连接产物转化克隆菌株(Trans-T1),挑选阳性单菌落送样测序。测序正确的重组表达载体转化表达菌株BL21(DE3)感受态细胞,挑取阳性单菌落用于原核小量表达。小量表达的菌液以1∶100接种至LB培养基(含终浓度0.5μL/mLAMP)中,37℃下200 r/min培养,当菌液在600 nm处的吸光度(OD600)达到0.7时,在培养体系中加入0.5 mmol/LIPTG,37℃下200r/min继续诱导6 h。之后,离心(4℃下8000 r/min持续11 min)收集菌体。为明确表达蛋白的可溶性,收集的菌体经裂解缓冲液处理后超声破碎,低温高速离心(4℃下12000 r/min持续10 min)后分别收集上清液和沉淀,SDS-PAGE电泳检测目的蛋白表达情况。后续蛋白纯化参照Ni-NTA His·Bind Resin纯化树脂说明书进行。使用Bradford蛋白浓度测定试剂盒检测PSTROBP-SC蛋白的浓度;利用重组肠激酶切除His-Tag标签,以此获得不含His-Tag的目的蛋白PSTROBP-SC。
1.7融合蛋白的Western bbotting检测
纯化后的融合蛋白经SDS-PAGE分离后,将分离胶、PVDF膜和滤纸板裁剪成适当大小,并在转膜夹负极面上按照海绵、滤纸板、凝胶、PVDF膜、滤纸板、海绵的顺序由下向上组装,在低温条件下恒流300 mA,进行2 h转膜;转膜完毕后,使用1×TBST Buffer对PVDF膜进行漂洗去除膜上的转膜缓冲液;使用丽春红染色液染色,判断蛋白是否转移到膜上,检测完毕后使用纯水漂洗PVDF膜上残留的丽春红染色液;将PVDF膜用封闭液(BSA Blocking Buffer)在摇床上室温封闭2 h,封闭结束后使用1×TBST Buffer洗去PVDF膜上的封闭液;用稀释好的鼠抗6×His单克隆抗体4℃静置过夜孵育;用1×TBST Buffer洗膜5次,加入羊抗鼠二抗,室温孵育1 h;1×TBST Buffer洗膜5次,加入ECL工作液室温孵育1 min,在Tanon 5200化学发光成像系统中检测免疫印记。
1.8 PSTROBP-SC荧光竞争结合试验
使用F-7000荧光分光光度计进行荧光结合试验。试验中设定的激发光波长、发射光谱范围和狭缝宽度参数参照Cheng等(2019)的方法。本试验所用的荧光探针N-苯基-1-萘胺(1-N-phenyl-naphtylamine,1-NPN)使用色谱级甲醇配置成终浓度为1 mmol/L的工作液。蛋白溶液使用常温20mmol/LTris-HCL(pH 7.4)缓冲液稀释至终浓度为2μmol/L。在含有2 mL稀释蛋白溶液的四通比色皿中依次加入终浓度为2、4、6、8、10、12、14、16、18、20、22和24μmol/L的1-NPN,分别测定荧光强度。试验重复3次。
2结果与分析
2.1 PstrOBP-sc基因克隆和序列分析结果
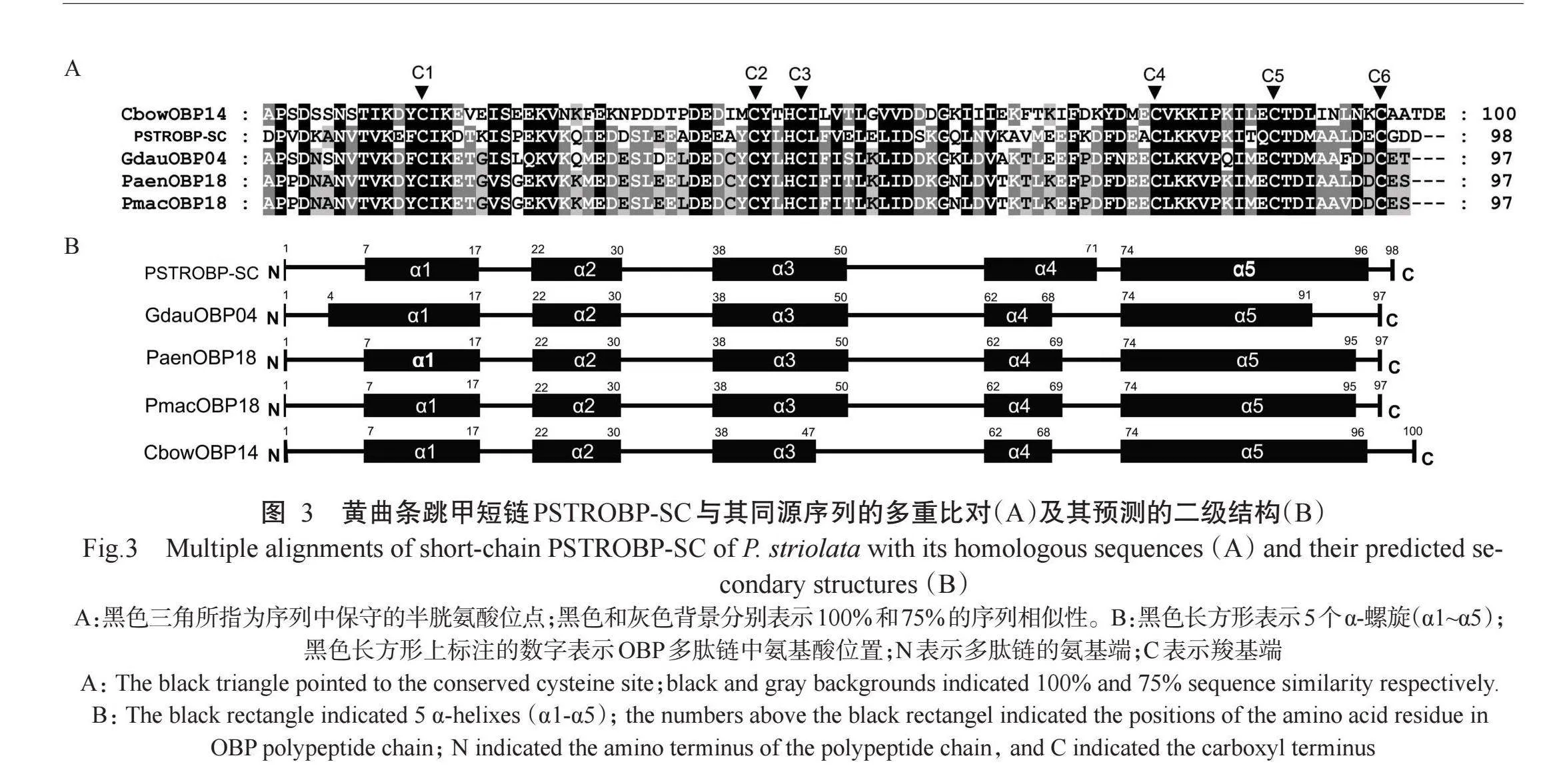
根据PstrOBP-sc基因克隆测序验证结果,PstrOBP-sc基因开放阅读框全长357bp,编码118个氨基酸残基,N末端具有1个由20个氨基酸残基组成的信号肽,成熟蛋白仅由98个氨基酸组成,其中包含6个保守的半胱氨酸残基,属于典型的短链ClsssicOBP(图1)。成熟PSTROBP-SC分子量11.1 kD,理论等电点(pI)4.26,亲水性总平均值(GRAVY)-0.437,无跨膜结构域,表明PSTROBP-SC是亲水性小分子量分泌蛋白,符合昆虫Clsssic OBP家族特征。
2.2 PSTROBP-SC系统发育分析
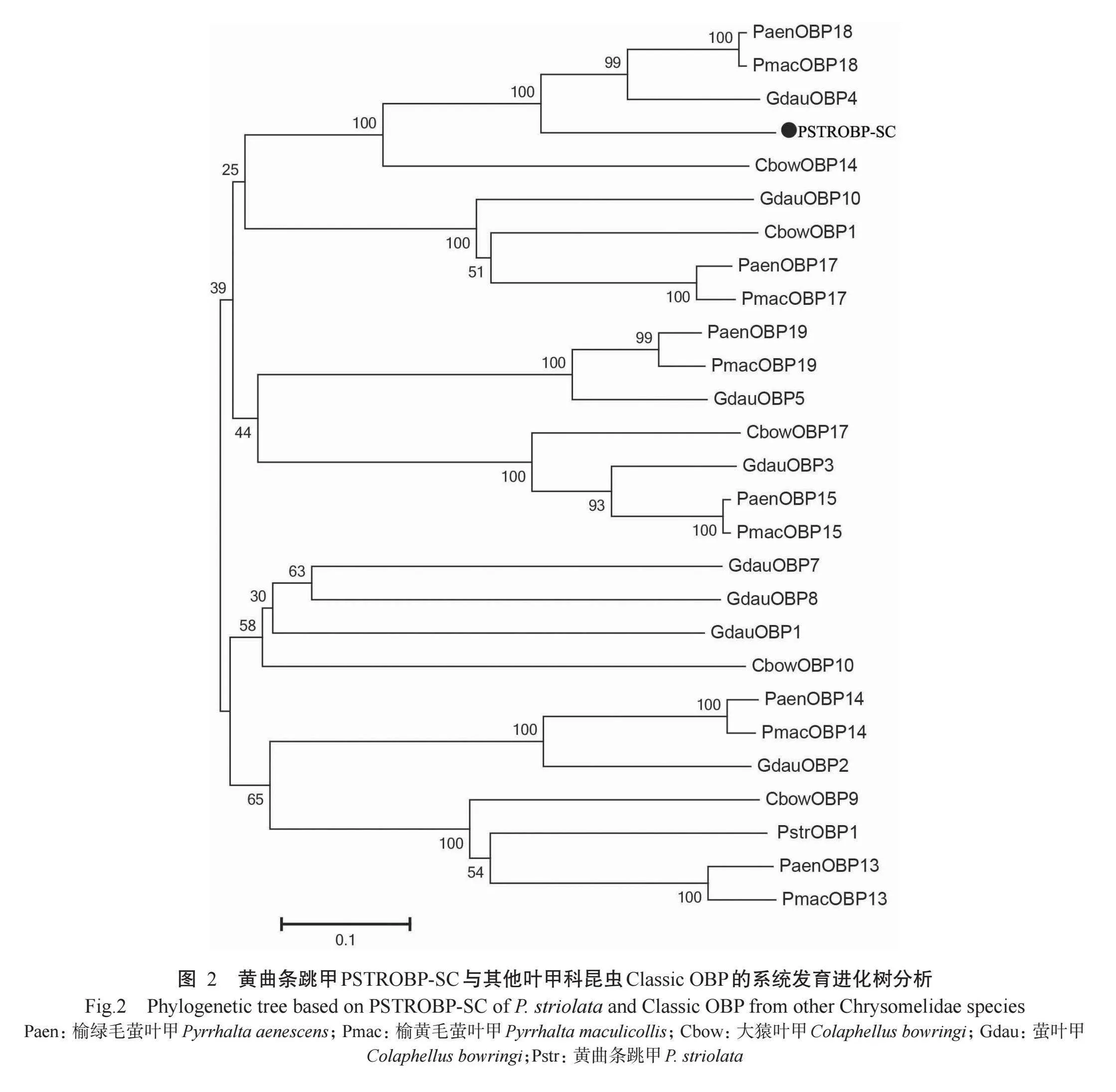
对27个叶甲科昆虫Classic OBP的系统发育分析显示(图2)[图中各OBP的氨基酸序列及登录号来自相关文献(Li etal.,2015;Zhang et al.,2016;Li et al.,2017;张国辉等,2023)],PSTROBP-SC与榆绿毛萤叶甲PaenOBP18、榆黄毛萤叶甲PmacOBP18、萤叶甲GdauOBP4和猿叶虫CbowOBP14聚在同一个分支上,表明其亲缘关系较近。进一步分析表明,与PSTROBP-SC聚在一起的4个OBP均是短链Classic OBP(97~100个氨基酸残基),且相似性很高,为76.6%(图3-A);对上述5个短链Classic OBP的二级结构预测结果(图3-B)显示,序列中只存在5个“-螺旋,且在靠近C端的第5个“-螺旋后仅存在2个氨基酸残基。本研究的5个短链Classic OBP特征与昆虫中长链Classic OBP存在明显差异。
2.3 PstrOBP-sc基因在成虫不同组织中的表达
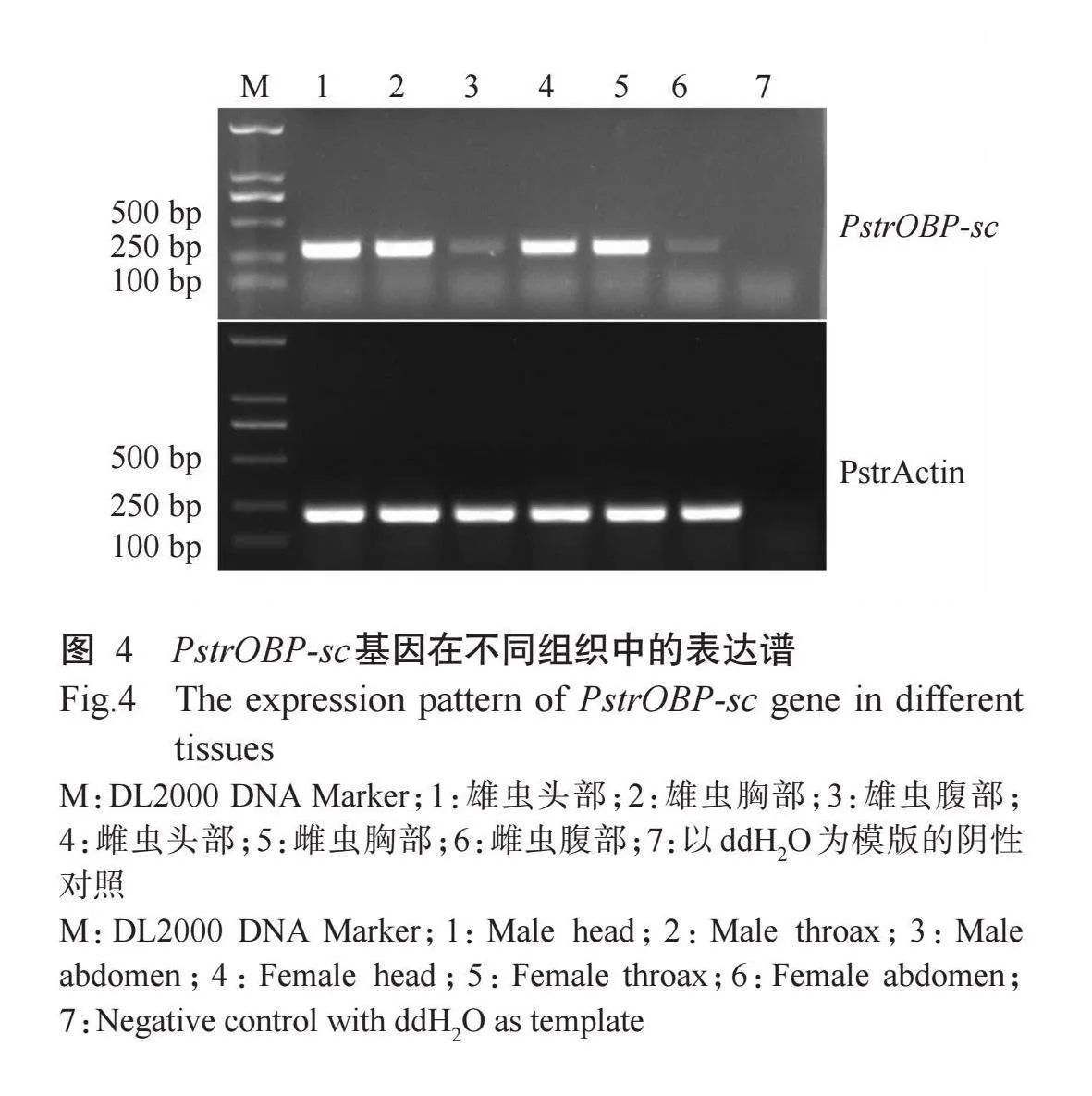
通过半定量PCR分析PstrOBP-sc基因在黄曲条跳甲雌、雄成虫不同组织中的表达情况,结果(图4)发现,该基因在供试所有组织中均有表达,并非特异性表达在含有嗅觉器官的头部组织。
2.4 PstrOBP-sc基因原核表达与纯化
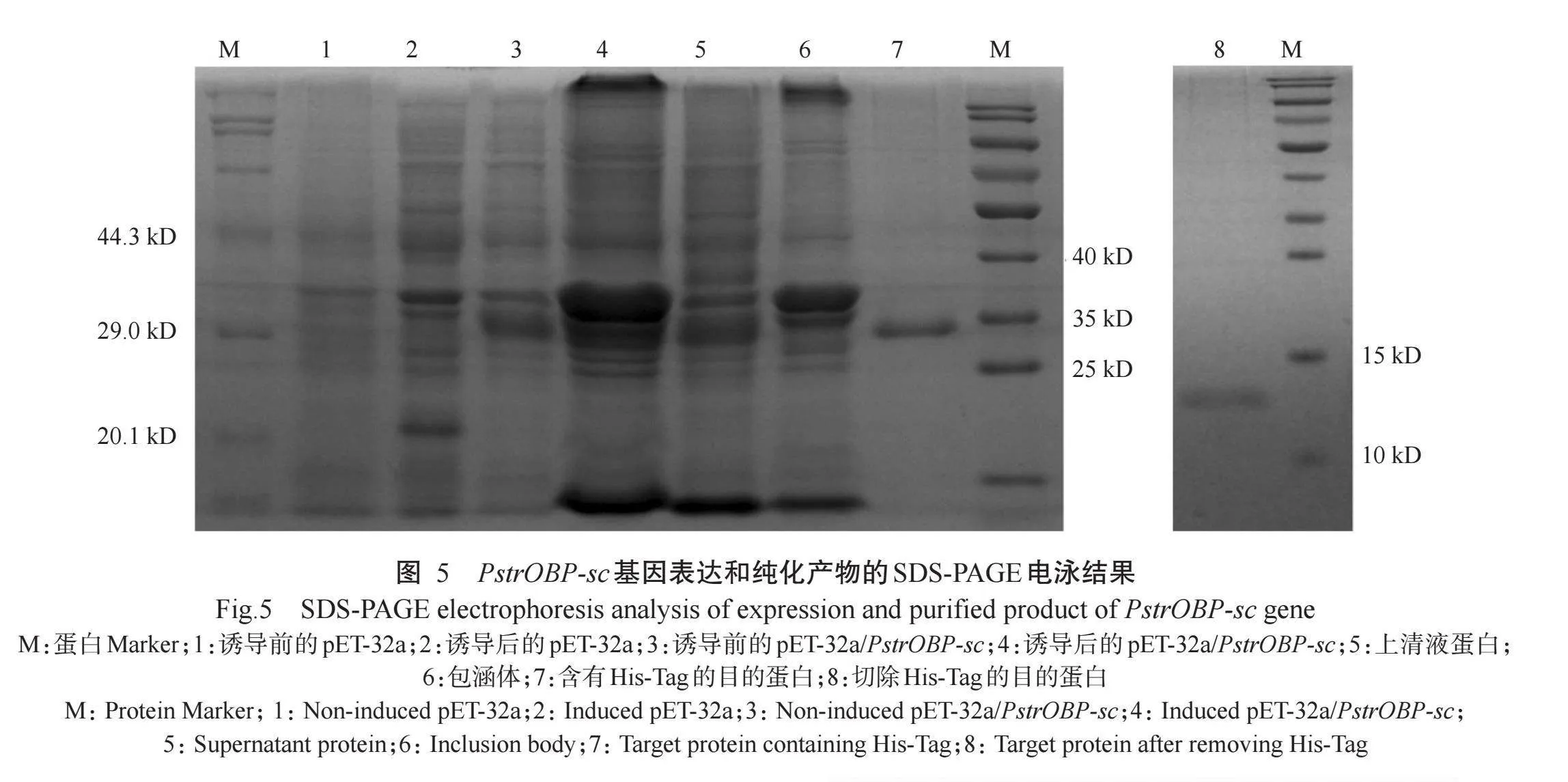
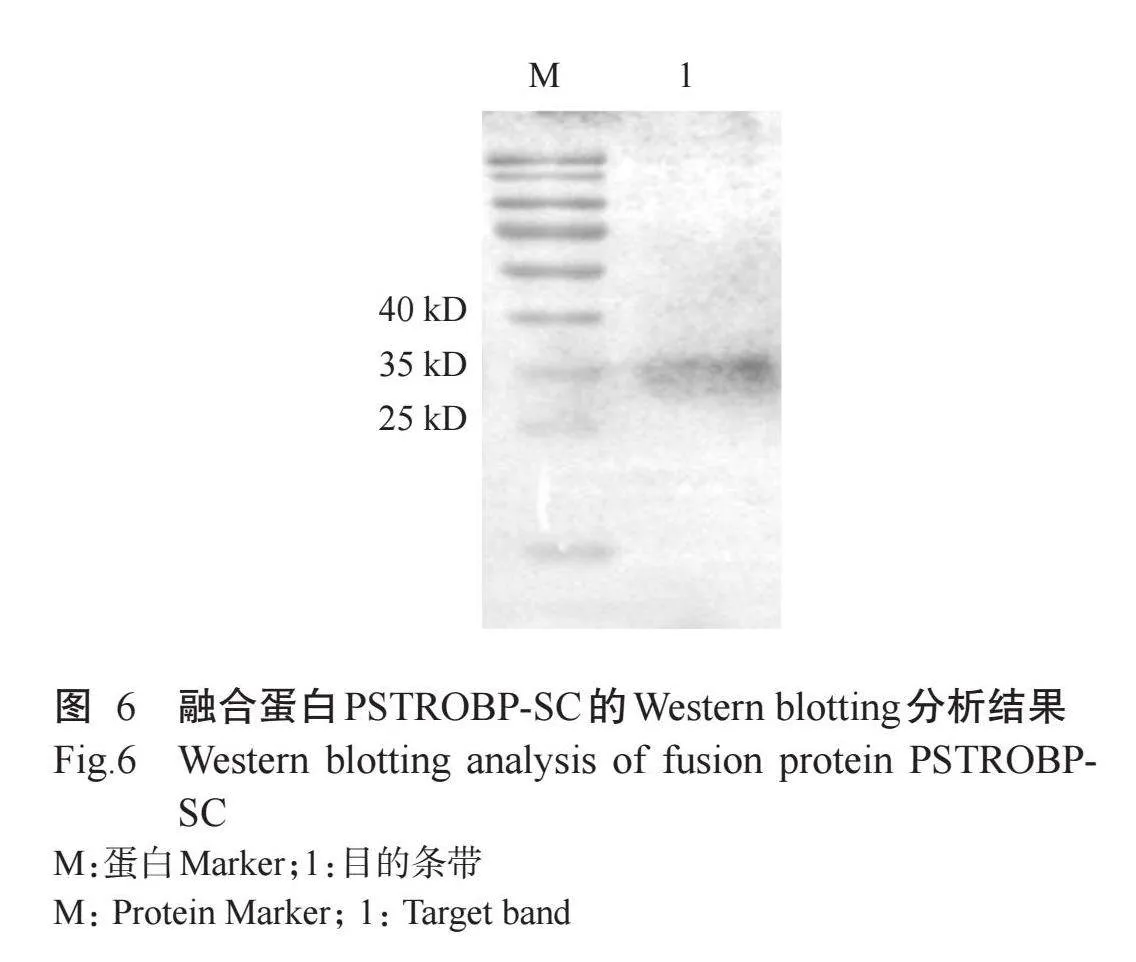
SDS-PAGE检测融合表达载体pET-32a/PstrOBP-sc在大肠杆菌中的表达,结果显示,经IPTG诱导的含有pET-32a/PstrOBP-sc的细菌细胞目的融合蛋白大量表达(图5,泳道4),而经IPTG诱导的含有空载体pET-32a的BL21菌株在相应位置未产生条带(图5,泳道2);为进一步检测表达产物是否为目的融合蛋白,对表达产物的Western blotting印迹分析显示,处于29 kD左右的蛋白条带与6×His抗体发生免疫反应(图6,泳道1),与预期大小一致,说明表达出目的融合蛋白。SDS-PAGE检测超声破损后的菌体显示,目的融合蛋白主要在上清液中表达,说明表达出的蛋白溶解性很好。利用Ni-NTA His·Bind Resin纯化树脂对上清液进行纯化,获得高纯度的目的融合蛋白(图5,泳道7);为了避免His标签对后续试验的影响,利用重组肠激酶切除目的融合蛋白的His标签,获得不含His-Tag的目的蛋白(图5,泳道8)。使用Bradford蛋白浓度测定试剂盒测得PSTROBP-SC蛋白溶液浓度为20.1μmol/L,可用于后续荧光竞争结合试验。
2.5 PSTROBP-SC的荧光结合试验结果
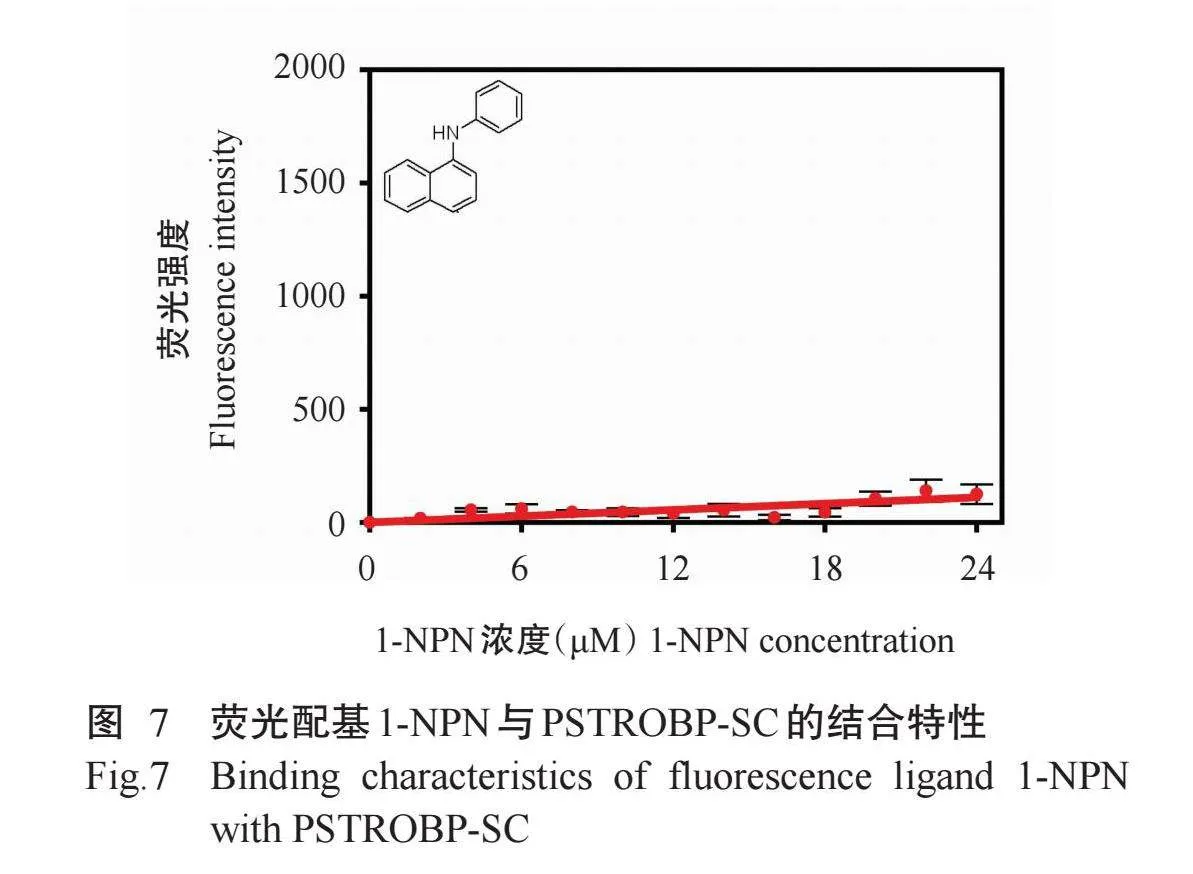
在PSTROBP-SC蛋白溶液中分别滴加终浓度为2、4、6、8、10、12、14、16、18、20、22和24μmol/L的1-NPN,发现其荧光强度均非常微弱(图7),表明PSTROBP-SC不能结合1-NPN于疏水结合腔内。
3讨论
本研究通过转录组数据筛选和逆转录聚合酶链反应克隆获得1条黄曲条跳甲短链OBP基因PstrOBP-sc。预测的成熟PSTROBP-SC蛋白仅由98个氨基酸残基组成,其中包含6个保守的半胱氨酸残基,属于典型的短链Clsssic OBP(Rihani et al.,2021)。当前对OBP的研究主要集中在Classic OBP家族中的中长链OBP,中长链OBP的一级结构长度通常在120~160个氨基酸残基(Rana et al.,2024)。其高级结构中通常存在6个“-螺旋,这些“-螺旋经过折叠在空间上形成1个疏水的气味配体结合腔(Sandler et al.,2000)。然而,PSTROBP-SC二级结构预测结果显示,PSTROBP-SC仅存在5个“-螺旋。这种二级结构元件组成上的差异势必会导致PSTROBP-SC与中长链OBP在三级结构上的差异,进而表现为功能上的差异。此外,本研究发现在PSTROBP-SC第5个“-螺旋的下游仅剩2个氨基酸残基组成C末端,与中长链OBP的C末端明显不同,中长链OBP通常具有较长的C末端(Lautenschlager et al.,2005;Wogulis et al.,2006;Leite et al.,2009;Mao et al.,2010)。OBP的C末端长度与OBP气味配体结合和释放过程密切相关。如家蚕BmorPBP1属于长链OBP,具有长C末端,pH为4.5时,C末端盘旋折叠成1个α-螺旋占据结合腔,挤出存在于结合腔中的配体;当pH为7.0时,C末端形成的α-螺旋解旋并离开结合腔,此时配体可进入(Lautenschlager et al.,2005)。对于中链OBP来说,其C末端虽然没有长链OBP长,但仍在OBP配体结合和释放过程中发挥重要作用。如属于中链OBP的冈比亚按蚊(Culex quinquefasciatus)AgamOBP1、埃及伊蚊(Aedes aegypti)AaegOBP1和致倦库蚊(Culex quinquefas-ciatus)CquiOBP1的C末端如同盖子,一旦配体结合于结合腔,C末端上的氨基酸残基通过pH敏感的分子内氢键将配体关在结合腔内。当环境变为酸性时,氢键断裂导致盖子开启配体被释放(Wogulisetal.,2006;Leite et al.,2009;Mao et al.,2010)。然而,黄曲条跳甲PSTROBP-SC二级结构预测结果显示,PSTROBP-SC在第5个α-螺旋后几乎没有C末端,仅有2个氨基酸残基组成1个非常短的C末端。由此推测,PSTROBP-SC配体结合与释放的机制与中长链OBP截然不同。由上述可知,短链PSTROBP-SC与中长链OBP的分子特征存在明显差异,这势必会导致二者间的功能差异。但这种短链OBP的具体结构信息仍有必要通过其晶体结构解析给出更具说服力的证据。
为分析PstrOBP-sc基因功能,本研究对PstrOBP-sc的组织表达模式进行了研究。组织表达模式显示PstrOBP-sc在黄曲条跳甲成虫的所有供试组织中均有表达,而非特异性表达于嗅觉器官集中的头部组织。那些在昆虫主要嗅觉器官触角中特异性或显著上调表达的OBP通常可结合、识别气味信息化合物调控昆虫嗅觉行为(Cheng et al.,2019;Lun et al.,2023;Yuan et al.,2024)。PstrOBP-sc在不同组织的广谱性表达暗示该基因在黄曲条跳甲体内很可能行使嗅觉以外的其他生理功能。为进一步验证PSTROBP-SC蛋白是否具备结合配体的能力,本研究利用荧光结合试验测试了细菌表达的PSTROBP-SC的配体结合能力,结果显示,PSTROBP-SC不能结合荧光探针1-NPN。1-NPN是目前研究OBP配体结合能力的通用探针,广泛应用于各种OBP功能研究(张玉等,2019)。本研究结果从侧面反映出短链PSTROBP-SC结合腔与当前广泛研究的中长链OBP的结合腔差异很大,进一步证明PSTROBP-SC很可能行使嗅觉以外的其他生理功能。事实上,随着研究的深入,研究者发现昆虫OBP并非总是结合配体化合物在昆虫化学感受过程中行使功能,而是还有很多其他生理功能。以目前研究比较深入的果蝇OBP为例,果蝇的OBP28a在造血和伤口愈合过程中发挥重要作用(Benoit et al.,2017);OBP59a在果蝇成虫湿度感受过程中扮演重要角色(Sun et al.,2018);OBP56g对于果蝇雄成虫交配突(Mating plug)的形成及其生育能力保持至关重要(Brown et al.,2023)。因此,PSTROBP-SC完全有可能在黄曲条跳甲体内行使嗅觉以外的其他生理功能,PSTROBP-SC的具体生理功能有待探究。
4结论
PstrOBP-sc基因的分子和功能特点不同于当前广泛研究的中长链OBP基因,其在生物体内很可能行使嗅觉以外的其他生理功能。
参考文献(References):
刘伟,王桂荣.2020.昆虫嗅觉中枢系统对外周信号的整合编码研究进展[J].昆虫学报,63(12):1536-1545.[Liu W,Wang G R.2020.Research progress of integrated coding of peripheral olfactory signals in the central nervous sys-tem of insects[J].Acta Entomologicab Sinica,63(12):1536-1545.]doi:10.16380/j.kcxb.2020.12.012.
杨成都,牛李阳,谷晓行,肖泽宇,李欣,尹新明,白素芬.2024.棉铃虫齿唇姬蜂的寄主识别与寄主选择[J/OL].河南农业大学学报.https://doi.org/10.16445/j.cnki.100-2340.20240305.002.[Yang C D,Niu LY,Gu X X,Xiao Z Y,Li X,Yin X M,Bai S F.2024.Host discrimination and host selection of Campoletischlorideae[J/OL].Journal of Henan Agricultural University.https://doi.org/10.16445/j.cnki.100-2340.20240305.002.]doi:10.16445/j.cnki.1000-2340.20240305.002
张国辉,杨岭,夏根,田晓丽,毛永娜.2023.黄曲条跳甲气味结合蛋白基因PstrOBP1克隆测序及其原核表达[J].南方农业学报,54(7):2015-2022.[Zhang G H,Yang L,Xia G,Tian X L,Mao YN.2023.Cloning,sequencing and pro-karyotic expression of an odorant-binding protein gene PstrOBP1 from Phyllotretastriolata(Fabricius)[J].Jour-nal of Southern Agriculture 54(7):2015-2022.]doi:10.3969/j.issn.2095-1191.2023.07.013.
张玉,杨斌,王桂荣.2019.昆虫嗅觉相关可溶性蛋白的研究进展[J].环境昆虫学报,41(2):229-240.[Zhang Y,Yang B,Wang G R.2019.Research progress of soluble proteins on chemosensationin insects[J].Journal of Environmental Entomology,41(2):229-240.]doi:10.3969/j.issn.1674-0858.2019.02.1.
Benoit J B,Vigneron A,Broderick N A,Wu Y,Sun J S,Carl-son J R,Aksoy S,Weiss B L.2017.Symbiont-induced odorant binding proteins mediate insect host hematopoiesis[J].eLife,6:e19535.doi:10.7554/eLife.19535.001.
Brown N C,Gordon B,McDonough-Goldstein C E,Misra S,Findlay G D,Clark A G,Wolfner M F.2023.The seminal odorant binding protein Obp56g is required for mating plug formation and male fertility in Drosophila melanogas-ter[J].eLife,12:e86409.doi:10.7554/eLife.86409.
Cheng W N,Zhang Y D,Liu W,Li G W,Zhu-Salzman K.2019.Molecular and functional characterization of three odorant-binding proteins from the wheat blossom midge,Sitodiplosismosellana[J].Insect Science,27(4):721-734.doi:10.1111/1744-7917.12677.
da Costa K S,Galúcio J M,da Costa C H S,Santana A R,dos Santos Carvalho V,do Nascimento L D,e Lima A H L,Cruz J N,Alves C N,Lameira J.2019.Exploring the potentiality of natural products from essential oils as inhibitors of odorant-binding proteins:A structure-and ligand-based virtual screening approach to find novel mos-quito repellents[J].ACS Omega,4(27):22475-22486.doi:10.1021/acsomega.9b03157.
Dippel S,Oberhofer G,Kahnt J,Gerischer L,Opitz L,Schachtner J,Stanke M,Schütz S,Wimmer EA,Angeli S.2014.Tissue-specific transcriptomics,chromosomal localization,and phylogeny of chemosensory and odorant binding proteinsfrom the red flour beetle Triboliumcastaneum reveal sub-group specificities for olfaction or more general functions[J].BMC Genomics,15:1141.doi:10.1186/1471-2164-15-1141.
Gong D P,Zhang H J,Zhao P,Xia Q Y,Zhong H X.2009.The odorant binding protein gene family from the genome of silkworm,Bombyx mori[J].BMC Genomics,10:332.doi:10.1186/1471-2164-10-332.
Hekmat-Scafe D S,Scafe C R,McKinney A J,Tanouye M A.2002.Genome-wide analysis of the odorant-binding pro-tein gene family in Drosophila melanogaster[J].Genome Research,12(9):1357-1369.doi:10.1101/gr.239402.
Hull J J,Perera O P,Snodgrass G L.2014.Cloning and expres-sion profiling of odorant-binding proteins in the tarnished plant bug,Lygus lineolaris[J].Insect Molecular Biology,23(1):78-97.doi:10.1111/imb.12064.
Kaissling K E.2009.Olfactory perireceptor and receptor events in moths:A kinetic model revised[J].Journal of Compara-tive Physiology A,195(10):895-922.doi:10.1007/s00359-009-0461-4.
Lautenschlager C,Leal W S,Clardy J.2005.Coil-to-helix tran-sition and ligand release of Bombyx mori pheromone-binding protein[J].Biochemical and Biophysical Research Communications,335(4):1044-1050.doi:10.1016/j.bbrc.2005.07.176.
Leal W S.2013.Odorant reception in insects:Roles of rece-ptors,binding proteins,and degrading enzyme[J].Annual Review of Entomology,58(1):373-391.doi:10.1146/annurev-ento-120811-153635.
Leite N R,Krogh R,Xu W,Ishida Y,Iulek J,Leal W S,Oliva G.2009.Structure of an odorant-binding protein from the mosquito Aedes aegypti suggests a binding pocket covered by a pH-sensitive“Lid”[J].PLoS One,4(11):e8006.doi:10.1371/journal.pone.0008006.
Li D X,Li C B,Liu D G.2021.Analyses of structural dyna-mics revealed flexible binding mechanism for the Agrilus mali odorant binding protein 8 towards plant volatiles[J].Pest Management Science,77(4):1642-1653.doi:10.1002/ps.6184.
Li L,Zhou Y T,Tan Y,Zhou X R,Pang B P.2017.Identifica-tion of odorant-binding protein genes in Galerucadaurica(Coleoptera:Chrysomelidae)and analysis of their expres-sion profiles[J].Bulletin of Entomological Research,107(4):550-561.doi:10.1017/S0007485317000402.
Li X M,Zhu X Y,Wang Z Q,Wang Y,He P,Chen G,Sun L,Deng D G,Zhang Y N.2015.Candidate chemosensory genes identified in Colaphellusbowringi by antennal tran-scriptome analysis[J].BMC Genomics,16:1028.doi:10.1186/s 12864-015-2236-3.
Lun X Y,Xu X X,Zhang Y,Zhang R R,Cao Y,Zhang X Z,Jin M N,Zhang Z Q,Zhao Y H.2023.An antennae-enriched odorant-binding protein EonuOBP43 mediate the behavioral response of the tea green leafhopper,Empoascaonukiimatsuda to the host and nonhost volatiles[J].Jour-nal of Agricultural and Food Chemistry,71(50):20000-20010.doi:10.1021/acs.jafc.3c07144.
Mao Y,Xu X Z,Xu W,Ishida Y,Leal W S,Ames J B,Clardy J.2010.Crystal and solution structures of an odorant-binding protein from the southern house mosquito com-plexed with an oviposition pheromone[J].Proceedings of the National Academy of Sciences of the United States of America,107(44):19102-19107.doi:10.1073/pnas.10122 74107.
Rana A,Sharma D,Choudhary K,Kumari P,Ruchika K,Yangchan J,Kumar S.2024.Insight into insect odorantbinding proteins:An alternative approach for pest manage-ment[J].Journal of Natural Pesticide Research,8:100069.doi:10.1016/j.napere.2024.100069.
Rihani K,Ferveur J F,Briand L.2021.The 40-year mystery of insect odorant-binding proteins[J].Biomolecules,11(4):509.doi:10.3390/biom 11040509.
Sandler B H,Nikonova L,Leal W S,Clardy J.2000.Sexual attraction in the silkworm moth:Structure of the pheromone-binding-protein-bombykol complex[J].Chemi-stry&Biology,7(2):143-151.doi:10.1016/s 1074-5521(00)00078-8.
Sun J S,Larter N K,Chahda J S,Rioux D,GumasteA,Carlson J R.2018.Humidity response depends on the small soluble protein Obp59a in Drosophila[J].eLife,7:e39249.doi:10.7554/eLife.39249.
Tian Z,Liu J Y,Zhang Y L.2016.Structural insights into Cydia pomonella pheromone binding protein 2 mediated prediction of potentially active semiochemicals[J].Scien-tific Reports,6:22336.doi:10.1038/srep22336.
Wogulis M,Morgan T,Ishida Y,Leal W S,Wilson D K.2006.The crystal structure of an odorant binding protein from Anopheles gambiae:Evidence for a common ligand release mechanism[J].Biochemical and Biophysical Research Communications,339(1):157-164.doi:10.1016/j.bbrc.2005.10.191.
Vogt R G,Riddiford L M.1981.Pheromone binding and inacti-vation by moth antennae[J].Nature,293:161-163.doi:10.1038/293161a0.
Yuan W Q,Rao X J,Zhong B Z,Chen M R,Ali H,LüC J.2024.Exploring the functional profiles of odorant binding proteins crucial for sensing key odorants in the new leaves of coconut palms in Rhynchophorus ferrugineus[J].Inter-national Journal of Biological Macromolecules,261(P2):129852.doi:10.1016/j.ijbiomac.2024.129852.
Zhang B,Zhang W,Nie R E,Li W Z,Segraves K A,Yang X K,Xue H J.2016.Comparative transcriptome analysis of chemosensory genes in two sister leaf beetles provides insights into chemosensory speciation[J].Insect Bioche-mistry and Molecular Biology,79:108-118.doi:10.1016/j.ibmb.2016.11.001.
Zhao X C,Berg G B,Wang G R.2023.Editorial:Recent advances in insect olfaction:Characterization of neural cir-cuits from sensory input to motor output[J].Frontiers in Cellular Neuroscience,17:1282499.doi:10.3389/fncel.2023.1282499.
Zhou J J,Huang W S,Zhang GA,Pickett J A,Field L M.2004.‘Plus-C’odorant-binding protein genes in two Drosophila species and the malaria mosquito Anopheles gambiae[J].Gene,327(1):117-129.doi:10.1016/j.gene.2003.11.007.
(责任编辑麻小燕)

