2种食蚊鱼属入侵鱼类线粒体基因组密码子偏好性分析







摘要:【目的】分析西部食蚊鱼(Gambusia affinis)和东部食蚊鱼(G.holbrooki)线粒体基因组组成和密码子使用偏好性,为食蚊鱼属入侵鱼类的早期预警和防控提供理论依据。【方法】基于线粒体基因组序列数据,通过生物信息学方法分析西部食蚊鱼和东部食蚊鱼线粒体基因组组成特征和密码子使用模式。【结果】西部食蚊鱼和东部食蚊鱼线粒体基因组大小分别为16614和16610 bp,碱基组成均呈现明显的AT偏好,蛋白编码基因密码子的适应指数(CAI)范围分别为0.14~0.20和0.13~0.20,偏好性指数(CBI)分别为-0.09~0.09和-0.09~0.07,有效密码子数(ENC)分别为35.19~50.50和39.03~50.06。中性绘图分析结果表明,2种食蚊鱼线粒体编码基因的第1、2位和第3位碱基GC含量相关性不明显;ENC-plot分析发现2种食蚊鱼线粒体基因ENC均位于标准曲线下方;PR2-plot分析发现2种食蚊鱼线粒体基因密码子第3位的4种碱基使用频率不相等,A和C的使用频率高于T和G。在西部食蚊鱼和东部食蚊鱼线粒体基因组中分别确定了16和18个最优密码子。【结论】2种食蚊鱼线粒体基因组均偏好使用以C或A结尾的密码子,无以G结尾的密码子。西部食蚊鱼和东部食蚊鱼分别筛选出16和18个最优密码子,主要以C或A结尾。2种食蚊鱼线粒体基因组密码子均为弱偏好性密码子,自然选择和突变压力均是影响2种食蚊鱼线粒体密码子偏好性形成的因素,其中自然选择起主导作用。
关键词:食蚊鱼;线粒体基因组;密码子偏好性;自然选择
中图分类号:S917.4文献标志码:A文章编号:2095-1191(2024)08-2514-09
Codon bias in the mitochondrial genomes of two invasive Gambusia species
XIE Jia-yan CHEN Yue SUN He-ying2*
(1School of Life Science and Technology,Wuhan Polytechnic University,Wuhan,Hubei 430023,China;2ChangjiangBasin Ecology and Environment Monitoring and Scientific Research Center,Changjiang Basin Ecology andEnvironment Administration,Ministry of Ecology and Environment,Wuhan,Hubei 430019,China)
Abstract:【Obj8a3935be9374272aaffddf2a5ee580b95770ef08d46eb4aa28c09ad1e015e485ective】The mitochondrial genome composition and codon usage bias of Gambusia affinis and G.hol-brooki were studied in order to provide theoretical basis for early warning,prevention and control of invasive Gambusia species.【Method】Based on the mitochondrial genome sequence data,the mitochondrial genome composition characteris-tics and codon usage patterns of G.affinis and G.holbrooki were analyzed by bioinformatics methods.【Result】The mito-chondrial genome sizes of G.affinis and G.holbrooki were 16614 and 16610 bp with obvious AT bias in their base compo-sition.The codon adaptation index(CAI)ranged for protein-coding genes were 0.14 to 0.20 for G.affinis and 0.13 to 0.20 for G.holbrooki.The values of codon bias index(CBI)ranged from-0.09 to 0.09 and-0.09 to 0.07,respectively.Theef-fective number of codon(ENC)ranged from 35.19 to 50.5 and 39.03 to 50.06,respectively.There was no obvious correla-tion between GC contents of the 1st,2nd,and 3rd bases of mitochondrial coding genes in both species based on neutralityplotanalysis.ENC-plot revealed that ENC values of mitochondrial genes in two species were both located below the stan-dard curve.PR2-plot analysis found that the usage frequency of the four bases at the 3rd codon position in mitochondrial genes was not equal,the usage frequency of A and C was higher than T and G.Sixteen and eighteen optimal codons in the mitochondrial genome of G.affinis and G.holbrooki were identified,respectively.【Conclusion】The mitochondrial ge-nomes of the two Gambusia species prefer to use codons ending with C or A,and without G endings.Sixteen and eigh-teen optimal codons in the mitochondrial genome of G.affinis and G.holbrooki are identified,mainly ending with C or A.The results suggest that codons in the mitochondrial genome of G.affinis and G.holbrookishow weak bias.Naturalselec-tion and mutation pressure play an important role in the forming codon bias in the mitochondrial genome for the two Gam-busia species,with natural selection being the more influencing factor.
Key words:Gambusia;mitochondrial genome;codon bias;natural selection
Foundation items:National Natural Science Foundation of China(32101410);Collaborative Education Project of Industry University Cooperation of the Ministry of Education(202102102111);Scientific Research Plan Funding Project of Hubei Education Department(B2017076)
0引言
【研究意义】随着经济全球化,生物入侵已成为全球性的生态环境问题(Bernery et al.,2022),对我国淡水生物多样性和生态系统稳定性也存在严重威胁(Liu et al.,2017)。食蚊鱼属鱼类因捕食蚊类幼虫可防止蚊媒疾病传播,东部食蚊鱼(Gambusia hol-brooki)和西部食蚊鱼(Gambusia affinis)在全世界范围内被广泛引种,已入侵60多个国家和地区(Pyke,2005)。东部食蚊鱼分布于美国东海岸,经西班牙传入意大利、欧洲及澳大利亚(Pyke,2008;Vidalet al.,2010);西部食蚊鱼分布于美国中西部和墨西哥东北部,经夏威夷引入其他太平洋地区(Pyke,2008)。通过资源竞争和捕食,东部食蚊鱼入侵可对西班牙鮰(Valencia hispanica)的存活(Caiola and de Sostoa,2005)及欧洲两栖类物种幼体丰度产生影响(Van-niniet al.,2018)。西部食蚊鱼已在我国长江流域迅速扩张,并进一步向高原水域扩散(陈银瑞等,1989;Gao et al.,2017),导致我国濒危鱼类唐鱼(Tanich-thysalbonubes)(陈国柱,2010)和中华青鳉(Oryzias sinensis)的种群数量急速下降,甚至促使云南地区中华青鳉局部灭绝(陈银瑞等,1989)。因此,开展入侵食蚊鱼遗传组成和特性分析,揭示其生物入侵的内在遗传机制,以期为食蚊鱼属入侵鱼类的早期预警和防控提供理论依据。【前人研究进展】食蚊鱼为杂食性鱼类,除捕食蚊虫幼虫外,还以多种小型水生生物为食,通过捕食和栖息环境竞争而对引入地土著种的生存产生威胁(Pyke,2008;Lau etal.,2019),于21世纪初被世界自然保护联盟(World Conserva-tion Union)列为世界百种恶性入侵物种(Lowe et al.,2000)。已有研究发现,西部食蚊鱼种群在入侵地仍具有较高的遗传多样性水平(Sanz ete25deb5ae6a09d3fd3f82b33fe6446acbc5e94e8e6ce95443ee3eaeadf301e09al.,2013),短移殖期内可形成快速进化(Stockwell and Weeks,1999)。这些种群遗传结构特征对食蚊鱼在入侵早期建群起重要作用,通过快速进化,食蚊鱼能迅速适应入侵地的异质环境,以确保入侵种群的成功定殖(Mack et al.,2000)。线粒体是细胞进行氧化磷酸化及合成腺嘌呤核苷三磷酸(ATP)的场所,其DNA具有进化速率快等遗传特性(陈星等,2012)。密码子是生命遗传信息传递过程中的基本单位,在翻译过程中会出现高频使用某些同义密码子进行编码的偏好,而自然选择和突变压力是导致同义密码子使用偏好的主要进化因素,因此这种偏好反映了自然选择和突变压力间的平衡及遗传漂变等因素对遗传信息的影响(Bulmer,1991)。孟乾等(2020)通过对斑石鲷(Oplegnathuspunctatus)和条石鲷(O.fasciatus)线粒体基因编码序列密码子的使用模式和特性分析发现两者亲缘关系较近;Chamani Mohasses等(2020)发现干旱胁迫下水稻(Oryza sativa)中高表达的基因GC含量较高,有效密码子数较低,部分氨基酸的偏好密码子发生变化;任童童等(2022)研究发现法螺(Charoniatritonis)线粒体基因组密码子偏好性形成过程中自然选择起主导作用。可见,研究密码子使用模式及其影响因素,可进一步理解基因组进化和生物体对不同生境的适应性。【本研究切入点】目前,对入侵食蚊鱼生物学、种群遗传结构及其对本土物种和生态系统的影响等方面已有相关研究(Pyke,2008;Gao et al.,2017),但对有关线粒体基因组的研究报道较少。【拟解决的关键问题】通过比较分析西部食蚊鱼和东部食蚊鱼线粒体基因组组成、密码子使用偏好性及其影响因素,深入理解入侵鱼类对生境快速适应的线粒体基因组特征,为入侵物种的早期预警提供理论支持。
1材料与方法
1.1线粒体基因组序列获取与筛选
在NCBI数据库中检索下载西部食蚊鱼和东部食蚊鱼的线粒体基因组序列(AP004422和OP882646),为降低误差选取长度大于300bp的蛋白编码序列用于密码子偏好性分析。
1.2密码子碱基组成参数计算
使用CodonW 1.4.2分析食蚊鱼线粒体基因组的碱基组成、密码子第1~3位的GC含量(GC1、GC2和GC3)、同义密码子第3位的GC含量(GC3s)、密码子适应指数(Codon adaptation index,CAI)、密码子偏好性指数(Codon bias index,CBI)、最优密码子使用频率(Frequency of optimal codons,FOP)、有效密码子数(Effective number of codon,ENC)及同义密码子相对使用度(Relative synonymous codon usage,RSCU)。采用SPSS 20.0进行各参数间的统计分析。
1.3中性绘图分析
以GC3为横坐标、GC12(GC1和GC2的平均值)为纵坐标作图进行中性绘图分析,以分析密码子偏好性受选择压力影响的情况(Sueoka,1988)。
1.4 PR2-plot分析
PR2-plot分析密码子第3位碱基组成是否对密码子偏好性产生影响,平面图中心点表示A=T及G=C,此时密码子偏好性完全受突变的影响,由中心点至图中各点发出的矢量分别表示各基因的偏好性方向及程度(Sueoka,1999)。
1.5 ENC-plot分析
以每个基因的GC3s为横坐标、ENC为纵坐标,构建散点图进行ENC-plot分析。ENC的标准曲线方程为ENC=2+GC3+29/[GC3s2+(1-GC3s)2](Wright,1990)。若密码子偏好性受突变作用的影响,ENC将分布于标准曲线附近;当密码子偏好性受较强选择作用影响时,ENC将分布在离曲线下方较远的位置(Wright,1990)。同时,计算ENC比值(ENC ratio)进一步评估影响密码子偏好性的因素,公式为ENC ratio=(ENC期望−ENC实际)/ENC期望(Kawabe and Miyashita,2003)。
1.6最优密码子分析
以ΔRSCU法确定最优密码子,根据ENC对基因进行排序,两端各选出30%的基因分别构建高、低偏好性库,计算两库的RSCU差值(ΔRSCU),选择ΔRSCU>0.08且在高表达基因库中RSCU>9f12e8a4dde3fc6469d1cba33bb5822fe47421d416e52620dbd983cc66a54ae41.00的密码子为最优密码子(Liu et al.,2004a,2004b)。
2结果与分析
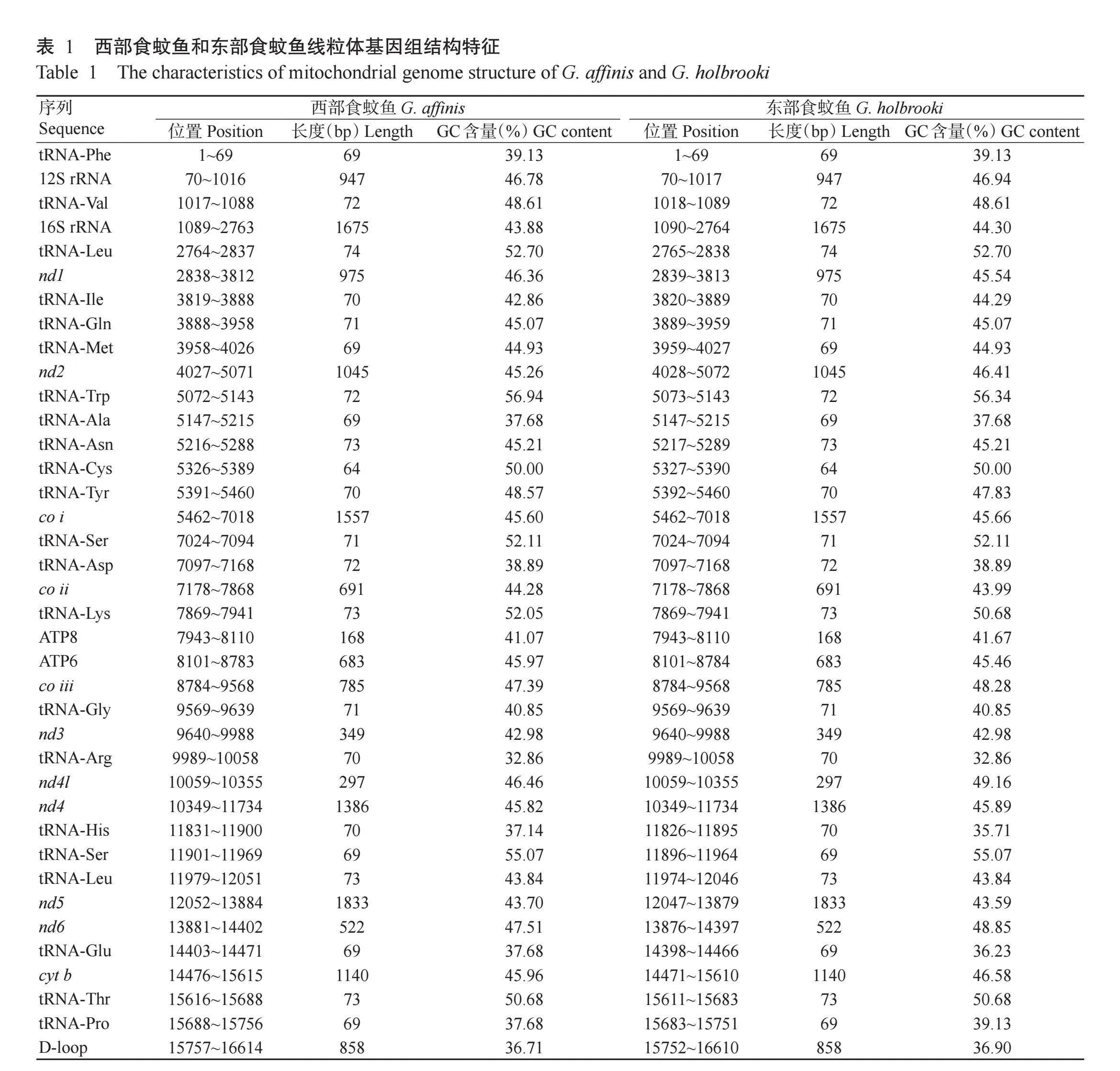
2.1 2种食蚊鱼线粒体基因组结构特征
结果如表1所示,西部食蚊鱼和东部食蚊鱼线粒体均为双链闭合环状DNA,基因组大小分别为16614和16610 bp,均包含13个蛋白、22个tRNA及2个rRNA。西部食蚊鱼整个基因组碱基A、T、G和C的含量分别为28.00%、27.10%、15.67%和29.23%,东部食蚊鱼的分别为27.90%、27.00%、15.68%和29.42%,均呈现明显的AT偏好和反G偏倚。西部食蚊鱼和东部食蚊鱼的线粒体不同结构组分GC含量范围分别为32.86%~56.94%和32.86%~56.34%。
2.2 2种食蚊鱼线粒体基因组核苷酸组成分析结果
对2种食蚊鱼线粒体基因组核苷酸组成进行分析,结果如表2所示。西部食蚊鱼和东部食蚊鱼蛋白编码区中碱基A、T、G、C的平均含量分别为25.89%、28.65%、14.47%、30.99%和25.82%、28.54%、14.47%、31.17%,总GC含量范围分别为42.98%~47.51%和42.98%~48.85%,与整个基因组类似,呈现明显的AT偏好和反G偏倚。西部食蚊鱼和东部食蚊鱼的CAI分别为0.14~0.20和0.13~0.20,不同密码子的CBI分别为-0.09~0.09和-0.09~0.07,FOP分别为0.33~0.45和0.31~0.46,ENC分别为35.19~50.50和39.03~50.06。
2.3 2种食蚊鱼线粒体基因组RSCU分析结果
对2种食蚊鱼线粒体基因组RSCU进行分析,结果如图1所示。在64个密码子中,2种食蚊鱼均有31个密码子的RSCU大于1.00,其中西部食蚊鱼和东部食蚊鱼以A、U、G、C结尾的密码子分别有12、5、0、14个和12、4、0、15个,表明2种食蚊鱼线粒体基因组均偏好使用以C或A结尾的密码子,无以G结尾的密码子。
2.4 2种食蚊鱼线粒体基因组中性绘图分析结果
对2种食蚊鱼线粒体基因组基因进行中性绘图分析,结果如图2所示。西部食蚊鱼和东部食蚊鱼密码子3个位置上的GC含量类似,其含量排序依次为GC1>GC3>GC2。西部食蚊鱼和东部食蚊鱼的GC12范围分别为0.43~0.49和0.44~0.49,GC3范围分别为0.38~0.53和0.38~0.56,基因主要分布于对角线两侧。其中,西部食蚊鱼基因nd5与cyt b分布在对角线附近,nd6等基因距对角线较远;东部食蚊鱼基因nd5与nd2分布在对角线附近,nd6等基因距对角线较远。2种食蚊鱼的相关系数分别为0.155和0.037,相关性均不明显,表明突变压力对2种食蚊鱼线粒体基因组碱基组成的影响较弱。
2.5 2种食蚊鱼线粒体基因PR2-plot分析结果
对2种食蚊鱼线粒体基因进行PR2-plot分析,结果(图3)显示,2种食蚊鱼的PR2-plot分布较类似,线粒体基因未均匀地分布于平面图的4个区域,仅分布于中线左上方。密码子第3位的4种碱基使用频率不相等,A和C的使用频率高于T和G。自然选择明显驱动了线粒体基因密码子的使用偏倚性。
2.6 2种食蚊鱼线粒体基因ENC-plot分析结果
对2种食蚊鱼线粒体基因进行ENC-plot分析,结果如图4所示。西部食蚊鱼和东部食蚊鱼线粒体基因均分布于标准曲线下方,距标准曲线最远的基因分别为nd6和nd2。对线粒体密码子的ENC比值进行分析,结果(图5)显示,2种食蚊鱼线粒体基因的ENC比值均呈单峰分布,西部食蚊鱼和东部食蚊鱼的ENC比值范围分别为0.14~0.41和0.15~0.35,50%基因的ENC比值频数分布于0.15~0.25,说明ENC的实际观察值与期望值频数差异较大,即实际值低于期望值。
2.7 2种食蚊鱼线粒体最优密码子分析结果
采用ΔRSCU法识别2种食蚊鱼线粒体的最优密码子,结果(表3)表明,2种食蚊鱼线粒体基因组最优密码子组成不同,西部食蚊鱼和东部食蚊鱼线粒体基因组中ΔRSCU>0.08的高表达密码子分别为17和18个。以RSCU>1.00确定最优密码子,西部食蚊鱼共确定获得16个最优密码子,以A、U、G、C结尾的密码子分别有4、3、0和9个,以C和A结尾的密码子占其总数的81.25%;东部食蚊鱼共确定获得18个最优密码子,以A、U、G、C结尾的密码子分别有8、2、1和7个,以C和A结尾的密码子占其总数的83.33%。
3讨论
生物入侵已成为全球性重大生态环境问题,入侵物种的治理极其困难,多个国家和地区均已加大对生物入侵早期预警的防控管理(Liu et al.,2017;Bernery et al.,2022)。入侵食蚊鱼属鱼类早期主要通过人工大量引入当地水体进行生物防控(Pyke,2005)。东部食蚊鱼的引入导致引入地生物群落中无脊椎动物、鱼类和两栖动物的丰度明显下降(Pyke,2005;Vanniniet al.,2018);而具高度入侵风险的西部食蚊鱼也已扩散至我国长江以南流域(Gao et al.,2017),对当地土著种的生存及生物多样性水平产生了明显影响(陈银瑞等,1989;陈国柱,2010)。因此,亟待解析入侵物种的遗传组成和特性,进而揭示生物入侵的内在遗传机制(Sanz etal.,2013)。
GC碱基含量是基因组结构形成与进化过程中DNA序列稳定的重要指标(Bellgardetal.,2001;韩春丽等,2023)。脊椎动物线粒体基因组中富含AT碱基,因此基因间进化速率高于核基因(陈星等,2012)。在鱼类中,青海湖裸鲤(Gymnocyprisprzew-alskiia42yY9qbtQRoODUh80QhnDmbg6HJn0vD5lCeqQKXs4k=)线粒体基因组的GC含量为44.01%(保长虹等,2023),银色裂腹鱼(Schizothorax argentatus)为44.48%(刘玉萍等,2023)。本研究中2种食蚊鱼线粒体基因组的总GC含量均小于46.00%,呈现明显的AT偏好性,与其他硬骨鱼类类似。大部分生物线粒体基因组使用密码子的偏好性较类似,但密码子在选择使用过程中,仍存在一定的物种保守性,致使相近物种的密码子使用偏好性较相似(宋乔乔和钟金城,2013)。本研究中,西部食蚊鱼和东部食蚊鱼线粒体基因组编码蛋白偏好以碱基C或A结尾,避免使用G结尾,与孟乾等(2020)的研究结果类似。RSCU分析结果表明,西部食蚊鱼和东部食蚊鱼线粒体中以碱基C和A结尾的最优密码子占其总数的80.00%以上,进一步说明2种食蚊鱼线粒体密码子偏好以碱基C或A结尾。密码子偏好性作为基因和基因组的一种静态特征,是生物在长期演化过程中适应和选择的结果(任桂萍等,2019;张扬等,2022)。密码子偏好性参与转录、翻译、蛋白折叠等过程的调控,还与mRNA稳定、细胞减数分裂中同源重组等过程存在协同进化(Pouyet et al.,2017)。因此,密码子偏好性是决定基因转录表达的关键因子,既影响蛋白表达和基因功能,也可显示物种的进化和突变。
碱基突变和自然选择是影响密码子偏好性的主要因素(Sharp etal.,2010)。ENC可衡量群体遗传过程中某一基因的密码子偏好性程度。本研究结果表明,西部食蚊鱼和东部食蚊鱼线粒体基因组编码蛋白基因的ENC均大于35.00,而CAI较低,说明2种食蚊鱼线粒体基因组密ejHv6CdbPMmJPHfqZFPutAkYcam46GH2v1t3yS9bAlo=码子为弱偏好性密码子。中性绘图分析表明,线粒体基因组密码子用法不能明显反映突变偏好,密码子的使用更多是受翻译选择压力的限制,表明自然选择可能是影响2种食蚊鱼线粒体基因组密码子偏好性形成的主要因素(Sueoka,1988)。ENC-plot分析同样表明,除中性突变造成的序列背景差异外,与弱选择效应相关联的基因表达水平可能同时影响着2种食蚊鱼线粒体中密码子用法的偏好(Wright,1990)。PR2-plot分析也证实突变并非是2种食蚊鱼线粒体基因组密码子偏好性形成的唯一驱动力,自然选择也进一步促进其偏好性的形成。自然选择和突变压力均是影响2种食蚊鱼线粒体基因组密码子偏好性的重要因素,而自然选择是其中最关键的影响因素。这与斑石鲷和条石鲷线粒体基因组密码子偏好性所受影响(孟乾等,2020)一致。除鱼类外,其他动物物种线粒体基因组密码子偏好性也多受选择压力的影响(Uddin et al.,2017),表明生物类群中线粒体密码子偏好性的驱动力类似。此外,不同物种密码子偏好性的差异还与物种所处环境和所受压力相关,可能是突变和选择在物种间存在差异的主要原因(Seward and Kelly,2016)。
4结论
2种食蚊鱼线粒体基因组均偏好使用以C或A结尾的密码子,无以G结尾的密码子。西部食蚊鱼和东部食蚊鱼分别筛选出16和18个最优密码子,主要以C或A结尾。2种食蚊鱼线粒体基因组密码子均为弱偏好性密码子,自然选择和突变压力均是影响2种食蚊鱼线粒体密码子偏好性形成的因素,其中自然选择起主导作用。
参考文献(References):
保长虹,李昭楠,关却多杰,李长忠,尹格玛,贺彩霞,金文杰,周叶吉,陈艳霞.2023.青海湖裸鲤和花斑裸鲤线粒体基因组比较及其系统进化分析[J].华北农学报,38(3):213-226.[Bao C H,Li Z N,Guanque D J,Li C Z,Yin G M,He C X,Jin W J,Zhou Y J,Chen Y X.2023.Compari‐son and phylogenetic analysis of mitochondrial genomes between Gymnocyprisprzewalskii and Gymnocypriseck-loni[J].Acta AgriculturaeBoreali-Sinica,38(3):213-226.]doi:10.7668/hbnxb.20193633.
陈国柱.2010.入侵种食蚊鱼与土著濒危物种唐鱼的种间关系研究[D].广州:暨南大学.[Chen G Z.2010.Interspe‐cific relationship between the invasive species Gambusia affinis and the native endangered species Tanichthysalbo-mubes[D].Guangzhou:Jinan University.]
陈星,沈永义,张亚平.2012.线粒体DNA在分子进化研究中的应用[J].动物学研究,33(6):566-573.[Chen X,Shen Y Y,Zhang Y P.2012.Review of mtDNA in molecular evolution studies[J].Zoological Research,33(6):566-573.]doi:10.3724/SP.J.1141.2012.06566.
陈银瑞,宇和纮,褚新洛.1989.云南青鳉鱼类的分类和分布(鳉形目:青鳉科)[J].动物分类学报,14(2):239-246.[Chen Y R,Yu H H,Chu X L.1989.Taxonomy and distri‐bution of the genus Oryzias in Yunnan,China(Cyprino-dontiformes:Oryzhdae)[J].Zoological Systematics,14(2):239-246.]
韩春丽,杨果豪,李天香,王健宇,熊忠萍,许尤厚,朱鹏,杨家林,王鹏良.2023.方格星虫线粒体全基因组密码子偏好性分析[J].南方农业学报,54(9):2604-2613.[Han C L,Yang G H,Li T X,Wang J Y,Xiong Z P,Xu Y H,Zhu P,Yang J L,Wang P L.2023.Codon bias analysis of mito‐chondrial genome in Sipunculusnudus[J].Journal of Sou-thern Agriculture,54(9):2604-2613.]doi:10.3969/j.issn.2095-1191.2023.09.011.
刘玉萍,胡建勇,宁子君,肖佩怡,杨天燕.2023.银色裂腹鱼线粒体基因组序列特征与系统进化分析[J].生物工程学报,39(7):2965-2985.[Liu Y P,Hu J Y,Ning Z J,Xiao PY,Yang TY.2023.Mitochondrial genome sequencecharacteristics and phylogebetic analysis of Schizothorax argentatus[J].Chinese Journal of Biotechnology,39(7):2965-2985.]doi:10.13345/j.cjb.220780.
孟乾,张志勇,张志伟,张曹进,陈淑吟,祝斐,贾超峰,曹广勇.2020.斑石鲷和条石鲷线粒体基因组密码子使用分析[J].水产科学,39(5):702-709.[MengQ,Zhang Z Y,Zhang Z W,Zhang C J,Chen S Y,Zhu F,Jia C F,Cao G Y.2020.Codon bias and cluster analysis on coding regions of mtDNA in rock porgy Oplegnathuspunctatus and roke bream O.fasciatus[J].Fisheries Science,39(5):702-709.]doi:10.16378/j.cnki.1003-1111.2020.05.008.
任童童,喻达辉,翟子钦,谭传港,何积翠,尚东维,陈移波,白丽蓉.2022.法螺线粒体全基因组密码子偏好性分析[J].南方农业学报,53(5):1379-1387.[Ren T T,Yu D H,Zhai Z Q,Tan C G,He J C,Shang D W,Chen Y B,BaiL R.2022.Analysis of codon usage bias in mitochondrial genome of Charoniatritonis[J].Journal of Southern Agri‐culture,53(5):1379-1387.]doi:10.3969/j.issn.2095-1191.2022.05.020.
任桂萍,董璎莹,党云琨.2019.密码子中的密码:密码子偏好性与基因表达的精细调控[J].中国科学(生命科学),49(7):839-847.[Ren G P,Dong Y Y,Dang Y K.2019.Codon codes:Codon usage bias influences many levels of gene expression[J].Science in China(Series C),49(7):839-847.]doi:10.1360/SSV-2019-0103.
宋乔乔,钟金城.2013.牛亚科动物线粒体基因密码子偏好性及聚类分析[J].西北农业学报,22(10):1-8.[Song QQ,Zhong J C.2013.Codon usage bias and cluster analysis of mitochondrial genes from Bovinae animals[J].Acta Agri‐culturaeBoreali-occidentalis Sinica,22(10):1-8.]doi:10.7606/j.issn.1004-1389.2013.10.001.
张扬,孙曙光,李晴,魏珍.2022.莴苣线粒体基因组密码子使用偏好性分析[J].河南农业科学,51(10):114-124.[Zhang Y,Sun S G,Li Q,Wei Z.2022.Codon usage bias analysis of cultivated and wild lettuce mitochondrial genomes[J].Journal of Henan Agricultural Sciences,51(10):114-124.]doi:10.15933/j.cnki.1004-3268.2022.10.013.
Bellgard M,Schibeci D,Trifonov E,Gojobori T.2001.Early detection of G+C differences in bacterial species inferred from the comparative analysis of the two completely sequenced Helicobacter pylori strains[J].Journal of Mo-lecular Evolution,53(4-5):465-468.doi:10.1007/s002390 010236.
Bernery C,Bellard C,Courchamp F,Brosse S,Gozlan R E,JarićI,Telethea F,Leroy B.2022.Freshwater fish inva‐sions:A comprehensive review[J].Annual Reviews,53:427-456.doi:10.1146/annurev-ecolsys-032522-015551.
Bulmer M.1991.The selection-mutation-drift theory of synony‐mous codon usage[J].Genetics,129(3):897-907.doi:10.1093/genetics/129.3.897.
Caiola N,de Sostoa A D.2005.Possible reasons for the decline of two native toothcarps in the Iberian Peninsula:Evi‐dence of competition with the introduced Eastern mosqui‐tofish[J].Journal of Applied Ichthyology,21(4):358-363.https://doi.org/10.1111/j.1439-0426.2005.00684.x.
Chamani Mohasses F,Solouki M,Ghareyazie B,Fahmideh L,Mohsenpour M.2020.Correlation between gene expres‐sion levels under drought stress and synonymous codon usage in rice plant by in-silico study[J].PLoS One,15(8):e0237334.doi:10.1371/journal.pone.0237334.
Gao J C,Ouyang X,Chen B J,Jourdan J,Plath M.2017.Molecular and morphometric evidence for the widespreadintroduction of western mosquitofish Gambusia affinis(Baird and Girard,1853)into freshwaters of mainland China[J].Bioinvasions Records,6(3):281-289.doi:10.3391/bir.2017.6.3.14.
Kawabe A,Miyashita N T.2003.Patterns of codon usage biasin three dicot and four monocot plant species[J].Genes&Genetic Systems,78(5):343-352.doi:10.1266/ggs.78.343.
Lau E T C,Leung K M Y,Karraker N E.2019.Native amphi-bian larvae exhibit higher upper thermal limits but lowerperformance than their introduced predator Gambusia affi-nis[J].Journal of Thermal Biology,81:154-161.doi:10.1016/j.jtherbio.2019.02.014.
Liu C L,He D K,Chen Y F,Olden J D.2017.Species inva-sions threaten the antiquity of China’s freshwater fish fauna[J].Diversity and Distributions,23(5):556-566.doi:10.1111/ddi.12541.
Liu Q P,Feng Y,Xue Q Z.2004a.Analysis of factors shaping codon usage in the mitochondrion genome of Oryza sativa[J].Mitochondrion,4(4):313-320.doi:10.1016/j.mito.2004.06.003.
Liu Q P,Feng Y,Zhao X A,Dong H,Xue Q Z.2004b.Synony-mous codon usage bias in Oryza sativa[J].Plant Science,167(1):101-105.doi:10.1016/j.plantsci.2004.03.003.
Lowe S,Browne M,Boudjelas S,De Poorter M.2000.100 of the world's worst invasive alien species:A selection from the global invasive species database[M].Gland:Invasive Species Specialist Group,World Conservation Union.
Mack R N,Simberloff D,Lonsdale W M,Evans H,Clout M,Bazzaz FA.2000.Biotic invasions:Causes,epidemiology,global consequences,and control[J].Ecological Applica-tion,103):689-710.doi:10.1890/1051-0761(200010[0689:BICEGC]2.0.CO;2.
Pouyet F,Mouchiroud D,Duret L,Sémon M.2017.Recombi-nation,meiotic expression and human codon usage[J].Elife,6:e27344.doi:10.7554/eLife.27344.
Pyke G H.2005.A review of the biology of Gambusia affinis and G.holbrooki[J].Reviews in Fish Biology and Fishe-ries,15:339-365.doi:10.1007/s 11160-006-6394-x.
Pyke G H.2008.Plague minnow or mosquito fish?A review ofthe biology and impacts of introduced Gambusia species[J].Annual Review of Ecology Evolution and Systema-tics,39(1):171-191.doi:10.1146/annurev.ecolsys.39.110707.173451.
Sanz N,Araguas R M,Vidal O,Diez-del-Molino D,Fernández-Cebrián R,García-Marín J L.2013.Genetic characteriza-tion of the invasive mosquitofish(Gambusia spp.)intro-duced to Europe:Population structure and colonizationroutes[J].Biological Invasions,15:2333-2346.doi:10.1007/s 10530-013-0456-5.
Seward E A,Kelly S.2016.Dietary nitrogen alters codon bias and genome composition in parasitic microorganisms[J].Genome Biology,17(1):226.doi:10.1186/s 13059-016-1087-9.
Sharp P M,Emery L R,Zeng K.2010.Forces that influence the evolution of codon bias[J].Philosophical Transactions of the Royal Society London Series B.Biological Sciences,365(1544):1203-1212.doi:10.1098/rstb.2009.0305.
Stockwell CA,Weeks S C.1999.Translocations and rapid evo-lutionary responses in recently established populations of western mosquitofish(Gambusia affinis)[J].Animal Con-servation,2(2):103-110.doi:10.1111/j.1469-1795.1999.tb00055.x.
Sueoka N.1988.Directional mutation pressure and neutral molecular evolution[J].Proceedings of the National Aca-demy of Sciences,85(8):2653-2657.doi:10.1073/pnas.85.8.2653.
Sueoka N.1999.Two aspects of DNA base composition:G+C content and translation-coupled deviation from intra-strand rule ofA=Tand G=C[J].Journal of Molecular Evo-lution,49(1):49-62.doi:10.1007/pl00006534.
Uddin A,Choudhury M N,Chakraborty S.2017.Factors influ-encing codon usage of mitochondrial ND1 gene in pisces,aves and mammals[J].Mitochondrion,37:17-26.doi:10.1016/j.mito.2017.06.004.
Vannini A,Bruni G,Ricciardi G,Platania L,Mori E,Tricarico E.2018.Gambusia holbrooki,the“tadpolefish”:The impact of its predatory behaviour on four protected species of European amphibians[J].Aquatic Conservation:Ma-rineand Freshwater Ecosystems,28(2):476-484.doi:10.1002/aqc.2880.
Vidal O,García-Berthou E,Tedesco P A,García-Marín J L.2010.Origin and genetic diversity of mosquitofish(Gam-busiaholbrooki)introduced to Europe[J].Biological In-vasions,12(4):841-851.doi:10.1007/s 10530-009-9505-5.
Wright F.1990.The‘effective number of codons’used in a gene[J].Gene,87(1):23-29.doi:10.1016/0378-1119(90)90491-9.
(责任编辑兰宗宝)

