狂犬病病毒GX074株P-MS20F突变体构建及其生长特性鉴定
摘要:【目的】明确狂犬病病毒(RABV)强毒株GX074的P蛋白和M蛋白第20位苯丙氨酸(Phe)替换至弱毒株rRC-HL的重组突变体在宿主细胞内的生长特性,为P蛋白和M蛋白转录复制机理研究提供理论依据。【方法】运用反向遗传技术以强毒株GX074的M蛋白第20位Phe替换弱毒株rRC-HL(GX074P)的M蛋白第20位丝氨酸(Ser),构建rRC-HL(GX074P-MS20F)突变体感染性cDNA克隆并拯救病毒。以重组突变体感染BSR/T7-9细胞,进行多步生长曲线测定,比较重组突变体与亲本毒株的生长能力,采用Western blotting检测N蛋白、P蛋白和M蛋白相对表达水平,利用实时荧光定量PCR检测N基因、P基因和M基因的相对表达量。【结果】经RT-PCR和测序鉴定,拯救的突变体rRC-HL(GX074P-MS20F)M蛋白第20位Ser成功替换为Phe。多步生长曲线测定结果显示,构建的重组突变体在感染24、48、72和96h后,病毒滴度均高于亲本弱毒株rRC-HL和对照弱毒株rRC-HL(GX074PM1),平均约为亲本弱毒株rRC-HL的10倍。在蛋白表达水平上,rRC-HL(GX074P-MS20FF)毒株在感染48h后,N蛋白和M蛋白相对表达水平均极显著高于亲本弱毒株rRC-HL(Plt;0.01),说明强毒株GX074的P蛋白联合M蛋白第20位Phe替换至弱毒株rRC-HL后,增加了突变体N蛋白和M蛋白表达。感染24和48h后,rRC-HL(GX074P-MS20F)毒株N基因、P基因和M基因的相对表达量均高于亲本弱毒株rRC-HL和对照弱毒株rRC-HL(GX074PM1)。【结论】强毒株GX074的M蛋白第20位Phe替换可提高病毒的增殖能力,同时增强病毒的复制与转录能力,M蛋白第20位Phe可能是影响病毒复制和转录的关键位点。
关键词:狂犬病病毒(RABV);P蛋白;M蛋白;生长特性;反向遗传
中图分类号:S852.659.5文献标志码:A文章编号:2095-1191(2024)01-0253-10
Construction of rabiesvirus GX074P-MS20Fmutant and identification of its growth characteristics
PENG Jing¹2,LI Wen-fang¹2,LU Li-ying¹2,WEI Xian-kai¹2, LI Xiao-ning¹·2*,LUO Ting-rong¹·2*
('College of Animal Science and Technology,GuangxiUniversity,Nanning,Guangxi530004,China;2State Key Laboratory for Conservation and Utilization of Subtropical Agro-bioresources,Nanning,Guangxi530004,China)
Abstract:[Objective]To clarify the growth characteristics of the recombinant mutant with the replacement of Ppro-teinand the20\"phenylalanine(Phe)of Mprotein of the strong strain of rabies virus(RABV)strainGX074to the attenua-ted strain rRC-HL in host cells,and to provide theoretical basis for the study of the transcription and replication mecha-nism ofthe Pand Mproteins.【Method]The20#serine(Ser)of Mprotein of attenuated strain rRC-HL(GX074P)was re-placedby the20Pheof Mprotein of strong strainGX074by reverse genetic technique toconstruct the infectious cDNA clone of rRC-HL(GX074P-MS20FF)mutant and rescue the virus.BSR/T7-9cell was infected by recombinant mutant,and multi-step growth curve determination was performed to compare the growth ability ofthe recombinant mutant and that of the parent strain.The relative expression levels of N,P and Mproteins were detected by Western blotting,and the rela-tive expression levels of N,P and Mgenes were detected by real-time fluorescence quantitative PCR.【Result]RT-PCR and sequencing confirmed that the20#Ser of the Mprotein of the rescued mutant rRC-HL(GX074P-MS20FF)was success-fully replaced by the20Phe.The results of the multiple-step growth curve demonstrated that the viral titer of therecombi-nant mutant was higher than that of both the parental attenuated strain rRC-HL and the control attenuated strain rRC-HL(GX074PM1)at24,48,72and96h after infection,with10times of the parent attenuated rRC-HL.In terms of protein expression,the relative expression levels of Nprotein and Mprotein of rRC-HL(GX074P-MS20F)strain were extremely significantly higher than those of the parental attenuated strain rRC-HL at48h after infection(Plt;0.01).These results indi cated that the substitution of the Pprotein of strong strain GX074with the20#Phe of Mprotein to the attenuated strain rRC-HL increased the expression of Nand Mproteins in the mutant.The relative expression levels of N,Pand Mgenes of rRC-HL(GX074P-MS0)strain were higher than those of parental attenuated strain rRC-HL and control attenuated strain rRC-HL(GX074PM1)strain at24and48h after infection.【Conclusion]The replacement of Phe at the20#posi-tion of Mprotein of strong strain GX074can improve the proliferation ability of virus,and enhance the replication and transcription ability of virus.Phe atthe20\"position of Mprotein maybe akey site affecting the replication and transcrip-tion of virus.
Keywords:rabies virus(RABV);Pprotein;M protein;growth characteristics;reverse genetics
Foundation items:National Natural Science Foundation of China(32070161);Guangxi Natural Science Foundation(2020GXNSFAA297212)
0引言
【研究意义]狂犬病是最古老的全球性人兽共患传染病之一,人类、家畜和野生动物中存在种内和种间的狂犬病病毒(Rabies virus,RABV)感染,持续威胁世界公共卫生健康。中国报告的狂犬病死亡人数在全球排名第2,仅次于印度(Tan et al.,2017),尽管近10年死亡人数每年下降20%,狂犬病仍是中国儿童第二大最常见的传染病死因(Dong et al.,2020)。据统计,2016—2020年中国狂犬病病例总数排名前5的省份为湖南、河南、广西、贵州和湖北,占中国狂犬病病例总数48%(覃晴等,2020)。目前,狂犬病无药物可治疗,而狂犬病死亡人数的下降主要依靠实施暴露后预防(Post-exposure prophylaxis,PEP)措施(Zhang et al.,2017)和家犬免疫预防措施(Wei et al.,2018b)。但农村和城市的犬疫苗接种率差异较大,从近乎0到90%不等(Wang et al.,2011)。因技术相对落后和官方政策未得到严格执行等,中国针对流浪犬和野生动物的口服疫苗仍处于实验室研究水平,疫苗接种率较低,因此寻找高效、强有力的狂犬病预防控制措施十分必要(Miao et al.,2021)。深入了解和研究RABV基因结构、感染后发病机制和遗传变异机制是有效控制和治疗狂犬病的基本前提。【前人研究进展】RABV形似子弹状,长约180nm,直径约90nm,由1个内部核衣壳和1个外部膜结构连接形成;内部螺旋状核衣壳由核蛋白(N)、磷蛋白(P)和病毒RNA依赖的RNA聚合酶(L)、单链基因组RNA组成(孙玉章等,2015);外部病毒囊膜由病毒糖蛋白(G)的棘状突起和从宿主细胞获得的双层脂膜形成;2个部分由基质蛋白(M)连接,M蛋白与G蛋白相互作用,形成致密的病毒囊膜(Liu et al.,2021)。RABV的P蛋白是一种多功能蛋白,帮助RABV的复制、转录和运输,可沿着微管轨迹操纵动力蛋白(Liu et al.,2022)。RABV的M蛋白是病毒成熟和出芽所必需的最小病毒蛋白(Riedel et al.,2020),其在病毒囊膜和核糖核蛋白(RNP)间形成结构桥并调节病毒和宿主蛋白的表达(Liu et al.,2021)。M蛋白调节病毒转录和复制间的平衡,参与病毒的装配和出芽、病毒基因组的调节和mRNA的合成(Finke et al.,2003),被证实与病毒致病性有关(Faber et al.,2004)。RABV的M蛋白氨基酸位点突变可能对病毒复制产生重要影响。Finke和Conzelmann(2003)构建了重组病毒SAD M(R58G),与SAD L16毒株相比,SAD M(R58G)毒株失去下调病毒转录和刺激复制能力,其转录水平上调15倍,但病毒的装配及出芽不受影响,说明RABV的M蛋白仅影响RNA合成的调节而不影响装配与出芽,M蛋白的差异表达表明其在RNA合成中的调节功能独立于病毒装配,第58位精氨酸(Arg)具有特殊作用。Wu等(2013)以PV疫苗株(M13215)为参照,鉴定出中国各地分离株中20个氨基酸变异,根据M蛋白比对结果将突变序列分为4组,第Ⅲ组含Q17H、S20F和P2IS特异性变化位点,包括来自安徽、浙江、广西、湖南、山东和上海的分离株,M基因高变区位于潜在抗原区,可能影响毒株的抗原性和致病性。GX074为广西大学动物科学技术学院亚热带家畜疫病致病机理和防治研究团队2003年从广西百色市德保县无临床症状犬分离获得的街毒株,属于广西流行RABVⅡ群毒株,其M蛋白也存在Q17H、S20F和P21S特异性变化位点,Wei等(2018c)研究发现,单独将强毒株GX074的P基因或M基因替换至标准弱毒株RC-HL相应位置构建的重组毒株未引起小鼠死亡,而联合突变GX074毒株P-M基因的突变体对小鼠具有致病性。陆丽莹等(2022)进一步研究影响GX074毒株致病性的M蛋白相关区域,将GX074毒株M蛋白的M1(1~22位氨基酸)、M2(44~46位氨基酸)区域与P蛋白联合替换至rRC-HL毒株,结果表明重组毒株rRC-HL(GX074PM1)、rRC-HL(GX074PM12)生长趋势与亲本弱毒株rRC-HL相似,但复制和增殖能力略高于毒株rRC-HL,远高于亲本强毒株GX074和强毒株CVS-11,推测GX074的M蛋白M1和M2区域与病毒致病性相关。【本研究切入点】RABV的P蛋白和M蛋白联合突变后的致死性是否与M蛋白某个位点相关尚未明确。GX074毒株M蛋白第20位氨基酸为苯丙氨酸(Phe),RC-HL毒株M蛋白第20位氨基酸为丝氨酸(Ser),将中性氨基酸Ser转变为疏水氨基酸Phe,会导致氨基酸的净电荷发生变化,可能影响病毒蛋白的功能。【拟解决的关键问题]运用反向遗传技术以强毒株GX074毒株M蛋白第20位Phe替换弱毒株rRC-HL(GX074P)的M蛋白第20位Ser构建重组突变体,进行多步生长曲线测定、Western blotting和实时荧光定量PCR检测,探究重组突变体在宿主细胞内的生长能力、复制和转录水平,为P蛋白和M蛋白转录复制机理研究提供理论依据。
1材料与方法
1.1试验材料
街毒株GX074为前期分离获得,弱毒株rRC-HL和重组弱毒株rRC-HL(GX074P)、rRC-HL(GX074PM1)为前期通过反向遗传技术拯救获得,适应细胞的标准强毒株CVS-11由华中农业大学赵凌教授惠赠;稳定表达T7RNA聚合酶的BSR/T7-9细胞由亚热带家畜疫病致病机理和防治研究团队保存;感染性cDNA克隆质粒pRC-HL(GX074P)和pRC-HL(GX074PM1)为前期通过反向遗传技术构建;辅助质粒pT7IRES-RN、pT7IRES-RP和pT7IRES-RL由日本岐阜大学源宣之教授惠赠。高保真酶Prim-erSTAR MAX DNA Polymerase、反转录酶Reverse Treanscriptase(M-MLV)、RNase Inhibitor、DL15000DAN Marker、DL5000DAN Marker和DL1000DAN Marker购自日本TaKaRa公司,限制性内切酶AgeI、SacⅡ购自美国NEB公司,ClonExpress®Ⅱ一步法克隆试剂盒、总RNA提取试剂盒和胎牛血清(FBS)购自诺唯赞生物科技有限公司,大肠杆菌TOP10感受态细胞购自天根生化科技有限公司,无内毒素质粒抽提试剂盒购自美国Omega Bio-Tek公司,DMEM培养基、Opti-MEM减血清培养基和LipofectaminelM2000转染试剂购自美国Life Technologies公司,AlexaFluor488标记山羊抗小鼠IgG、荧光二抗显色试剂盒、碱性磷酸酶标记马抗小鼠IgG、BCIP/NBT碱性磷酸酯酶显色试剂盒和RIPA裂解液购自碧云天生物技术有限公司,RABVN蛋白单克隆抗体购自杭州大毒生物科技有限公司,RABVM蛋白单克隆抗体购自武汉华美生物工程有限公司,RABV P蛋白单克隆抗体由亚热带家畜疫病致病机理和防治研究团队保存,鼠抗β-actin单克隆抗体购自江苏康为世纪生物科技股份有限公司,二硫苏糖醇(DTT)购自赛默飞世尔科技有限公司,脱脂奶粉购自北京索莱宝科技有限公司,PVDF膜(0.45μm)购自美国Millipore公司。主要仪器设备:S1000热循环PCR仪(美国Bio-Rad公司)、荧光倒置显微镜(日本Nikon公司)、ImageQuant LAS500成像仪(美国GE HealthCare公司)、NanoDrop1000分光光度计(赛默飞世尔科技有限公司)、LightCycler96实时荧光定量PCR仪[罗氏诊断产品(上海)有限公司]和恒温培养箱(上海跃进医疗器械厂)。
1.2引物设计与合成
从GenBank下载标准弱毒株RC-HL(GenBank登录号AB009663.2)及广西街毒株GX074(GenBank登录号MG201923.1)全基因组序列,采用DNAMAN进行碱基序列比对,使用Primer5.0设计包含M蛋白第20位氨基酸突变位点的上、下游引物;含有Age I和SacⅡ酶切位点的引物根据ClonExpress®Ⅱ克隆试剂盒说明设计;引物信息见表1,所有引物均委托南宁捷尼斯生物科技有限公司合成。毒株RC-HL、CVS-11和GX074的N基因、P基因、M基因及β-actin内参基因的实时荧光定量PCR引物由亚热带家畜疫病致病机理和防治研究团队保存。
1.3重组突变体感染性cDNA克隆构建
以前期构建的感染性cDNA克隆质粒pRC-HL(GX074P)为模板,用高保真酶进行PCR扩增,以Age I-F和S20F-R为引物扩增N蛋白AgeI酶切位点至M蛋白第20位氨基酸突变位点的Age I~S20F片段,以S20F-F和Sac II-R为引物扩增M蛋白第20位氨基酸突变位点至M蛋白间隔区SacⅡ酶切位点的S20F~Sac IⅡ片段,以Age I~S20F和S20F~Sac IⅡ片段为模板,Age I-F和Sac II-R为上、下游引物进行重叠PCR,获得包含M蛋白第20位氨基酸突变(S20F)的Age I酶切位点至Sac IⅡ酶切位点完整片段Age I~Sac IⅡ。将质粒pRC-HL(GX074P)进行双酶切,获得线性化载体,通过一步法克隆试剂盒将重叠片段连接至pRC-HL(GX074P)线性化载体,以连接后的产物转化TOP10感受态细胞,进行菌液PCR鉴定,经琼脂糖凝胶电泳、测序鉴定,获得rRC-HL(GX074P-MS20F)突变体感染性cDNA克隆。
1.4重组突变体拯救
将构建成功的突变体感染性cDNA克隆进行无内毒素质粒抽提,转染前将BSR/T7-9细胞接种至12孔细胞板,待细胞长至70%~80%,按比例将突变体感染性cDNA克隆质粒、pT7IRES-RN、pT7IRES-RL、pT7IRES-RP辅助质粒加入Opti-MEM减血清培养基,脂质体和质粒混合物按400μL每孔加入细胞板中,5h后用含2%FBS的DMEM更换孔中液体,置于二氧化碳培养箱37℃培养6d,将12孔细胞板置于-80℃反复冻融3次,收集第1代病毒上清液及细胞置于1.5mL EP管中,4℃下12000r/min离心5min,-80℃保存;进行间接免疫荧光(IFA)试验,以未接种病毒的正常细胞为阴性对照,以RABVN蛋白单克隆抗体(1:2000)为一抗,Alexa Fluor488标记山羊抗小鼠IgG(1:1000)为二抗,通过荧光二抗显色试剂盒显色,使用荧光倒置显微镜观察病毒在BSR/T7-9细胞上有无特异性荧光灶。亲本弱毒株rRC-HL、对照弱毒株rRC-HL(GX074PM1)、强毒株CVS-11、亲本强毒株GX074的接种及测定方法同上。经IFA鉴定成功后,取盲传第3代的突变体培养液及细胞进行RNA抽提,并反转录合成cDNA,用引物Age I-F和Sac II-R进行PCR扩增,PCR扩增产物经琼脂糖凝胶电泳鉴定后,进行胶回收并测序,通过SnapGene将测序结果与参考序列进行比对,鉴定突变体序列是否与预期一致。
1.5多步生长曲线测定
将BSR/T7-9细胞接种至24孔细胞板,测定重组突变体病毒滴度后,按0.01MOI接种于长至70%~80%的BSR/T7-9细胞,置于二氧化碳培养箱37℃孵育2h后,换含2%FBS的DMEM继续培养,分别于24、48、72和96h收集细胞上清液置于1.5mL EP管中,-80℃保存备用。采用IFA试验测定各培养时间的病毒滴度,绘制病毒多步生长曲线。亲本弱毒株rRC-HL、对照弱毒株rRC-HL(GX074PM1)、强毒株CVS-11和亲本强毒株GX074的接种及测定方法同上。
1.6Western blotting检测
以0.1MOI将重组突变体接种于长至70%~80%的BSR/T7-9细胞,二氧化碳培养箱37℃孵育2h后,换含2%FBS的DMEM继续培养,分别于24、48h收集经RIPA裂解细胞后的蛋白样品,-80℃保存备用。蛋白样品经超声破碎,4℃下12000r/min离心15min,每管样品加入1/5体积5×Buffer和1/10体积DTT,轻微混匀后煮沸5min。12%SDS-PAGE凝胶电泳后,分离的蛋白通过半干转膜法转印至PVDF膜中。配置5%脱脂奶粉液,封闭2h;分别按1:20000、1:8000、1:2000、1:1000比例,以TBST稀释RABVN蛋白单克隆抗体、P蛋白单克隆抗体、M蛋白单克隆抗体和鼠抗β-actin内参抗体,将稀释好的一抗加入PVDF膜中塑封,置于恒温培养箱37℃孵育1.5h。将塑封的PVDF膜取出,置于含有TBST的玻璃皿中,在水平摇床上洗涤3次,每次10min。按1:1000比例稀释碱性磷酸酶标记马抗鼠IgG,将稀释好的二抗加入PVDF膜中塑封,置于恒温培养箱37℃孵育1h,将PVDF膜置于含有TBST的玻璃皿中,水平摇床上洗涤3次,每次10min。按BCIP/NBT碱性磷酸酯酶显色试剂盒说明对PVDF膜进行显色,检测病毒N蛋白、P蛋白和M蛋白相对表达水平。亲本弱毒株rRC-HL、对照弱毒株rRC-HL(GX074PM1)、强毒株CVS-11和亲本强毒株GX074的接种及测定方法同上,以DMEM为空白对照。
1.7实时荧光定量PCR检测
病毒接种方法同1.6,分别于24和48h收集细胞裂解样品,使用总RNA提取试剂盒对病毒RNA进行抽提及RNA浓度测定,经RT-PCR后进行实时荧光定量PCR检测,采用LightCycler96实时荧光定量PCR仪记录数据,通过2-440法计算病毒N基因、P基因和M基因的相对表达量。亲本弱毒株rRC-HL、对照弱毒株rRC-HL(GX074PM1)、强毒株CVS-11和亲本强毒株GX074的接种及测定方法同上。
1.8统计分析
采用GraphPad Prism8.0进行单因素方差分析(One-way ANOVA),比较重组突变体rRC-HL(GX074P-MS20F)与亲本弱毒株rRC-HL的Western blotting和实时荧光定量PCR检测结果,分析差异显著性,并进行作图。
2结果与分析
2.1重组突变体感染性cDNA克隆构建结果
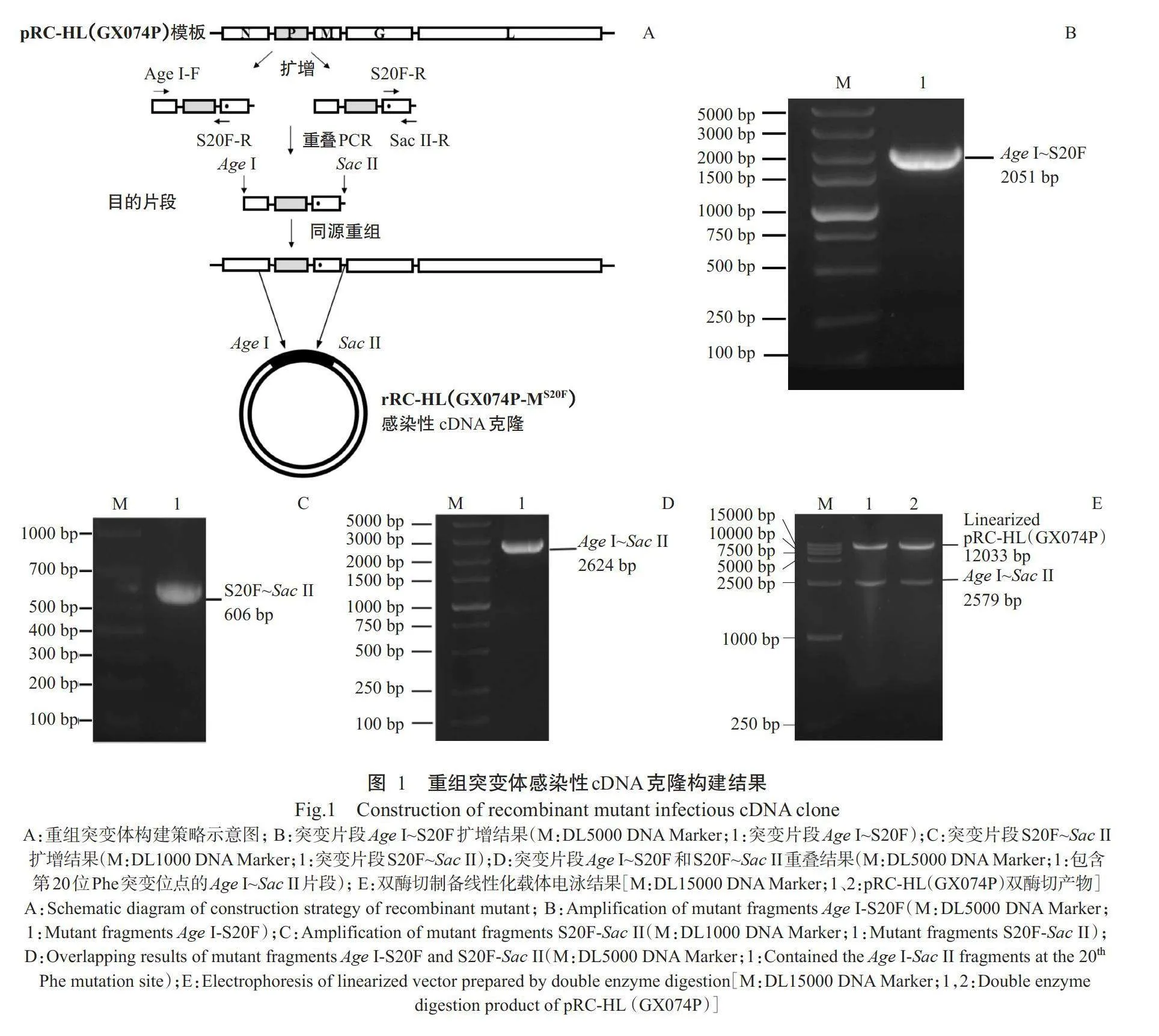
2.1.1重组突变体片段扩增及重叠PCR结果构建策略如图1-A所示,以质粒pRC-HL(GX074P)为模板,用引物Age I-F和S20F-R扩增出N蛋白Age I酶切位点至M蛋白第20位氨基酸的突变片段Age I~S20F,大小约2051bp(图1-B)。用引物S20F-F和Sac II-R扩增出M蛋白第20位突变位点至M蛋白间隔区SacⅡ酶切位点的突变片段S20F~Sac II,大小约606bp(图1-C)。以突变片段Age I~S20F、S20F~Sac IⅡ为模板,Age I-F和Sac IⅡ-R为上、下游引物进行重叠PCR,获得包含M蛋白第20位氨基酸突变(S20F)的Age I酶切位点至Sac IⅡ酶切位点完整序列片段Age I~SacⅡ,大小约2624bp(图1-D)。
2.1.2重组突变体片段的连接将质粒pRC-HL(GX074P)进行双酶切,获得pRC-HL(GX074P)线性化载体,大小为12033bp(图1-E),将线性化载体与重叠后包含M蛋白第20位氨基酸突变(S20F)的Age I酶切位点至SacⅡ酶切位点完整序列片段进行连接,以连接后产物转化TOP10感受态细胞,经菌液PCR鉴定为阳性,成功构建rRC-HL(GX074P-MS20F)突变体感染性cDNA克隆。
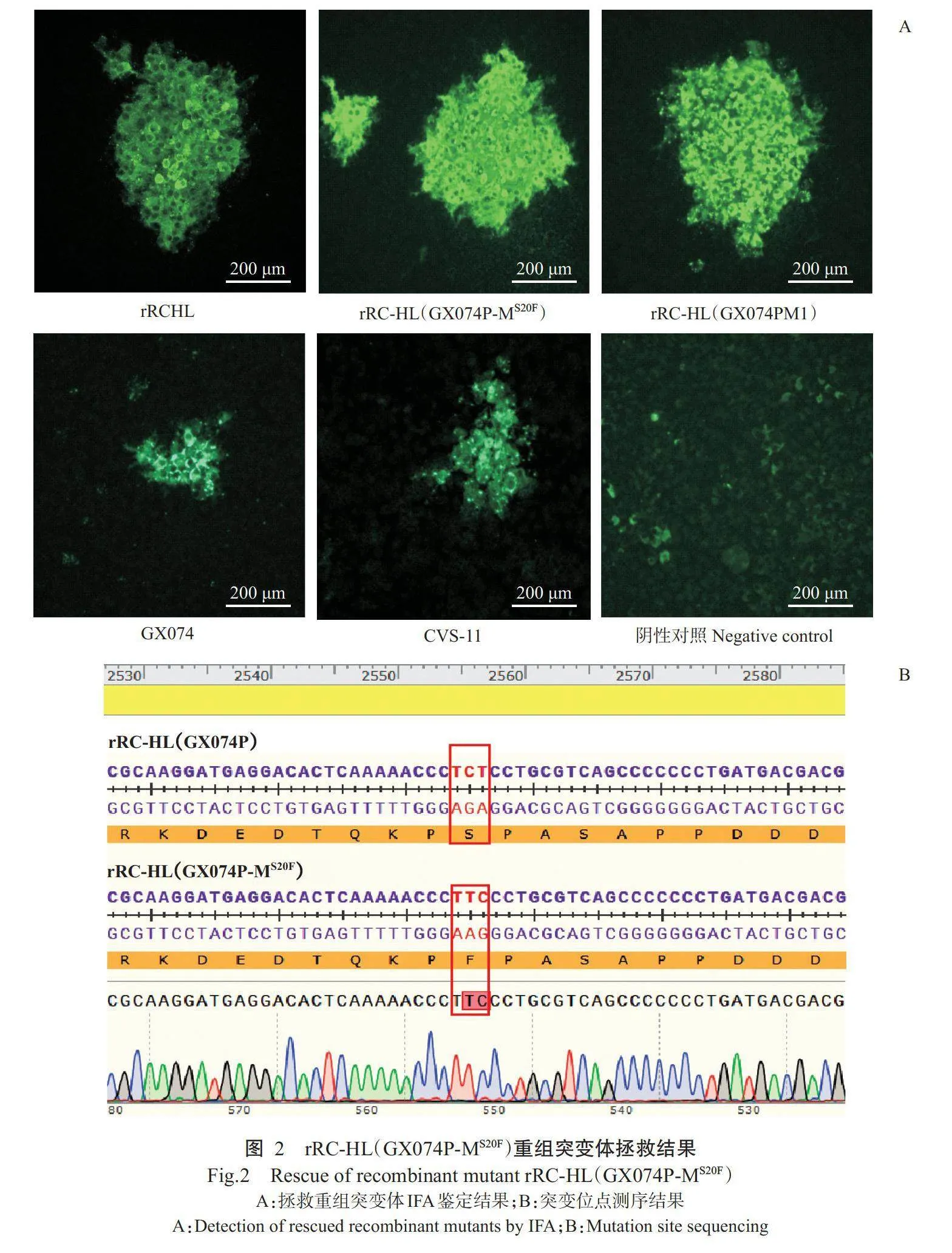
2.2重组突变体拯救结果
以突变体感染性cDNA克隆质粒与辅助质粒共转染BSR/T7-9细胞,收集细胞样品进行IFA试验,结果如图2-A所示,通过荧光倒置显微镜观察到rRC-HL(GX074P-MS20F)感染BSR/T7-9细胞有特异性荧光,且形成荧光灶。经IFA鉴定成功后,取盲传第3代的突变体培养液及细胞进行RNA抽提,反转录合成cDNA并进行PCR扩增,PCR扩增产物经琼脂糖凝胶电泳鉴定合格后,进行测序并通过Snap-Gene比对,拯救的重组突变体rRC-HL(GX074P-MS20F)M蛋白第20位Ser成功替换为Phe(图2-B)。
2.3多步生长曲线测定结果
病毒多步生长曲线测定结果如图3所示,亲本强毒株GX074在BSR/T7-9细胞上的生长能力最弱,强毒株CVS-11适应细胞,在BSR/T7-9细胞上的生长能力与弱毒株rRC-HL相似。对照弱毒株rRC-HL(GX074PM1)与亲本弱毒株rRC-HL的生长趋势相似,而重组突变体rRC-HL(GX074P-MS20F)在感染24、48、72和96h后,病毒滴度均高于亲本弱毒株rRC-HL和对照弱毒株rRC-HL(GX074PM1),平均病毒滴度约为亲本弱毒株rRC-HL的10倍,说明与将GX074毒株M蛋白第1~22位氨基酸替换至rRC-
HL毒株构建的rRC-HL(GX074PM1)相比,M蛋白第20位Phe替换提高了弱毒株rRC-HL的增殖能力。
2.4Western blotting检测结果
如图4所示,与亲本弱毒株rRC-HL相比,突变体rRC-HL(GX074P-MS20F)在感染细胞24h后,N蛋白、P蛋白和M蛋白相对表达水平呈不同程度上调,分别是亲本弱毒株rRC-HL的1.2、1.3和1.7倍。rRC-HL(GX074P-MS20F)毒株在感染48h后,N蛋白和M蛋白相对表达水平均极显著高于亲本弱毒株rRC-HL(Plt;0.01),分别是其1.8和2.2倍。说明强毒株GX074的P蛋白联合M蛋白第20位Phe突变入弱毒株rRC-HL后,增加了突变体N蛋白和M蛋白表达。
2.5实时荧光定量PCR检测结果
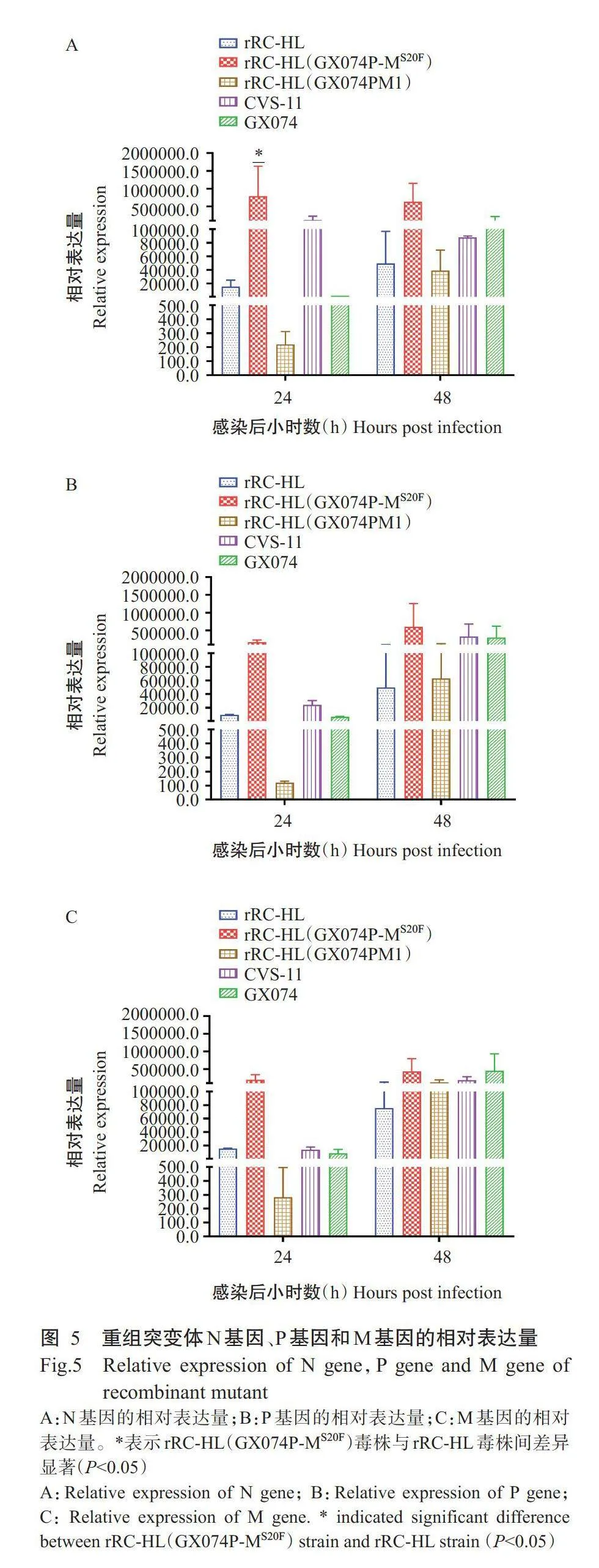
由图5可知,构建的突变体rRC-HL(GX074P-MS20F)感染BSR/T7-9细胞24h后,N基因、P基因、M基因的相对表达量均高于亲本弱毒株rRC-HL、对照弱毒株rRC-HL(GX074PM1)、亲本强毒株GX074和强毒株CVS-11,其中N基因的相对表达量显著高于亲本弱毒株rRC-HL(Plt;0.05),是其51.3倍;P基因和M基因的相对表达量分别是亲本弱毒株rRC-HL的18.3和12.5倍。感染48h后,rRC-HL(GX074PM1)毒株的P基因和M基因的相对表达量与亲本毒株rRC-HL相比呈不同程度上调,而rRC-HL(GX074P-MS20F)毒株N基因、P基因和M基因的相对表达量分别是亲本弱毒株rRC-HL的12.8、12.1和5.7倍。说明与将GX074毒株M蛋白第1~22位氨基酸替换至rRC-
HL构建的rRC-HL(GX074PM1)相比,将GX074毒株M蛋白第20位Phe替换至rRC-HL可提高病毒的转录与复制水平,与病毒多步生长曲线测定结果一致。
3讨论
狂犬病是一种神经性、致死性的人畜共患病,通过患狂犬病的动物咬伤传播。中国至少95%人类狂犬病病例是由患狂犬病的犬咬伤引起(Zhou et al.,2016)。对犬进行狂犬病疫苗接种是预防犬感染RABV,进而降低被咬伤人类发病率的重要措施(韦显凯等,2016),但中国的犬疫苗接种率较低,导致RABV传播频繁并具有遗传多样性。近年来,RABV反向遗传学的发展推动了对不同病毒和宿主细胞因子在病毒致病性中所起作用的研究(杜加亮和唐青,2008)。通过反向遗传技术可对病毒基因进行突变、缺失、插入、互补等操作,推动了对RABV分子特征、基因复制和表达、致病机理及病毒与宿主间相互作用等方面的研究(齐瑛琳等,2010)。此外,反向遗传学在RVBV疫苗研发方面也具有独特优势,为降低RABV病原性和增强免疫原性提供了有效途径(谭业平等,2010)。RABV的M蛋白具有多种功能,参与病毒的转录与复制,M蛋白和G蛋白与RNP的膜内结构域相互作用,分别参与病毒出芽和RNA复制(Zhang et al.,2022)。M蛋白在复制的中晚期作为一种调节开关,可使最初高水平mRNA的合成转变为有利于病毒粒子基因组RNA的装配,以此来调节病毒转录与复制间的平衡(Finke et al.,2003)。本研究通过反向遗传技术将rRC-HL的P和M蛋白的第20位Ser替换为GX074的P蛋白和M蛋白第20位Phe,结果表明M蛋白第20位氨基酸突变对病毒复制和转录能力产生影响。
GX01和GX074均为前期分离获得的广西街毒株,陆专灵(2012)根据氨基酸的差异将GX01毒株M蛋白分成4个区域(1~27、43~47、137~139和57~202),并分别替换至弱毒株rRC-HL,结果发现重组病毒rRC-HL(GX01M2)的复制和出芽能力显著降低。钟一治(2015)针对GX01毒株M蛋白位点研究发现,M蛋白F44L和S46G位点联合突变能显著影响病毒的复制能力,诱导产生更高水平的干扰素,使细胞具有抗病毒作用。曾兰(2007)、Wei等(2018a)对GX074毒株进行全基因组测序和遗传进化分析发现,GX074属于广西流行RABVⅡ群毒株,Ⅱ群毒株的特异性变化位点为Q17H、S20F和P21S,且GX074毒株可100%致死成年小鼠。Wei等(2018c)将GX074的P基因和M基因分别替换至弱毒株rRC-HL相应位置,发现不能引起小鼠死亡,而联合突变P基因和M基因能引起成年小鼠死亡。陆丽莹等(2022)研究GX074毒株M蛋白区域功能,利用反向遗传技术将强毒株GX074的P基因和部分M基因替换至弱毒株rRC-HL相应位置,发现重组病毒rRC-HL(GX074PM1)和rRC-HL(GX074PM12)的生长趋势与亲本弱毒株rRC-HL相似,但复制和增殖能力略高,重组病毒的复制能力远高于亲本强毒株GX074和强毒株CVS-11。周桂全等(2023)研究GX074毒株P蛋白不同区域功能,将GX074毒株P蛋白第48~78位氨基酸和GX074毒株M蛋白替换至弱毒株rRC-HL相应位置,成功构建突变体rRC-HL(GX074P48-78M),发现突变体病毒滴度略低于rRC-HL毒株,但略高于GX074毒株和CVS-11毒株,说明突变体rRC-HL(GX074P48-78M)的复制能力比亲本弱毒株rRC-HL减弱,但比亲本强毒株GX074的复制能力稍强,推测是由于GX074毒株P蛋白第48~78位氨基酸与M蛋白协同作用,影响了突变体的复制能力。但RABV的P蛋白和M蛋白联合突变后的致死性是否与M蛋白某个位点相关尚未明确。
本研究以前期研究为基础,进一步分析P蛋白和M蛋白联合突变后的致死性是否与M蛋白某个位点有关,经序列比对发现rRC-HL和GX074毒株的M蛋白存在S20F特异性变化位点,推测将rRC-HL毒株M蛋白第20位中性氨基酸Ser转变为疏水氨基酸Phe,会导致氨基酸的净电荷发生变化,影响蛋白功能。因此,本研究利用反向遗传技术将GX074毒株M蛋白第20位Phe替换至rRC-HL(GX074P)毒株相应位置,结果表明与亲本弱毒株rRC-HL和对照弱毒株rRC-HL(GX074PM1)相比,重组突变体的多步生长曲线明显上调,约为亲本弱
毒株rRC-HL的10倍,N基因、P基因和M基因的相对表达量,N蛋白、P蛋白和M蛋白相对表达水平均呈不同程度上调,GX074毒株M蛋白第20位Phe可能是影响病毒复制和转录的关键位点,表明M蛋白的单位点氨基酸突变能对病毒复制和转录产生影响,与Finke和Conzelmann(2003)的研究结果相似。在传统进化分析中,单位点氨基酸突变易被忽略,但其在RABV传染性和抗原进化中起关键作用,RABV的P蛋白和M蛋白联合突变后的致死
性与M蛋白第20位Phe有关,MS20F对病毒在BSR/T7-9细胞的复制和转录有重要影响,为后续针对P蛋白和M蛋白的转录复制机理研究提供了理论依据。
4结论
强毒株GX074的M蛋白第20位Phe替换可提高病毒的增殖能力,同时增强病毒的复制与转录能力,M蛋白第20位Phe可能是影响病毒复制和转录的关键位点。
参考文献:
杜加亮,唐青.2008.狂犬病病毒反向遗传学及其应用[J].病毒学报,24(6):478-482.[Du JL,Tang Q.2008.Reverse genetics of rabies virus and its application[J].Chinese Journal of Virology,24(6):478-482.]doi:10.3321/j.issn1000-8721.2008.06.011.
陆丽莹,彭璟,李文芳,韦显凯,李晓宁,罗廷荣.2022.狂犬病病毒GX074野毒株M基因重组病毒的拯救与鉴定[J].广西畜牧兽医,38(5):195-197.[Lu LY,Peng J,LiWF,WeiXK,Li XN,Luo TR.2022.Rescue and identifica-tion of Mgene recombinant virus of rabies virus GX074wild strain[J].Guangxi Journal of Animal Husbandryamp;Veterinary Medicine,38(5):195-197.]doi:10.3969/j.issn.1002-5235.2022.05.001.
陆专灵.2012.狂犬病病毒M基因功能探索及L基因聚合酶活性部位的多态性分析[D].南宁:广西大学.[Lu ZL2012.Functional research on rabies virus Mgene and poly-morphism analysis of Lgene polymerase activity module[D].Nanning:Guangxi University.]
齐瑛琳,杨松涛,金宏丽,赵丽丽,冯娜,夏咸柱.2010.反向遗传学技术在狂犬病病毒研究中的应用[J].中国病原生物学杂志,5(11):865-868.[Qi YL,Yang ST,Jin HL,Zhao LL,Feng N,Xia XZ.2010.Use of reversegenetics in the study of the rabies virus[J].Journal of Pathogen Biology,5(11):865-868.]doi:10.13350j.cjpb.2010.11.002.
覃晴,梁星雪,曹晗,韦显凯,梁晶晶,李晓宁,罗廷荣.2020.2000—2019年广西狂犬病病毒流行株的基因组遗传稳定性分析[J].南方农业学报,51(3):645-651.[Qin Q,Liang XX,Cao H,Wei XK,Liang JJ,LiXN,Luo TR.2020.Genetic stabilityanalysis based on complete genome of wild rabies virus epidemic inGuangxi during2000-2019[J].Journalof Southern Agriculture,51(3):645-651.]doi:10.3969/j.issn.2095-1191.2020.03.022.
孙玉章,何彪,许运斌,丛彦龙,Conzelmann KK.2015.狂犬病病毒单质粒拯救系统的建立及应用[J].畜牧兽医学报,46(11):2020-2024.[Sun YZ,He B,Xu YB,Cong YL,Conzelmann KK.2015.Generation of asingle infec-tious RABV cDNA clone by inserting IRES sequences[J]Acta Veterinaria et Zootechnica Sinica,46(11):2020-2024.]doi:10.11843/j.issn.0366-6964.2015.11.014.
谭业平,祝艳蕾,郭霄峰.2010.狂犬病病毒反向遗传学研究进展[J].中国兽医学报,30(2):282-288.[TanYP,Zhu YL,Guo XF.2010.Research advance on reverse genetics of rabies virus[J].Chinese Journal of Veterinary Science,30(2):282-288.]doi:10.16303/j.cnki.1005-4545.2010.02.035.
韦显凯,郑敏,郑列丰,苏姣秀,尹彦文,梁晟,兰斌.2016.2012—2013年广西犬狂犬病监测及免疫效果分析[J].中国兽医杂志,52(2):71-72.[WeiXK,Zheng M,Zheng LF,Su JX,Yin YW,Liang S,Lan B.2016.Surveillance ofcanine rabies in Guangxi in2012-2013and analysis of immune effect[J].Chinese Journal ofVeterinary Medicine,52(2):71-72.]
曾兰.2007.广西狂犬病野毒株L基因3'端的克隆和序列分析[D].南宁:广西大学.[Zeng L.2007.Cloning and sequenceanalysis of Lgene3'terminal of rabies virus iso-lates from Guangxi[D].Nanning:Guangxi University.]
钟一治.2015.狂犬病病毒M蛋白与细胞传播性相关的功能位点分析[D].南宁:广西大学.[Zhong YZ.2015.Analy-sis of functional sites of rabies virus Mprotein related with cell-to-cell spread capacity[D].Nanning:Guangxi Univer sity.]
周桂全,陈俊蓉,韦显凯,李晓宁,罗廷荣.2023.狂犬病病毒GX074株P蛋白48~78位区域氨基酸联合M蛋白突变株的构建及特性研究[J].广西畜牧兽医,39(1):3-7[Zhou GQ,Chen JR,WeiXK,LiXN,Luo TR.2023.
Construction and characterization of rabies virus GX074strain with48-78amino acids combined with Mprotein mutant[J].Guangxi Journal of Animal Husbandryamp;Vete-rinary Medicine,39(1):3-7.]
Dong YH,WangLP,Burgner DP,Miller JE,SongY,Ren X,Li ZJ,Xing Y,MaJ,Sawyer SM,Patton GC.2020.Infectious diseases in children and adolescents in China:Analysis of national surveillance data from2008to2017[J].The British Medical Journal,369:m1043.doi:10.1136/bmj.m1043.
Faber M,Pulmanausahakul R,Nagao K,Prosniak M,Rice AB,Koprowski H,Schnell MJ,Dietzschold B.2004.Identifi-cation of viral genomic elements responsible for rabies virus neuroinvasiveness[J].Proceedings of the National Academy of Sciences of the United States of America,101(46):16328-16332.doi:10.1073/pnas.0407289101.
Finke S,Conzelmann KK.2003.Dissociation of rabies virus matrix protein functions in regulation of viral RNA synthe-sis and virusassembly[J].Journal ofVirology,77(22):12074-12082.doi:10.1128/jvi.77.22.12074-12082.2003.
Finke S,Mueller-Waldeck R,Conzelmann KK.2003.Rabies virus matrix protein regulates the balance of virus tran-scription and replication[J].Journal of General Virology84(6):1613-1621.doi:10.1099/vir.0.19128-0.
Liu XL,Nawaz Z,Guo CX,Ali S,Naeem MA,Jamil T,Ahmad W,Siddiq MU,Ahmed S,Asif Idrees M,Ahmad A.2022.Rabies virus exploits cytoskeleton network to cause early disease progression and cellular dysfunction[J].Frontiers in Veterinary Science,9:889873.doi:103389/fvets.2022.889873.
Liu X,LiF,Zhang JW,Wang LL,Wang JL,Wen ZY,Wang ZL,Shuai L,Wang XJ,Ge JY,Zhao DM,BuZ G.2021.The ATPase ATP6V¹A facilitates rabies virus replication by promoting virion uncoating and interacting with the"viral matrix protein[J].Journal of Biological Chemistry296:100096.doi:10.1074/jbc.RA120.014190.
Miao F,Li N,Yang JJ,Chen T,Liu Y,Zhang SF,Hu RL.2021.Neglected challenges in the control of animal rabies in China[J].One Health,12:100212.doi:10.1016/j.one-hlt.2021.100212.
Riedel C,Hennrich AA,Conzelmann KK.2020.Components and architectureof the rhabdovirus ribonucleoprotein com-plex[J].Viruses,12(9):959.doi:10.3390/v12090959.
Tan JM,Wang RY,JiS L,Su S,ZhouJY.2017.One Health strategies for rabies control in rural areas of China[J].The Lancet Infectious Diseases,17(4):365-367.doi:10.1016/S1473-3099(17)30116-0.
Wang C,Wang Y,Du XY,Zeng L,Dong G,Wu YH,Lu J,Wei DY,Zhu X,Liu GS,Zhao TY,Chen ZW.2011.Rabies immunization status of dogs,Beijing,China[J].Emerging Infectious Diseases,17(6):1129-1130.doi:10.3201/eid1706.101590.
Wei XK,He XX,Pan Y,Liu C,TangH B,Zhong YZ,LiX N,Liang JJ,Luo TR.2018a.Evolutionary analysis of rabies virus isolates from Guangxi Province of southern China[J].BMC Veterinary Research,14(1):188.doi:10.1186/s12917-018-1514-0.
Wei XK,Xiong Y,LiXN,Zheng M,Pan Y,He XX,Liang JJ,Liu C,Zhong YZ,Zou LB,Zheng LF,Guo JG,LiC T,Huang SB,GanJZ,Meng ZM,Yang J,Tang HB,Liu Q,Luo TR.2018b.Vaccination demonstration zone suc-cessfully controls rabies in Guangxi Province,China[J]BMC Infectious Diseases,18(1):386.doi:10.1186/s12879-018-3301-8.
Wei XK,Zhong YZ,Pan Y,Li XN,Liang JJ,Luo TR.2018c.TheN and Pgenes facilitate pathogenicity of the rabies virus Ggene[J].Veterinární Medicina,63(12):561-570.doi:10.17221/63/2018-VETMED.
Wu H,Wang LH,Tao XY,Li H,Rayner S,Liang GD,Tang"Q.2013.Genetic diversity and molecular evolution of the rabies virus matrix protein gene in China[J].Infection Genetics and Evolution,16:248-253.doi:10.1016/j.meegid.2013.02.009.
Zhang HL,Huang JB,Song YM,Liu XQ,Qian MC,Huang P,LiYY,Zhao L,Wang HL.2022.Regulation of innate immune responses by rabies virus[J].Animal Models and Experimental Medicine,5(5):418-429.doi:10.1002/ame2.12273.
Zhang JY,Zhang B,Zhang SF,Zhang F,LiN,Liu Y,HuR L.2017.Dog-transmitted rabies in Beijing,China[J].Bio-medical andEnvironmental Sciences,30(7):526-529.doi:10.3967/bes2017.069.
Zhou H,Vong S,Liu K,LiY,Mu D,Wang LP,Yin WW,Yu HJ.2016.Human rabies in China,1960-2014:A descrip-tive epidemiological study[J].PLoS Neglected Tropical Di-seases,10(8):e0004874.doi:10.1371/journal.pntd.0004874.
(责任编辑 刘可丹)

