茶树大面白叶绿体基因组特征、密码子偏好性及其系统发育分析
尹明华 张嘉欣 乐芸 何凡凡 黄添慧 张牧彤
摘要:茶树大面白(Camellia sinensis cv. ‘Damianbai)在1985年被全国农作物品种审定委员会认定为国家品种,其起源以及与其他茶树品种之间的进化关系尚不清晰。以茶树大面白为试验材料,采用高通量测序技术对茶树大面白叶绿体全基因组进行测序、组装、注释,采用生物信息学软件对其叶绿体的基因组特征、系统发育和密码子偏好性进行分析。结果表明,茶树大面白叶绿体基因组全长157 129 bp,为典型的四分体结构,包括1个LSC区(86 687 bp)、1个SSC区(18 282 bp)和2个IR区(包括IRa和IRb,均为26 080 bp)。大面白叶绿体基因组共注释到135个功能基因,包括90个CDS基因;8个rRNA基因和37个tRNA基因。共检测到52个SSR和50个Longrepeat,SSR只有A/T单核苷酸重复序列,Longrepeat只存在正向重复和回文重复2种类型。茶树大面白叶绿体基因组密码子使用偏性主要受自然选择的影响,受内部突变压力的影响小。茶树大面白叶绿体基因有14个最优密码子(AAU、GAU、UGU、AAA、UAA、GCA、GCU、GGU、CCU、GUA、CGU、CUU、AGU、UCU)。茶树大面白与凤凰单丛茶白银(Camellia sinensis isolated Baiyin cultivar Phoenix Dancong Tea,OL690374)亲缘关系较近。本研究分析了大面白茶树叶绿体基因组序列特征及系统发育进化关系,为加强茶树大面白种质鉴定及其资源多样性的开发利用提供了参考依据。
关键词:茶树大面白;叶绿体基因组;序列特征;密码子偏好性;最优密码子;系统发育分析
中图分类号:S571.1;S326 文献标识码:A 文章编号:1000-369X(2024)03-411-20
Genomic Characteristics, Codon Preference, and Phylogenetic Analysis of Chloroplasts of Camellia sinensis cv. ‘Damianbai
YIN Minghua1,2,3,4, ZHANG Jiaxin1, LE Yun1, HE Fanfan1, HUANG Tianhui1, ZHANG Mutong1
1. College of Life Sciences, Shangrao Normal University, Shangrao 334001, China; 2. Shangrao Agricultural Technology Innovation Research Institute, Shangrao 334001, China; 3. Majiayou Industry Research Institute of Shangrao Normal University, Shangrao 334001, China; 4. Key Laboratory of Protection and Utilization of Medicinal and Edible Plant Resources in Shangrao City, Shangrao 334001, China
Abstract: ‘Damianbai was approved as a national tea cultivar by the National Crop Variety Approval Committee in 1985, but its origin and evolutionary relationship with other tea resources are still unclear. Using ‘Damianbai as the experimental material, high-throughput sequencing technology was used to sequence, assemble and annotate the entire chloroplast genome of ‘Damianbai. In order to provide a basis for studying its phylogenetic evolutionary relationship, bioinformatics software was used to analyze the characteristics, phylogeny, and codon preference of its chloroplast genome. The results show that the chloroplast genome of the tea cultivar ‘Damianbai had a total length of 157 129 bp and was a typical tetrad structure, including 1 LSC region (86 687 bp), 1 SSC region (18 282 bp), and 2 IR regions (including IRa and IRb, both of which were 26 080 bp). A total of 135 functional genes were annotated in the chloroplast genome of ‘Damianbai, including 90 CDS genes, 8 rRNA genes, and 37 tRNA genes. A total of 52 SSRs and 50 Longrepeat sequences were detected in the chloroplast genome of ‘Damianbai. The SSRs had only A/T single nucleotide repeat sequences, while Longrepeat sequences had only two types: forward repeat and palindrome repeat. The codon usage bias in the chloroplast genome of tea cultivar ‘Damianbai was mainly influenced by natural selection, and was less affected by internal mutation pressure. The chloroplast gene of tea cultivar ‘Damianbai had 14 optimal codons (AAU, GAU, UGU, AAA, UAA, GCA, GCU, GGU, CCU, GUA, CGU, CUU, AGU, UCU). The Camellia sinensis cv. ‘Damianbai had a close genetic relationship with Camellia sinensis isolate Baiyin cultivar Phoenix Dancong Tea (OL690374). This study analyzed the chloroplast genome sequence characteristics and phylogenetic relationships of ‘Damianbai, which provided a reference basis for strengthening the identification of tea cultivar ‘Damianbai and the development and utilization of its resource diversity.
Keywords: Camellia sinensis cv. ‘Damianbai, chloroplast genome, sequence characteristics, codon usage bias, optimal codons, phylogenetic analysis
茶树(Camellia sinensis)为山茶科山茶属多年生、自交不亲合、异花授粉、高度遗传异质性的常绿植物,是古老的非酒精饮料经济作物[1-2]。我国茶树种质资源丰富,种间杂交频繁,核基因组大,重复序列多,基因组杂合度高,常多倍体化,起源、进化和分类复杂,保护、鉴定、开发和利用难度较大[3-4]。茶树大面白(C. sinensis cv. ‘Damianbai)是江西省上饶市广信区上沪公社红区林场于1968—1984年从广信区上沪乡洪水坑群体茶园通过系统选育而成,1985年全国农作物品种审定委员会认定为国家品种(编号:GS13018-1985)[5-7]。大面白茶树一般发芽早(3月上旬),开采早(4月上中旬),育芽强,芽肥壮,持嫩性强,夏梢旺盛,新梢轮次多,叶背密生白色茸毛,适合制作绿茶、红茶和乌龙茶,如上饶白眉和仙台大白等[8]。大面白扦插繁殖能力较强,河南、湖南、安徽、广东等省份均有引种[9]。但对于大面白的进化和发展历程,以及与其他茶树品种之间的进化关系尚不清晰。
叶绿体是绿色植物进行代谢活动的活性中心,也是植物进行光合作用和次生代谢物合成的重要场所[10]。植物的绿色组织中一般均具有叶绿体,叶绿体是植物细胞内最重要、最普遍的质体,也是三大遗传物质载体之一,更是细胞内独立且具有半自主性的细胞器之一[11]。叶绿体内独立的遗传物质——叶绿体基因组,为100~250 kb的双链环状DNA,可编码120~130个基因,结构较为简单,普遍母系遗传,与光合作用、转录和翻译等相关,目前引起了学者的广泛关注[12]。叶绿体基因组一般具有四分体结构,由大单拷贝区(Large single copy,LSC)、小单拷贝区(Small single copy,SSC)以及2个反向重复区(Inverted repeat,IRa和IRb)组成,规模小,拷贝数多,结构稳定,序列高度保守,遗传重组率低,已被广泛应用于植物的DNA条形码开发、物种鉴定、物种演化和系统发育等[13]。植物叶绿体基因组进化的一个重要特征是其密码子的偏好性[14]。叶绿体基因组中编码同一种氨基酸的不同密码子视为同义密码子,植物种类的叶绿体基因组不同,或同一叶绿体基因组的基因不同,同义密码子的使用频率存在差异,这种使用同义密码子不均衡的现象被称为同义密码子使用偏好性[15]。研究叶绿体基因组密码子的偏好性,有助于探究植物分子进化的效率以及外源蛋白质表达的效率[16]。影响叶绿体基因组密码子偏好性的因素主要有环境选择、碱基突变、基因漂变等[17]。叶绿体基因工程已经成为植物基因工程十分重要的研究领域,目前广泛应用于基因工程、分子标记开发以及系统发育分析等方面[18]。
目前,有关茶树叶绿体基因组结构特征、系统发育以及密码子偏好性方面的研究多有报道。赵许朋等[19]研究表明,贵定鸟王茶叶绿体基因组全长157 071 bp,共注释基因138个,与茶亲缘关系较近,其中与信阳10号的亲缘关系最近;佟岩等[20]研究表明,大理茶(C. taliensis)叶绿体密码子偏好性的主要因素是自然选择,与秃房茶(C. gymnogyna)聚为一支,是栽培茶的近缘种;黎巷汝等[21]研究表明,6个武夷名丛品种的叶绿体基因组的长度为157 024~157 126 bp,6个武夷名丛品种并未聚为一支,其中大红袍和肉桂与龙井43的分支较近,半天妖与安化茶聚为一支,水金龟与铁观音聚为一支,白鸡冠和铁罗汉分别聚成单个分支;刘振等[22]研究表明,三倍体茶树品种西莲1号基因组全长为157 038 bp,西莲1号与其他栽培型茶树品种(C. sinensis var. sinensis)处于系统发育树的末端,属于进化的茶树资源类型;闫明慧等[4]研究表明,茶树信阳10号叶绿体基因组大小为157 041 bp,与福建铁罗汉关系最近,推测可能来源于相同母本,与韩国茶Chamnok和Sangmok、福建白鸡冠、云南德宏茶的亲缘关系也较近;叶晓倩等[23]的研究表明,龙井43叶绿体基因组大小为157 085 bp,龙井瓜子、龙井长叶、龙井圆叶和中茶102聚为一支,自展支持率为100%,叶绿体基因片段可有效区分茶树品种间的亲缘关系;Li等[1]的研究表明,全球易危物种、我国特有野生山茶资源大苞山茶(C. granthamiana)叶绿体总基因组大小为157 001 bp,共鉴定出131个基因,与茶树(C. sinensis)聚为一支,相似值为100%;Li等[24]的研究表明,大红袍茶树叶绿体总基因组大小为157 077 bp,共鉴定出137个基因,与茶树龙井(C. sinensis cv. ‘Longjing)聚为一支;Peng等[25]对栽培型和野生型茶树进行了比较和进化分析,发现栽培型茶树的叶绿体基因组大小为157 025~157 100 bp,栽培型比野生型茶树更保守。栽培型茶树的核苷酸多样性高于野生型,变化最大的基因为ycf1,ycf1在栽培型茶树中已经分化。但关于茶树大面白的叶绿体基因组组装、注释、基因组特征、系统发育以及密码子偏好性方面的研究尚未见报道。
本研究利用高通量测序技术对大面白茶树进行叶绿体全基因组测序组装,对叶绿体基因组图谱和序列特征进行表征,分析大面白叶绿体基因组密码子使用的偏好性,探究茶树大面白与其近缘物种的IR边界差异,明确其在山茶属植物系统发育中的位置。本研究不仅可为大面白茶树物种鉴定、遗传多样性分析以及分子育种等研究提供理论基础,也可为山茶属植物的系统发育研究提供更丰富的叶绿体基因组数据。
1 材料与方法
1.1 材料
大面白茶树(代号:DMB)地栽苗由江西茗龙实业集团有限公司提供。
1.2 方法
1.2.1 DNA提取和测序
大面白茶树叶片的总DNA提取、DNA纯度的检测、DNA文库的初步定量、DNA文库插入片段的检测、DNA文库有效浓度的准确定量以及DNA文库的测序分别采用改良的CTAB法[26]、NanoDrop 2000分光光度计(美国,Thermo Scientific公司)法、美国Invitrogen Qubit? 2.0荧光定量仪法、Agilent 2100生物分析仪系统法、实时荧光定量PCR (Real-time quantitative PCR,RT-qPCR)法和DNBSEQ-T7测序仪平台(华大智造)法(广州佰数生物科技有限公司,Bio&Data Biotechnologies)。
1.2.2 叶绿体全基因组的组装与注释
利用Fastp v.0.20.1软件将大面白茶树叶片DNA文库原始数据进行过滤,去除低质量数据,即可获得Clean Data。大面白茶树叶绿体基因组的组装采用Noveplastys软件[27],利用GenBank上全部山茶属的完整叶绿体作为种子序列,过滤掉非叶绿体基因组序列,K-mer设置为49、59和69(类似两个序列之间的overlap)进行组装,组装结果表明序列一致且自动环化,说明组装结果没有受到核基因组污染[28]。茶树大面白叶绿体基因组图谱的制作采用GeSeq[29]、tRNAscan-SE[30]和OGDRAW软件[31]。大面白叶绿体基因组序列提交至NCBI(https://www.ncbi.nlm.nih.gov),登录号为PP024603。
1.2.3 叶绿体基因组特征分析
大面白叶绿体基因组LSC、SSC、IRa和IRb的GC含量的分析和统计、SSR分析、Longrepeat分析、同义密码子相对使用度(Relative synonymous codon usage,RSCU)的计算和分析分别采用CGViewServer软件[32]、MISA(MIcroSAtellite identification tool)软件[33]、REPuter软件[34]、CodonW软件[35]。茶树大面白及其16个近缘种(表1序号1~17)叶绿体基因组变异圈图的绘制和序列相似性的计算均采用Gview软件[36];叶绿体基因组IR结构变异的绘图以及核苷酸多态性(Nucleotide polymorphism,Pi)的计算分别采用IRscope软件[37]和NADnaSP 6.0软件[38];茶树大面白及其51个山茶科山茶属(Camellia)近缘种和山茶科木荷属(Schima)2个外群种(表1)叶绿体基因组的序列比对和建树分别采用MAFFT 7.0软件[39]和FastTree 2.1.10软件[40]。近缘种选取的原则:进化树采用从NCBI中能够获得的全部山茶属51个物种叶绿体序列进行分析,其他特征比较则从全部山茶属51个物种中不同进化树分支上选择16个(含大面白共17个)代表序列进行分析。
1.2.4 叶绿体基因组密码子使用偏好性分析
GC3-GC12(Neutrality-plot)、ENC-plot、PR2-plot和最优密码子参考佟岩等[20]的方法进行分析。
2 结果与分析
2.1 大面白茶树叶绿体基因组的质控数据
利用Fastp v.0.20.1软件可将茶树大面白叶片DNA文库Raw Data原始数据进行过滤,去除低质量Reads(表2)。由表2可知,经过过滤后,可获得茶树大面白叶片DNA文库高质量Clean Data。
2.2 大面白茶树叶绿体基因组的基本结构
大面白茶树叶绿体基因组全长157 129 bp,结构呈现为双链环状分子,为典型的四分体结构,包括1个LSC区(86 687 bp)、1个SSC
区(18 282 bp)和2个IR区(包括IRa和IRb,均为26 080 bp)(图1)。茶树大面白叶绿体基因组平均GC含量为37.29%,其中IR区的GC含量(42.96%)最高,其次是LSC区(35.30%),GC含量最低的是SSC区(30.53%)。
2.3 茶树大面白叶绿体基因组的基因组成
茶树大面白叶绿体基因组共注释到135个功能基因(表3),包括90个编码蛋白(Coding sequence,CDS)基因,8个核糖体RNA(rRNA)基因和37个转运RNA(tRNA)基因。trnK-UUU、trnI-GAU(2个拷贝)、
trnA-UGC(2个拷贝)、trnG-UCC、trnV-UAC、trnL-UAA、rpoC1、ndhB(2个拷贝)、ndhA、rpl2(2个拷贝)、rpl16、petB、atpF、petD、rps16、rps12(2个拷贝)基因具有2个外显子;rps12(2个拷贝)、clpP1、ycf3(pafI)基因具有3个外显子;ycf3(pafI)和clpP1基因具有2个内含子;trnK-UUU、ndhA、rpl16、trnI-GAU(2个拷贝)、rps16、trnA-UGC(2个拷贝)、clpP1、petB、rpoC1、petD、atpF、trnG-UCC、ndhB(2个拷贝)、rpl2(2个拷贝)、trnV-UAC、rps12(2个拷贝)、trnL-UAA基因具有1个内含子;trnK-UUU内含子最大(2 488 bp),trnL-UAA内含子最小(525 bp);有83个基因(23个tRNA基因和60个CDS基因)完全在LSC区;有12个基因(1个tRNA基因和11个CDS基因)完全在SSC区;有18个基因(4个rRNA基因,7个tRNA基因和7个CDS基因)完全在IRB和IRA区,7个CDS基因为ycf2、ndhB、rpl2、rps7、rpl23、ycf15(2个拷贝);有1个CDS基因(ycf1)在SSC-IRB连接处;有1个CDS基因(rps19)在LSC-IRB连接处;有1个CDS基因(ycf1)在SSC-IRA连接处。
2.4 茶树大面白叶绿体基因组SSR和Longrepeat分析
茶树大面白叶绿体基因组共检测到52个SSR(表4),只包括单核苷酸重复序列,其中23个为A重复(44.23%),29个为T重复(55.77%)。
茶树大面白16个近缘种叶绿体基因组(MN086819、MZ817088、MT773373、MT773375、OL690374、OL690348、OL690347、OL690350、OL690357、OL690369、OL690351、OL690370、OL690366、OL690360、KF562708、OL840043)的SSR数量分别为51、51、53、52、53、54、54、54、54、52、52、52、51、53、56、56,SSR数量平均为53,也以A/T单核苷酸重复序列为主,说明茶树大面白及其16个近缘种叶绿体基因组SSR偏好A/T单核苷酸重复。茶树大面白叶绿体基因组共检测到50个Longrepeat(表5),包括分散重复(Dispersed repeats,D)和回文重复(Palindromic repeats,P),其中分散重复22个(30~82 bp),回文重复28个(30~26 080 bp)。分散重复只包括正向重复(Forward repeats,F)、反向重复(Reverse repeats,R)和互补重复(Complement repeats,C)3种类型。茶树大面白叶绿体基因组分散重复只有正向重复一种类型。
2.5 叶绿体基因组比对分析
茶树大面白及其16个近缘种叶绿体基因组的变异圈图(图2)、mVIST结构变异图(图
3)和Pi多样性指数分析图(图4)表明,茶树大面白及其16个近缘种的叶绿体基因组序列高度保守,但LSC和SSC区的基因还是存在较大的序列差异。由图4可知,茶树大面白叶绿体基因组核苷酸多样性的变化范围为0~0.003 06;茶树大面白叶绿体基因组非编码区的变异程度高于基因编码区,4个区的变异性从高到低依次为LSC区>SSC区>IR区,其中IR区最为保守;通过Pi(≥0.001 38)筛选出10个高变异区域,均位于LSC和SSC区,LSC区有9个高变异区域:matK、trnK-UUU、matK_rps16、psbZ_trnG-GCC、trnG-GCC、trnG-GCC_trnfM-CAU、trnF-GAA、rbcL_accD、accD;SSC区有1个高变异区域:ycf1-2。
2.6 叶绿体基因组的SC/IR边界分析
茶树大面白及其16个近缘种叶绿体基因组四分体结构的SC/IR边界收缩扩张情况(图5)表明,茶树大面白及其16个近缘种叶绿体基因组的4个边界较为保守。LSC/IRb(JLB)边界均位于rps19基因内,rps19基因在IRb区内长为46 bp,在LSC区内长为233 bp;IRb/SSC(JSB)边界均位于ycf1基因内,ycf1基因在SSC区内长为2 bp,在IRb区内长为1 071 bp。SSC/IRa(JSA)边界均位于ycf1基因内,ycf1基因在SSC区长为4 545~4 553 bp,ycf1基因在IRa区长为1 069 bp。IRa/LSC(JLA)边界均位于IRa区的rpl2基因和LSC区的trnH基因之间,trnH基因距离IRb/SSC(JLA)边界均为1 bp。
2.7 茶树大面白叶绿体基因组密码子使用偏性分析
2.7.1 同义密码子的偏性分析
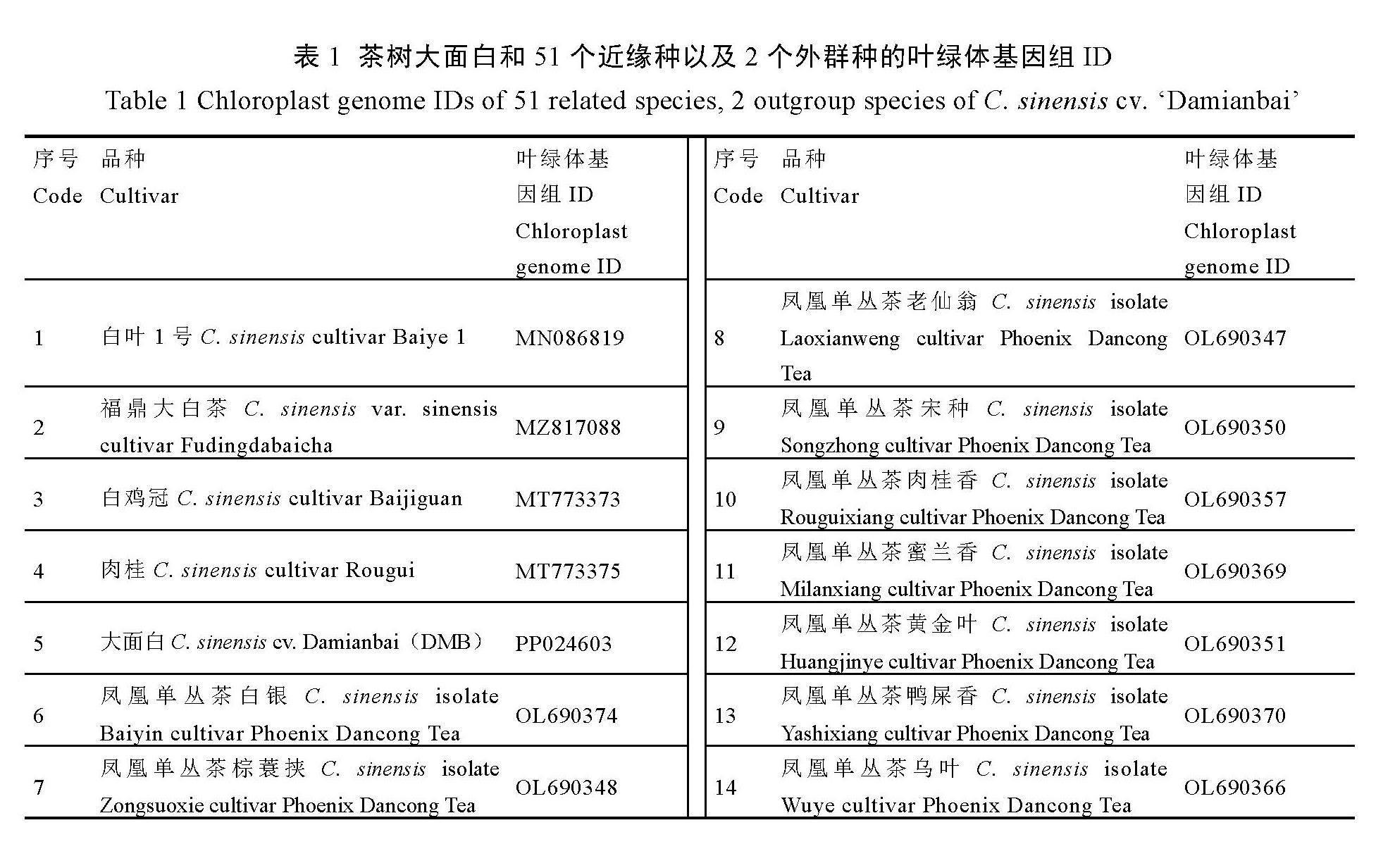
茶树大面白及其16个近缘种叶绿体基因组CDS基因密码子3个位置GC含量的平均值为37.90%,GC1、GC2、GC3的平均值分别为45.84%、39.41%、28.44%,3个位置GC含量的变化趋势为GC3<GC2<GC1(图6);茶树大面白叶绿体基因组90个CDS基因密码子的ENC值介于24.103~61.000,平均值为46.46,87个基因的ENC值>35,3个基因的ENC值<35,密码子偏性较弱(图7);茶树大面白16个近缘种叶绿体基因组CDS基因密码子的ENC值也介于24.103~61.000,平均值为46.50,1 392个基因的ENC值>35,48个基因的ENC值<35,密码子偏性同样较弱(图7)。茶树大面白叶绿体基因组90个CDS基因序列共有31个RSCU>1的密码子,在这31个密码子中,除UUG、AUG外,其余都以A、U结尾;茶树大面白16个近缘种叶绿体基因组CDS基因序列共有496个RSCU>1的密码子,在这496个密码子中,除UUG、AUG外,其余也均以A、U结尾,密码子同样偏好以A、U结尾。
2.7.2 中性绘图分析(GC3-GC12分析)
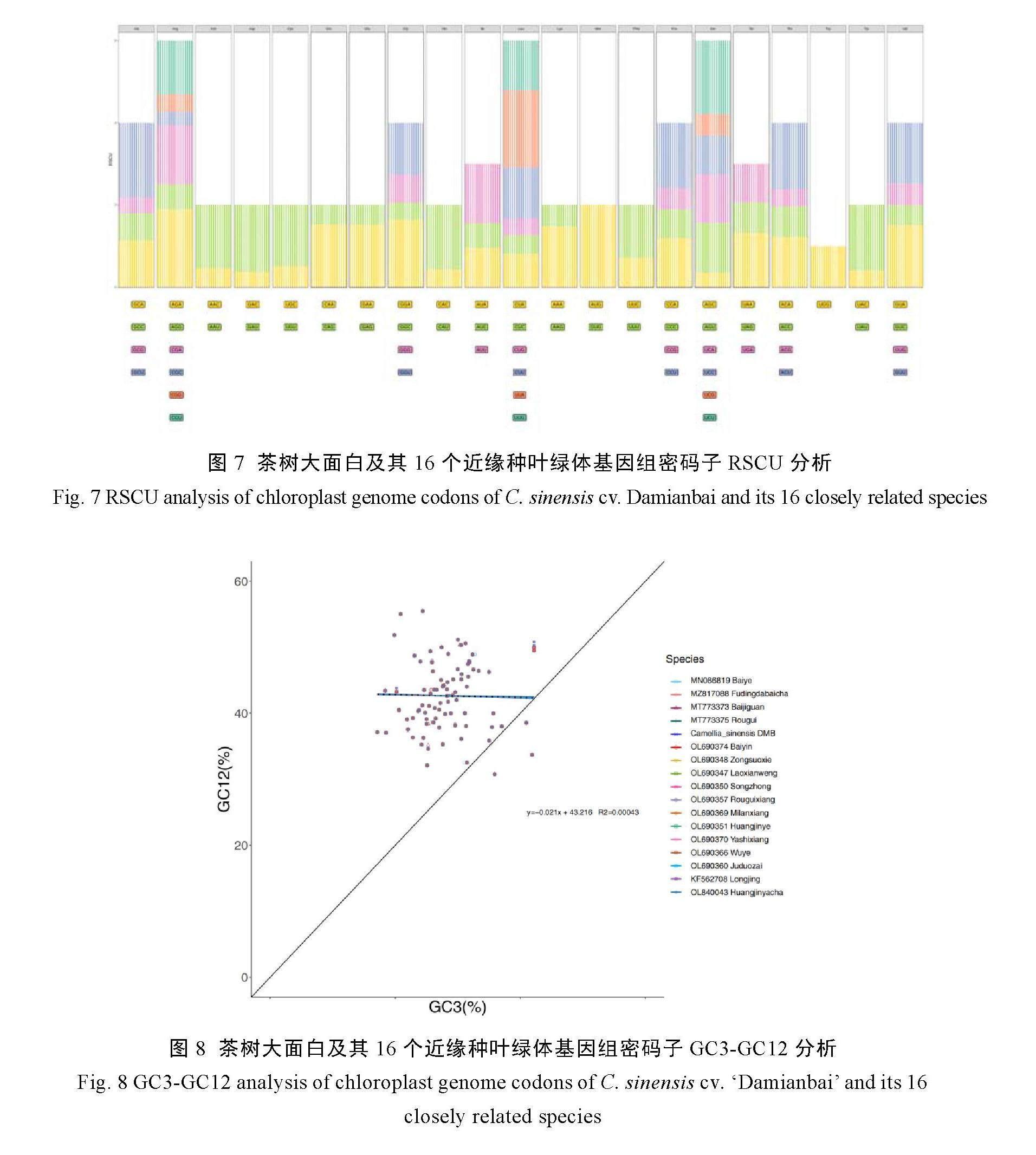
大面白茶树及其16个近缘种叶绿体基因的GC3含量在17.14%~42.19%,GC12含量在24.70%~62.50%,二者绝大多数沿对角线上方分布(图8)。两者的相关系数r=0.020 784 6(R2=0.000 432),说明GC12与GC3显著相关。回归系数为0.021,内部突变贡献率仅2.1%,自然选择贡献率为97.9%,表明茶树大面白及其16个近缘种叶绿体基因组密码子使用偏性主要受自然选择的影响,而受内部突变压力的影响小。
2.7.3 ENC-plot分析
茶树大面白及其16个近缘种叶绿体基因组大部分基因的有效密码子数(ENC)大于35,主要集中在40~55,大部分基因位于标准曲线的下方(图9),表明大部分基因ENC的实际值与预期值存在较大差异,且GC3值分布较为集中,可见自然选择是影响茶树大面白叶绿体基因组密码子使用偏性的重要原因,密码子使用偏性受突变的影响较小。
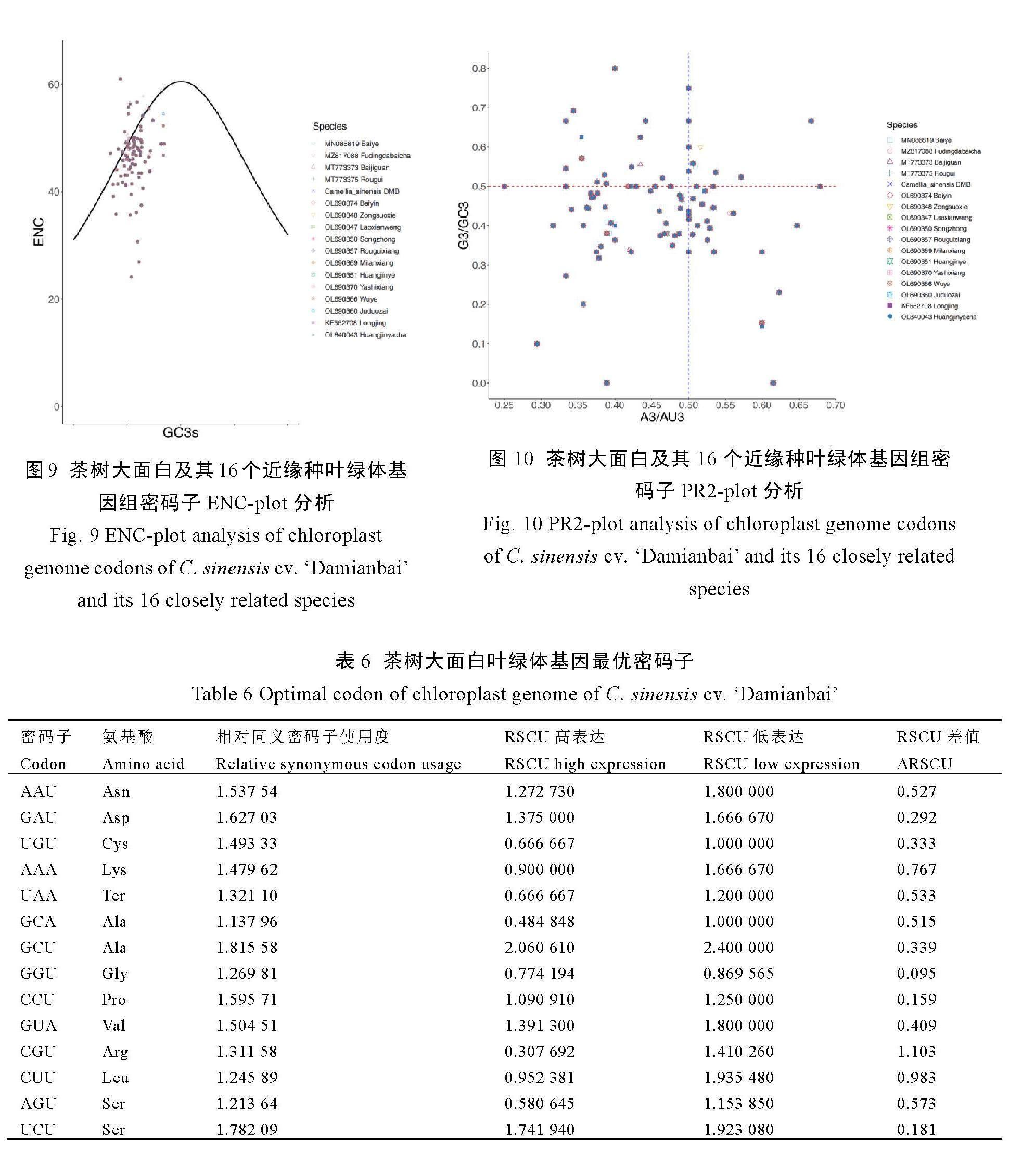
2.7.4 PR2-plot分析
从G3/GC3轴看,茶树大面白及其16个近缘种叶绿体基因组中较多的基因分布于PR2-plot图的下部分(图10),说明4种碱基在同义密码子第3位上存在C>G现象。从A3/AU3轴看,茶树大面白及其16个近缘种叶绿体基因组中较多的基因分布于PR2-plot图的左部分(图10),说明4种碱基在同义密码子第3位上存在T>A现象。如密码子使用存在偏性只受内部突变压力影响时,C和G以及A和T在第3位上的分布应相等,说明茶树大面白及其16个近缘种叶绿体基因组密码子使用偏好性同时受突变和自然选择的影响,且受自然选择影响更大。
2.7.5 最优密码子确定
茶树大面白叶绿体基因满足相对同义密码子使用度(Relative synonymous codon usage,RSCU)>1(高频率密码子)且ΔRSCU(=RSCU高表达-RSCU低表达)≥0.08的最优密码子有AAU、GAU、UGU、AAA、UAA、GCA、GCU、GGU、CCU、GUA、CGU、CUU、AGU、UCU(表6),共14个,均以A、U结尾。说明茶树大面白叶绿体基因组密码子偏好性是以A和U结尾。
2.8 系统发育分析
基于大面白茶树及其51个山茶科山茶属(Camellia)近缘种和山茶科木荷属(Schima)2个外群种叶绿体基因组构建的系统发育树(图11)表明,茶树大面白[DMB,Camellia sinensis cv. Damianbai]与凤凰单丛茶白银C. sinensis isolate Baiyin cultivar Phoenix Dancong Tea(OL690374)、凤凰单丛茶棕蓑挟C. sinensis isolate Zongsuoxie cultivar Phoenix Dancong Tea(OL690348)、凤凰单丛茶老仙翁C. sinensis isolate Laoxianweng cultivar Phoenix Dancong Tea(OL690347)、凤凰单丛茶宋种C. sinensis isolate Songzhong cultivar Phoenix Dancong Tea(OL690350)、凤凰单丛茶肉桂香C. sinensis isolate Rouguixiang cultivar Phoenix Dancong Tea(OL690357)单独聚为一分支。在这个分支中,茶树大面白[DMB,Camellia sinensis cv. Damianbai]又与凤凰单丛茶白银C. sinensis isolate Baiyin cultivar Phoenix Dancong Tea(OL690374)单独成一小分支。说明茶树大面白[DMB,Camellia sinensis cv. Damianbai]与凤凰单丛茶白银C. sinensis isolate Baiyin cultivar Phoenix Dancong Tea(OL690374)亲缘关系较近。
3 讨论
植物叶绿体基因组一般有4个区,包括LSC、SSC、IRa和IRb,大面白茶树叶绿体基因组的结构与绝大多数植物叶绿体基因组的结构一样,呈闭合环状双链结构,具有典型的四分体结构[41]。本研究对大面白茶树及其16个近缘种的叶绿体基因组进行了比较。结果表明,大面白及其16个近缘种之间叶绿体基因组高度相似,基因组长度为156 691~157 100 bp(大面白茶树茶树最长,为157 129 bp),总GC含量均为37.90%,IR区的GC含量高于LSC区和SSC区,IR区具有4个rRNA(rrn16、rrn23、rrn4.5、rrn5)基因,这可能是导致IR区GC含量较高的原因[42]。研究表明,叶绿体基因组中最保守的区域是IR区,IR边界的收缩与扩张决定着植物叶绿体基因组进化过程中的叶绿体基因组长度[43],也可反映物种在进化过程中的关系[44]。本研究对茶树大面白及其16个近缘种的叶绿体基因组进行了详细比较,发现非编码区的变异程度高于基因编码区,4个区的变异性从高到低依次为LSC区>SSC区>IR区,其中IR区是最为保守的区域,IR区的收缩或扩张对基因组长度影响较小。因此,推测茶树大面白及其16个近缘种叶绿体基因组大小的差异可能是由LSC区和SSC区的基因间隔区的插入或缺失所引起的[45]。
研究表明,茶树叶绿体基因组一般可编码97~141个基因,其中有60~100个CDS基因、24~47个tRNA基因和8个rRNA基因。大多CDS基因只含1个内含子,但cloP、clpP和ycf3等基因含2个内含子[46]。本研究表明,茶树大面白叶绿体基因组的ycf3(pafI)和clpP1基因具有2个内含子,ycf3(pafI)是光系统Ⅰ复合物稳定积累所必需的,茶树大面白的ycf3(pafI)内含子增益可能有助于诠释光合作用的演化机制[2];茶树大面白叶绿体基因组的ycf15基因具有4个拷贝,武夷名丛叶绿体基因组的ycf15基因也具有4个拷贝[21],而贵定鸟王茶[19]和三倍体茶树西莲1号[22]叶绿体基因组的ycf15基因只有2个拷贝,信阳10号[4]、印度阿萨姆茶和云抗10号[25]叶绿体基因组无ycf15基因。可见,山茶属植物叶绿体基因ycf15虽有完整的编码框,但转录后无任何功能,推测叶绿体DNA转录后加工可能涉及非功能基因的复杂剪接。
植物基因组的重要组成部分是重复序列[19]。叶绿体基因组中的SSR复制率高,遗传共显性,多态性高,母系遗传,广泛应用于植物基因表达、遗传调控、物种鉴定及群体遗传多态性等方面[47]。本研究对茶树大面白及其16个近缘种叶绿体基因组的SSR进行比较分析表明,茶树大面白及其16个近缘种的SSR类型(主要为A/T)和数目(51~56)基本一致,进一步证明了植物叶绿体基因组一般由短polyA或polyT重复构成SSR位点、且SSR位点很少具有C或G串联重复的观点。叶绿体基因组除了SSR,还有Longrepeat。Huang等[48]发现山茶属存在正向重复序列、回文序列及反向重复序列共3种Longrepeat类型,赵许朋等[19]发现贵定鸟王茶叶绿体基因组Longrepeat只存在正向重复和回文重复2种类型树。本研究中,茶树大面白叶绿体基因组共检测到50个Longrepeat,包括分散重复D(30~82 bp)22个和回文重复P(30~26 080 bp)28个,其中分散重复D只包括正向重复,茶树大面白叶绿体基因组Longrepeat也只存在正向重复和回文重复2种类型,与贵定鸟王茶的研究结果一致。说明茶树大面白和贵定鸟王茶的Longrepeat重复类型丰富度略低于其他茶树品种。Longrepeat重组活性较强,可以介导叶绿体基因组的可逆重组,调节叶绿体基因组的分子构象,使其结构呈现多样性,导致高等植物叶绿体基因组不断扩大和复杂化,从而推动了高等植物的不断进化[19]。茶树大面白和贵定鸟王茶由于包含较少数量的长重复序列,其叶绿体基因组更小且不易形成多环结构,导致其叶绿体基因组在进化过程中的重组频率较低[48]。因此,与其他茶树品种相比,茶树大面白和贵定鸟王茶Longrepeat重复类型丰富度略低,表明茶树大面白和贵定鸟王茶这2个地方茶树品种的进化水平较低。
遗传密码是识别和传递生物体遗传信息的载体,在生物遗传和变异中起着重要作用[49]。在进化过程中植物叶绿体基因组密码子会通过自然选择、基因突变、蛋白质结构变化等手段产生一定的偏好性,影响密码子偏好性的因素一般有基因的GC含量、长度、翻译效率、表达水平及tRNA丰度等[20]。分析植物叶绿体基因组密码子的偏好性对于研究叶绿体基因工程、物种间的亲缘关系及物种的进化具有十分重要的作用[50]。茶树大面白中14个最优密码子大部分以A/U结尾,与大理茶(C. taliensis)[20]的研究结果一致。ENC-plot、GC3-GC12和PR2-plot等分析结果表明,茶树大面白叶绿体基因密码子偏性主要受自然选择的影响,受内部碱基突变的影响较少,这也与大理茶(C. taliensis)[20]的研究结果一致。茶树大面白的野生种、半野生种和栽培种的生长环境较为稳定,上饶广信区栽培和管理模式日趋生态化,茶树大面白叶绿体基因组的密码子偏好性受人工选择、基因突变等影响较小,其叶绿体基因组的密码子偏性较弱。如长期经过人工栽培驯化,叶绿体基因组的密码子偏好性可由基因突变和自然选择共同发挥作用。
山茶属植物为异花授粉,种间甚至属间杂交十分普遍,再加上自然杂交和人工选育,山茶属植物的种间界限不是很清晰,传统的形态解剖学依据很难明确山茶属植物的分类[20],植物的叶绿体基因组可用于山茶属植物的系统发育及其物种鉴定等[51]。本研究结果显示,山茶属物种聚为1支,木荷属2个物种单独聚为1支,该结果与Rawal等[52]的研究结果一致,表明叶绿体基因组序列能够作为区分山茶属与其他属亲缘关系的分类依据。为了进一步揭示茶树大面白在山茶属种间的亲缘关系,本研究选取了茶树大面白及其51个山茶科山茶属(Camellia)近缘种和山茶科木荷属(Schima)2个外群种,利用FastTree软件GTR模型(Generalized time-reversible model)构建ML系统发育树。结果表明,茶树大面白[DMB,Camellia sinensis cv. Damianbai]与凤凰单丛茶白银C. sinensis isolate Baiyin cultivar Phoenix Dancong Tea(OL690374)、凤凰单丛茶棕蓑挟C. sinensis isolate Zongsuoxie cultivar Phoenix Dancong Tea(OL690348)、凤凰单丛茶老仙翁C. sinensis isolate Laoxianweng cultivar Phoenix Dancong Tea(OL690347)、凤凰单丛茶宋种C. sinensis isolate Songzhong cultivar Phoenix Dancong Tea(OL690350)、凤凰单丛茶肉桂香C. sinensis isolate Rouguixiang cultivar Phoenix Dancong Tea(OL690357)单独聚为一分支。在这个分支中,茶树大面白[DMB,Camellia sinensis cv. Damianbai]又与凤凰单丛茶白银[C. sinensis isolate Baiyin cultivar Phoenix Dancong Tea(OL690374)]单独成一小分支。说明茶树大面白[DMB,Camellia sinensis cv. Damianbai]与凤凰单丛茶白银[C. sinensis isolated Baiyin cultivar Phoenix Dancong Tea(OL690374)]亲缘关系较近。
综上所述,大面白茶树叶绿体基因组全长157 129 bp,为典型的四分体结构,包括1个LSC区(86 687 bp)、1个SSC区(18 282 bp)和2个IR区(包括IRa和IRb,均为26 080 bp)。茶树大面白叶绿体基因组共注释到135个功能基因,包括90个CDS基因,8个rRNA基因和37个tRNA基因。大面白茶树叶绿体基因组共检测到52个SSR和50个Longrepeat,SSR只有A/T单核苷酸重复序列,Longrepeat只存在正向重复和回文重复2种类型。大面白茶树叶绿体基因组密码子使用偏性主要受自然选择的影响,而受内部突变压力的影响小。大面白茶树叶绿体基因具有14个最优密码子(AAU、GAU、UGU、AAA、UAA、GCA、GCU、GGU、CCU、GUA、CGU、CUU、AGU、UCU)。茶树大面白(C. sinensis cv. Damianbai)与凤凰单丛茶白银C. sinensis isolate Baiyin cultivar Phoenix Dancong Tea(OL690374)亲缘关系较近。本研究结果揭示了大面白茶树叶绿体基因结构及与其他茶树进化关系,可为大面白茶树遗传多样性、种群历史动态以及其系统发育与亲缘关系研究奠定理论基础。随着基因组测序的发展,结合基因组、细胞器基因组深入研究茶树大面白密码子使用规律,并结合形态、细胞、化学等研究共同揭示大面白茶树的起源、进化,以及茶树大面白如何参与山茶属种间的起源进化也是后续研究的重要方向之一。
参考文献
79Li W X, Shi X G, Guo W X, et al. Characterization of the complete chloroplast genome of Camellia granthamiana (Theaceae), a Vulnerable species endemic to China [J]. Mitochondrial DNA B Resource, 2018, 3(2): 1139-1140.
80杨雨青, 谭娟, 汪芳, 等. 茶树叶绿体基因组的研究与应用进展[J]. 生物技术通报, 2024, 40(2): 1-11.
Yang Y Q, Tan J, Wang F, et al. Research and application progress in chloroplast genome of tea plant (Camellia sinensis) [J]. Biotechnology Bulletin, 2024, 40(2): 1-11.
81Xia E H, Tong W, Wu Q, et al. Tea plant genomics: achievements, challenges and perspectives [J]. Horticulture Research, 2020, 7(1): 7. doi: 10.1038/s41438-019-0225-4.
82闫明慧, 刘柯, 王满, 等. 信阳10号叶绿体基因组及其系统进化[J]. 茶叶科学, 2021, 41(6): 777-788.
Yan M H, Liu K, Wang M, et al. Complete chloroplast genome of Camellia sinensis cv. Xinyang 10 and its phylogenetic evolution [J]. Journal of Tea Science, 2021, 41(6): 777-788.
83王士圻. 名茶新魁——上饶白眉[J]. 蚕桑茶叶通讯, 1986(1): 31-32.
Wang S Q. Famous tea new leader: Shangrao white eyebrow [J]. Newsletter of Sericulture and Tea, 1986(1): 31-32.
84王士圻. 上饶“白眉”炒制技术要点[J]. 茶业通报, 1983(6): 44.
Wang S Q. Key points of frying techniques for Shangrao white eyebrows [J]. Journal of Tea Business, 1983(6): 44.
85黄奋文, 黄积安. 福丁大毫、上饶大面白不同行距种植对茶叶产量的影响[J]. 蚕桑茶叶通讯, 1988(4): 4-7.
Huang F W, Huang J A. The effect of different row spacing planting on tea yield in Fuding Damao and Shangrao Damanbai [J]. Newsletter of Sericulture and Tea, 1988(4): 4-7.
86王士圻. 茶树良种——大面白的选育及其鉴定报告[J]. 茶业通报, 1981(5): 40-45.
Wang S Q. Breeding and identification report of a good tea tree variety: Damianbai [J]. Journal of Tea Business, 1981(5): 40-45.
87王士圻.茶树良种——上饶大面白[J].蚕桑茶叶通讯, 1978(2): 4-5.
Wang S Q. Excellent tea tree variety: Shangrao Damianbai [J]. Newsletter of Sericulture and Tea, 1978(2): 4-5.
88Cao P H, Wang D, Gao S, et al. OsDXR interacts with OsMORF1 to regulate chloroplast development and the RNA editing of chloroplast genes in rice [J]. Journal of Integrative Agriculture, 2023, 22(3): 669-678.
89Zhang G L, Feng C, Kou J, et al. Phylogeny and divergence time estimation of the genus Didymodon (Pottiaceae) based on nuclear and chloroplast markers [J]. Journal of Systematics and Evolution, 2023, 61(1): 115-126.
90Park T. Complete chloroplast genome sequence of Solanum iopetalum, one of the tuber-bearing wild potato relatives [J]. Mitochondrial DNA Part B, 2023, 8: 347-351.
91Li B Z, Li Y, Li Z F, et al. The complete chloroplast genome of Impatiens mengtszeana (Balsaminaceae), an endemic species in China [J]. Mitochondrial DNA Part B, 2022, 7(2): 367-369.
92Bourret J, Borvet? F, Bravo I G. Subfunctionalisation of paralogous genes and evolution of differential codon usage preferences: the showcase of polypyrimidine tract binding proteins [J]. Journal of Evolutionary Biology, 2023, 36(10): 1375-1392.
93Tan F, Banerjee A K, Deng J, et al. Characterization of the complete chloroplast genome of Firmiana hainanensis (Malvaceae), an endemic and vulnerable tree species of China [J]. Mitochondrial DNA Part B, 2023, 8(1): 57-60.
94Singhal S, Kumar U, Alqahtani T, et al. An insight into codon pattern analysis of autophagy genes associated with virus infection [J]. Current Pharmaceutical Design, 2023, 29(14): 1105-1120.
95Chu D, Wei L. Characterizing the heat response of Arabidopsis thaliana from the perspective of codon usage bias and translational regulation [J]. Journal of Plant Physiology, 2019, 240: 153012. doi: 10.1016/j.jplph.2019.
153012.
96Zhu Y A, Wang S, Xie J, et al. The complete chloroplast genome of Rubus ellipticus var. obcordatus, an edible and medicinal dual-purpose plant [J]. Mitochondrial DNA Part B, 2022, 7(2): 406-408.
97赵许朋, 崔奎, 耿苗苗, 等. 贵定鸟王茶的叶绿体基因组特征[J]. 西南农业学报, 2023, 36(11): 2348-2357.
Zhao X P, Cui K, Geng M M, et al. Chloroplast genome characteristics of Guiding Niaowang tea [J]. Southwest China Journal of Agricultural Sciences, 2023, 36(11): 2348-2357.
98佟岩, 黄荟, 王雨华. 森林茶园古茶树大理茶叶绿体基因组密码子偏好性及系统发育研究[J]. 茶叶科学, 2023, 43(3): 297-309.
Tong Y, Huang H, Wang Y H. Analysis of codon usage bias and phylogenesis in the chloroplast genome of ancient tea tree Camellia taliensis in forest-tea garden [J]. Journal of Tea Science, 2023, 43(3): 297-309.
99黎巷汝, 赵雅琦, 张艳, 等. 武夷名丛叶绿体基因组序列特征及系统发育分析[J]. 南方农业学报, 2023, 54(5): 1352-1362.
Li X R, Zhao Y Q, Zhang Y, et al. Chloroplast genome sequence features and phylogenetic analysis of Wuyi Mingcong [J]. Journal of Southern Agriculture, 2023, 54(5): 1352-1362.
100刘振, 赵洋, 杨培迪, 等. 三倍体茶树‘西莲1号叶绿体基因组特征及系统发育分析[J]. 茶叶通讯, 2023, 50(2): 166-175.
Liu Z, Zhao Y, Yang P D, et al. Characterization and phylogenetic analysis of the complete chloroplast genome of triploid tea plant Xilian 1 [J]. Journal of Tea Communicatio, 2023, 50(2): 166-175.
101叶晓倩, 赵忠辉, 朱全武, 等. 茶树‘龙井43叶绿体基因组测序及其系统进化[J]. 浙江大学学报(农业与生命科学版), 2014, 40(4): 404-412.
Ye X Q, Zhao Z H, Zhu Q W, et al. Entire chloroplast genome sequence of tea (Camellia sinensis cv.Longjing 43): a molecular phylogenetic analysis [J]. Journal of Zhejiang University (Agriculture & Life Sciences), 2014, 40(4): 404-412.
102Li L, Hu Y F, Wu L , et al. The complete chloroplast genome sequence of Camellia sinensis cv. Dahongpao: a most famous variety of Wuyi tea (Synonym: Thea bohea L.) [J]. Mitochondrial DNA Part B, 2021, 6(1): 3-5.
103Peng J, Zhao Y L, Dong M, et al. Exploring the evolutionary characteristics between cultivated tea and its wild relatives using complete chloroplast genomes [J]. BMC Ecology and Evolution, 2021, 21(1): 71. doi: 10.1186/s12862-021-01800-1.
104Doyle J J. A rapid DNA isolation procedure for small quantities of fresh leaf tissue [J]. Phytochem Bull, 1987, 19(1): 11-15.
105Dierckxsens N, Mardulyn P, Smits G. NOVOPlasty: de novo assembly of organelle genomes from whole genome data [J]. Nucleic Acids Research, 2016, 45(4): e18. doi: 10.1093/nar/gkw955.
106Chen S F, Zhou Y Q, Chen Y R, et al. Fastp: an ultra-fast all-in-one FASTQ preprocessor [J]. Bioinformatics, 2018, 34(17): i884-i890.
107Tillich M, Lehwark P, Pellizzer T, et al. GeSeq: versatile and accurate annotation of organelle genomes [J]. Nucleic Acids Research, 2017, 45(W1): W6-W11.
108Lowe T M, Eddy S R. tRNAscan-SE: a program for improved detection of transfer RNA genes in genomic sequence [J]. Nucleic Acids Research, 1997, 25(5): 955-964.
109Lohse M, Drechsel O, Bock R. OrganellarGenomeDRAW (OGDRAW): a tool for the easy generation of high-quality custom graphical maps of plastid and mitochondrial genomes [J]. Current Genetics, 2007, 52(5/6): 267-274.
110Grant J R, Stothard P. The CGView Server: a comparative genomics tool for circular genomes [J]. Nucleic Acids Research, 2008, 36: 181-184.
111Thiel T, Michalek W, Varshney R, et al. Exploiting EST databases for the development and characterization of gene-derived SSR-markers in barley (Hordeum vulgare L.) [J]. Theoretical & Applied Genetics, 2003, 106(3): 411-422.
112Kurtz S, Choudhuri J V, Ohlebusch E, et al. REPuter: the manifold applications of repeat analysis on a genomic scale [J]. Nucleic Acids Research, 2001, 29(22): 4633-4642.
113Sharp P M, Li W H. Codon usage in regulatory genes in Escherichia coli does not reflect selection for ‘rare codons [J]. Nucleic Acids Research, 1986, 14(19): 7737-7749.
114Domselaar G V. Interactive microbial genome visualization with GView [J]. Bioinformatics, 2010, 26(24): 3125-3126.
115Amiryousefi A, Hyv?nen J, Poczai P. IRscope: an online program to visualize the junction sites of chloroplast genomes [J]. Bioinformatics, 2018, 34(17): 3030-3031.
116Rozas J, Rozas R. DnaSP, DNA sequence polymorphism: an interactive program for estimating population genetics parameters from DNA sequence data [J]. Bioinformatics, 1995, 11(6): 621-625.
117Katoh K, Standley D M. MAFFT multiple sequence alignment software version 7: improvements in performance and usability [J]. Molecular Biology and Evolution. 2013, 30(4): 772-780.
118Price M N, Dehal P S, Arkin A P. FastTree 2-approximately maximum-likelihood trees for large alignments [J]. Plos One, 2010, 5(3): e9490. doi: 10.1371/journal.pone.0009490.
119Zhang C, Li S Q, Xie H H, et al. Comparative plastid genome analyses of Rosa: Insights into the phylogeny and gene divergence [J]. Tree Genetics & Genomes, 2022, 18(3): 20. doi: 10.1007/s11295-022-01549-8.
120纵丹, 黄嘉城, 段晓盟, 等. 无籽刺梨及其近缘种叶绿体基因组序列比较分析[J]. 福建农林大学学报(自然科学版), 2024, 53(1): 39-47.
Zong D, Huang J C, Duan X M, et al. Comparative analyses on chloroplast genome sequence of Rosa sterilis and its related species [J]. Journal of Fujian Agriculture and Forestry University (Natural Science Edition), 2024, 53(1): 39-47.
121Kaila T, Chaduvla P K, Saxena S, et al. Chloroplast genome sequence of pigeonpea (Cajanus cajan (L.) Millspaugh) and Cajanus scarabaeoides (L.) Thouars: genome organization and comparison with other legumes [J]. Frontiers in Plant Science, 2016, 7: 1847. doi: 10.3389/fpls.2016.01847.
122Park I, Yang S, Kim W J, et al. The Complete chloroplast genomes of six Ipomoea species and indel marker development for the discrimination of authentic pharbitidis semen (seeds of I. nil or I. purpurea) [J]. Frontiers in Plant Science, 2018, 9: 965. doi: 10.3389/fpls.2018.00965.
123Yang Y, Dang Y Y, Li Q, et al. Complete chloroplast genome sequence of poisonous and medicinal plant Datura stramonium: organizations and implications for genetic engineering [J]. Plos One, 2014, 9(11): e110656. doi: 10.1371/journal.pone.0110656. eCollection 2014.
124Fan L, Li L, Hu Y F, et al. Complete chloroplast genomes of five classical Wuyi tea varieties (Camellia sinensis, Synonym: Thea bohea L.), the most famous oolong tea in China [J]. Mitochondrial DNA Part B, 2022, 7(4): 655-657.
125Li X W, Gao H H, Wang Y T, et al. Complete chloroplast genome sequence of Magnolia grandiflora and comparative analysis with related species [J]. Science China Life Sciences, 2013, 56(2): 189-198.
126Huang H, Shi C, Liu Y, et al. Thirteen Camellia chloroplast genome sequences determined by high-throughput sequencing: genome structure and phylogenetic relationships [J]. BMC Evolutionary Biology, 2014, 14(1): 151. doi: 10.1186/1471-2148-14-151.
127Geng X S, Huang N, Zhu Y L, et al. Codon usage bias analysis of the chloroplast genome of cassava [J]. South African Journal of Botany, 2022, 151: 970-975.
128Ramesh G A, Mathew D, John K J, et al. Chloroplast gene matK holds the barcodes for identification of Momordica (Cucurbitaceae) species from Indian subcontinent [J]. Horticultural Plant Journal, 2022, 8(1): 89-98.
129Yang C J, Wang K, Zhang H, et al. Analysis of the chloroplast genome and phylogenetic evolution of three species of Syringa [J]. Molecular Biology Reports, 2023, 50(1): 665-677.
130Rawal H C, Kumar P M, Bera B, et al. Decoding and analysis of organelle genomes of Indian tea (Camellia assamica) for phylogenetic confirmation [J]. Genomics, 2020, 112(1): 659-668.

