病毒宏基因组学检测猪博卡病毒及其遗传进化分析
孙晓瑜 , 单 虎 , 黄 娟
(1.青岛农业大学动物医学院 , 山东 青岛 266109 ; 2.山东省预防兽医学重点实验室 , 山东 青岛 266109 ;3.山东省兽药诊断试剂工程技术研究中心 , 山东 青岛 266109)
猪博卡病毒(Porcine bocavirus,PBoV)于2009年首次被发现,是从瑞典患有仔猪断奶多系统衰竭综合征的病猪体内检测到的[1]。不少学者推测PBoV与猪的腹泻疾病和呼吸道疾病有关[2],但目前与其致病性相关的研究较少,适合PBoV体外培养的传代细胞系也尚未见报道[3]。PBoV常与猪流行性腹泻病毒(Porcine epidemic diarrhea virus,PEDV)混合感染[4],与猪瘟病毒(Classical swine fever virus,CSFV)、猪繁殖与呼吸综合征病毒(Porcine reproductive and respiratory syndrome virus,PRRSV)和猪圆环病毒2型(Porcine circovirus 2,PCV2)等也存在较为普遍的共感染现象[5]。目前,我国对于该病毒的研究相对较少,病毒基因组学信息也不够丰富,因此,对PBoV进行分子流行病学调查和遗传学分析,对其防控具有重要意义。
PBoV属于细小病毒科、博卡病毒属[6],是一种单链线性DNA病毒,无囊膜,二十面体对称,基因组大小约为5 kb[7]。PBoV基因组由3个开放阅读框(Open reading frame,ORF)组成。ORF1编码非结构蛋白NS1,ORF2编码结构蛋白VP1和VP2,ORF3位于ORF1和ORF2之间,编码非结构蛋白NP1[8]。根据NS1蛋白的差异,曾将PBoV分为PBoV 1~5型共5个基因型,且各个基因型之间的同源性较低[9]。随后有研究提出,将PBoV分为3个不同的群,即PBoV G1、PBoV G2和PBoV G3[2]。也有研究者提议,如果PBoV的NS1蛋白氨基酸序列同源性低于85%,则将其指定为新毒株[10]。VP1和VP2在基因组上存在序列重叠,VP1序列包含整个VP2序列和1个N-末端区域(约227个氨基酸),该N-末端区域称为VP1独特区域(VP1 unique region,VP1u)[11]。PBoV的VP1u存在分泌型磷脂酶A2(Phospholipase A2,PLA2)基序[12]。序列分析发现,与PBoV同科的大多数细小病毒在该区域中都含有保守结构域,在催化位点中具有保守的HDXXY基序,在钙结合环中具有保守的YXGXF基序,具有PLA2活性[13]。
传统的核酸检测方法往往需要已知病毒的基因组序列才能进行检测,Illumina平台可以对未知病毒进行深度测序,这对于发现新病原、检测混合感染和诊断复杂病例具有重要意义[14]。本试验利用病毒宏基因组学技术在山东地区检测到了PBoV,获得了3 801 bp的基因组序列,并进行了基因序列特征分析,为未来PBoV全基因组测序和流行病学调查提供了基因组数据信息。
1 材料与方法
1.1 主要试剂 杜氏磷酸缓冲盐溶液(Dulbecco's phosphate-buffered saline,DPBS)、乙二醇双(2-氨基乙基醚)四乙酸[Ethylene glycol bis(2-aminoethyl ether)tetraacetic acid,EGTA]和三乙酰基乙酸缓冲液(Tris acetate-EDTA buffer,TAE),均为北京索莱宝科技有限公司产品;微球菌核酸酶,New England Biolabs(NEB)公司产品;病毒核酸提取试剂盒,广州美基生物科技有限公司产品;PremixTaq和DNA Marker,均为TaKaRa公司产品;琼脂糖,生工生物工程(上海)股份有限公司产品。
1.2 主要仪器 离心机,德国艾本德(Eppendorf)公司产品;水浴锅,江苏科析仪器有限公司产品;0.45 μm过滤器和Amicon®Ultra-15离心式过滤器,美国密理博(Millipore)公司产品;高速组织研磨器,天根生化科技有限公司产品;PCR仪,杭州柏恒科技有限公司产品;核酸电泳凝胶成像系统,上海勤翔科学仪器有限公司产品。
1.3 试验方法
1.3.1 样本采集 山东省平度市某养猪场仔猪发生不明原因腹泻,剖检其中1只患病仔猪,采集仔猪的肺脏、肾脏、肝脏、小肠、脾脏和淋巴结,分别各取0.5 g上述样本放置于1.5 mL离心管中,使用研磨器研磨,然后向每个试管中添加200 μL DPBS进行稀释,并于-80 ℃保存。
1.3.2 样本处理和核酸提取 将样本研磨稀释液反复冻融3次,于4 ℃、12 000 r/min离心3 min,取上清。将所有样本的上清液混合在一起以形成样本池。使用0.45 μm过滤器过滤上清混合液,并使用离心式过滤器浓缩。使用微球菌核酸酶在37 ℃下消化经浓缩的样本混合液2 h(样本∶酶∶Buffer=100∶1∶10)。消化结束时,加入浓度为500 mmol/L、pH为8的EGTA(样本体积的0.045倍)以灭活微球菌核酸酶,使用病毒核酸提取试剂盒分别提取DNA和RNA,送至上海探普生物科技有限公司进行测序。
1.3.3 病毒宏基因组测序 使用Illumina测序技术对1.3.2中提取的样本核酸进行测序,并构建Illumina PE文库。利用BBmap软件去除核糖体RNA(Ribosomal RNA,rRNA)、宿主和细菌序列,并利用Kraken2软件进行病毒注释,使用SPAdes 和SOAPdenovo软件进行Denovo组装,将从组装中获得的重叠簇与BLAST的Viral NT数据库(V2.10.0+)进行比较,确定进化上亲缘关系最密切的候选参考序列,确定匹配序列所属的物种,并将符合匹配标准的序列判断为候选序列,选择长度≥1 500 bp的片段重叠群(Contigs)进行深度统计。
1.3.4 病毒基因序列比对和系统发育分析 将测序得到的病毒序列提交到GenBank获得登录号,从GenBank数据库下载21株PBoV参考毒株基因组序列(G1、G2和G3群各选几株代表株),使用MegAlign软件的Clustal W方法与参考毒株进行多序列比对分析。应用MEGA 11软件,选择邻接法(Neighbor-joining,NJ),Bootstrap值设为1 000个重复,构建系统发育进化树。
1.3.5 病毒基因组序列分析 使用MegAlign软件的Clustal W方法,将获得的病毒基因组序列与参考毒株序列进行同源性分析。氨基酸比对结果用BioEdit 7.0软件呈现。
1.3.6 样本中PBoV的PCR检测 PBoV PCR检测引物序列参照参考文献[15],引物序列为PBoV-F:GGTGATCCTGTCAATAAAG(VP1基因核苷酸第82~100位),PBoV-R:TGCAAAGAGTCGATAAAGT(VP1基因核苷酸第194~212位),预期扩增片段大小为131 bp,引物由生工生物工程(上海)股份有限公司合成。PCR反应体系(25 μL):PremixTaq12.5 μL,DNA模板3 μL,上、下游引物各0.5 μL(50 μmol/L),去离子水8.5 μL。PCR反应程序:94 ℃预变性2 min;94 ℃变性30 s,62 ℃退火20 s,72 ℃延伸20 s,共进行35个循环;最后再72 ℃延伸2 min。PCR扩增产物经3.0% 琼脂糖凝胶电泳检测。
2 结果
2.1 病毒宏基因组测序 原始数据的质量控制结果良好。RNA样本病毒种水平注释结果显示,只有A型流感病毒(H1N1和H3N2),读数(Reads counts)共682次,读数比为6.8%,共得到35条重叠群序列,长度为136 ~1 777 bp,未发现其他猪病相关RNA病毒。DNA样本测序数据去除rRNA、宿主和细菌序列等污染数据后,读数共827 395次,读数比为4.9%。病毒注释后,在科水平上,读取次数最多的是细小病毒科(Parvoviridae)(图1A),为2 443次,读数比为59.3%;在种水平上,读取次数最

图1 DNA样本的病毒注释
多的是PBoV(图1B),为1 056次,读数比为34.5%;其他猪病相关病毒包括猪巨细胞病毒(Porcine cytomegalovirus)、猪乳腺腺病毒(Porcine mastadenovirus)和猪细环病毒(Porcine torque teno virus,TTV),读数比均<1.0%,拼接比对得到的重叠群序列长度均短于280 bp,所以不予进一步分析。因为PBoV读数比最高,是优势病毒,又是猪腹泻相关病原,故后续重点分析PBoV。
2.2 病毒基因序列比对和系统发育分析 通过测序获得1段PBoV的基因序列,将其命名为PBoV-SDPD-2022,全长为3 801 bp,包括完整的NS1、NP1基因和不完整的VP1、VP2基因(VP1基因N端968 bp序列,47%覆盖率;VP2基因N端560 bp序列,34%覆盖率),序列提交至GenBank,获得登录号为OQ860988。通过序列比对,与PBoV-SDPD-2022同源性最高的毒株是PBoV3_VIRES_HuB01_C1(MK378121.1),同源性为97.4%。从GenBank中下载了21株参考毒株的基因序列,与PBoV-SDPD-2022进行比对,分别基于全基因组、NS1基因和VP1基因构建了系统发育进化树(图2),PBoV毒株主要分为3个分支,即PBoV的3个群,PBoV-SDPD-2022与PBoV3_VIRES_HuB01_C1处于同一分支,属于PBoV G3群。

图2 PBoV-SDPD-2022与PBoV参考毒株的系统发育进化树
2.3 病毒基因组序列分析
2.3.1 同源性分析 将毒株PBoV-SDPD-2022的NS1、VP1和NP1蛋白的编码基因核苷酸和氨基酸序列分别与21株参考毒株进行比对,同源性分析结果见表1。针对NS1蛋白,PBoV-SDPD-2022与G1群参考毒株核苷酸序列同源性为49.6%~51.3%,氨基酸序列同源性为40.6%~41.4%;与G2群参考毒株核苷酸序列同源性为52.9%~54.3%,氨基酸序列同源性为38.5%~39.0%;与G3群参考毒株核苷酸序列同源性为79.2%~97.2%,氨基酸序列同源性为79.4%~95.3%。针对VP1蛋白,PBoV-SDPD-2022与G1群参考毒株核苷酸序列同源性为47.9%~51.1%,氨基酸序列同源性为34.8%~39.7%;与G2群参考毒株核苷酸序列同源性为54.9%~55.1%,氨基酸序列同源性为47.0%~47.6%;与G3群参考毒株核苷酸序列同源性为83.4%~97.9%,氨基酸序列同源性为85.0%~98.8%。针对NP1蛋白,PBoV-SDPD-2022与G1群参考毒株核苷酸序列同源性为35.2%~43.6%,氨基酸序列同源性为26.9%~27.4%;与G2群参考毒株核苷酸序列同源性为45.0%~46.4%,氨基酸序列同源性为33.8%~34.2%;与G3群参考毒株核苷酸序列同源性为79.1%~98.5%,氨基酸序列同源性为72.6%~97.4%。结果表明,PBoV-SDPD-2022与G3群参考毒株同源性较高,且在G3群毒株中,NP1蛋白同源性比NS1和VP1蛋白低。
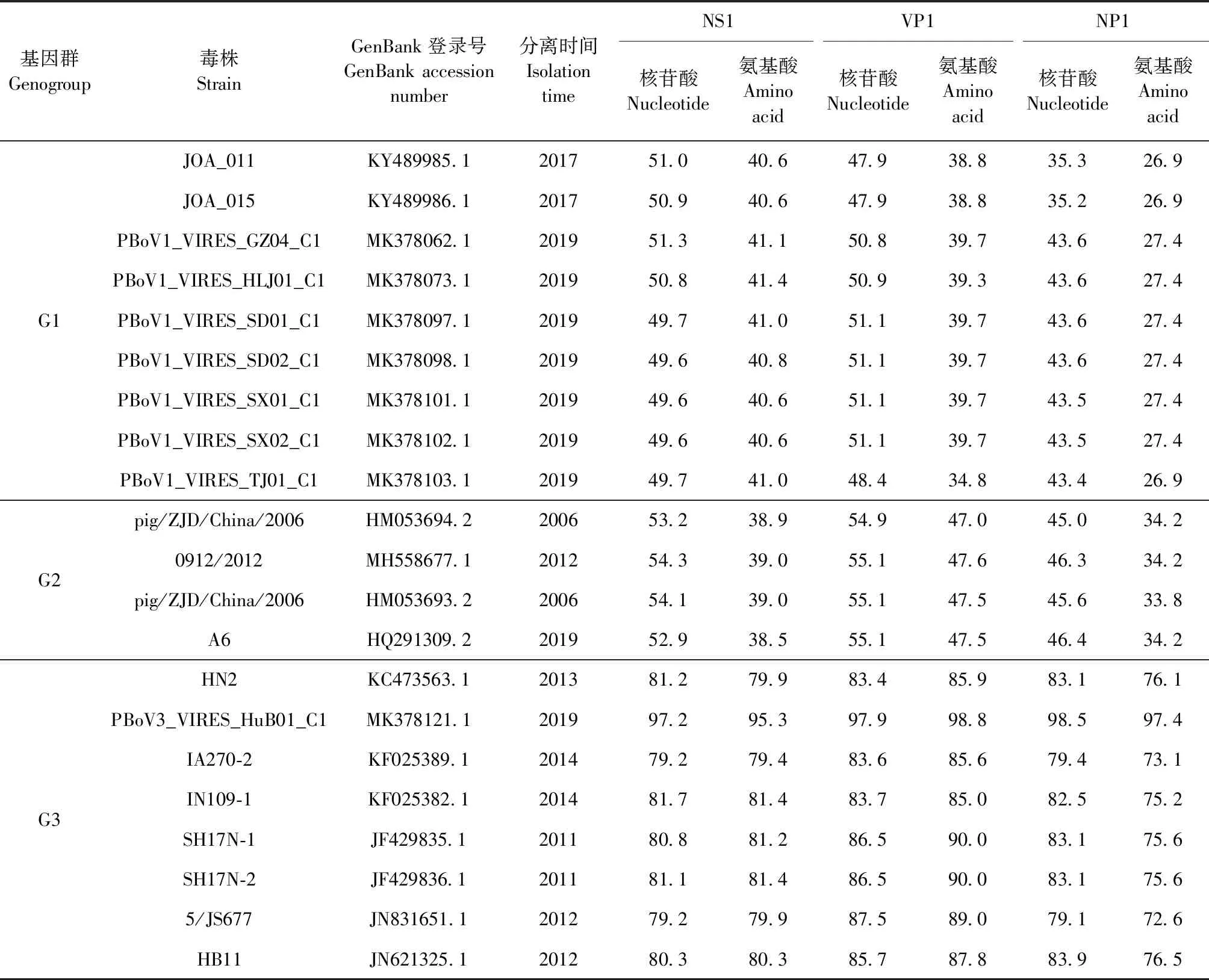
表1 PBoV-SDPD-2022与参考毒株的同源性分析
2.3.2 NS1蛋白氨基酸序列分析 与G3群参考毒株相比,PBoV-SDPD-2022 NS1蛋白没有氨基酸插入现象,但在第654位点处有1个氨基酸的缺失(图3)。

图3 PBoV-SDPD-2022与G3群PBoV参考毒株NS1蛋白的氨基酸分析
2.3.3 VP1蛋白氨基酸序列分析 YXGXF基序在PBoV G2和G3群毒株(包括PBoV-SDPD-2022)中均为YLGPF(VPl aa 18~22)(图4A)。HDXXY基序在PBoV G2和G3群其他毒株均为HDQAY,但在PBoV-SDPD-2022中变为HDLAY(VP1 aa 41~45)(图4A)。与G3群其他毒株相比,PBoV-SDPD-2022和PBoV3_VIRES_HuB01_C1的VP1蛋白在第151和152位点处有2个氨基酸(151G、152P)的插入(图4B)。

图4 PBoV-SDPD-2022与PBoV参考毒株VP1蛋白的氨基酸分析
2.3.4 NP1蛋白氨基酸序列分析 与G3群其他毒株相比,PBoV-SDPD-2022 NP1蛋白在第15、16、35、36、212位点处发生氨基酸的缺失,在第95位点处有1个氨基酸(P)的插入(图5)。

图5 PBoV-SDPD-2022与G3群PBoV参考毒株NP1蛋白的氨基酸分析
2.4 样本中PBoV的PCR检测 通过PCR扩增得到大小为131 bp的单一条带(图6),与预期片段大小相符。

图6 样本中PBoV的PCR检测
3 讨论
PBoV自2009年发现以来,已在我国多次发生疫情,并且在健康猪和腹泻猪体内均可检测到PBoV,说明PBoV在我国猪群中的感染已经较为广泛。许雅茹等[16]调查了我国猪群中PBoV的流行情况,对我国部分地区89份腹泻病猪粪便样本进行PCR检测,结果显示,PBoV阳性率为47.19%,其中上海和江西的样品阳性率最高,山东的样品阳性率最低,并且鉴定的5株PBoV均属于PBoV 3型。另有许多研究在腹泻样本中检测到PBoV[17-18],说明PBoV与猪腹泻具有一定的相关性。Aryal等[19]针对PBoV的致病机理进行了研究,用无细菌和其他病毒的PBoV阳性肠内容物饲喂缺初乳仔猪,接种PBoV的仔猪出现腹泻,接种后18 h即可在直肠拭子中检测到病毒,并且小肠出现肉眼和显微镜下可观察到的病变,通过常规PCR检测到了小肠中的病毒,证实了PBoV具有致病性,并将其定性为缺乏初乳的新生仔猪腹泻的病原体。近年来有研究表明,我国PBoV 3、4和5型(G3群)的感染力要强于PBoV 1和2型[20]。本试验从腹泻仔猪体内检测到了PBoV,经分析属于PBoV G3群,在一定程度上也支持了以上观点。
对于PBoV的分型依据,目前较为混乱,缺乏公认且权威的分型方法,国际病毒分类委员会曾依据NS1蛋白氨基酸同源性大于95%为一个基因型的标准将PBoV分为PBoV 1~5型共5个基因型,后又将该分型标准调整为NS1蛋白氨基酸同源性大于85%为1个基因型[10]。随着更多新毒株的发现,这一分型方法已经难以区分PBoV。根据VP1基因将PBoV分为G1、G2和G3 共3个基因群[21],可将所有的毒株划分在内,且同群内毒株基因序列同源性较高,应用较为广泛。余蕊等[22]提到常用的PBoV分型方法有2种:依据病毒NS1和VP1蛋白的编码基因序列进行基因型划分。因此,本试验基于全基因组、NS1和VP1基因分别构建了系统发育进化树,结果显示,3种方法构建的系统发育进化树基本一致,PBoV G2群与G3群处于同一大分支,处于G3群的PBoV-SDPD-2022与G2群的同源性也相比于G1群高,猜测G2群与G3群可能由共同祖先重组进化而来。Lau等[23]研究发现,PBoV 4型可能存在重组事件,表明同一宿主内的不同毒株可能由重组产生。Zhou等[24]在PBoV G3群的NS1区发现了1个天然重组断点。但在本试验中,PBoV-SDPD-2022并未检测到重组事件。Leisi等[25]研究发现,VP1u的N端区域负责病毒的内化,并且VP1u的二聚体化可导致细胞结合和内化大大增强。本试验分析了PBoV VP1u的PLA2基序YXGXF和HDXXY,这2个序列的变化不仅影响病毒DNA复制,还与PBoV的致病性有关[12]。与PBoV G2和G3群毒株相比,PBoV G1群毒株缺少这2个基序,这可能表明PBoV G1群病毒不能激活PLA2。
本试验通过病毒宏基因组学的方法对未知原因的仔猪腹泻病例进行了病毒检测,旨在了解腹泻病例中存在哪些猪源病毒,检测可能的未知致病病原。虽然之前的报道表明,PBoV常与PEDV、CSFV、PRRSV和PCV2混合感染[4-5],但在本病例中并未检测到上述病原,主要检测到了PBoV,虽然也检测到A型流感病毒(H1N1和H3N2)、猪巨细胞病毒、猪乳腺腺病毒和猪细环病毒,但病毒丰度很低,因此,PBoV可能为引起该养猪场仔猪腹泻的主要病原。通过对病毒进行遗传进化分析,以及结合相关报道,目前PBoV G3群在我国流行最为广泛,但检测到的完整基因序列较少,未来应加强PBoV全基因组测序和流行病学调查。

