牛科物种THRSP 基因的分子特征及功能比较分析*
吴 宇,王荣平,朱 伟,范新阳,滕晓红,苗永旺**
(1.云南农业大学 动物科学技术学院,云南 昆明 650201;2.云南农业大学 动物遗传育种研究所,云南 昆明 650201;3.云南农业职业技术学院 畜牧兽医学院,云南 昆明 650212)
甲状腺激素应答(thyroid hormone responsive,THRSP)基因的编码蛋白Spot14 (又称S14)最初由SEELIG 等[1]鉴定自大鼠肝脏的体外翻译产物,是Spot14 家族的重要成员之一。在该家族中,还有1 个与其序列高度同源的蛋白——Spot14 相关蛋白(Spot14-R,MIG12)[2]。Spot14 家族成员包含3个保守区域,即N 末端的疏水区、中间的疏水区和C 末端的亮氨酸拉链区[2-4]。Spot14 和Spot14-R 依赖亮氨酸拉链结构形成同二聚体(Spot14/Spot14 和Spot14-R/Spot14-R)或异二聚体(Spot14/Spot14-R)参与基因的表达调控[4-6]。目前NCBI数据库中已有普通牛、水牛、瘤牛、野牛、绵羊、山羊等主要牛科动物及单峰驼、双峰驼、马、驴、人、小鼠、鸡等非牛科动物的THRSP基因序列信息,有研究表明该基因参与奶牛和山羊的乳脂合成过程,并与奶牛乳脂率和乳品质有关[7-10]。
虽然已有一些关于牛科物种THRSP基因的研究,但对于牛科家畜THRSP 分子特征及功能的深入比较分析鲜有报道。本研究从NCBI 和Ensembl 数据库下载了主要牛科物种和非牛科物种THRSP基因及对应的编码氨基酸序列,进一步采用比较基因组学和生物信息学分析方法对牛科物种该基因转录区的结构、编码区(coding sequence,CDS)的核苷酸组成、密码子使用偏好性 (codon usage bias,CUB)、编码产物的氨基酸组成、理化特性、结构特征、功能修饰位点、参与的生物学路径、分子功能和系统发育关系进行全面预测和分析,旨在阐明牛科物种THRSP基因的分子特征及功能差异,为其表达调控研究提供参考。
1 材料与方法
1.1 序列及基因组注释文件的下载
从NCBI (https://www.ncbi.nlm.nih.gov/)和Ensembl (https://asia.ensembl.org/index.html)数据库中下载普通牛、水牛、牦牛、杂交牛、绵羊、山羊等牛科物种的THRSP基因及对应的编码氨基酸序列;利用NCBI 网站中的在线Blast 程序(https://blast.ncbi.nlm.nih.gov/Blast.cgi)进行同源搜索,获取与牛科THRSP同源的非牛科物种序列;从NCBI网站的Genome 数据库(https://www.ncbi.nlm.nih.gov/genome/?term=)中下载各物种的基因组GTF格式数据用于分析。对所有序列进行比对和核对,筛除不完整或可能存在错误的序列,进一步对剩余的可靠序列(表1)进行分析。
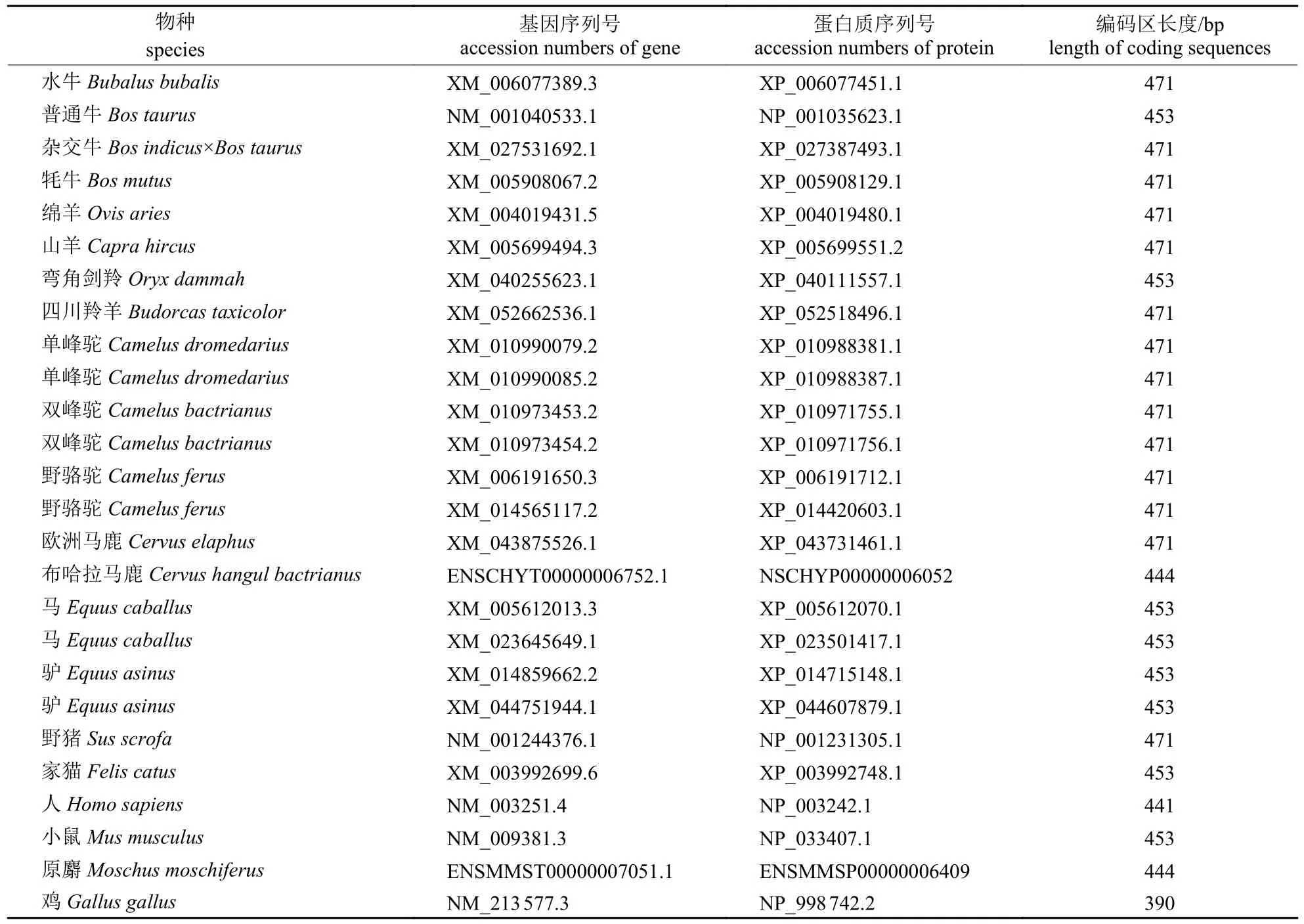
表1 本研究所用序列Tab.1 Sequences used in this study
1.2 核苷酸组成和转录区结构分析
使用NCBI 数据库中的ORF Finder 程序(ht-tps://www.ncbi.nlm.nih.gov/orffinder/)查找已下载的各物种THRSP基因开放阅读框(open reading frame,ORF),以确定每条序列的完整CDS,使用Lasergene 软件包(DNAStar Inc.,USA)中的Edit-Seq 程序分析各物种THRSP基因CDS 的碱基组成。在NCBI 数据库中下载牛科和非牛科物种的基因组数据,并提取THRSP基因的转录本信息;使用TBtools[11]软件对各物种THRSP基因的转录本信息进行完整化处理,进一步使用在线软件Gene Structure Display Server 2.0 (http://gsds.gaolab.org/)呈现各物种THRSP基因的转录区结构,包括非翻译区(untranslated region,UTR)、外显子和内含子。
1.3 THRSP 基因的 CUB 分析
使用Codon 程序(http://codonw.sourceforge.net/)进行各物种THRSP基因的 CUB 分析,包括相对同义密码子使用度(relative synonymous codon usage,RSCU)、有效密码子数 (effective number of codons,ENc 值)、GC 含量和密码子第3 位的GC 含量(GC of silent 3rd codon position,GC3s),其中,GC3s 为除了蛋氨酸、色氨酸和终止密码子外,G 和C 出现在密码子第3 个位置的频率。
1.4 编码蛋白的理化特性和结构特征分析
利用1.1 节的编码氨基酸序列,使用在线程序ProtParam (http://web.expasy.org/protparam/)预测不同物种THRSP 蛋白的分子质量和理论等电点;使用SignalP5.0 (https://services.healthtech.dtu.dk/service.php?SignalP-5.0)预测信号肽;使用TMHMM version 2.0 (http://www.cbs.dtu.dk/services/TMHMM/)预测跨膜结构域;使用ProtScale (http://web.expasy.org/ protscale/)预测疏水性;使用PROSITE (https://prosite.expasy.org/prosite.html)预测功能修饰位点;使用ProtComp 9.0 (http://linux1.softberry.com/berry.phtml)分析各物种THRSP蛋白的亚细胞定位;分别使用SOPMA (https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_sopma.html)和SWISS-MODEL (https://beta.swissmodel.expasy.org/)分析牛科物种THRSP 蛋白的二级结构和三维结构,蛋白三维结构基于同源建模法,以小鼠Spot 14 蛋白模板(3ont.1.A)为最佳模型构建;使用STRING (https://cn.string-db.org/)分析蛋白相互作用关系;使用NCBI Batch Web CD-Serach Tool (https://www.ncbi.nlm/cdd/)预测各物种THRSP 的保守结构域;使用在线软件Meme Suite[12](https://meme-suite.org)分析各物种THRSP 保守基序;使用TBtools[11]软件的Gene Structure View (Advance)工具整合上述分析结果。
1.5 序列一致性和系统发育分析
使用Lasergene 软件包(DNAStar Inc.,USA)中的MegAlign 程序分析各物种THRSP基因CDS及编码氨基酸的序列一致性;使用MEGA 7[13]软件输出序列间的差异位点,进一步选用该软件中的最大似然法,基于优化出的T92+G 和GTT+G+F 模型分别构建核苷酸序列和氨基酸序列的系统发育树,每个节点处的支持率采用Bootstrap 方法进行评估(10 000 次重复)。
2 结果与分析
2.1 THRSP 基因的核苷酸组成、转录区结构及CUB 分析
2.1.1 CDS 核苷酸组成和转录区结构
牛科与非牛科哺乳动物间THRSP基因CDS的核苷酸组成相似,但与非哺乳动物鸡的THRSP基因CDS 核苷酸组成差异较大,且表现出属内相似性较高的特点;牛科动物中牛属和羊属动物的属内核苷酸组成相似性更高,水牛与牛属和羊属动物之间存在较大差异(表2)。不同物种THRSP基因的转录区结构存在差异,尤其是内含子长度和UTR 长度的差异;除了骆驼科和马科物种包含2 个可变剪接转录本外,其他物种都只包含1 个可变剪接转录本;驴THRSP基因包含3 个外显子和2 个内含子,只有外显子2 编码蛋白质,而其他物种的THRSP基因都由2 个外显子和1 个内含子组成,且只有外显子1 编码蛋白质(图1)。
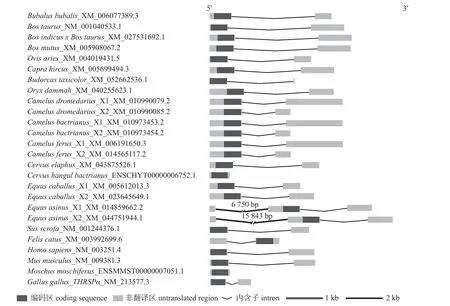
图1 牛科物种及非牛科物种THRSP 转录区的结构Fig.1 Structure of THRSP gene in the species of Bovidae and non-Bovidae
2.1.2THRSP基因CDS 的CUB 分析
由表3 可知:水牛THRSP基因对20 种密码子具有偏好性(RSCU>1),普通牛THRSP基因RSCU>1 的密码子有22 种,杂交牛有18 种,牦牛有20 种,绵羊和山羊分别有22 和21 种,人和鸡都有23 种,小鼠有24 种。水牛与其他牛科物种共同偏好使用的密码子有20 种,且对CUG、AGC、CCC、CGG、ACC、GUG、AUC等密码子的偏好性较强,水牛与其他牛科物种的RSCU 值差异较大。牛科物种编码蛋氨酸和色氨酸的密码子RSCU 值都为1。由表4 可知:THRSP基因ENc 值呈现属内相似、属间差异大的特点,且ENc 值总体偏小,范围在33.95~44.22。牛科物种THRSP基因ENc 值接近20,与其他非牛科动物存在较大差异,表明THRSP基因在牛科物种中的CUB较强;THRSP基因GC3s 值范围为0.741~0.832,均大于0.5,说明THRSP基因中的密码子比较偏好以G/C 结尾;GC 含量范围为0.581~0.625,表明各物种THRSP基因CDS 序列中G+C 碱基的含量大于A+T 碱基的含量。
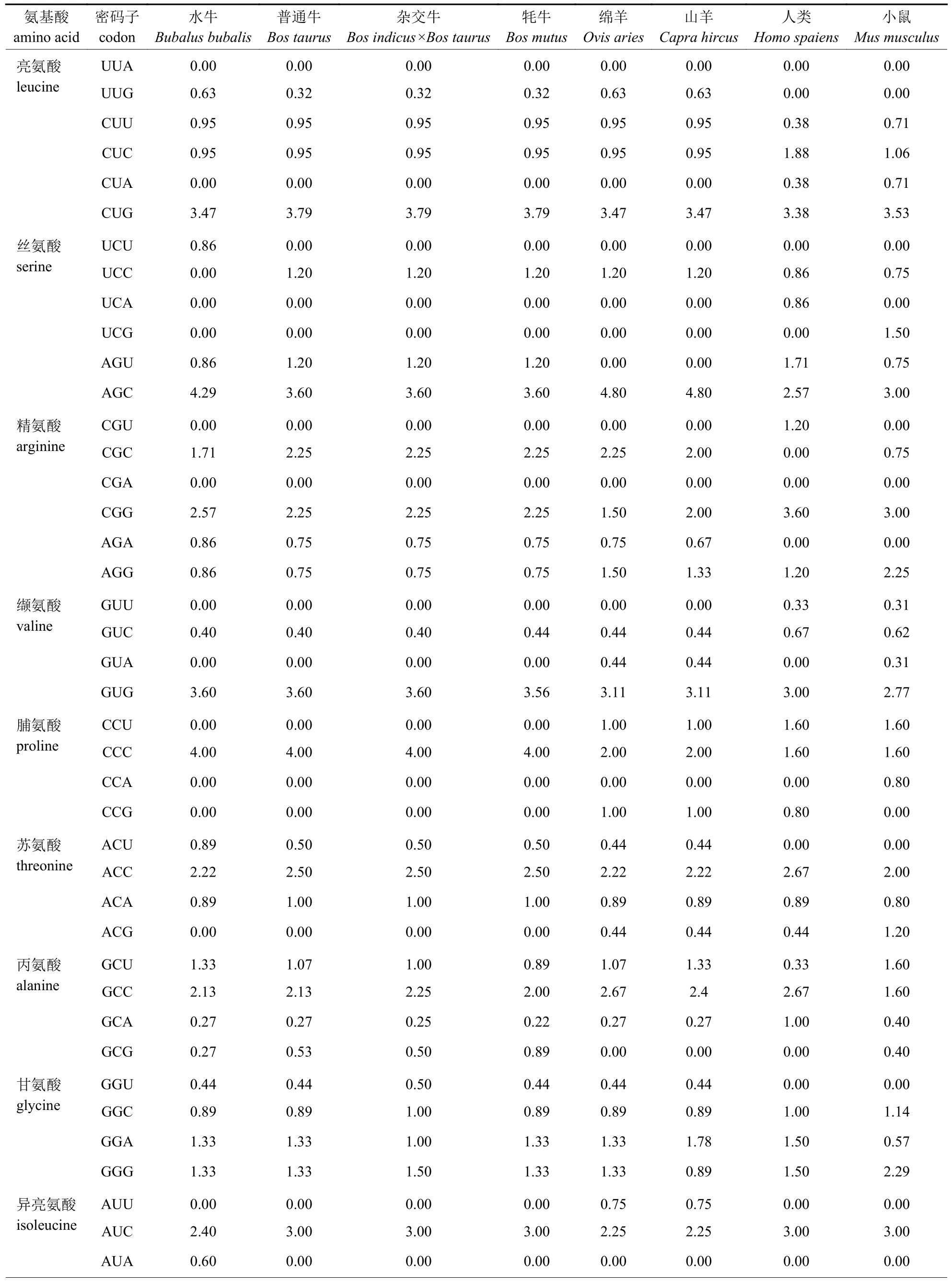
表3 牛科及非牛科物种THRSP 基因密码子的RSCU 值Tab.3 RSCU values of THRSP gene codons in Bovidae and non-Bovidae
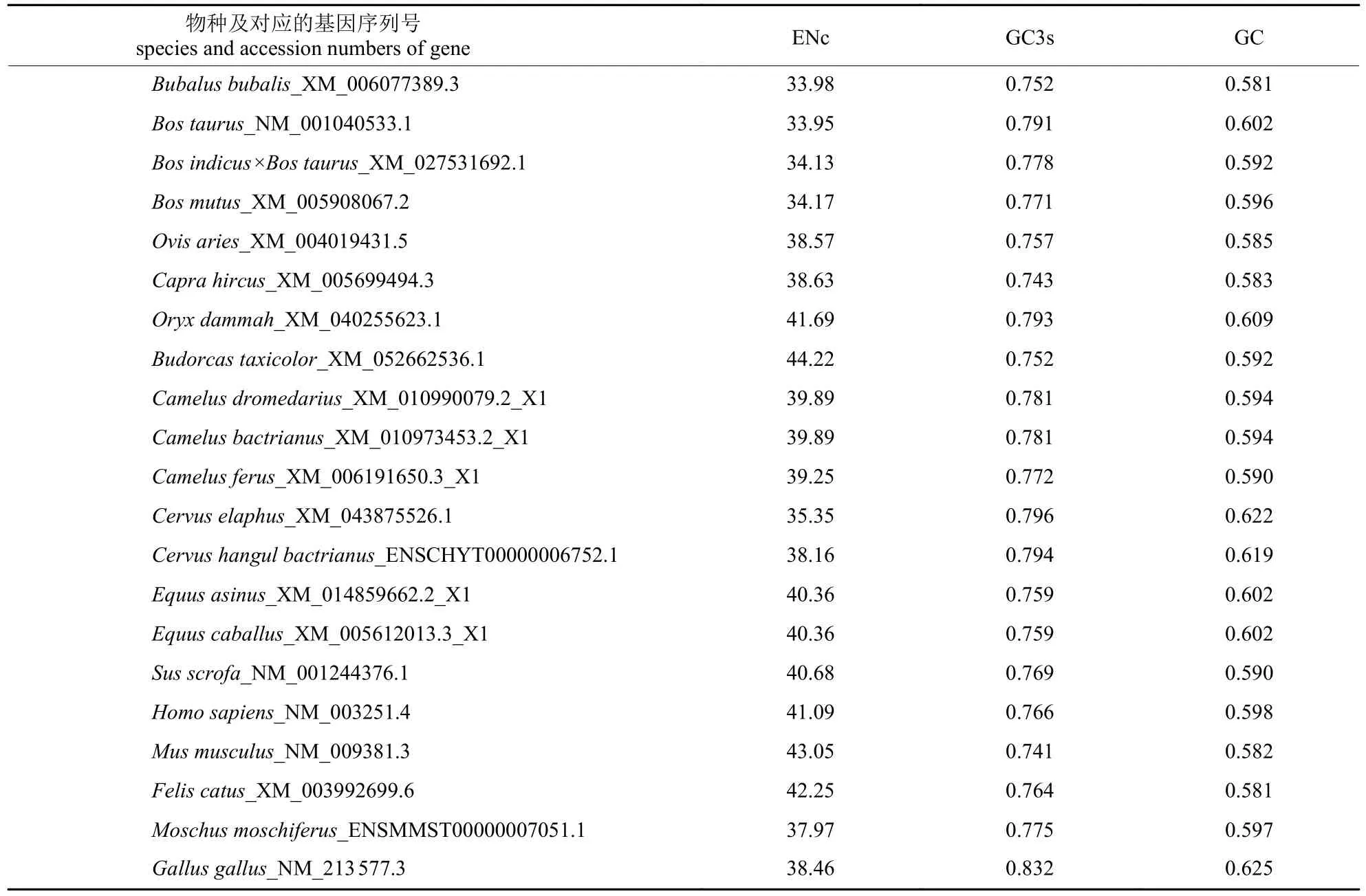
表4 THRSP 基因密码子ENc、GC3s 频率和GC 含量Tab.4 ENc,GC3s frequency and GC content of THRSP gene codons
2.2 THRSP 蛋白的理化特性和结构
2.2.1 理化特性
牛科物种THRSP 蛋白的理化特性较为相似,都为定位于细胞核的亲水性蛋白质,不含跨膜区和信号肽,水牛与其他牛科物种的极性氨基酸含量、脂肪族指数、亲水性总平均值等指标差异较大;牛科物种THRSP 的理化特性与鸡的差异最大,其次是人和小鼠(表5)。
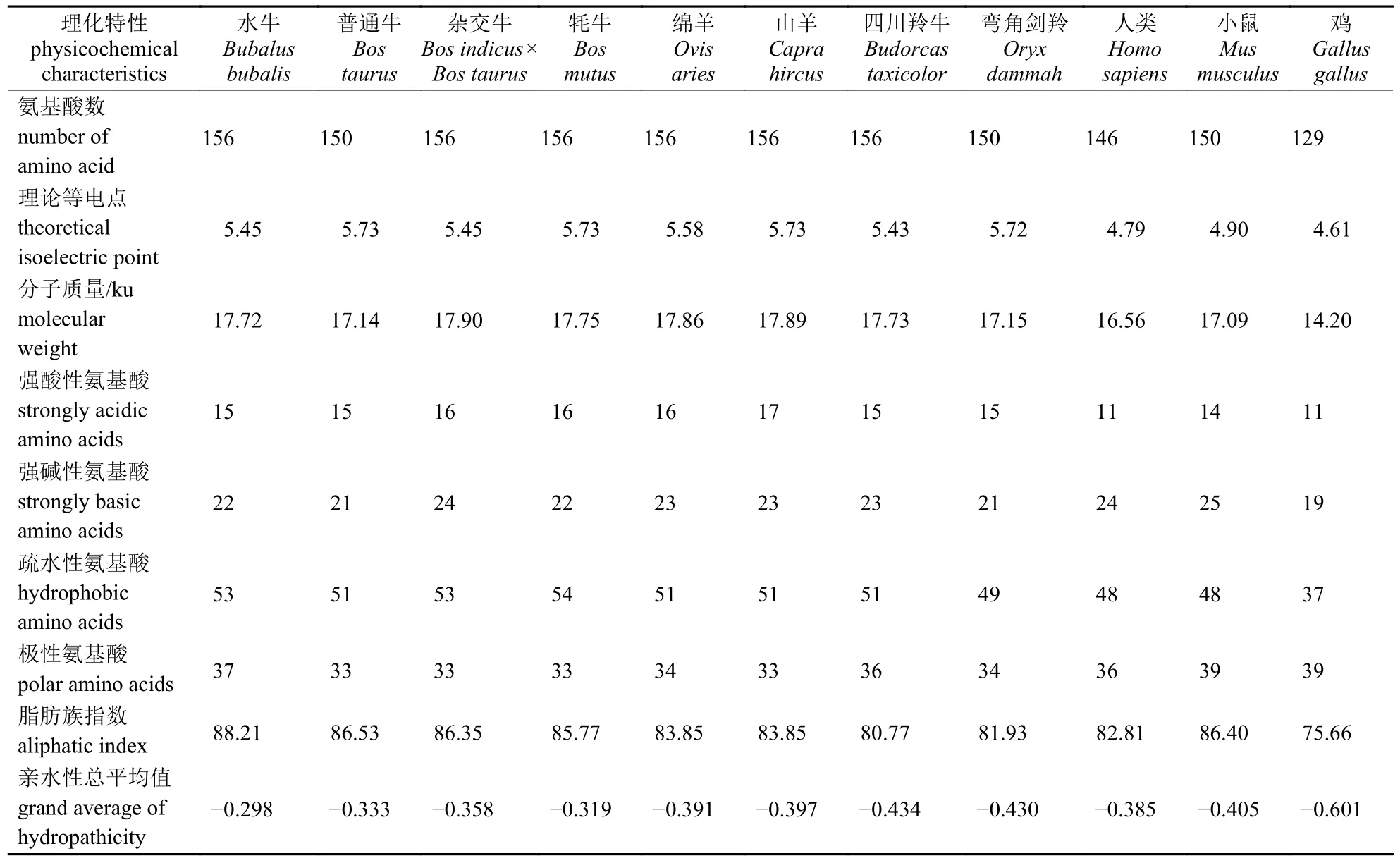
表5 牛科物种及非牛科物种THRSP 理化特性Tab.5 Physicochemical characteristics of THRSP in the species of Bovidae and non-Bovidae
2.2.2 THRSP 基序组成模式和保守结构域
THRSP 具有4 种不同类型的基序组成模式(图2):模式a 包含motif1~5,模式b 比模式a 缺少motif5 和motif3,模式c 包含motif1~4,模式d只包含motif1 和motif2。除普通牛和弯角剑羚外,其他牛科物种THRSP 基序组成都为模式a类型,与非牛科哺乳动物THRSP 基序模式相似,但牛科与鸡差异较大。THRSP 蛋白包含2 种保守结构域,牛科物种中普通牛、杂交牛和牦牛以及除了原麝和鸡以外的其他非牛科物种的THRSP 保守结构域为Spot_14,水牛、山羊、绵羊、弯角剑羚、四川羚羊、原麝和鸡的保守结构域为Spot_14 超家族,牛科物种及其他非牛科动物保守结构域的位置大致相同。

图2 牛科和非牛科物种THRSP 的Motif 组成和保守结构域Fig.2 Motif composition and conserved domains of THRSP in the species of Bovidae and Non-Bovidae
2.2.3 牛科物种THRSP 功能修饰位点
牛科物种THRSP 蛋白功能修饰位点有2 种,即酪蛋白激酶Ⅱ磷酸化位点和蛋白激酶C 磷酸化位点;人和小鼠比牛科物种多了N-豆蔻酰化位点和N-糖基化位点,鸡比牛科物种多了N-豆蔻酰化位点、亮氨酸拉链结构以及cAMP 和cGMP 依赖性蛋白激酶磷酸化位点;牛科物种及其他非牛科哺乳动物相同功能磷酸化位点所处的位置相似,与鸡的差异较大(图3)。
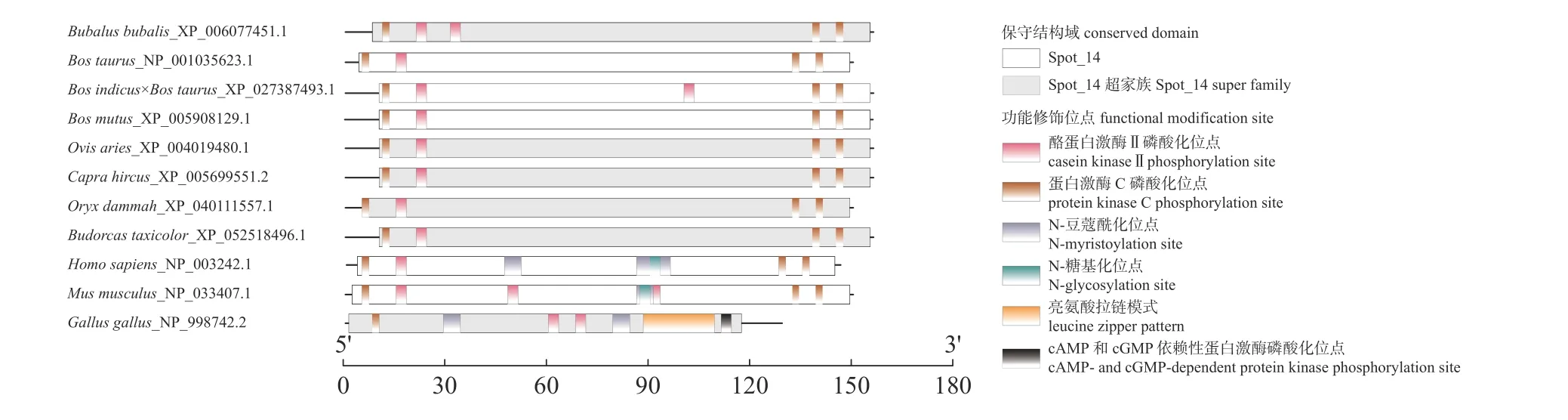
图3 牛科物种THRSP 蛋白功能修饰位点Fig.3 Functional modification site of THRSP protein in the species of Bovidae
表2THRSP基因CDS 核苷酸组成
Tab.2 Nnucleotide composition ofTHRSPCDS %

注:物种及其基因序列号对应的物种中文名见表1;下同。Note: The Chinese names of species and accession number of gene sequences are shown in Tab.1;the same as below.
2.2.4 二级结构和三维结构
牛科物种间THRSP 蛋白二级结构元件的氨基酸比例相似,与其他非牛科动物的差异较大(表6)。水牛、牦牛和四川羚羊的THRSP 蛋白三维结构与模板3ont.1.A 的覆盖率为96% (AA:7~156),一致性为72.00%;杂交牛、绵羊和山羊的三维结构与模板3ont.1.A 的一致性为73.33%,覆盖率为96% (AA:1~156);普通牛和弯角剑羚的三维结构与模型模板3ont.1.A 的一致性和覆盖率分别为73.33%和100% (AA:1~150)(图4)。

图4 牛科物种THRSP 蛋白的三维结构Fig.4 Three-dimensional structures of THRSP protein in the species of Bovidae
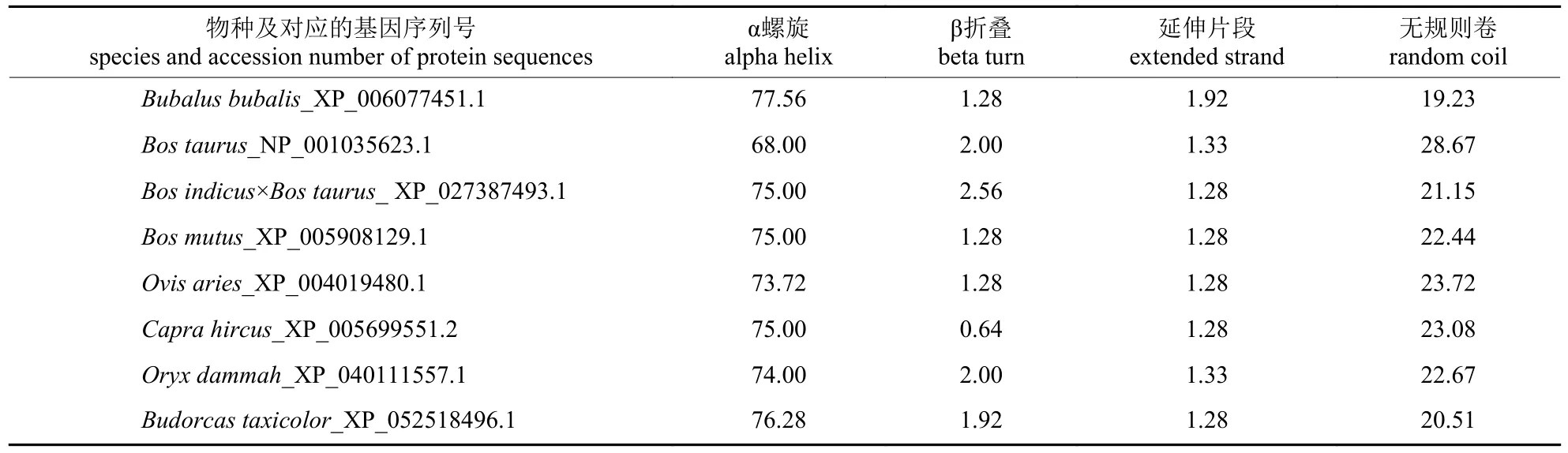
表6 THRSP 蛋白4 种二级结构的氨基酸占比Tab.6 Amino acid proportion of four secondary structures in THRSP protein %
2.2.5 蛋白互作、参与的生物学路径和分子功能
各牛科物种与THRSP 相互作用的蛋白质不尽相同,其中脂肪酸合成酶、甘油-3-磷酸酰基转移酶、乙酰辅酶A 合成酶2 和RAS 癌基因家族成员是与THRSP 互作的蛋白质(图5)。这些蛋白分子主要与脂肪酸的合成和β-氧化、蛋白质代谢和转运、跨细胞膜或细胞器的物质转运、溶酶体与吞噬体的融合、囊泡介导的物质运输等生物学过程有关。THRSP 蛋白主要参与脂类的生物合成(GO:0046890)、脂肪酸代谢(GO:0006631)、乙酰辅酶A 代谢(GO:0006084)、酰基辅酶A 的生物合成(GO:0071616)等生物学过程,其分子功能主要与脂肪酸合酶活性(GO:0004312)和蛋白质同源二聚化活性(GO:0042803)相关。

图5 牛科物种THRSP 互作蛋白网络Fig.5 Interaction protein network of THRSP in the species of Bovidae
2.2.6 序列一致性和系统发育分析
牛科物种THRSP基因包含2 种长度的CDS(471 和453 bp),其编码产物相差6 个氨基酸。牛科物种间THRSP基因CDS 及编码氨基酸序列的一致性高,其中核苷酸序列一致性为93.2%~96.9%,氨基酸序列一致性为91.3%~94.0%;牛科物种与非牛科哺乳动物的序列一致性较低,核苷酸和氨基酸的序列一致性分别为78.6%~95.3%和72.0%~94.6%;牛科与鸡的序列一致性最低,核苷酸和氨基酸的序列一致性分别为51.0%~52.8%和32.2%~35.5% (图6)。序列差异位点分析表明:位点c.79C、c.162T、c.191A、c.226A、c.423G 和c.466G 是区分牛属动物与水牛、羊属动物的核苷酸位点;位点c.91A、c.157A、c.180T、c.222A、c.253C、c.288C 和c.423T 是区分水牛与牛属、羊属动物的核苷酸位点;水牛特有的氨基酸位点为p.31S、p.59T、p.74I 和p.85Q;p.54H、p.64N、p.76M、p.96E 和p.156V 是区分牛属动物与水牛、羊属动物的氨基酸位点;p.57T 和p.90L是区分羊属动物与水牛、牛属动物的氨基酸位点(图7)。
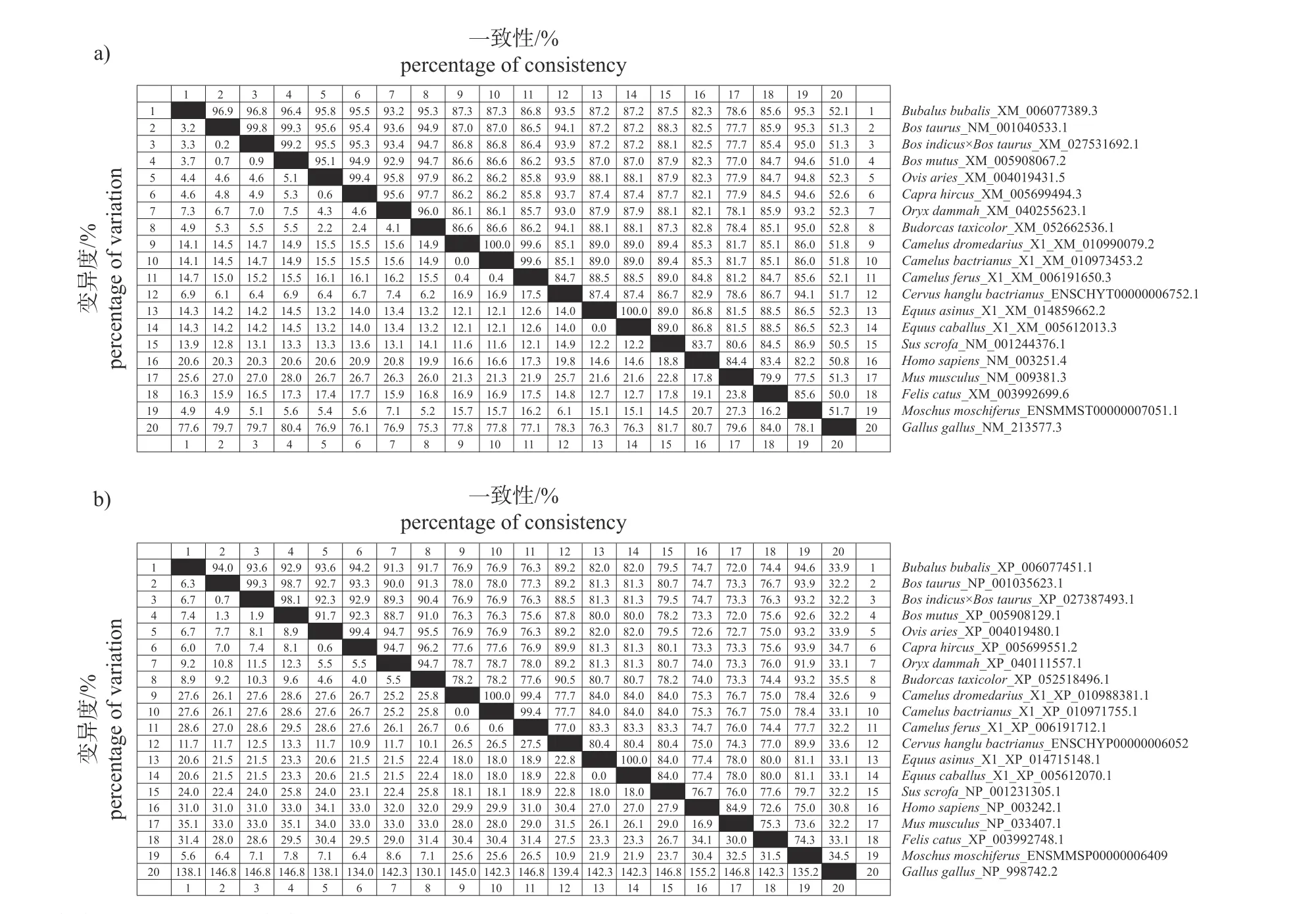
图6 牛科和非牛科物种THRSP 基因核苷酸(a)及编码氨基酸(b)的序列一致性Fig.6 Consistency of THRSP nucleotides (a) and their encoded amino acid (b) sequences in the species of Bovidae and non-Bovidae

图7 牛科和非牛科物种THRSP 基因核苷酸(a)及编码氨基酸(b)序列的差异位点Fig.7 Difference sites in THRSP nucleotides (a) and their encoded amino acid (b) sequences in the species of Bovidae and non-Bovidae
基于各物种THRSP基因核苷酸和编码氨基酸序列构建的系统发育树聚类关系相似,牛科物种聚为一大支,其中水牛和牛属动物聚为一小支,羊属动物聚为单独的一小支;非牛科哺乳动物聚为一支(图8)。
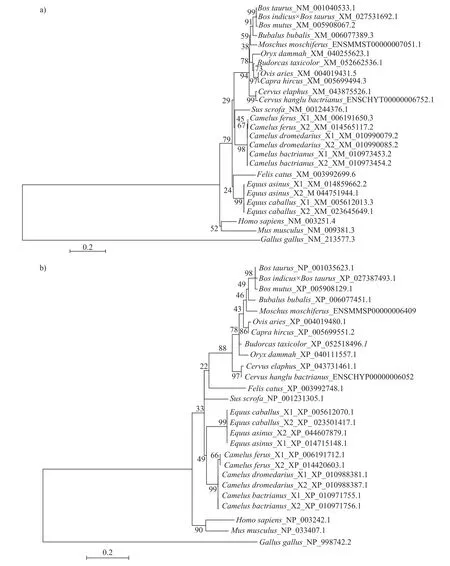
图8 基于核苷酸序列(a)和氨基酸序列(b)构建的牛科和非牛科物种THRSP 系统进化树Fig.8 Phylogenetic trees of THRSP between the species of Bovidae and non-Bovidae based on the sequences of nucleotide (a) and amino acid (b)
3 讨论
牛科家畜能为人类提供丰富的肉类、乳制品等畜产品,同时具有役用价值,在农业生产中具有重要地位。研究表明:THRSP基因与奶牛和山羊乳脂肪酸的从头合成、杂交牛的肌内脂肪和韩国牛的肌肉脂肪酸种类及胴体性状相关,是调节牛科家畜脂合成的重要功能基因,然而,目前关于该基因的研究主要涉及普通牛和山羊等少部分牛科家畜[7-8,14-15],对其他牛科家畜的研究鲜有报道。基于此,本研究采用比较基因组学和生物信息学分析方法对牛科家畜THRSP基因及其编码产物的理化特性和结构特征、蛋白互作以及参与的生物学路径与功能等进行了深入的比较分析,旨在阐明牛科动物THRSP 在分子特征及功能上的差异。
本研究表明:牛科物种间THRSP基因CDS的长度存在差异,但碱基组成和CUB 一致性高,其编码蛋白的理化特性、二级结构、3-D 结构、基序组成模式、保守结构域、功能位点等高度相似,但呈现属间差异,与其他非牛科物种也具有一定的相似性;系统发育分析表明:牛科THRSP 蛋白序列聚为一大支,表明牛科该蛋白的功能更为相似。可见,THRSP基因在牛科及非牛科哺乳动物中功能保守,但牛科家畜间的功能更为相似。已有研究发现:THRSP基因主要表达于肝脏、乳腺、皮下脂肪等组织,它能调节脂肪酸合成基因的转录,参与这些组织中脂肪酸的从头合成过程[2-3,8,16-17]。敲低Spot14 小鼠的乳腺中三酰甘油(triacylglycerol,TAG)含量显著降低,表明Spot14 是泌乳期小鼠乳腺脂肪酸从头合成所必需[2];Spot14 还与泌乳期小鼠乳腺中中链脂肪酸(medium chain fatty acids,MCFA)的合成以及脂肪从头合成关键酶(fatty acid synthase,FASN)基因的表达相关[18];Spot14 对人乳腺癌细胞的增殖和乳脂合成具有促进作用,敲低Spot14 还会诱导细胞凋亡[19]。THRSP基因的表达还与牛肌肉[15]、脂肪[14]以及奶牛乳品质有关[7,9-10]。如:韩国牛THRSP基因的多态性位点与肌肉大理石纹评分、脂肪酸组成及胴体性状相关联[15];夏洛莱牛×荷斯坦牛肌内脂肪酸含量与THRSP基因的表达量正相关[14];与低乳脂率奶牛相比,高乳脂率奶牛乳腺THRSP的mRNA和蛋白的相对表达量显著升高,且过表达牛乳腺上皮细胞中的THRSP基因,TAG含量显著增加,并上调FASN、过氧化物酶体增殖物激活受体γ (peroxisome proliferator-activated receptor γ,PPARγ)和固醇调节元件结合蛋白1(sterol regulatory element binding protein 1,SREBP1)等重要脂肪生成酶基因的表达,提示THR-SP基因能通过调节脂代谢基因的表达进而调节乳脂合成过程,其在乳腺中的表达量可能是奶牛乳脂率的标志[7];THRSP基因在山羊皮下脂肪组织的表达量最高,在乳腺的相对表达水平极低,但泌乳高峰期的表达量显著高于干奶期,对调节山羊乳腺上皮细胞的脂肪酸从头合成和去饱和过程具有重要作用[8]。
本研究还表明:牛科物种THRSP 定位于细胞核,无跨膜区和信号肽序列,包含1 个Spot_14或Spot_14 超家族保守结构域,参与的生物学过程主要与脂类的生物合成、脂肪酸代谢、乙酰辅酶A 代谢、酰基辅酶A 的生物合成等相关,分子功能主要与脂肪酸合酶活性及蛋白质同源二聚化活性相关;与之互作的蛋白质分子主要与脂肪酸的合成和β-氧化、蛋白质代谢和转运、跨细胞膜或细胞器的物质转运、溶酶体与吞噬体的融合、囊泡介导的物质运输等生物学过程有关。因此,推测THRSP基因可能与牛科物种脂肪酸的合成过程相关,特别是参与了乳脂合成过程的调节。
4 结论
牛科物种THRSP基因转录区的结构、CDS长度、碱基组成及其编码产物的理化特性和结构有一定差异,但具有较高的一致性。牛科THRSP 蛋白均是定位于细胞核的亲水蛋白质,含有1 个Spot_14 或Spot_14 超家族保守结构域,参与的生物学过程主要与脂类的生物合成有关,分子功能主要与脂肪酸合酶活性及蛋白质同源二聚化活性相关。推测THRSP 蛋白参与了牛科物种脂合成(包括乳脂合成)的调节。

