A型塞内卡病毒序列元件与蛋白功能研究进展
南福龙,孟海蓝,李紫薇,,沙 洲,尼 博,刘拂晓
(1. 青岛农业大学动物医学院,山东 青岛 266109;2. 中国动物卫生与流行病学中心,山东 青岛 266032)
2007年在加拿大发现了一种可引起猪水疱性疾病的新发病毒[1]。该病毒最初被命名为塞内卡谷病毒,现重命名为A型塞内卡病毒(Senecavirus A,SVA)。随后,美国、巴西等多个国家相继发现该病毒感染的病例[2]。SVA多感染育肥猪和新生仔猪,无明显季节性,临床症状为病猪口鼻部、蹄部和黏膜处出现水疱样病变,后期发生溃疡甚至糜烂[2];病猪出现厌食、发热和呕吐等症状,与口蹄疫及其他水疱病症状十分相似;感染SVA的新生仔猪死亡率高达70%,部分痊愈仔猪生长发育不良,给我国乃至世界养猪业造成了较大的经济损失。我国于2015年首次发现SVA感染病例[3],其传播十分迅速。自2015年以来,我国湖北、黑龙江等多个省市均成功分离到SVA毒株[2]。2019年,Liu等[4]从广东省分离出4株SVA,经重组分析表明,SVA发生重组变异,具有遗传多样性。SVA较高的变异性和毒株的多样性是其难以防控的主要原因,目前SVA感染尚没有针对性的防治方案和有效的商品化疫苗,给我国养猪业带来较大的挑战。因此,本文对 SVA 的基因组结构和功能以及SVA结构蛋白和非结构蛋白的相关研究进展进行了总结,旨在为进一步研究SVA致病机制、建立新的防控方法提供参考。
1 病原学
SVA属于小RNA病毒科(Picornaviridae)、塞内卡病毒属(Senecavirus),且是该属唯一的成员[2]。早在2008年就有研究表明,SVA结构与其他小RNA病毒科成员相似[5]。SVA病毒粒子为二十面体对称的球形颗粒(图1),直径大约为27 nm,无囊膜[2]。SVA含有4种结构蛋白,基因组为单股正链、不分节段的RNA,全长约7 300 nt,无5′ 帽子结构,但具有3′ 多聚A尾。基因组包含约668 nt的5′ 非编码区(Untranslated region,UTR),1个多聚蛋白开放阅读框(Open reading frame,ORF)和长约68 nt的3′ UTR。
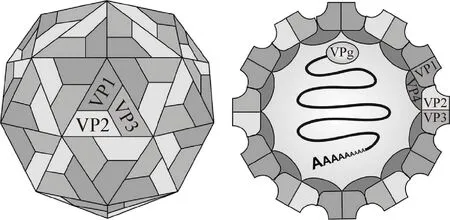
图1 SVA病毒粒子结构示意图[2]
2 SVA基因组序列元件
2.1 编码区序列 SVA多聚蛋白ORF长度为6 546 nt,可编码2 181个氨基酸。如图2所示,该ORF由L序列和中间体P1、P2、P3区组成。P1区可分为VP1、VP2、VP3和VP4 四个结构基因,P2区分为2A、2B和2C三种非结构基因,P3区分为3A、3B、3C和3D四种非结构基因。SVA基因组与其他小RNA病毒相似,都具有典型的“L-4-3-4”布局[2]。

图2 SVA基因组示意图[2]
2.2 5′ UTR序列 SVA基因组的5′ 末端与病毒连接蛋白(Viral genome-linked protein,VPg)共价相连。如图3所示,SVA 5′ UTR包含1个重要的内部核糖体进入位点(Internal ribosome entry site,IRES)[6],在病毒增殖过程中,该结构可通过与起始因子结合参与病毒RNA的翻译。小RNA病毒的

图3 SVA IRES示意图[6]
IRES区域含有多个复杂的二级结构,其中在起始密码子AUG上游的假结(Pseudoknot,PK)结构中,假结颈部I(Pseudoknot stem I,PKS-I)形成基序的完整性影响着核糖体40S亚基的结合,而假结颈部II(PKS-II)形成基序的突变,使IRES在核糖体P位点定位起始密码子的能力减弱[7]。SVA IRES区域可以招募核糖体,使病毒以不依赖于5′ 帽子结构的方式启动蛋白翻译[8],因此IRES对SVA启动蛋白翻译至关重要。
Willcocks等[9]研究表明,SVA 5′ UTR下游假结的PKS-II两个相邻位点突变(CC→GG),将严重降低IRES活性。然而,Liu等[10]通过微型基因组转染方法,证明了该结构的任何点突变都不会对IRES启动蛋白表达的活性产生显著影响;同时借助反向遗传技术,证明了假结受到破坏的IRES也不会对SVA反基因组复制产生干扰,但会导致重组SVA拯救失败。由此得出一种假说,即假结的破坏可能使SVA基因组的衣壳化信号受到抑制。Liu等[6]还证明,SVA能够完全耐受PKS-Ia和非配对间隔序列的突变,相比而言,PKS-Ib可能是SVA拯救所必需的保守序列 。
SVA含有1个丙型肝炎病毒(Hepatitis C virus,HCV)样IRES[11],主要包括2个区域(Domain,D),即DII和DIII,后者包括DIIIa、DIIIb、DIIIc、DIIId1、DIIId2、DIIIe和DIIIf,这是8种不同的茎环结构。Liu等[12]构建了8个各含有1个茎环突变的SVA cDNA克隆,结果显示,这些突变的cDNA克隆都不能单独拯救出具有复制能力的SVA,表明SVA IRES中每个假定的结构域对于病毒复制都是必不可少的,但在成对转染8个cDNA克隆的28个组合中,成功复活出4个野生型SVA,说明2个复制缺陷型不同的SVA在胞内发生了RNA重组,即SVA基因组之间存在重组现象,除此之外,该研究还提出了“选择性复制(Copy-choice)”的病毒重组模型。
SVA IRES假结与AUG之间有一段13 nt序列,Liu等[13]将SVA cDNA克隆的13 nt序列进行了逐一敲除,试图拯救核苷酸缺陷型重组病毒,结果表明,SVA的13 nt序列最多容忍5个核苷酸的缺失,即便如此,由于RNA病毒的不保真特性,只有1 nt和2 nt缺失型SVA可以稳定传代,其他3株拯救的病毒在13 nt序列处皆有核苷酸突变或增加。该研究揭示了1、2和3 nt缺失型SVA的复制特性,并证明当13 nt序列缺失2个核苷酸时,已经对病毒的生长造成影响。该研究最终提出假想模型,即SVA IRES-AUG距离影响病毒起始密码子在核糖体中的准确定位。
SVA的翻译起始于IRES近端的起始密码子,而起始密码子通过直接定位在核糖体亚基上启动翻译。Wang等[14]研究发现,从含有ATG侧翼为3个核苷酸倍数的外源序列的cDNA克隆中可拯救出具有复制能力的重组SVA,不是3的倍数的外源序列则导致病毒拯救失败,可能是IRES近端的人工AUG取代真正的AUG,进而引导多聚蛋白翻译,最终致使病毒拯救失败。
小RNA病毒5′ UTR通常有1个二级结构不同的功能结构域,其对病毒的复制至关重要。Meng等[15]通过网络服务器对SVA 5′ UTR进行分析,建立了其RNA的二级结构,并利用反向遗传技术对其进行基因修饰,以拯救重组病毒,结果发现大部分基序都不能忍受额外的基因修饰,说明该结构可能是SVA合成RNA等机制的重要顺式作用元件;同时该研究还发现,SVA可以修复其5′ 末端的核苷酸缺陷,其自我修复的极限是5个连续的核苷酸。
2.3 3′ UTR序列 SVA的3′ UTR高度结构化,下游连接多聚A尾结构,长度较短。多聚A尾可以避免病毒在细胞中被核酶降解,并且对病毒的感染起着非常关键的作用。结构分析表明,肠道病毒的3′ UTR吻环结构具有1个同轴螺旋结构域,该螺旋结构域与其他各个结构域形成复制原点的整体结构,启动负链RNA的合成,若将相关基序突变,则会扰乱吻环间的相互作用,从而抑制肠道病毒合成RNA[16]。SVA 3′ UTR具有2种高级RNA结构:吻环和H型假结,但这2种结构不能在3′ UTR共存。Liu等[17]利用反向遗传技术对SVA 3′ UTR中的吻环或假结基序进行一个或多个点突变,结果表明,若在3′ UTR中存在假定的吻环结构,其对于SVA的复制是非必要的;若存在假结结构,其突变将对SVA的复制产生“致命”影响。
3 SVA蛋白
3.1 结构蛋白 SVA的结构蛋白,也称为衣壳蛋白,由VP1、VP2、VP3和VP4 四种蛋白组成,对应的基因分布见图2,结构蛋白的功能见表1。4种结构蛋白主要参与病毒粒子的包装,结构蛋白是SVA主要的保护性抗原,存在中和表位,可刺激机体产生中和抗体,常作为血清学检测方法和疫苗研发的靶蛋白;此外,结构蛋白还可以与细胞受体结合,介导病毒进入细胞,使病毒对某些有神经内分泌特征的肿瘤细胞产生选择性细胞毒性[18]。

表1 SVA结构蛋白的功能
VP1、VP2和VP3蛋白分布在病毒颗粒的最外层,其中,VP1蛋白具有最强的免疫原性,可用于病毒血清学鉴定[5,18]。此外,VP1蛋白含有潜在的B细胞表位和T细胞表位,且高抗原指数的21GELAAP26完全暴露在VP1蛋白表面,可能是B细胞重要的线性表位[19]。Zhao等[20]通过SVA的VP1蛋白对其遗传进化进行分析,鉴定出4个核苷酸突变(A65T、N94S、A114P和S229G),并假设其与当前我国发现的SVA致病性的改变有关。研究表明,SVA的非结构蛋白参与细胞自噬,并利用自噬机制促进病毒感染[21]。但最近的一项研究表明,SVA的结构蛋白VP1同样参与激活信号通路以诱导自噬[22]。
VP2蛋白与血清中的抗体具有较高的结合反应性[23]。有研究评估了猪对SVA感染的细胞免疫反应,发现相比于VP1和VP3蛋白,VP2蛋白可引起更高的T细胞反应,表明VP2蛋白含有免疫显性T细胞表位[24]。炭疽毒素受体1(Anthrax toxin receptor 1,ANXTR1)是SVA的高亲和力细胞受体,可与衣壳表面的关键残基相互作用,研究发现,大部分ANXTR1分布于VP2蛋白表面[25]。
VP3蛋白可以在SVA感染的早期阶段诱导中和抗体的产生[24]。VP3蛋白表面具有线性表位,但迄今为止只报道过其B细胞表位[26]。研究表明,VP3蛋白还可与其他蛋白协同作用以参与细胞自噬[22,27]。
VP4蛋白位于蛋白质衣壳内部,在4种结构蛋白中相对保守。小RNA病毒科的一些成员在感染期间会形成天然的空颗粒,但由于VP0蛋白无法正确切割或VP4蛋白可能与衣壳解离等原因,这些空颗粒稳定性较差。相反,SVA VP4蛋白的终端特异性β链部分附着在VP2蛋白上稳定其结构,并裂解VP0蛋白产生VP2和VP4蛋白,进而组装成稳定的前体衣壳,表明SVA前体衣壳可作为病毒样颗粒或癌症靶向药物递送的工具[8]。SVA是一种无囊膜病毒,其感染细胞不依赖于膜融合,而是通过其他机制将其基因组运送到细胞中,VP4蛋白在此过程中起着主要作用。现已证明,同属小RNA病毒科的人鼻病毒,其小豆蔻酰化衣壳蛋白VP4从病毒中释放出来并与细胞膜相互作用,导致细胞膜上形成孔隙,提高了细胞膜的通透性,进而将病毒RNA基因组递送到细胞质中以启动病毒复制[28]。
3.2 非结构蛋白 SVA的非结构蛋白有L蛋白、2A、2B、2C、3A、3B、3C和3D 八种,对应的基因分布见图2。非结构蛋白比结构蛋白更保守,不与病毒粒子结合,主要参与病毒复制、基因表达调控和与宿主之间的相互作用[5],不同非结构蛋白的功能见表2。
表2 SVA非结构蛋白的功能
L蛋白是小RNA病毒最先表达的蛋白。在口蹄疫病毒和心病毒等小RNA病毒中,L蛋白被证明是具有特殊“锌指”构象的木瓜蛋白酶样半胱氨酸蛋白酶,主要参与抑制宿主细胞蛋白质的合成,切割真核起始因子,调节宿主细胞中病毒的转录和翻译等过程[29-30]。SVA L蛋白的不同之处在于其缺乏蛋白水解活性所需的催化残基,且没有“锌指”构象或酪氨酸磷酸化基序,表明其功能可能与口蹄疫病毒和心病毒不同,确切功能仍需进一步研究[5]。
SVA 2A蛋白是只有9个氨基酸的短肽,其末端存在NPG/P保守基序,具有核糖体跳跃功能,即多肽合成的中断发生在P1-2A和2BC-P3之间。若2A蛋白附着在VP1蛋白的羧基末端或与VP1蛋白分离,可能会导致多聚蛋白合成中断,但尚未有研究证明2A蛋白是否附着在VP1蛋白的羧基末端或与VP1蛋白分离[5]。
小RNA病毒2B蛋白具有在细胞感染过程中增强细胞膜通透性的能力,SVA 2B蛋白的一级结构与其他已知的小RNA病毒的一级结构不同,但其二级结构与其他小RNA病毒2B蛋白的二级结构非常相似,因此推测SVA 2B蛋白可作为病毒孔蛋白,在增强细胞膜通透性方面发挥重要作用。除此之外,2B蛋白不切割线粒体抗病毒信号蛋白(Mitochondrial antiviral signaling protein,MAVS),也不诱导细胞凋亡,但可与MAVS相互作用以降低MAVS的表达,导致I型干扰素的表达量降低[31]。
2C蛋白是最保守的病毒非结构蛋白之一,在病毒生命周期和免疫逃逸中起着不可或缺的作用[32]。2C蛋白可以靶向定位于线粒体,使细胞色素C释放到细胞质中,从而在一系列信号级联反应中激活半胱天冬酶-9和半胱天冬酶-3以诱导细胞凋亡的发生[33]。2C蛋白还可以借助半胱天冬酶介导的蛋白降解,靶向作用于视黄酸诱导基因Ⅰ(Retinoic acid-inducible gene-I,RIG-I),并阻断干扰素诱导基因56(Interferon-stimulated gene 56,ISG56)和干扰素β(Interferon-β,IFN-β)的转录以削弱宿主先天免疫反应[34]。
小RNA病毒的3A蛋白高度分化,在不同属的小RNA病毒复制周期中发挥不同的功能[35]。关于SVA 3A蛋白的确切功能尚不清楚,但最近有研究表明,口蹄疫病毒的3A蛋白可以通过自噬抑制IFN信号传导并抑制宿主先天性抗病毒反应[36]。
SVA 3B区域编码一种VPg蛋白,其经尿苷酰化后可作为病毒RNA合成的引物[37]。先前的研究证实,小RNA病毒基因组的ORF中存在顺式作用复制元件(cis-acting replication element,cre),而口蹄疫病毒的cre位于5′ UTR[38]。cre是一种茎环结构,在环结构靠近5′末端的那一半环序列中具有保守的 AAACA(或 AAACG)基序,AAACA基序可作为将U残基添加到VPg的模板,从而产生VPg-pUpU,其将作为小RNA病毒合成RNA的引物[39]。Meng等[40]研究发现,SVA的VP2 ORF中存在1个含有AAACA的茎环,且结构上与脑心肌炎病毒的cre高度相似,通过试验证明此茎环在功能上与其他小RNA病毒的cre相似。
3C蛋白是一种多功能半胱氨酸蛋白酶,含有半胱氨酸-组氨酸-天冬氨酸催化三联体,其与蛋白酶和RNA结合活性相关的结构是保守的,有助于病毒多聚蛋白的切割和SVA复制过程中RNA复制复合物的组装[41]。此外,SVA 3C蛋白对抑制宿主先天免疫至关重要,可调节宿主不同的信号通路和细胞死亡形式[33,42],抑制先天免疫信号级联反应[34]和宿主翻译机制[43]等多种途径,进而促进病毒的复制。
3D蛋白相对保守,可以与3A、3B和3C蛋白相互作用,调节病毒复制和尿苷酰化。3D蛋白本质是病毒的RNA依赖性RNA聚合酶(RNA-dependent RNA polymerase,RdRp),在病毒复制和mRNA合成中发挥重要作用[44]。RdRp的保真性较差,缺乏校对活性,使得病毒在复制过程中较易发生突变和重组,这反而有助于SVA快速适应不断变化的环境[45]。功能上,3D蛋白与VPg结合形成3Dpol-VPg复合物,促使VPg 尿苷酰化形成VPg-pUpU,随后作为触发全长基因组或反义基因组合成的引物[46]。
4 小结与展望
位于SVA 5′ UTR的IRES和顺式作用元件对病毒启动蛋白翻译和基因组复制至关重要,3′ UTR中的多聚A尾对病毒感染起着关键作用,且其中存在的二级结构对病毒的复制有“致命”影响。SVA蛋白在病毒增殖过程中的功能不同,结构蛋白除了形成病毒衣壳和保护病毒基因组免受恶劣环境的影响外,对粘附和侵袭宿主细胞也是必不可少的;非结构蛋白参与调节病毒复制,并通过不同途径诱导细胞死亡,抑制宿主的免疫应答,还可与其他蛋白协同调控信号通路。识别和理解SVA蛋白及其基因组元件的功能对研究病毒的复制、免疫逃避和致病性的分子机制十分必要,可为进一步了解SVA基因组结构和蛋白功能与病毒感染和发病机制的关系提供理论依据。
目前,对SVA蛋白及其基因组元件的功能有了初步了解,但仍有许多蛋白的功能仍需进一步阐明,例如:(1)系统分析SVA蛋白与宿主细胞之间的相互作用网络以发现新的信号通路;(2)研究结构蛋白在病毒组装和释放中的作用并揭示它们与细胞膜和囊泡运输系统的相互作用;(3)解析非结构蛋白的晶体结构,特别是2C和3D蛋白,其关键催化基序的鉴定有助于阐明非结构蛋白参与病毒复制和转录的机制;(4)鉴定SVA的其他受体,已知SVA具有多种组织嗜性,但除了ANTXR1之外,病毒进入细胞的其他受体尚未明确,鉴定SVA感染的靶细胞和受体对于理解其多组织嗜性至关重要。这些机制的阐明将为SVA抗病毒制剂的开发提供理论支持。

