穿心莲内酯对耐甲氧西林金黄色葡萄球菌关键耐药蛋白PBP2a的抑制作用
李彩霞,刘盼盼,陈晓慧,罗小凤,王桂琴
(宁夏大学动物科技学院,宁夏 银川 750021)
耐药菌株引起的高发病率和病死率已经严重威胁到人和动物的生命安全,并已成为全球关注的公共卫生问题[1]。金黄色葡萄球菌(Staphylococcusaureus)是引起奶牛乳房炎主要的致病菌之一,抗菌药物的使用仍为奶牛乳房炎主要治疗手段。我国奶牛乳房炎金黄色葡萄球菌分离株耐药性严重,80%~90%菌株对β-内酰胺类药物耐药,远远高于其他国家,部分省区90%以上金黄色葡萄球菌分离株耐药[2]。地区不同,耐药也有很大区别。从新疆地区分离出的菌株对青霉素耐药率高达93.0%,浙江地区分离出的菌株对青霉素耐药率为77.35%[3]。
耐甲氧西林金黄色葡萄球菌(methicillin-resistantS.aureus,MRSA)的耐药机制包括靶位改变、钝化酶产生、主动外排等,其中抗生素作用靶位的改变是最主要的耐药机制。细菌细胞壁的肽聚糖合成是由青霉素结合蛋白(penicillin binding proteins,PBPs)催化完成,金黄色葡萄球菌通常可产生4种PBPs,即PBP1、PBP2、PBP3和PBP4,它们参与细胞壁的合成并在细菌细胞周期中发挥至关重要的作用。PBPs具有转肽酶(transpeptidase,TPase)活性和转糖基酶(glycosyltransferase,TGase)活性,而β-内酰胺类药物能与PBPs转肽酶功能域选择性结合,使TPase失活[4]。MRSA菌株中存在可移动遗传元件葡萄球菌盒式染色体mec(SCCmec),该元件携带mecA基因,编码具有转肽酶活性的青霉素结合蛋白2a(penicillin binding protein 2a,PBP2a)蛋白[5]。PBP2a蛋白包含3个功能区,其中N末端跨膜区(1~23 aa)将PBP2a固定在细胞膜表面,非青霉素结合区(24~326 aa)主要负责蛋白质的延伸和固定,转肽酶区(327~668 aa)发挥转肽酶活性。PBP2a的转肽酶区含有β-内酰胺类抗生素作用的低亲和力位点,与β-内酰胺类药物难以结合,当其他PBPs被抑制时,PBP2a可代替其功能,完成肽聚糖合成,维持细菌正常生长。
穿心莲内酯(andrographolide,AP)是一类多靶点的萜类小分子化合物,具有多种生物活性,如抗炎和抗菌等作用[6-7]。AP与β-内酰胺类抗生素联合使用可以增强MRSA对β-内酰胺类抗生素的敏感性[8]。而PBP2a为MRSA关键耐药蛋白,那么是否AP通过影响PBP2a表达及功能进而使金黄色葡萄球菌对β-内酰胺类药物增敏?为探究这一科学问题,本研究首先通过生物信息学及分子模拟等手段分析PBP2a的蛋白分子特性及AP与PBP2a蛋白的作用及其结合模式,探究AP对PBP2a蛋白功能的影响;随后,测定了AP作用下PBP2a转录水平变化;通过原核表达纯化PBP2a蛋白,制备多克隆抗体,检测AP对PBP2a表达量的影响。
1 材料与方法
1.1 菌株
金黄色葡萄球菌参考菌株ATCC33591购自中国药品与生物制品检定所,试验菌株为本实验室从宁夏不同地区奶牛场乳房炎乳样中分离出的MRSA(WLD10、WLD1、XF2)。
1.2 实验动物
8周龄成年新西兰大白兔,购自四川里来思诺生物科技有限公司。
1.3 药品与试剂
穿心莲内酯,纯度99.89%,购自美国Med Chem Express公司;TRIzol试剂购自日本TaKaRa公司;二甲基亚砜(DMSO)和0.2 μm PVDF膜均购自德国Merck公司;荧光定量试剂购自美国ABclonal公司;内切酶BamHⅠ和EcoRⅠ购自纽英伦生物技术(北京)有限公司;ECL化学发光显色液试剂购自上海雅酶生物医药科技有限公司;辣根过氧化物(HRP)标记山羊抗兔IgG购自亚科因(武汉)生物技术有限公司;异丙基-D-硫代半乳糖苷(IPTG)购自北京索莱宝科技有限公司;Ni2+亲和层析柱购于上海生工生物工程有限公司;BCA蛋白含量检测试剂购自江苏凯基生物技术股份有限公司;脱脂奶粉购自美国BD公司。
1.4 荧光定量PCR检测AP对mecA基因转录水平的影响
选择4株MRSA菌株ATCC33591、WLD10、WLD1、XF2划线培养,挑取单菌落过夜培养至对数生长期,按1∶100的比例稀释,然后分别加至终浓度为0、64、128、256 μg/mL的AP,继续培养12 h。使用TRIzol法提取总mRNA,使用逆转录试剂盒将mRNA转录为cDNA。以gyrB为内参基因,使用SYBR Green Real-time荧光PCR预混液在20 μL反应体系进行qPCR反应。利用2-ΔΔCt法对结果进行处理[9],引物序列信息见表1。
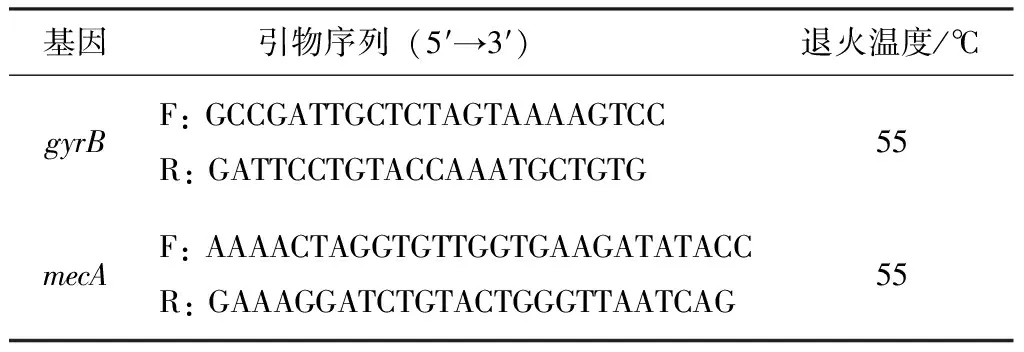
表1 引物序列信息
1.5 蛋白分子结构分析
利用在线软件NPS@:SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)对PBP2a蛋白二级结构进行预测和分析。采用在线服务器Consurf Web Serve(http://consurf.tau.ac.il/)进行同源性建模和保守性分析[10]。利用PyMOL软件将保守性氨基酸映射到PBP2a蛋白的三维结构上,并分析氨基酸保守性与蛋白结构和功能的联系。
1.6 分子对接
采用SWISS-MODEL进行同源建模,获得PBP2a蛋白的三级结构。将能量最小化的优势构象作为PBP2a蛋白对接的初始构象。AP小分子结构文件来源于PubChem数据库(https://pubchem.ncbi.nlm.nih.gov/),随后利用PyMOL软件将结构文件转化为PDB格式。使用AutoDock对蛋白去水、加氢、计算电荷以及设置小分子的旋转键等预处理后进行半柔性分子对接。最终选取兼顾打分和构象契合的最优结果,使用BIOVIR Discovery Studio 2020进行可视化[11]。
1.7 动力学模拟
进一步验证AP与PBP2a蛋白的结合稳定性。分子对接后,以PBP2a-AP复合物作为初始构象,使用Gromacs 2020版本对复合物进行分子动力学模拟(molecular dynamics simulation,MD)[12]。此处,MD模拟的分子力场选择为OPLS3e,并使用TIP3水模型对系统进行溶剂化,通过添加离子对系统电荷进行中和,整个系统的能量最小化是使用全原子型OPLS3e力场来实现[13]。水分子的几何结构、重原子的键长和键角都是通过SHAKE算法来约束[14]。通过应用周期性边界条件对连续系统进行模拟,并通过粒子网格Ewald方法维持长程静电[15]。在进行动力学模拟之前先对模型进行优化:(1)限制溶剂,进行能量最小化;(2)没有限制,能量最小化;(3)温度10 K,Berendsen热力学方法,运行12 ps,每1 ps重新采样1次;(4)NPT系综,温度300 K,压力1.01×105Pa,正常压力释放系数,运行24 ps[16];(5)MD成品:前期准备结束后,以1.2 fs的时间进行100 ns的运行,每25 ps进行一次轨迹记录,总计记录4 000帧。计算PBP2a蛋白及复合物主链原子的均方根偏差(RMSD),衡量复合物体系的稳定性;以均方根波动(RMSF)反映蛋白氨基酸残基的结构柔性;通过回旋半径(Rg)考量AP对PBP2a蛋白结构折叠紧密度的影响,捕捉PBP2a与AP间氢键数目及持续时间主链原子的RMSD,以了解蛋白质-配体相互作用的性质。
1.8 PBP2a蛋白的原核表达
1.8.1mecA基因的PCR扩增
从NCBI基因数据库中下载mecA基因序列,根据基因序列设计引物,并送至生工生物工程股份有限公司合成。在引物两端加入限制性酶切位点保护碱基,目的基因上游引物序列为:5′-CGGGATCCACTATTGATGCTAAAGTTCA-3′,下游引物序列为:5′-GGAATTCGCAACCCACGTTACCGGATTG-3′,Prime STAR Max DNA Polymerase 进行PCR扩增,并胶回收目的片段。引物序列下划线处分别为BamHⅠ和EcoRⅠ酶切位点。
1.8.2 重组质粒pET28a-mecA的构建
将回收目的片段及质粒pET28a经BamHⅠ和EcoRⅠ双酶切,酶切产物回收后使用T4 DNA连接酶16 ℃过夜连接。转化至DH5α感受态,经卡那霉素筛选及PCR鉴定获得阳性菌落,提取质粒进行酶切鉴定,鉴定正确的质粒送至生工生物工程股份有限公司进行测序验证。
1.8.3 重组质粒转化
将测序正确的重组质粒pET28a-mecA转化至BL21(DE3)感受态细胞,经卡那霉素抗性筛选,PCR鉴定获得阳性菌落,于-80 ℃保存备用。
1.8.4 重组蛋白PBP2a的诱导表达
将新鲜的单菌落挑取至5 mL含有卡那霉素(50 μg/mL)的LB过夜培养,1∶100转接至100 mL卡那霉素抗性LB。37 ℃ 200 r/min震荡培养3 h,当菌液OD600值为0.6~0.8时,加入IPTG(1 mmoL,1∶1 000)诱导剂,160 r/min,25 ℃诱导24 h后进行SDS-PAGE分析。
1.8.5 重组蛋白的鉴定
同1.8.4方法制备样品经SDS-PAGE分离后,120 V转印PVDF膜,用5%脱脂奶4 ℃封闭过夜,一抗为抗His标签抗体(1∶2 000稀释),孵育1 h后TBST缓冲液洗4次,每次10 min;二抗为HRP标记的山羊抗兔IgG(1∶2 000稀释),孵育1 h,TBST洗涤4次,ECL显色。
1.9 PBP2a多克隆抗体的制备
1.9.1 重组蛋白纯化
同1.8.4方法小量诱导表达目的蛋白,离心取上清液和沉淀进行SDS-PAGE鉴定,以相同条件大量诱导表达重组蛋白,使用Ni2+亲和层析柱进行纯化。然后进行最适洗涤浓度确定,20、50、75、100、150、250 mmol/L咪唑缓冲液洗涤,确定目的蛋白不被洗脱的最大咪唑浓度为最佳洗涤浓度。
1.9.2 免疫程序
第一次免疫使用弗氏完全佐剂与0.2 mg的重组蛋白进行等体积乳化,背部皮下多点注射,在一免后14 d进行二免,28 d进行三免,二免、三免使用弗氏不完全佐剂。
1.9.3 蛋白质免疫印迹检测抗体特异性
含有pET28a-mecA的质粒并诱导后的大肠杆菌作为对照,同时使用MRSA菌株制备样品,以制备的抗重组蛋白PBP2a的多克隆抗体作为一抗,HRP标记的山羊抗兔IgG作为二抗,鉴定多克隆抗体对重组PBP2a蛋白和MRSA源PBP2a的特异性。
1.10 蛋白质免疫印迹检测AP作用下PBP2a蛋白含量的变化
过夜培养MRSA菌株WLD10,1∶100接种至TSB培养基,培养至对数生长期,加入AP使各管浓度分别为0、64、128、256 μg/mL,37 ℃、200 r/min过夜培养。次日将菌液浊度调整至OD600=1.0,用超声仪破碎。使用BCA法测定蛋白含量,将蛋白总含量稀释一致后使用5×loading Buffer进行金属浴加热10 min,样品制备完成进行SDS-PAGE,转膜,封闭,孵育一抗(PBP2a蛋白多克隆抗体),孵育二抗(HRP标记的山羊抗兔IgG),ECL显影检测蛋白表达量。
2 结果与分析
2.1 荧光定量PCR分析mecA基因转录水平
经64、128、256 μg/mL浓度的AP处理,MRSA菌株ATCC33591、WLD10、WLD1、XF2中mecA基因的转录水平均显著下调(P<0.01),且随着AP浓度的升高,4株菌mecA的转录水平均逐渐降低(图1)。

**表示与对照组(0 μg/mL AP)比较P<0.01。下同。
2.2 PBP2a蛋白分子特征分析
二级结构预测分析显示,PBP2a蛋白为单体及二聚体结构,含有33.98% α螺旋、18.11% 延伸链、7.93% β转角、39.97% 无规则卷曲。利用Consurf服务器进行保守性分析,通过PyMOL软件将保守性和溶剂可及性赋值投影至三维结构上,从而关联保守性氨基酸与结构和功能的关系。结果显示,Met1-Lys3、Asp156-Asn158、Ser261-Lys265、Thr354-Leu360、Val361-Tyr369、Ser462-Arg469、Leu514-Gly520、Ile595-Lys604、Gln613-Ile618区域和GLU 284、ASP288残基高度保守。氨基酸溶剂可及性通过卷积神经网络算法预测,其中包含多个埋藏于蛋白内部用于维持结构特异性的高度保守性残基,它们分别是Val45、Tyr46、Trp127、Ile154、Leu264、Tyr373、Phe395、Ala471等氨基酸残基;Met1、Arg65、Thr103、Gly106、Ile132、Arg150、Gly151、ASP288、Gln333、Ser437等保守残基位于PBP2a蛋白表面,可能在生物学功能发挥中起到至关重要的作用。
2.3 AP和PBP2a蛋白的结合模式分析
对接结果的3D、2D结合构象可视化见图2,AP与蛋白PBP2a的GLU170、GLU239和THR238残基形成4个氢键,并通过烷基间分子作用力与残基VAL277、PRO258结合、与残基TYR373形成π-烷基堆积作用力,结合能显示PBP2a蛋白与AP小分子具有较强结合能力。

图2 分子对接结果3D(A)、2D(B)展示
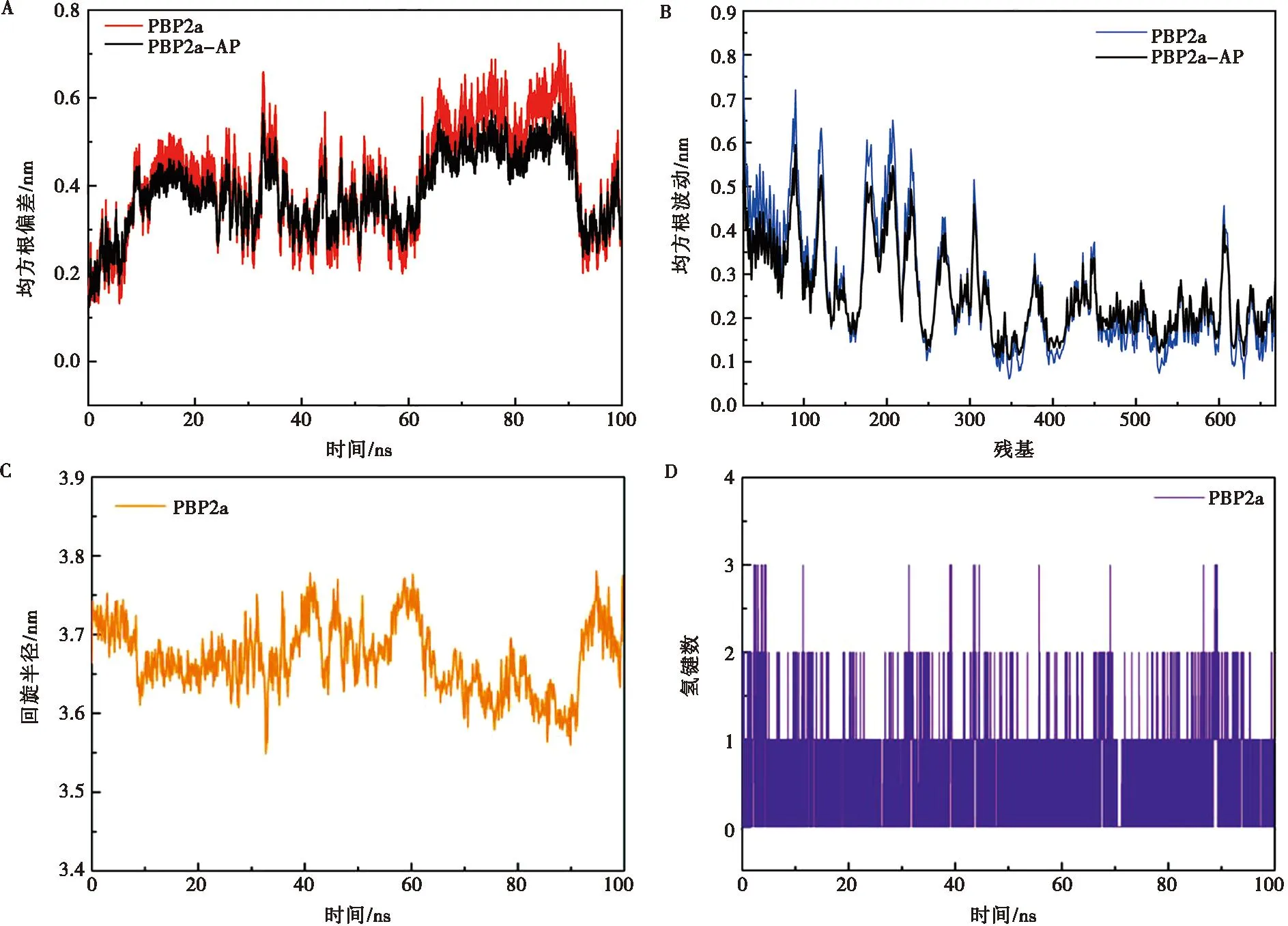
A.PBP2a-AP复合物的RMSD;B.表示复合物的RMSF;C.PBP2a-AP配合物的Rg;D.表示PBP2a和AP的氢键数。
2.4 分子动力学模拟
为进一步研究AP分子与PBP2a蛋白的相互作用,本研究对PBP2a-AP复合物进行了100 ns的分子动力学模拟。如图3A所示,复合物的RMSD小于4.5Å,而且复合物在较短的(18 ns)时间内达到了动态平衡,这表明AP与靶蛋白PBP2a能够形成稳定的复合物。根据图3B可知,大部分的氨基酸构象变化较小,AP分子可通过结合降低PBP2a蛋白中残基的结构柔性。这也是复合物RMSD变化较小的主要原因。氢键在复合物形成中的作用也很关键,形成的氢键越多,两者将具有更强的结合活性,再结合Rg,Rg越小,表明AP对蛋白结构折叠的影响越轻微。从图3C和图3D可知,小分子与蛋白结合良好,能够形成稳定的复合物从而实现小分子其活性功能。在整个分子动力学模拟过程,活性位点残基的结合情况可反映PBP2a蛋白与配体AP间更详尽的能量细节。根据结合能(表2)可以看到,蛋白的GLU145,GLU170,ASN164,ASP275,VAL277,GLU284,ASP288,ASP295,TYR373等氨基酸贡献的结合自由能最为突出,特别是与VAL277结合能达到 -6.78 kJ/mol,位于PBP2a蛋白的功能区氨基酸TYR373结合自由能达到-5.199 4 kJ/mol(图4),对稳定蛋白口袋中的小分子有着重要作用,另一方面也表明了分子与蛋白位点存在的这些作用可有效促使小分子牢牢地锚定在蛋白活性位点,从而形成稳定的复合物。
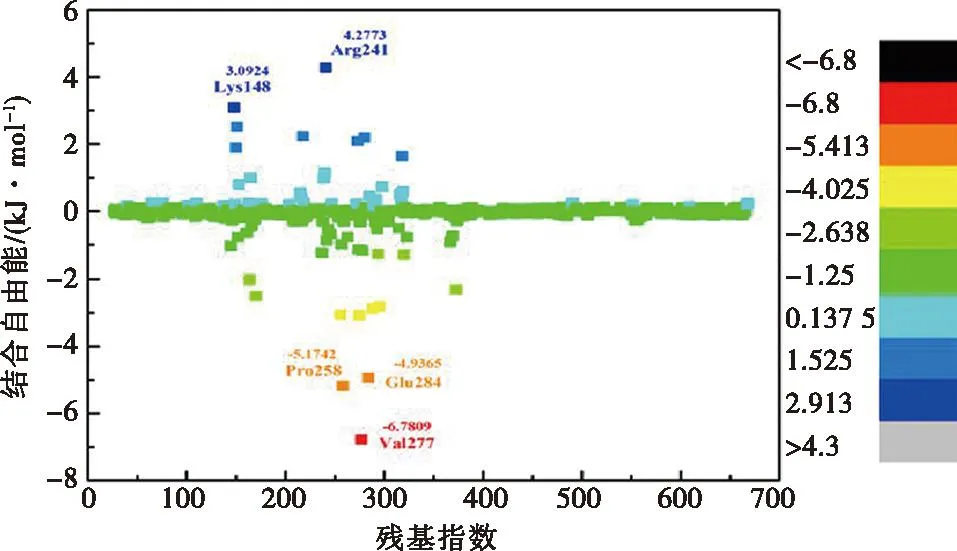
图4 与配体相互作用的每个蛋白质残基的结合自由能的分解
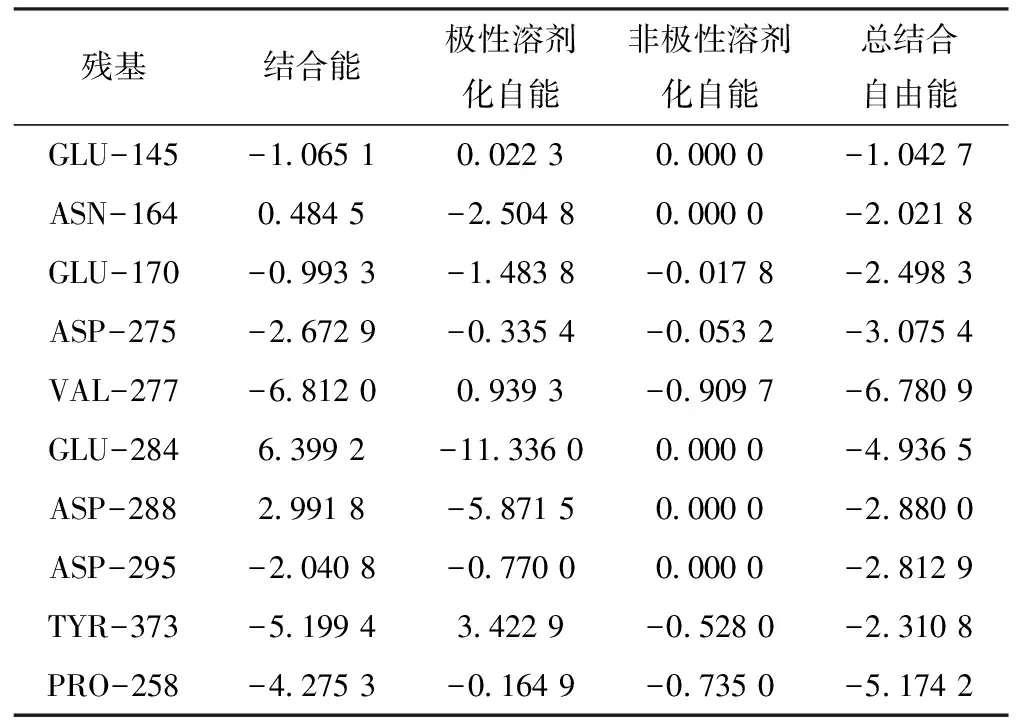
表2 结合能及其所选生物活性分子的组成能
2.5 PCR扩增mecA片段
mecA基因PCR扩增所得产物经1%琼脂糖凝胶电泳,结果如图5所示,在1 000 bp附近存在条带,与预期1 026 bp符合。

M. DL2000 Marker;1.PCR扩增产物。
2.6 重组质粒pET28a-mecA的酶切鉴定
提取的重组质粒经BamHⅠ和EcoRⅠ酶切鉴定结果如图6所示。重组质粒的mecA基因序列未出现突变和移码,表明原核表达载体pET28a-mecA成功构建。

M. DL10000 Marker;1.重组质粒双酶切;2.重组质粒。
2.7 重组蛋白PBP2a的表达
通过不断优化诱导表达条件,发现在1 mmol/L的IPTG浓度下,低温25 ℃诱导24 h可获得目的蛋白。经蛋白质免疫印迹检测PBP2a蛋白的N端His标签,特异性反应条带约为38 kDa,见图7,与预期结果相符。

M. 蛋白Marker;1.PBP2a重组蛋白。
2.8 重组蛋白PBP2a的纯化
重组蛋白经IPTG诱导,用超声破碎仪破碎后,离心分别取上清液和沉淀进行SDS-PAGE鉴定,发现沉淀和上清液都存在目的蛋白,取上清液经Ni2+纯化柱进行纯化,最佳洗涤的咪唑浓度为75 mmol/L,用150 mmol/L的咪唑对目的蛋白进行洗脱,获得大小约38 kDa的目的蛋白(图8)。

M. 蛋白Marker;1~6.75 mmol/L咪唑洗涤蛋白;7~9.150 mmol/L咪唑洗脱蛋白。
2.9 蛋白质免疫印迹分析PBP2a蛋白多克隆抗体的特异性
使用制备的抗重组蛋白PBP2a的多克隆抗体对PBP2a重组蛋白及MRSA菌株中的PBP2a蛋白进行免疫反应性分析。如图9所示,在38 kDa左右出现特异性目的条带,表明该多克隆抗体可与原核表达的PBP2a蛋白及MRSA菌株中的PBP2a蛋白发生特异性反应,进而实现对PBP2a蛋白表达量的检测。

M. 蛋白Marker;1.重组蛋白PBP2a;2.MRSA菌内PBP2a蛋白。
2.10 蛋白质免疫印迹检测AP作用下PBP2a蛋白含量的变化
将经0、64、128、256 μg/mL的AP处理的MRSA菌株,标定总蛋白含量制样,蛋白质免疫印迹测定PBP2a的蛋白含量变化。结果如图10所示,在38 kDa左右处出现目的条带,与未处理的MRSA菌株内PBP2a蛋白相比,经处理的PBP2a蛋白表达量显著降低(P<0.01),且随着AP浓度的升高,PBP2a的表达量逐渐降低。

图10 蛋白质免疫印迹检测PBP2a蛋白表达量
3 讨论
细菌耐药性已成为威胁全球公共卫生健康的重要因素之一,革兰阳性和阴性菌中的多重耐药模式导致传统抗菌剂难以治疗甚至无法治疗[17]。 MRSA几乎耐所有的β-内酰胺类药物,且对多种抗生素耐药,引起的感染也极难治疗。因此,开发有效的抗MRSA感染治疗策略或抑制剂对治疗至关重要。随着对植物抗微生物成分的探索,一些候选药物表现出潜在的抑菌活性,被认为是安全、无毒、不易产生副作用的天然化合物,因此被广泛应用于研究[18-19]。
PBPs在细菌细胞壁的肽聚糖合成中发挥作用[20]。 PBPs主要参与肽铰链桥的形成,催化邻近糖链,肽侧链之间的特定交联来辅助细胞壁的合成。当β-内酰胺类抗生素使PBPs失去活性时,PBP2a却能代替其功能继续完成肽聚糖的合成,使细菌可以继续生长,并呈现高度耐药性[21]。因此,抑制PBP2a的功能,进而抑制细菌细胞壁肽聚糖的合成是使MRSA对β-内酰胺类增敏的可能途径。
AP是穿心莲的主要活性成份,具有多种生物学效应,其抗炎、抗肿瘤、抗病毒、抗菌并治疗多种疾病,在体内和体外有广泛的治疗作用,具有大量药理学靶点[22]。本试验通过荧光定量PCR测定发现,不同浓度AP处理的MRSA菌株mecA基因的转录水平均有下调,且具有浓度依赖;进一步通过原核表达及抗体制备获得PBP2a重组蛋白及其多克隆抗体,经蛋白质免疫印迹验证,表明AP可以通过抑制mecA基因的转录和表达来抑制PBP2a蛋白。
分子模拟在很多研究中被用于筛选抗金黄色葡萄球菌的有效分子[23-24]。本试验采用半柔性分子对接及全原子动力学模拟来探究AP与PBP2a蛋白的相互作用及结合模式,其中分子对接结果显示,AP会与PBP2a形成4个氢键,还存在烷基间分子作用力,同时,会在π-烷基堆积作用力下形成复合物,这些氨基酸残基与AP结合可能会损害其生物学功能,且氢键的出现对复合物的形成至关重要,几乎起到决定性作用[22]。其中TYR373残基位于PBP2a蛋白的功能区,AP与TYR373残基的结合会使PBP2a蛋白无法发挥其转肽酶功能。这些结果都说明AP对PBP2a蛋白有一定抑制潜力。分子动力学模拟被认为是一项分析蛋白质配体相互作用的关键技术,为了分析AP和PBP2a蛋白结合的可靠性,将AP-PBP2a复合物进行了分子动力学模拟。分析结果发现复合物的RMSD小于4.5Å,复合物RMSD变化较小,Rg也在比较小的范围内波动,这些都说明AP-PBP2a复合物比较稳定。再结合活性位点残基的结合情况分析,小分子与蛋白位点的氨基酸存在很好的氢键以及疏水相互作用,其中残基GLU284,ASP288是高度保守的残基,AP与其结合可能使PBP2a蛋白的生物学功能受到影响;与VAL277残基的结合能达到 -6.78 kJ/mol,结合能越低对稳定蛋白口袋中的小分子有着重要作用,与功能区氨基酸TYR373结合能达到-5.199 4 kJ/mol。以上分析说明AP对MRSA主要耐药蛋白PBP2a存在抑制作用,并显示出巨大的潜力。
综上,本研究从分子模拟的角度提出了AP与PBP2a蛋白存在结合效应,揭示了AP对PBP2a的抑制潜力。同时发现,AP下调mecA转录水平,并抑制MRSA菌株中PBP2a蛋白的表达,且呈剂量依赖性。总之,AP可抑制PBP2a蛋白的转录和表达,并有可能会损坏其构象来提高MRSA对β-内酰胺类抗生素的敏感性。

