鲸偶蹄目RNase 11和RNase 12的分子进化研究
郎大田,李高银,张 毅,刘松菊*
(1.昭通学院 农学与生命科学学院,云南 昭通 657000;2.云南省昭通市昭阳区动物卫生监督所,云南 昭通 657000;3.云南省昭通市动物卫生监督所,云南 昭通 657000)
核糖核酸酶基因(ribonuclease A,简称RNaseA)超家族因频繁发生基因重复并在正选择驱动下发生功能分化,是基因重复与功能分化研究最为经典的模式[1-5].RNaseA 超家族成员由基因重复产生并分化出不同功能,且一些成员在不同物种中又会发生重复,也同样被证实与新功能产生密切相关[4-5].
在家牛中,RNaseA超家族共有15个成员RNase1~15,包含典型成员RNase1~8和非典型成员RNase9~15[1].过去的研究主要聚焦于与食性相关的典型成员RNase1,而对非典型成员RNase9~15的研究较少[6-8].有趣的是,与生殖功能相关的非典型成员RNase9在鲸偶蹄目中经历了独特的进化模式,高度保守的RNase9在反刍亚目中经历了基因重复和假基因化,时间发生在始新世中期[9].基因表达实验揭示RNase9仅在附睾、输精管或前列腺等生殖组织中表达,其基因重复有利于促进精子活力和雄性生殖率,从而使得反刍亚目动物更好地适应始新世中期气候季节性变化[9].相比之下,与RNase9在反刍亚目动物中发生基因重复截然不同,在鲸目的所有物种中发生基因丢失,进而有利于鲸目物种适应水环境和为生存需要繁殖后代,平衡海洋资源需求[9].与生殖功能相关的RNase10在鲸偶蹄目中为单个基因[10].值得注意的是,同在雄性生殖组织中表达,并紧邻于RNase9 且为非典型成员的RNase11,RNase12,在鲸偶蹄目中的分子进化尚未清楚.
笔者以大型哺乳动物中物种最丰富的鲸偶蹄目4个亚目21科92个物种为研究对象,基于系统发育树、等电点、选择压力分析、蛋白质结构等方法深入探讨RNase11和RNase12的分子进化研究,进一步拓展人们对RNaseA超家族非典型成员的认知,丰富RNase11和RNase12分子进化研究的多样性.
1 材料与方法
1.1 研究材料
笔者的研究取材于NCBI(/www.ncbi.nlm.nih.gov/)已公布鲸偶蹄目的反刍亚目、胼足亚目、猪形亚目、河马形亚目4个亚目21科92个物种基因组,反刍亚目(Ruminantia)包含了牛科(Bovidae)、鹿科(Cervidae)、长颈鹿科(Giraffidae)、麝科(Moschidae)、叉角羚科(Antilocapridae)的59个物种,胼足亚目(Tylopoda)包含了骆驼科(Camelidae)的4个物种,猪形亚目(Suoidea)包含了猪科(Suidae)、西猯科(Tayassuidae)的2个物种,河马形亚目(Cetancodonta)包含了河马科(Hippopotamidae)、海豚科(Delphinidae)、鼠海豚科(Phocoenidae)、一角鲸科(Monodontidae)、剑吻鲸科(Ziphiidae)、抹香鲸科(Physeteridae)、白鱀豚科(Lipotidae)、河豚科(Platanistidae)、亚河豚科(Iniidae)、拉河豚科(Pontoporiidae)、须鲸科(Balaenopteridae)、灰鲸科(Eschrichtiidae)、露脊鲸科(Balaenidae)的27个物种.
1.2 研究方法
1.2.1 获取基因序列
利用过去研究经常使用的本地blastn和tblastn方法[1,5],分别对每个物种进行分析,分析结果按Scaffold进行分类排序,根据查询序列比对在Scaffold上的序列长度和相似度,获取每个物种RNase11和RNase12的基因序列.
1.2.2 系统发育分析
追溯生物进化历史并以系统发育树显示生物类群之间的进化关系,是进化生物学研究的重要内容.笔者分别对RNase11和RNase12基因序列的功能基因和假基因进行鉴定,若在序列编码框中出现终止密码子或非三联体的插入/缺失,则认定为假基因,反之为完整的功能基因[11].利用ClustalX软件比对DNA序列并人工矫正,以DAMBE转换DNA 序列格式[12],选择奇蹄目的家驴(Equusasinus)、马(Equuscaballus)、普氏野马(Equusprzewalskii)、白犀(Ceratotheriumsimumsimum)作为外群,采用j Modeltest软件计算核苷酸替代模型并确定最适模型参数[13],使用邻接法(neighbor-joining,简称NJ)、贝叶斯法(Bayesian inference,简称BI)构建系统发育树,具体方法如下.
邻接法:使用MEGA11软件构建邻接树,选用Kimura双参数模型为核苷酸替代模型,对于序列缺口采用成对删除,并进行2 000次自展重抽样分析,构建NJ树[14].贝叶斯法:基于MrBayes v3.1.2软件[15],以j Modeltest 3.0.6[16]和赤池信息量准则(akaike information criterion,简称AIC)来确定最佳核苷酸进化模型,用4个马尔可夫链进行2次单独运行,每次5×106代,每100代取1次样.
1.2.3 选择压力分析
生物表型的适应性进化在分子水平上表现为DNA 序列的改变[17-18].非同义替换与同义替换比率(ω),可以反映1个基因在进化过程中受到的选择压力.若ω>1,表明基因受到了适应性(正)选择;若ω=1,表明基因经历了中性进化;若ω<1,表明基因受到了纯净化选择[19-20].笔者利用PAML 软件codeml程序的“位点模型”(M1a vs.M2a;M8 vs.M8a)和“枝-位点模型”中的test2[21-22],分别对鲸偶蹄目RNase11和RNase12功能基因计算选择压力.“位点模型”的M1a(近中性),假设仅有保守位点(0<ω<1)和中性位点(ω=1)而没有正选择位点(ω>1),这两类位点的比率分别为p0和p1,其对应的ω值分别为ω0和ω1;M2a(正选择),该模型在M1a基础上增加了第3类ω值,即假设除了保守位点和中性位点外,还存在处于正选择压力下的位点(ω>1),这3类位点的比率分别为p0,p1和p2,其对应的ω值分别为ω0,ω1和ω2;M8假设所有位点的ω属于矩阵(0,1)并呈beta分布,将ω值固定为大于1(ω>1),M8a与M8模型类似,而将ω值固定为1,p0=1-p1.在“位点模型”和“枝-位点模型”中,均以保守的贝叶斯经验值(Bayes empirical Bayes,简称BEB)[23]对可能受到的正选择位点和进化枝进行后验概率检测,并进行多重校正.
1.2.4 翻译比对氨基酸序列
虽然RNaseA各成员有不同功能,但典型成员(RNase1~8)拥有共同序列特征,其特征与功能密切相关[1,24].为此,在每个亚目中选择一定数量的代表物种,利用MEGA11软件[14]分别翻译RNase11和RNase12功能基因的氨基酸序列,在其序列上标注出该超基因家族序列典型特征及变异位点.
1.2.5 等电点预测
利用在线网站https://web.expasy.org/compute_pi/预测RNase11和RNase12等电点[25],为揭示鲸偶蹄目RNase11和RNase12可能拥有的生物学功能提供线索.
1.2.6 蛋白质的三维结构模拟
基于蛋白质结构数据库(PDB),利用3D-JIGSAW 和Py MOL软件模拟RNase11和RNase12的三维结构[26-28],将RNaseA超家族功能相关的位点(结构半胱氨酸、功能区CKXXNTF和正选择位点)比对到蛋白质三维结构上,查看受到选择的位点是否在维持蛋白结构稳定的半胱氨酸上或者其附近.
2 结果
2.1 鲸偶蹄目Nase11和RNase 12序列
对鲸偶蹄目的4个亚目21科92个物种进行分析,共获得69条RNase11序列,包括61条功能基因和8条假基因;在RNase12中获得88条序列,包括87条功能基因和1条假基因(表1).在反刍亚目59个物种和胼足亚目4个物种中,RNase11,RNase12均为单条完整的功能基因.在水生的河马形亚目27个物种中,RNase11序列均不完整,仅在6个物种中分析检测到6条假基因,其他21个物种中未分析到同源序列;而RNase12在多数物种为单条序列,仅在4个物种中未分析到同源序列,1个物种中为不完整的序列(表1).此外,RNase11,RNase12开放阅读框的长度分别为577~600 bp和435~438 bp,其功能基因蛋白序列比对后的长度分别为199 aa和145 aa.RNase11包含完整信号肽(1~16 aa)和成熟肽(17~199 aa),而RNase12包含完整信号肽(1~24 aa)和成熟肽(25~145 aa)(图1和图2).

图2 鲸偶蹄目RNase12蛋白的一级结构

表1 鲸偶蹄目RNase11和RNase12基因数量
2.2 鲸偶蹄目Nase11和RNase 12等电点分析
RNaseA超家族等电点与其生物学功能有着紧密联系,等电点高的成员有较多数量带正电荷的氨基酸残基,更能紧密接触带负电的细菌细胞壁,具有抗菌活性进而拥有宿主防御功能,而等电点低的成员失去核糖核酸酶的抗菌活性而具有其他生物学功能,如消化、生殖等[3,29].笔者对鲸偶蹄目RNase11的61条功能基因等电点进行预测,其数值为7.09~9.13,平均值是8.58;而RNase12的87条功能基因等电点为7.11~8.98,平均值为8.25(图1和图2).这些揭示RNase11和RNase12等电点明显高于同为非典型成员RNase9和RNase10[9-10],而与典型成员中拥有抗菌活性的RNase5和RNase6基本一致[5,30].
2.3 鲸偶蹄目Nase11和RNase 12的系统发育分析
基于RNase11和RNase12功能基因,以奇蹄目的4个物种作为外群,采用邻接法和贝叶斯法构建NJ树和BI树,其拓扑结构总体一致.在研究物种最多的反刍亚目中,鹿科、长颈鹿科、麝科、叉角羚科物种分别形成单系(图3和图4);基于RNase12构建的系统发育树显示,胼足亚目和猪形亚目形成姐妹群最先分歧,其次是河马形亚目,最后是反刍亚目(图4).
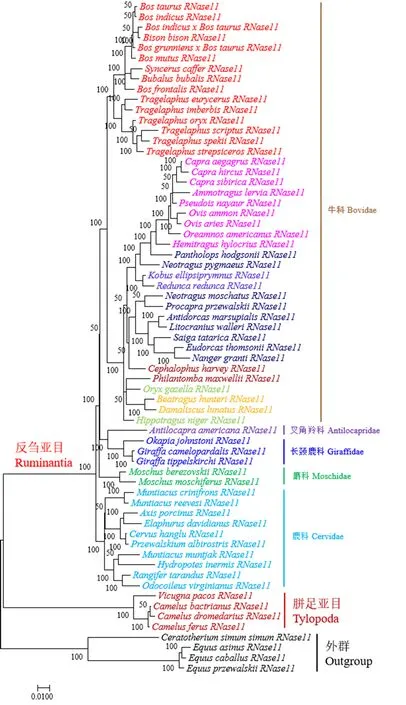
图3 基于鲸偶蹄目RNase11功能基因构建的BI树
2.4 鲸偶蹄目Nase11和RNase 12的选择压力分析
基于RNase11和RNase12功能基因构建的系统发育树(图3和图4),其进化枝长短不一,预示鲸偶蹄目RNase11和RNase12进化过程中经历的进化速率和选择压力可能不同.利用PAML软件包CODEML分别计算RNase11,RNase12所经历的选择压力,考虑到CODEML对系统树拓扑结构比较灵敏,基于NJ树分析也得到类似的结果.分析结果显示“位点-特异”模型中的正选择模型(M2a和M8)比中性模型(M1a和M8a)有显著的更好适合度(p<0.05)(表2),RNase11的7个位点受到正选择,比对到其蛋白一级结构,位置是86,87,89,125,129,151和153,其中位点87,89,125的后验概率大于0.95(表2和图1),将受到的正选择位点后验概率P值进行FDR多重矫正,位点87,89,125在FDR ≤0.1水平显著.类似于RNase11,RNase12中的位点25,41,79,83,92,120,123,129,144在“位点-特异”模型中受到正选择,其中位点79和120的后验概率大于0.95(表2和图2),同样进行FDR多重矫正,在FDR ≤0.1水平下未有显著位点.在“枝-位点”模型中,笔者分别对鲸偶蹄目4个亚目和反刍亚目5个科祖先枝的RNase11和RNase12进行选择压力分析,均未检测到受正选择的信号.
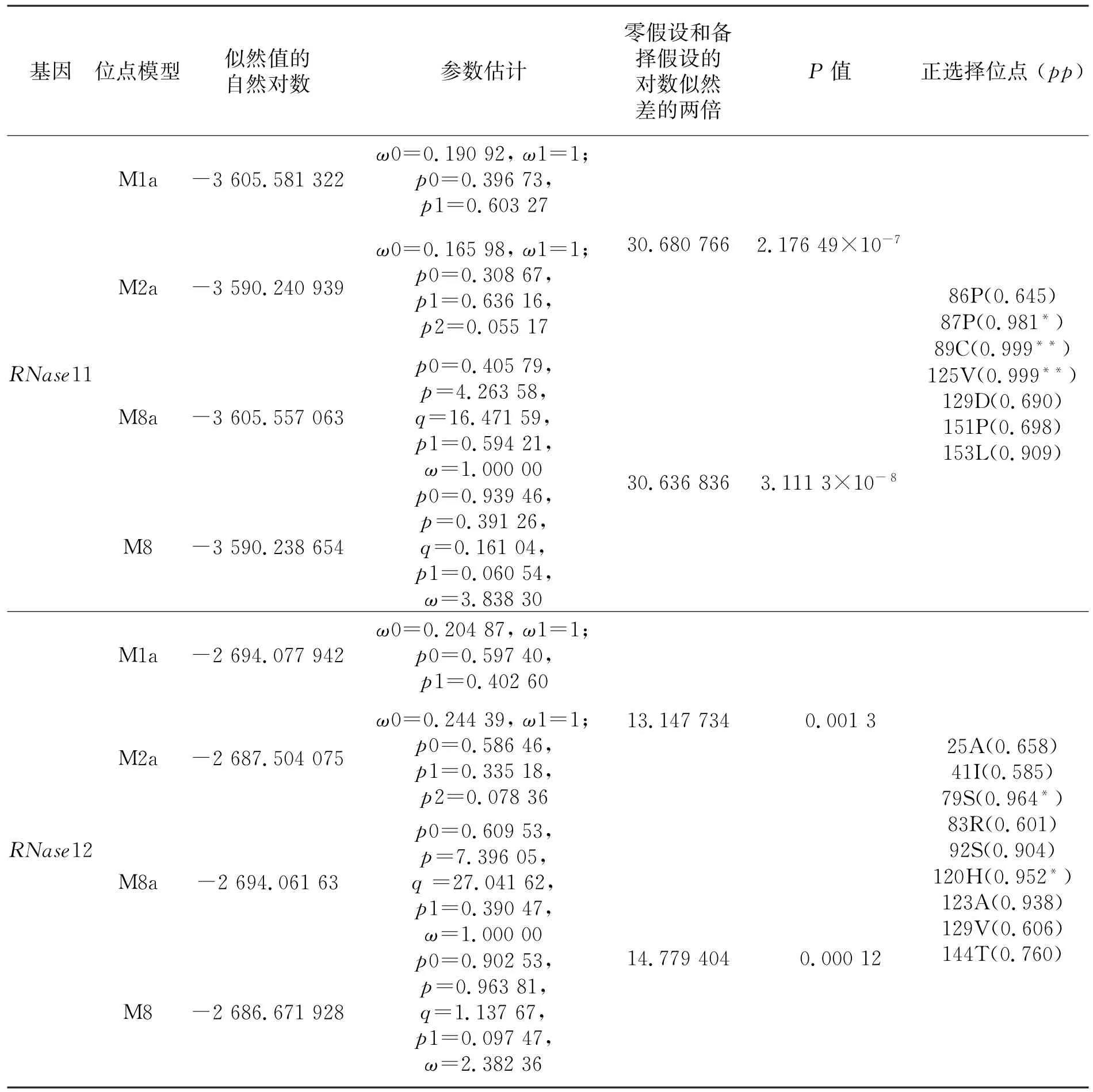
表2 鲸偶蹄目RNase 11和RNase 12选择压力分析
另外,蛋白质生物学功能与其空间结构密切相关,特定的结构是行使蛋白质生物学功能的基础.RNaseA超家族中,半胱氨酸形成二硫键进而在维持蛋白结构稳定中有着重要作用,在半胱氨酸上或附近位点发生改变,有可能影响蛋白的结构稳定致使功能发生改变.将检测出的正选择位点绘制到RNase11和RNase12蛋白质三级结构(图5和图6),发现RNase12中受选择的位点79,92,120在维持蛋白结构稳定的半胱氨酸附近(图6),揭示受到正选择的位点在RNase12进化过程中,有可能影响其蛋白的结构稳定,从而致使RNase12生物学功能发生了改变.
3 讨论
RNaseA超家族共有15个成员(RNase1~15),包含典型成员RNase1~8和非典型成员RNase9~15[1].典型成员RNase1~8具有核糖核酸酶活性,在基因序列上具有一些共同的特征,如单个外显子构成开放阅读框、编码区由信号肽和成熟肽组成、6~8个结构半胱氨酸形成3~4对二硫键、催化活性氨基酸H12-K41-H119和功能区“CKXXNTF”等特征,拥有消化、血管生成、精子成熟、抗菌和抗病毒等功能[24,31];而非典型成员RNase9~15失去了核糖核酸酶活性,仅有结构半胱氨酸,催化活性氨基酸和功能区发生了丢失[1].
笔者基于RNaseA超家族的非典型成员RNase11和RNase12,在占据水生和陆生不同生境、大型哺乳动物中物种最丰富的鲸偶蹄目92个物种中开展分子进化研究,共获得69条RNase11和88条RNase12序列.在反刍亚目5个科59个物种中,具体包含牛科43个物种、鹿科10个物种、长颈鹿科3个物种、麝科2个物种、叉角羚科1个物种,RNase11和RNase12均为单条基因序列.值得注意的是,RNase11在水生的27个河马形亚目物种中发生假基因化或基因丢失,与过去研究报道生殖功能相关RNase9在鲸目物种发生丢失的进化模式一致,其机制可能也与河马形亚目适应水环境和为生存需要繁殖后代、平衡海洋资源需求有关[9].类似发生基因丢失而有助于水生物种类群适应生存的海洋环境研究,在芳烷基胺N-乙酰转移酶[11,32]、栓形成基因F12和KLKB1[33]、表皮相关基因(DSG4,DSC1,TGM5和GSDMA)均有相应的研究报道[34].
RNase11和RNase12蛋白一级结构的序列长度不同,分别为199 aa和145 aa,维持蛋白结构稳定的半胱氨酸在RNase11 和RNase12 中极为保守,其等电点明显高于过去研究报道的RNase9 和RNase10[9-10],而与抗菌活性相关的RNase5和RNase6一致[5],揭示RNase11和RNase12在鲸偶蹄目中可能拥有与生殖相关的抗菌功能,但其在组织中的表达模式需后续深入开展研究,以进一步揭示它们的生物学功能.
此外,鲸偶蹄目4个亚目之间的系统发育关系一直存在争议[35-38],笔者基于RNase11,RNase12构建系统发育树,结果显示胼足亚目和猪形亚目形成姐妹群最先分歧.虽然RNase11和RNase12基因序列较短,构建的系统发育树显示的拓扑结构不一定接近4个亚目之间的真实系统发育关系,但能为后续基于基因组水平深入开展系统发育研究提供一定的基础.在选择压力分析中,RNase11和RNase12在“位点模型分别检测到7个和9个正选择位点,且一些位点在维持蛋白结构稳定的半胱氨酸附近,这些揭示鲸偶蹄目RNase11和RNase12在进化过程中功能可能发生了分化.
总之,笔者以鲸偶蹄目4个亚目21科92个物种为研究对象,基于系统发育树、等电点、选择压力分析、蛋白质结构深入系统开展RNase11和RNase12的分子进化研究,丰富了RNaseA超家族分子进化研究的多样性,为进一步认识RNaseA超家族非典型成员和占据水生和陆生不同生境鲸偶蹄目的进化提供了基础.

