基于罂粟基因组的SSR位点鉴定及其特征分析
凌立贞,杜毛毛,张书东
(六盘水师范学院生物科学与技术学院,贵州 六盘水 553004)
罂粟(Papaver somniferum L.)为罂粟科(Papaveraceae)罂粟属1~2 年生草本植物[1]。罂粟属全球约有100 种,其中中国有7 种[2],主要分布于中国的东北与西北地区,与大麻、古柯并称为世界“三大毒品原植物”[1]。该植物全草均有毒,未成熟果实内含乳白色汁液,去掉大量水分后形成黑色膏状物质便是鸦片。其主要成分有吗啡、可待因、蒂巴因、罂粟碱等生物碱,在医学上有着重要的研究价值。同时,这些物质具有很强的毒性,吸食后会使人产生强烈的依赖性[3],给自身和社会带来严重的危害。目前,罂粟属植物通常是通过花、果实、根、茎、叶等形态特征来进行鉴别,该方法大多需要较完整的植株[4]。另外,在一些毒品运输过程中,通常都以提取物或者其他形式运输,虽然可以通过化学成分进行鉴定,但是需要的仪器设备比较昂贵,操作比较复杂。一些幼苗期的罂粟属植物与毒品原植物罂粟形态上极为相近,但主要化学成分还没有达到检测的量,这些都为毒品原植物罂粟的鉴定增加了难度。因此,亟需从分子水平鉴定毒品原植物。
简 单 重复 序 列(Simple Sequence Repeat,SSR),又称微卫星脱氧核糖核酸(Microsatellite DNA),通常是以1~6个核苷酸为重复单元、反复出现的简单序列,如(AC)n、(AAG)n、(CATG)n等。与其他分子标记相比,SSR 具有数量大、多态性高、保守性强等诸多优点[4],被广泛应用于物种或品种的鉴定。赵丽霞等[5]利用SSR 分子标记对罗田8 个主栽板栗品种进行鉴别,该鉴别技术解决了生产中难以从外表鉴别相似品种的问题。谷方红等[6]借助微卫星技术将大麦及麦芽的8 个品种区分开来。李元元等[7]利用3 个特异性SSR 分子标记构建罂粟荧光复合扩增检验体系,将罂粟与虞美人、大麻等进行有效区分。然而,不同地域之间的罂粟植物也存在着多态性差异。因此,一套完善的罂粟种属、种间以及溯源推断体系的建立还需要在基因组范围内开发更多的特异性分子标记。
目前,通过表达序列标签(Expressed Sequence Tag,EST)序列[8]和简化基因组序列[1]筛选出特异性SSR 标记仅155 个(包括59 个EST-SSR引物及96 个基因组SSR 引物),但均表现出多态性较低、遗传分化区别能力较差和等位基因数量较少等缺点。目前,罂粟全基因组已公布,为全基因水平SSR 位点的分析及标记开发提供了可能。切利克(Celik)等[9]利用焦磷酸测序技术对罂粟基因组DNA 进行了测序,并对其进行SSR 技术检测,共获得长度为474 Mb 的1 244 412 条序列,得出以AAG/TTC为最丰富的三核苷酸重复,但是未见有关罂粟全基因组SSR的分析报道。本研究目的旨在对罂粟全基因组进行SSR 分析,通过对罂粟全基因组中SSR数量和重复出现的频率进行统计,分析SSR 重复基元组成和单碱基、二碱基、三碱基、四碱基、五碱基和六碱基重复次数占比以及组成与分布特征,对罂粟全基因组SSR 位点进行全面而系统的分析,为罂粟的鉴别和遗传多样性分析等提供分子标记信息。
1 材料与方法
1.1 数据来源
罂粟基因组数据包括11 条染色体的序列数据从美国国家生物技术信息中心基因数据库(GenBank)下载获取。
1.2 SSR位点的搜索
利用微卫星识别软件(MIcroSAtellite Identification,MISA)(https://webblast.ipk-gatersleben.de/misa/)对罂粟基因组不同染色体的1~6 bp的重复核苷酸进行查找,其最少重复数分别设定为10、6、4、3、3、3。最后形成以.statistics 为后缀的文件。里面记录了该基因组长度、微卫星的总数量、重复基元类型以及重复次数等信息。利用Geneious R9.0.2软件将罂粟的11条染色体整合为1个fasta文件,统计SSR位点。
1.3 统计分析
所有的数据分析和作图均利用Microsoft 2010 Excel软件完成。
2 结果分析
2.1 罂粟全基因组SSR的总体分布特征
罂粟全基因组总序列长度为2 216 804 860 bp,共检测出833 005 个SSR 位点,总长度为11 285 328 bp,占全基因组长度的0.51%。罂粟全基因组中SSR不同重复类型的分布特征如表1所示。
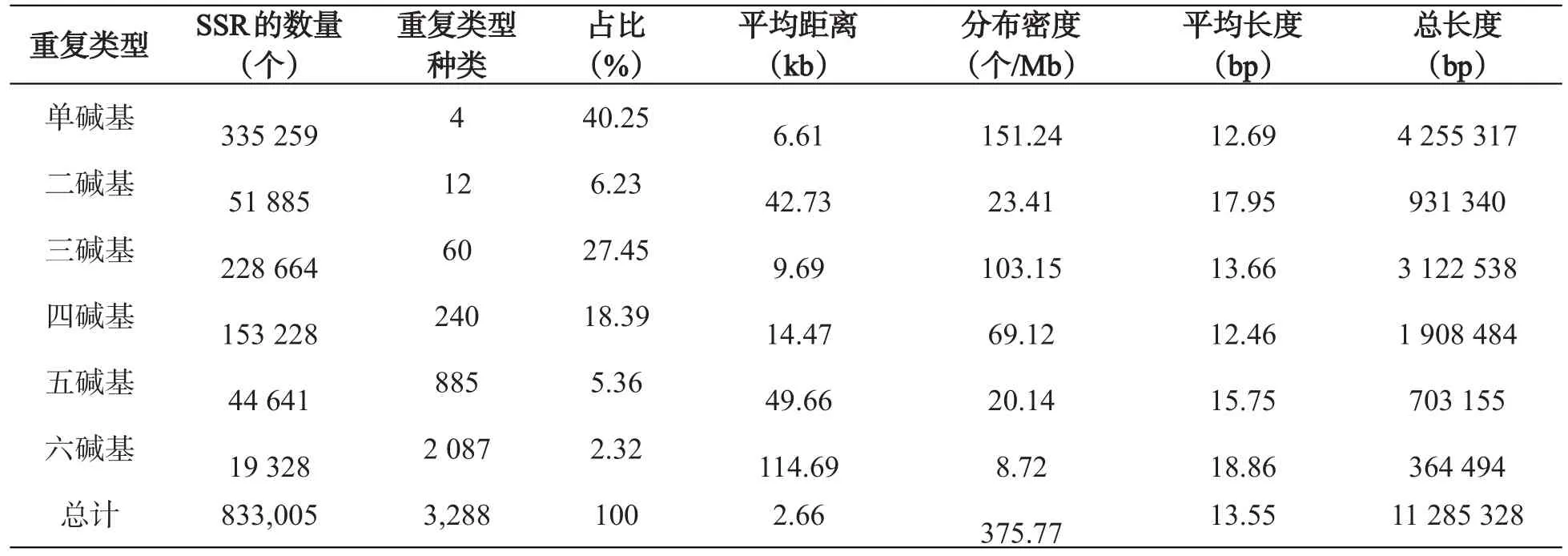
表1 罂粟全基因组中SSR不同重复类型的分布特征
由表1可知,单碱基的总长度最长,高达4 255 317 bp,其次为三碱基、四碱基、二碱基、五碱基,分别是3 122 538 bp、1 908 484 bp、931 340 bp、703 155 bp。SSR 总长度最短的是六碱基,只有364 494 bp。其平均长度最长的是六碱基(18.86 bp),最短的是四碱基(12.46 bp)。在罂粟基因组SSR中,平均距离最短的是单碱基,只有6.61 kb;其次分别是三碱基(9.69 kb)、四碱基(14.47 kb)、二碱基(42.73 kb)、五碱基(49.66 kb)和六碱基(114.69 kb)。从分布密度来看,罂粟基因组在每1 Mb序列中有315.8个SSR位点,其中单碱基的密度最高,平均每1 Mb有375.77个,而六碱基的只有8.72个。
2.2 罂粟全基因组SSR中各重复类型分析
在罂粟全基因组中,碱基的重复类型种类数量众多,共有3 288种,其中单碱基仅有4种,分别为A、T、C、G;重复类型种类数量最多的是六碱基,有2 087 种;二至五碱基的分别为12、60、240、885种(表1)。很明显,重复种类数量随着碱基重复单元数增加而增加。首先,单碱基重复单元数量最高,占40.25%,其次是三碱基重复单元和四碱基重复单元占比分别为27.45%和18.39%(表1)。六碱基重复单元占比最少,仅有2.32%。罂粟全基因组SSR中不同重复类型的重复次数频率分布如图1所示。
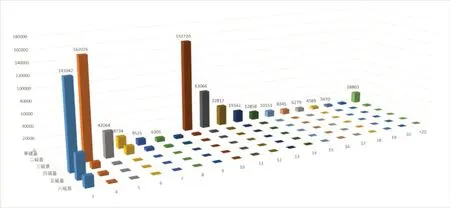
图1 罂粟全基因组SSR中不同重复类型的重复次数频率分布
由图1 可知,罂粟全基因组中重复序列的重复次数大多在3~20 次,少部分的重复次数大于20次,仅占总体的2.53%,且每种重复碱基都是随着重复次数的增多而减少。首先,碱基重复大于20次的单碱基共有18 803个,其次二碱基有2 094个,然后是三碱基和四碱基,分别是146 个和19个,最后,五碱基与六碱基重复次数均没有超过16次。
罂粟全基因中各重复类型不同基元的比例分布如图2所示。

图2 罂粟全基因中各重复类型不同基元的比例分布
由图2 可知在单碱基中,A/T 占绝对优势,为单碱基重复基元的96.68%,并在全部重复基元中排首位。二碱基重复碱基共有4 种(AT/AT、CG/CG、AG/CT、AC/GT),其中AT/AT的重复基元占比最高,为66.59%,在全部重复单元中占4.15%。最少的是CG/CG,仅有0.05%,剩余的是AG/CT、AC/GT,分别占重复基元两个碱基的23.92%、9.44%。三碱基重复碱基有10种,其中AAG/CTT是三碱基重复基元中占比最高的,为27.61%,位居全部重复基元SSR 位点的第二位。其次是AAT/ATT、AGC/CTG,分别占重复基元三个碱基的18.80%、15.06%。四碱基中1/3 的重复基元是AAAT/ATTT,但不在全部重复拷贝类型的前十位中。五碱基重复基元中占比最多是AAAAT/ATTTT,为37.23%,位居全部重复基元SSR 位点的第十位。而六碱基的占比最多是AAAAAT/ATTTTT,占重复基元六个碱基的23.19%。一至六碱基的重复类型中,优势重复类型分别为A/T、AT/AT、AAG/CTT、AAAT/ATTT、AAAAT/ATTTT、AAAAAT/ATTTTT,这6组数据的总和超过52%,大多为A、T碱基的序列。所有SSR 位点更倾向于腺嘌呤(A)与胸腺嘧啶(T)所构成的碱基。
2.3 SSR位点在染色体上的数量及分布情况
SSR在11条染色体上数量分布如图3所示。
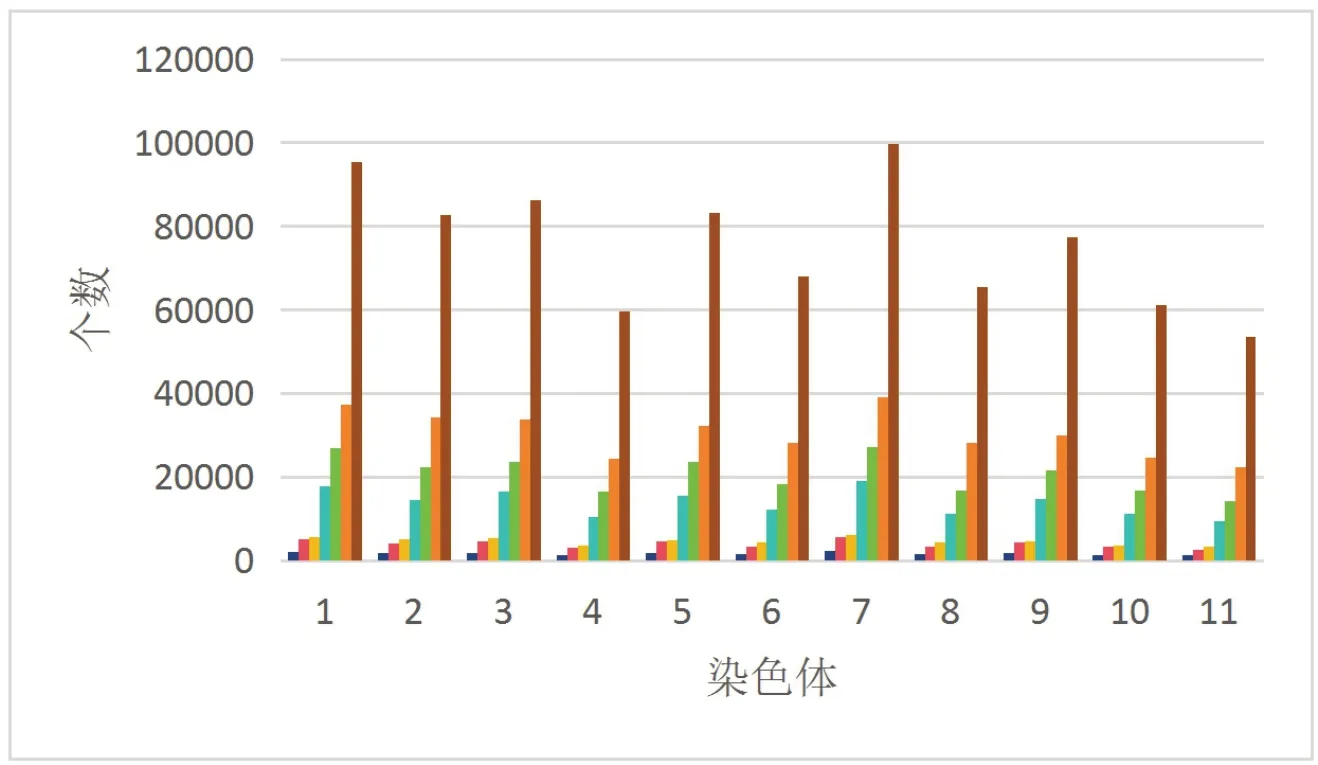
图3 SSR在11条染色体上数量分布
由图3 可知,SSR 位点在罂粟基因组11 条染色体上的分布是不均匀的。7 号染色体上的SSR位点数量最多,有99 794 个,其中单碱基SSR 数量最多,有39 251 个。而第11 号染色体上SSR 位点数量最少,为53 710 个,六碱基只有1 276 个。在11条染色体中,每条染色体的单碱基SSR都多于其他碱基的数目,而六碱基SSR 是最少的,二碱基的SSR 数量均少于单碱基、三碱基和四碱基。
SSR在染色体上的分布距离如图4所示。

图4 SSR 在染色体上的分布距离
由图4 可知,罂粟11 条染色体上的SSR 平均分布距离为2.66 kb,每一条染色体上的SSR 分布距离都不尽相同,但与染色体的长度相关性不大。第11 条染色体长度是最短的,但SSR 的分布距离为2.61 kb,比染色体长度较长的1 号和5号的都大。而在染色体长度第二短的4 号染色体SSR 分布距离是最大的,为2.74 kb,超出平均距离约3%的长度。1 号染色体上的SSR 分布距离最短,为2.61 kb,低于平均距离的2%。染色体长度最长是7 号染色体,SSR 分布距离为2.71 kb,是所有染色体中相对平均长度为第二的染色体。
2.4 染色体长度与重复类型相关性分析
染色体长度与重复类型相关性如图5所示。
由图5a 可知,在全基因组中检测出的3 288种重复基元不均等地分布在11 条染色体上。总体趋势是染色体长度越长,重复基元种类数量也越多。但在1、3和6号染色体上,重复类型的数量与染色体长度呈现出略微下降的趋势。在最长的7 号染色体上,SSR 重复类型种类达到了峰值,为1 538 种。为了探究碱基重复类型与染色体长度是否有相关性,进行线性回归分析,发现碱基重复类型与染色体长度的一元回归方程为y=330304x-3E+08,R2为0.96,拟合效果较好。这些结果说明重复类型的数量与其所在染色体的长度具有一定的线性关系(图5b)。
3 讨论与结论
随着高通量测序技术的发展,越来越多的植物、动物和微生物的基因组数据都在陆续公布。SSR 位点广泛分布在生物基因组中,分布模式一定程度上可以反映出不同染色体的特点[10]。从形态学上区分物种存在着巨大的限制,而SSR 标记具有多态性高、符合孟德尔遗传、保守性强等优点,可以解决形态学鉴定中难以解决的问题。
本研究通过GenBank数据库下载获取罂粟全基因组序列,并利用MISA软件对罂粟全基因组中的SSR 位点进行分析,一共得到微卫星序列833 005条,重复类型数量共有3 288种,这说明在罂粟全基因组中的重复类型数量丰富。其中单碱基的SSR 数量位点是最多的,占据总长度的40.25%。其结果与黑果枸杞(Lycium ruthenicum)[11]、大花序桉(Eucalyptus cloeziana)[12]、紫果西番莲(Passiflora edulis)[13]等植物的研究结果相似,但在壳斗科植物锥栗(Castanea henryi)[14]和闽楠(Phoebe bournei)[15]的研究中是以二核苷酸或三核苷酸重复单元为主的。这些研究说明在不同植物中SSR重复类型中最丰富的类型是不同的。这些SSR在基因组中的平均距离为2.66 kb,与大花序桉[12]平均距离(3.13 kb)较为接近。对樟树(Cinnamomum camphora)[16]SSR位点分析发现,每隔0.35 kb就有一个微卫星位点出现,在灌木椒(Capsicum frutescens)全基因组中两个重复序列之间相隔8.72 kb[17],黑枸杞的平均距离为5.28 kb[11]。因此,SSR在不同物种上的分布差异性很大。
以前研究表明碱基重复基元类型多以一类碱基为主,如在柳树(Salix caprea)中以二碱基(TC/AG、GA/CT 和CA/GT)为主,占复合微卫星位点的3/4[18]。而本研究中的结果表明罂粟全基因组SSR中数量较多的重复类型较复杂,其中位居前六位的重复基元有A/T、AAG/CTT、AAAT/ATTT、AAT/ATT、AT/AT 和AAC/GTT,占全基因组SSR 位点总数的52%。碱基偏好性的本质是反映密码子的使用偏好情况,不同的密码子翻译形成的氨基酸种类不同,从而使不同植物有着不同的生理表达。罂粟SSR 序列在一定程度上偏好于A 与T 所构成的碱基。而在小麦等植物中是以C和G为主要重复类型。另外,罂粟全基因组中,7号染色体分布的SSR位点最多。研究发现,在罂粟11条染色体中,染色体长度越长,其上分布的SSR位点的数量相对会越多,在毛果杨(Populus trichocarpa)[19]中也发现类似的现象。因此,物种内染色体长度的差异也是影响分布在染色体上SSR数量的原因之一。通过相关性分析,可以看出重复基元类型与染色体的长度有一定的关系。本研究对罂粟全基因组内的SSR 分析发现,SSR 上的分布是比较多的,其中单碱基的重复序列最多。这些SSR 的序列以A和T碱基为主,而重复基元种类比较多,含量高的重复基元体现在多种类型。另外,SSR 在罂粟11条染色体上的分布是不均等的,重复类型与染色体的长度体现出一定的线性关系。这些研究结果将为进一步筛选有效的SSR 提供数据基础。

