基于三联体和金门克隆的快速TALE 模块组装技术
周继曾,杨洋,鹿璇,李双鹏,邹庆剑,张焜,*
(1.广东工业大学 生物医药学院,广东广州 510530;2.五邑大学 生物科技与大健康学院,广东江门 529020)
转录激活因子样效应物(Transcription Activator-Like Effector,TALE)是一种能识别特定DNA 序列的蛋白,最早由科学家在植物病原菌黄单胞菌(Xanthomonas)中发现,其天然功能是直接调节宿主基因表达。在通过细菌Ⅲ型分泌系统侵染进入宿主细胞后,TALE 进入细胞核,与宿主基因启动子中的效应器特异性序列结合并激活转录[1]。TALE 主要由N 端、C 端和中间的DNA 结合域(DNA Binding Domain,DBD)三部分组成。DNA 结合域由含34 个氨基酸(Amino Acid,AA)重复序列串联组成,每段重复序列模块的第12 和13 位氨基酸为重复可变二残基(Repeat Variable Di-residues,RVD),其中4 种常见的RVD 是NI、NG、HD 和NN,分别识别核苷酸A、T、C 和G[2]。与CRISPR/Cas9 系统相比,TALE具有其独特优势。例如,TALE 可以灵活地融合各种蛋白,如与核酸内切酶Fok Ⅰ融合靶向目标序列进行DNA 双链切割[3]、与转录调控域融合进行基因激活[4],以及与核酸酶或脱氨酶融合实现CRISPR/Cas9 系统难以完成的线粒体基因组编辑,作为线粒体疾病治疗的潜在工具[5-8]。此外,TALE 核酸酶(TALENs)的识别范围为10 ~31 个碱基对(bp),具有脱靶风险低的优势[9-10],比CRISPR/Cas9 系统更适用于临床应用。然而,目前常用的TALE 组装方法,如金门(Golden Gate)克隆技术[11]、solid-phase 克隆技术[12]和 Gibson克隆技术[13]等都存在组装时间长、高成本、难操作的问题,限制了TALE 工具在基因打靶中的应用。
笔者开发了一种由Cas9/sgRNA 和TALE 组成的双导航系统的新型单碱基编辑器——TaC9-BE[14-15],它不仅能在不产生DNA 双链切割的情况下在靶位点实现精准高效的碱基C-T 或A-G 替换,而且没有可预测DNA 脱靶效应,为基因治疗和转基因生物的产生提供了一种安全的工具。然而,TALE 构建的复杂性成为TaC9-BE 应用的主要障碍。本研究通过新开发的TALE 三联体结合金门克隆技术建立一种新的TALE 构建技术——Trimer-Golden Gate(Tri-GG),该方法可高效、快速、简易地组装TALE,为TaC9-BE系统提供了便捷的TALE 服务。
1 材料与方法
1.1 材料
1.1.1 质粒与细胞
质粒EFS-ABE8EdelR153-TALE-EGFP(LacZ)和EFS-YE1-TALE-EGFP(LacZ)、nCas9-mCherry、UGI-nCas9-UGI-mCherry、gRNA 骨架载体、TALE三联体库等,均由五邑大学邹庆剑课题组实验室构建,并存于-20 ℃冰箱;大肠杆菌(Escherichiacoli)感受态细胞DH5α 菌株和实验所需所有引物,均购于广州艾基生物技术有限公司;HEK293T 细胞,购自美国ATCC 生物生物标准品资源中心。
1.1.2 试剂
DNA marker,日本Takara Bio 公司;Ⅱ型限制性核酸内切酶(Esp3I 和BbsI)、高糖培养基,美国Thermo Fisher Scientific 公司;T4 DNA 连接酶,美国New England Biolabs 公司;PCR 酶和质粒提取试剂盒,南京诺唯赞生物科技股份有限公司;转染试剂lipo 8000,上海碧云天生物技术有限公司;TE 缓冲液(Tris-EDTA)、0.5% NP-40、10 μg/mL 蛋白酶K,德国Merck 公司;10%胎牛血清,美国Cytiva 公司;1%胰蛋白胨、0.5%酵母粉(Gene Star),青岛金斯达生物技术有限公司;1%氯化钠,天津市大茂化学试剂厂。
1.1.3 仪器
LHG-3_G-F8 生物安全柜,新加坡Esco 公司;HERA cell 150i1 型细胞培养箱,美国Thermo Fisher Scientific 公司;OK-8D 三孔水浴锅,上海一恒科学仪器有限公司;IX73 荧光倒置显微镜,日本奥林巴斯公司;DYY-6C DNA 凝胶电泳仪,北京六一生物科技有限公司;2FD 超净工作台,瑞智精密股份有限公司;T100 PCR 仪,美国Bio-Rad 公司;LRH-70 生化培养箱,上海一恒科学仪器有限公司。5425R 常温离心机,德国Eppendorf 公司;3000 超微量分光光度计,美国Nanodrop 公司;1600 凝胶成像仪,上海天能生命科学有限公司;MoFlo™ XDP 流式分选仪,美国Beckman Coulter 公司。
1.2 实验方法
1.2.1 TALE 的构建
TALE 三联体由非重复的306 bp DNA 序列组成,其表达产物为3 个RVD 重复单元,可结合任意3 个连续DNA 核苷酸序列,如图1(a)所示。以TALE三联体为模板,设计具有特定末端序列的引物,通过PCR 扩增出三联体片段中末端具有Ⅱ型限制性核酸内切酶(Esp3I)的识别序列。不同引物对扩增出的片段,经酶切后产生突出末端可以按顺序首尾互补配对,如图1(b)和表1 所示。三联体扩增体系:反应总体系为20 μL,其中2×Phanta Max Master mix为10 μL,扩增三联体两条引物(10 μmol/L)分别加0.5 μL,三联体模板取10 ng,补充dd H2O 至20 μL。三联体PCR 扩增程序:98 ℃预变性1 min, 98℃变性10 s、60℃退火10 s、72 ℃延伸10 s,循环30 次。扩增片段胶回收纯化后,与TALE 骨架载体混合,反应体系为1 μL 连接缓冲液,Esp3I 和T4 连接酶各0.5 μL,混合PCR 产物100 ng,TALE 骨架载体60 ng,加ddH2O 补充至总体系10 μL。反应程序为37 ℃酶切5 min 和16 ℃连接5 min,循环10 次,最终实现在一个反应体系中实现多片段的有序连接,完成TALE模块组装,如图1(c)所示。

表1 三联体扩增引物序列

图1 简并密码三联体金门法组装TALE 的流程
1.2.2 菌液PCR 鉴定及测序鉴定
将金门克隆反应液转移至DH5α 感受态细胞中进行转化,冰浴30 min 后放入42 ℃水浴热激1 min后,继续冰浴3 min。最后加入含有X-Gal(200 μg/mL)和IPTG(1 mmol/L)的LB 平板上,放置37 ℃培养箱中12 h。第二天,观察平板菌落,挑取白色单菌落,加入含氨苄青霉素的LB 液体培养基中,37 ℃摇菌8 h。取10 μL 菌液高温裂解后,用作PCR 鉴定的扩增模板。PCR 鉴定引物序列如下,CEXU-F:A A C G T G G C G G C G T G A C C G C A;C E X U-R:GGTGGTCGTTGGTCAACGCGG。 菌液PCR 体系:2×Rapid Taq Master Mix 5 μL,两引物(10 μmol/L)各0.5 μL,裂解后的菌液0.5 μL,补充ddH2O 至总体系10 μL。PCR 扩增程序:98 ℃预变性1 min,98 ℃变性10 s、55 ℃退火10 s、72 ℃延伸20 s,循环30 次。PCR 产物凝胶电泳,初步鉴定正确的菌液样品,然后测序进一步验证载体序列是否正确。引物为CEXU-F和CEXU-FR。测序正确的TALE 载体用于后续细胞水平的功能性测试。
1.2.3 gRNA 载体的构建
根据5 个靶位点序列合成sgRNA 寡核苷酸链,将100 μmol/L 的正链和反链各取1 μL 混合,加ddH2O至20 μL,进行退火。退火程序:95 ℃孵育10 min后以0.1 ℃/s 的速度降低样品温度,直到16 ℃。退火后,与线性化gRNA 骨架载体(BbsI 酶切)混合,配制连接反应体系:连接缓冲液1 μL,T4 连接酶0.5 μL,退火产物5 μL,gRNA 骨架载体100 ng,加ddH2O 补充至10 μL,16 ℃孵育1 h。反应完成后,加入DH5α感受态细胞进行转化,转化过程同1.2.2步骤。最后提取质粒进行测序鉴定,测序正确的样品用于后续的细胞实验。sgRNA 的引物名称和序列见表2。

表2 sgRNA 引物序列
1.2.4 细胞转染和流式分选
用lipo8000 转染试剂将gRNA、TALE-EGFP和nCas9-mCherry 表达质粒按1 ∶1 ∶2 的比例共转染到293T 细胞中,同时将gRNA 和ABE8EdelR153-mCherry 或YE1-BE4max-mCherry 作为对照共转染到293T 细胞中。转染48 h 后,细胞通过流式分选仪进行分选收集带红色荧光和绿色荧光的细胞。
1.2.5 PCR 扩增和测序
将分选的细胞样品离心收集后,加入10 μL 的NP40/蛋白酶K 裂解液,裂解程序为56 ℃裂解1 h,95 ℃处理10 min 使酶失活。裂解产物作为PCR 鉴定的模板。PCR 反应体系:2×PhantaMax Master mix 10 μL,靶位点上下游引物(10 μmol/L)2 μL,细胞裂解物2 μL,ddH2O 补充至20 μL。PCR 扩增程序同1.2.1 步骤。收集PCR 产物,通过琼脂糖凝胶电泳及胶回收纯化目标条带。最后将目标产物送金维智公司进行一代测序。目的条带的扩增引物序列见表3。测序结果通过EditR(https://moriaritylab.shinyapps.io/editr_v10/)分析,并用GraphPad Prism8 软件进行数据统计分析。
表3 靶位点及其扩增引物序列
2 结果与分析
2.1 TALE 的构建
常用的TALE 靶序列长度为13 ~18 bp,笔者构建的TALE靶向AAVS1 site位点序列为GGGTACTTTTATC(13 bp),以GGG TAC TTT TAT CNN 对应的三联体为模板,分别用引物对1F+1R、2F+2R、3F+3R、4F+4R、5F+R+1 扩增出5 个三联体片段,其中1 ~4 的片段大小为306 bp,5 的片段大小为102 bp。TALE 靶向ABE site2、ABE site1、HEK site 和RNF site 的靶序列分别为14 bp、16 bp、17 bp 和18 bp。同理,分别按顺序每3 个核苷酸对应的三联体为模板,每个模板按顺序用相应的引物对进行扩增,扩增产物PCR 凝胶电泳图见图2(a)。

图2 扩增TALE 三联体片段及连接转化
PCR 产物回收后,与TALE 骨架载体通过金门克隆技术快速组装。转化后在通过蓝白斑筛选平板中长出的单菌落均为白斑[图2(b)],说明载体连接效率高,没有原载体转化克隆。对白色菌落进行PCR鉴定。由于TALE 模块的序列的重复性,具有正确模板的PCR 产物在凝胶电泳上形成了一系列DNA 阶梯带(约300 bp 间隔)。
5 个TALE 载体构建的电泳条带正确率平均超过50%(AAVS1 site TALE 7/10、ABE site1 TALE 4/10、ABE site2 TALE 5/10、HEK site TALE 5/10、RNF site TALE 6/10),如图3(a)所示。经进一步测序比对,证实PCR 条带为大小正确的载体。经测序比对,发现其中有约40%与预计序列完全相同[图3(b)],为成功构建的TALE 载体。

图3 菌液PCR 鉴定及测序统计
2.2 通过细胞基因编辑验证Tri-GG TALE 的功能
TaC9-BE 编辑系统为双向导基因编辑器,两个向导分别为sgRNA 和TALE。5 个TALE 载体已经成功构建的前提下,根据其识别序列的上游6 ~10 bp 处设计sgRNA靶向位点。gRNA和TALE识别序列见表4。经测序,5 个位点的gRNA 载体成功构建。

表4 基因位点的gRNA 和TALE 识别序列
在成功构建TALE 和sgRNA 载体的基础上,将TALE 与脱氨酶TADA8EdelR153融合,与nCas9/gRNA组成TaC9-ABEdelR153编辑系统,如图4(a)所示;同理,TALE 与YE1 融合,与UGI-nCas9-UGI/gRNA 组成TaC9-CBEYE1编辑系统,如图4(b)所示。TaC9-ABEdelR153和TaC9-CBEYE1载体系统分别转染试剂到HEK293T 细胞。收集细胞对目标位点进行测序证实,TaC9-ABEdelR153能在五个位点都具有高效A 到G 的单碱基转换,同样,TaC9-CBEYE1能在5 个位点也具有高效C 到T 的单碱基转换效率,如图4(c)所示。经统计,在所有5 个位点的编辑TaC9-BE 编辑系统与常规胞苷或腺苷单碱基编辑系统具有相同的编辑能力,如图4(d)和图4(e)所示,说明用Tri-GG 克隆技术构建的TALE,具有高效靶向DNA 结合能力,有效应用于基因编辑。
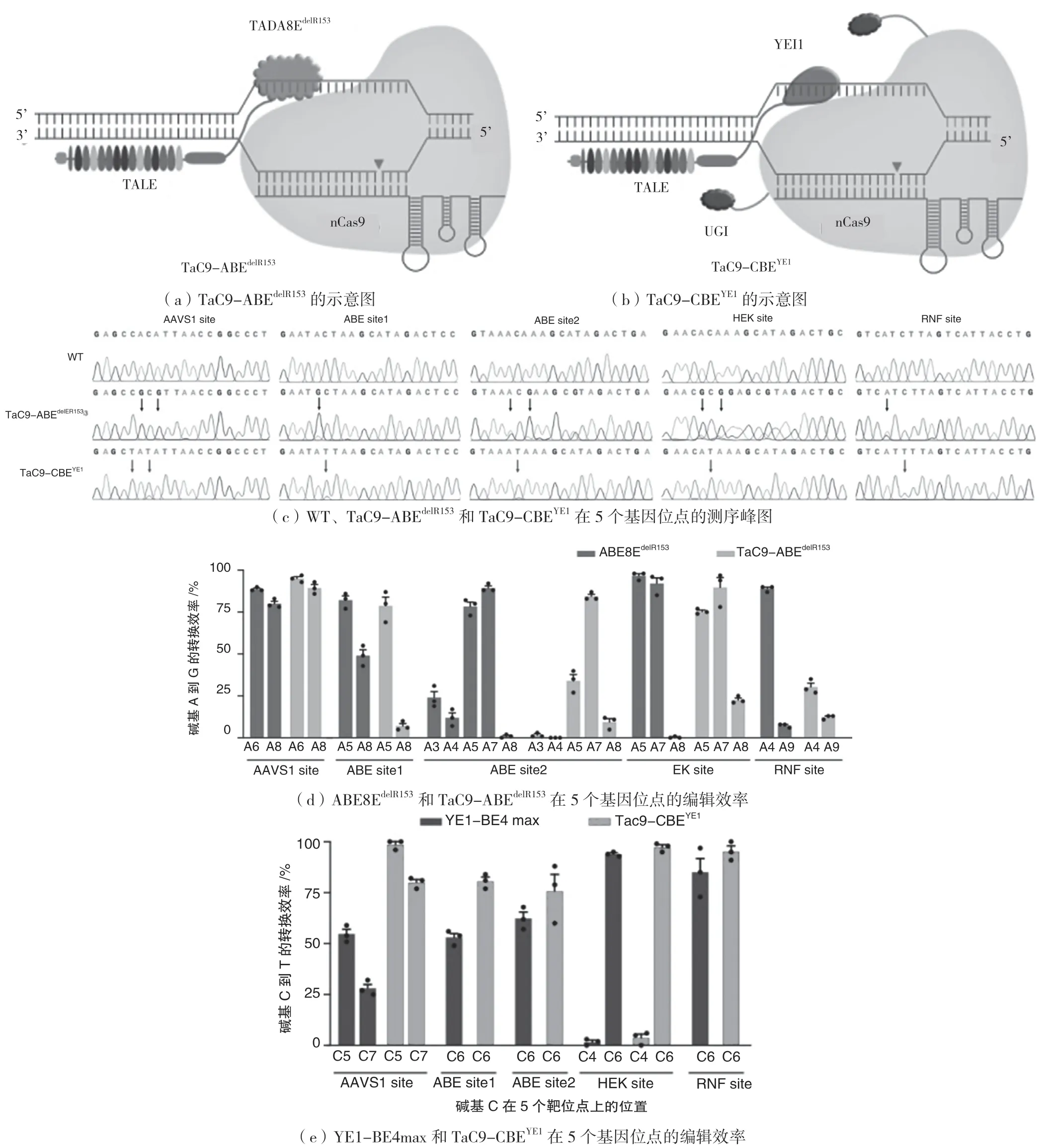
图4 TaC9-BE 系统的示意图和在5 个基因位点上的编辑效率
3 讨论
TALE 一直作为一项强大的DNA 靶向工具被广泛使用,但由于构建方法较为复杂、成本高、耗时长等限制,其近年来被CRISPR/Cas9 系统所替代。由于TALE 自身独特的优势,最近被用于消除可预测的脱靶效应的TaC9-BE 系统和线粒体单碱基编辑中。通过传统的Golden Gate 克隆技术组装TALE 需要分两步进行组装,复杂且耗时长。本研究通过TALE 三联体库结合Golden Gate 组成的新的克隆技术Tri-GG,具有易操作、低成本且仅需一步反应即可组装TALE的优势,且能保证TALE 靶向DNA 的功能性。
在用Tri-GG 法构建TALE 识别序列为13 bp、16 bp 时,扩增的最后一个三联体是单个TALE 模块,由于条带较小导致回收浓度较低。因此,建议在设计TALE 识别序列长度时,尽量选择3 的整数倍。在菌液PCR 鉴定正确而一代测序结果出现点突变时,可尝试更换高保真的PCR 酶或连接酶来提高正确率。对于构建识别序列长度超过18 bp 的TALE 时,建议扩大连接反应的循环数。
4 结论
本研究通过TALE 三联体结合金门克隆技术建立一种新的TALE 构建技术——Tri-GG,成功构建了5 个位点的TALE 载体。将TALE 与脱氨酶融合后,与nCas9/gRNA 组成Tac9-BE 系统,在293T 细胞中实现了高效的单碱基编辑。本技术为TALE 的构建和应用提供了一个新的简单快速的方法。

