面筋蛋白三维网络结构计算机重构研究
徐帅帅,张 斌,马 爽,赵萌迪,李 敏,巨明月,牛付阁,张影全,王效灵,张 波*,潘伟春*
(1 浙江工商大学信息与电子工程学院 杭州310018 2 浙江工商大学食品与生物工程学院 杭州310018 3 浙江方圆检测集团股份有限公司 杭州310018 4 中国农业科学院农产品加工研究所 北京 100193)
小麦是中国的三大主粮之一,是重要的植物蛋白资源[1]。小麦中面筋蛋白可以形成网络结构,从而使其制品具有特有的黏弹性和口感[2-4]。前人研究表明,面团流变性能主要由面筋蛋白所构建的三维网络决定[5-6]。挤压组织化植物蛋白的质构特性是决定产品品质的关键指标之一,主要来自于原料中所含蛋白质的展开、取向和交联所形成的网络结构,以求所得制品具有类似动物肌肉的纤维状结构。因此,定量表征植物蛋白所形成的网络结构就显得非常重要。
然而,该网络结构的表征,是一个亟待解决的问题。利用扩散波谱技术(DWS)能在微米尺度上实时测量面团形成过程中三维结构的变化[6],而试验结果主要通过散射光的相关函数,来获取示踪粒子在所研究体系中的均方差位移,再根据广义Einstein-Stokes 公式,求取体系的流变参数[7]。由于在此过程中,用到不少近似和假设,同时,需要大量计算,且结果呈现不够直观,同时所得结果须用其它技术手段来验证其可靠性。此外,固态植物蛋白制品具有高度透光性,因此,普通的光学方法无法观察到三维介观尺度下的图像。电镜技术需要很薄的切片,制样难度大;同时样品的前处理可能会影响样品本身的形貌;作为典型软物质代表的食品体系,对电流强度非常敏感,长时间或强电流都会破坏样品。原子力显微镜探针精度要求很高,振动速度限制了扫描速度,且表征范围很小,只能观察表面附近的图像。光共聚焦技术可以观察到2-维平面上蛋白纤维的分布[8]。受医学核磁共振成像技术发展的启发[9],本研究探究利用2-维图片,在计算机三维重构技术的协助下再现植物蛋白制品中的三维网络。
本研究以面筋蛋白形成的网络结构为标的,以面团为载体,通过三维重建技术,在一系列高度已知的二维照片的基础上,利用计算机三维重建技术,研究面团中蛋白纤维在三维空间的分布。
1 材料与方法
1.1 样品制备
使用商业小麦粉(Rosenmühle,550,德国)制备面团。每100 g 面粉含有(14.17±0.03)g 水分(AACCI 44-01),每100 g 干面粉含有(12.70±0.04)g 蛋白质(AACCI 46-16,N×5.7)、每100 g 干面粉含有(0.63±0.01)g 灰分(ICC 104/1)、每100 g 面粉含有(28.75±0.81)g 湿面筋(AACCI 56-81)。根据AACCI 54-70.01,在粉质仪(Perten Instruments,doughLAB,德国)中测量面团阻力和吸水率,来确定所需的揉制时间和添加比例。为了达到500 FU 的稠度,准确称取50.1 g 面粉,加入29.6 g 蒸馏水,置于和面机(克莱美斯机电科技(深圳)有限公司,HA-3480AS,中国)中以63 r/min 的转速搅拌揉和180 s,以备使用。
1.2 荧光显微镜下的显微结构分析
用罗丹明B 溶液(0.01 g/100 mL 水)代替5 mL 水对样品进行染色。在和面机中揉制后,置于-20 ℃冰箱中过夜,用10%甲醛磷酸缓冲液固定好面团,固定6 h 以上,固定的标本经4 个浓度梯度的(75%,85%,95%,100%)酒精和二甲苯脱水透明,用切片机(Leica,RM2235,德国)切成14 μm 薄片,用ZMN-7803 全自动组织包埋机(常州市华利电子有限责任公司,ZMN-7803,中国)做成石蜡切片,放在载玻片上,用目镜(20)×物镜(20)倍成像,绿光激发的荧光显微镜(Nexcope,NE910,美国)拍摄相片。为每个面团样品拍摄10 张不同的图像,每个面团样品重复3 次。
1.3 数据集与试验装置
本试验采用80 张小麦面团蛋白质结构切片图像,图像为TIFF 格式,试验采用的CPU 为Intel(R)Core(TM)i7-10700 CPU 4.8Ghz,GPU 为NVIDIA GeForce GTX TitanX。通过3DMed 以实现对二维切片的三维重建。序列断层图像三维重建就是从一系列平行断层图像数据中恢复被重建对象原有的三维面貌。主要涉及断层图像的获得、图像层间插值、绘制、数据的压缩[2]。
1.4 算法简介
MarchingCubes(MC)[10]是三维重建基于面绘制的算法,核心是在三维离散数据场中通过线性差值逼近等值面。该算法中,将多个平行等间距二维切片数据看作一个三维的数据场。
体素一般定义为8 个领域点所包含的区域[11]。假设采样点在3 个坐标轴(i,j,k)方向上是均匀分布的,以等间隔的间距采样为△i、△j、△k,体数据就可以用三维数据的形式表现。将8 个采样点当做立方体的8 个顶点,立方体区域就是1 个体素。体素模型如图1 所示。体素的角点坐标分别为(i,j,k),...,(i+1,j+1,k+1)。
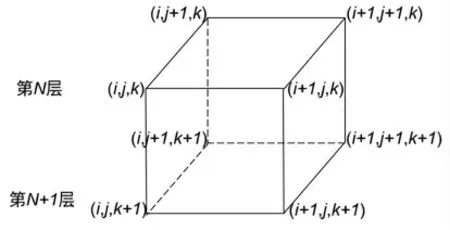
图1 体素模型Fig.1 Voxel model
等值面提取是将二维切片数据中具有某种阈值的物质提取出来,以某种连接方式构成三角形面片[12]。通过对立方体8 个顶点的值进行三线性插值计算得到体素顶点的标量值f(x,y,z),将等值面值C 和标量值进行比较,以此判断该顶点的位置。R 如果体素值大于等于等值面值,定义该顶点位于等值面外,标记为0,反之,则顶点在等值面之内,标记为1,见公式(1):
MC 算法通过对三维数据场中的每一个立方体的体素进行上述处理,将所有的三角形面片进行曲面拟合,最终形成三维立体图。
1.5 重建过程
通过对原始数据进行预处理,得到分割之后的图像,分割图像的目的是为了减少背景因素的干扰和提高重建速度,使得重建后的模型准确性更高。对原始数据集进行一系列的预处理过程:灰度化、降噪、分割等操作,将目标区域从背景中提取出来[13]。
灰度化的主要目的是为了避免图像条带失真,灰度化之后的图像只需要一个字节存放灰度值。灰度图像素通常采用像素8 位的非线性尺度保存,因此更易于编程[14]。图像噪声会对图像分割等操作产生不良影响,为了最终模型的准确性,采用高斯滤波器对图像进行平滑去噪[15]。通过图像灰度级的不同,将目标以外的背景干扰因素除去,只保留目标区域,为三维重建过程做好基础准备。
原始图像集和图像预处理过程如图2 所示。阈值分割对重建后的模型影响非常大,阈值选取不当会对目标区域造成过分割现象,使得目标区域上的一些蛋白质结构被切割掉,后续提取有用信息将会造成误差[16]。将处理好的图像输入到3DMed 软件中,建立体数据,并生成网格数据,最后生成三维模型。

图2 原始图像集和图像预处理过程:(a)原始图像集,(b)灰度图像集,(c)分割后的图像Fig.2 Original image set and image preprocessing process:(a)original image set,(b)grayscale image set,(c)image segmentation
2 结果与分析
2.1 原始照片及处理
图3 显示的一组垂直方向保持不变而水平方向相差14 倍数微米差的图像。图像采用等间隔、小间距采样方式,这对于三维重建至关重要,可以保证重建后的模型特征不丢失。图中的尺度标志为200 μm,非常明显看到蛋白面筋纤维在平面上的分布。

图3 一组在垂直方向一样的面团相片Fig.3 Pictures of dough with the same vertical orientation
图4 是用Opencv 处理后得到的灰度图像。在数字图像处理中,灰度图像中不同的目标以不同的灰阶显示,灰阶范围在0~255 之间。灰度图像的描述与彩色图像一样,反映了整幅图像的整体和局部的色度和亮度等级的分布和特征,相较于彩色图像,灰度图像占用更少的维度,运算速度大幅度提高,并且最重要的梯度信息得以保留。将彩色图像灰度化后,再根据灰度的不同,对图像做阈值分割,可以得到蛋白面筋纤维的二值图像。

图4 Opencv 处理后所得的灰度图像Fig.4 Grayscale image after Opencv processing
图5 为在图4 基础上进行二值化处理得到的图像。二值图像分析在对象识别和特征匹配中具有重要作用,基于灰度的不同设定一个阈值,将图像数据分为两部分,大于此阈值的像素群置为白色,小于此阈值的像素群置为黑色,经过二值化处理后,整幅图像只剩下黑、白两种颜色。二值化处理将重建目标以外的背景因素干扰去除,减少了三维重建过程所用时间。同时,二值图像又保留了目标区域的结构特征,确保了重建模型的准确性。

图5 二值图像Fig.5 Binary image
2.2 蛋白面筋纤维的粗细分布
通过上述操作,对80 张1024 px×800 px 间距为14 μm 的蛋白质结构进行三维重建,重建后的结果如图6 所示。生成的模型基于MC 算法,模型内部经过投影,生成三角形面片,三角形面片拟合成曲面,将模型放大后,可以更加细致的观察到内部结构如图7 所示。

图6 三维重建模型Fig.6 Three-dimensional reconstruction model

图7 模型内部图Fig.7 Internal diagram of the model
该方法能提供以下关键性参数,纤维的密度和在空间的分布;纤维的长度;每根纤维的粗细。
本试验所重建的小麦面粉团蛋白质模型是基于微米级的三维重建,三维立体模型可以更直观的从不同角度观察到样品特征(图8),快速的提取出所需要的物理信息。

图8 3 个相互垂直的方向上面筋蛋白纤维染色模型Fig.8 Gluten fiber staining model in three mutually perpendicular directions
根据图7 和图8,经过测量换算,整个模型内部面筋蛋白纤维的数量约3 000 个,纤维的粗细为2~14 μm(图9a),长度为140~420 μm(图9b)。

图9 面筋蛋白纤维长度分布图Fig.9 Distribution map of gluten protein fiber length
其中面筋蛋白纤维的粗细主要集中在较小的部分(〈7 μm),约50%面筋蛋白纤维的粗细在2 μm 左右,这和Don 等[5]利用激光共聚焦技术在2维平面上看得的结果有部分吻合。面团质构和这一参数的分布紧密相连。如果用虎克定律(弹簧拉伸)模型来简化面筋蛋白纤维的机械拉伸赋予面团的机械性能[17],因为弹簧的弹性系数式中,G——弹簧的切变模量(Pa);d——弹簧钢丝的直径(mm);DZ——弹簧中弹簧环的直径(mm);n——弹簧的环数[18]。因此,面团稠度和弹性可以近似用来估算,式中ni是直径为di蛋白面筋纤维的空间浓度。同时面团机械性能还与G 有关。而G 可能和面团中的营养成分分布、各种化学键(如二硫键等用于构建面团中的三位网状结构)在面团中的时空分布[19]、麦谷蛋白颗粒的变化[20]、水含量[21]、添加剂的种类和用量[22]等有关。
利用三维重构技术,使得量化上述参变量以研究对面团机械性能的影响成为可能;同时能更精细化的描述面粉储存等条件的影响。这些影响因素虽然在工业实践中是非常明显的,但其作用机制还存在巨大争议[23];同时这也为配粉等操作提供理论指导[24]。
2.3 面团中面筋蛋白纤维长度分布
面团中面筋蛋白纤维长度的分布如图9b 所示,约45%的面筋蛋白纤维的长度在140~210 μm 之间;约30%的在210~280 μm,剩下约24%长度大于280 μm。这个长度的分布非常重要,因为它决定了面团中纤维所分割的空间大小。Li 等[6]利用扩散波谱技术(DWS)分析了面团在揉制的不同时间段下,这些空间的变化,发现这一变化规律能揭示面团中面筋蛋白三维网络的发展,同时DWS 所得的蛋白面筋三维网络中的空间大小和本试验所得有一定的吻合。
2.4 存在的问题及解决策略
受算法的限制,目前还没有考虑面筋蛋白纤维的弯曲和缠绕,这是导致目前试验结果和DWS等技术手段所获取的结果存在一定差异的原因。目前,本课题组正在改进算法,优化重构程序,以期能与其它试验结果有更好的吻合度。如特征信息进行分类标记等技术的应用。
目前采用的荧光显微镜技术拍照片,位置控制和照片清晰度等存在很大的提升空间。后期将利用激光共聚焦技术,以期得到质量更好的照片(位置控制、分辨率、相片的清晰度等)。
3 结论
利用三维重构技术,可以了解面筋蛋白中纤维的数量、大小,及其在空间中的分布。然而目前所得的仅仅是初步结果,验证计算机三维重构在面团中再现面筋蛋白纤维的可能性,且存在不少局限性:如二维平面上蛋白纤维截点之间的连接存在较大的任意性,需要进一步优化算法;同时,该结果还需用扩散波谱等技术证明该方法的可靠性。

